A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
بروتوكول في المختبر لتقييم مستويات MicroRNA، وظائف، والجينات المستهدفة المرتبطة بها في الخلايا السرطانية
In This Article
Summary
ويستخدم هذا البروتوكول تفاعلاً متسلسلاً للبوليميراز في الوقت الحقيقي القائم على المسبار، ومسباراً من نوع سولفورهوامين B (SRB)، واستنساخ 3 مناطق غير مترجمة (3' UTR)، وإمكانية اختبار لوسيفيراز للتحقق من الجينات المستهدفة لميرنا ذات الأهمية ولفهم وظائف الميرنا.
Abstract
MicroRNAs (miRNAs) هي RNAs التنظيمية الصغيرة التي يتم التعرف عليها لتعديل العديد من مسارات الإشارات داخل الخلايا في العديد من الأمراض بما في ذلك السرطان. وتتفاعل هذه التقييمات التنظيمية الصغيرة أساساً مع المناطق غير المترجمة البالغ عددها 3 مناطق (3' من جمهورية ترانسبورت) من الرسل المستهدفين (mRNAs) مما أدى في نهاية المطاف إلى تثبيط عمليات فك رموز الـ mRNAs وزيادة تحلل الحمض اتّصال اتّصاليّة الهدف. استناداً إلى مستويات التعبير والوظائف داخل الخلايا، miRNAs قادرة على أن تكون بمثابة عوامل تنظيمية من mRNAs oncogenic والورم المثبطة. تحديد الجينات المستهدفة حسن النية من ميرنا بين مئات أو حتى الآلاف من الأهداف المتوقعة حسابيا هو خطوة حاسمة لتمييز الأدوار والآليات الجزيئية الأساسية لميرنا من الفائدة. تتوفر العديد من برامج التنبؤ بالأهداف miRNA للبحث عن تفاعلات ميرنا-ميرنا المحتملة. ومع ذلك، فإن السؤال الأكثر تحديا هو كيفية التحقق من صحة الجينات المستهدفة المباشرة من ميرنا ذات الفائدة. يصف هذا البروتوكول استراتيجية قابلة للاستنساخ من الأساليب الرئيسية حول كيفية تحديد أهداف ميرنا المتعلقة بوظيفة ميرنا. يقدم هذا البروتوكول دليلاً عملياً على الإجراءات خطوة بخطوة للكشف عن مستويات ميرنا، والوظائف، وما يتصل بها من mRNAs الهدف باستخدام تفاعل البوليميراز المتسلسل القائم على المسبار في الوقت الحقيقي (PCR)، اختبار sulforhodamine B (SRB) بعد اختبار محاكاة ميرنا للانف ، توليد منحنى الجرعة والاستجابة، والاختبار لوسيفيراز جنبا إلى جنب مع استنساخ 3 'UTR من الجين، وهو أمر ضروري لفهم صحيح لأدوار miRNAs الفردية.
Introduction
MicroRNAs (miRNAs) هي RNAs التنظيمية الصغيرة التي تعدل بشكل رئيسي عملية ترجمة وتدهور RNAs رسول (mRNAs) عن طريق الرد على 3 'المناطق غير المترجمة (3' UTR) في الجينات المستهدفة حسن النية1. ويمكن تنظيم التعبير عن الـ miRNAs من خلال آليات النسخ وما بعد النسخ. عدم التوازن في هذه الآليات التنظيمية يجلب مستويات التعبير miRNAs غير المنضبط والمميزة في العديد من الأمراض بما في ذلك السرطانات2. يمكن أن يكون لميرنا واحد تفاعلات متعددة مع mRNAs متنوعة. وفي المقابل، يمكن التحكم في الميرنا الفردية من قبل العديد من miRNAs. لذلك، تتأثر شبكات الإشارات داخل الخلايا بشكل معقد بـ miRNAs التي يتم من خلالها بدء الاضطرابات والأمراض الفسيولوجية وتدهورها2،3،4، 5 , 6.على الرغم من أن التعبير المتغير من miRNAs لوحظ في أنواع مختلفة من السرطانات، والآليات الجزيئية التي تعدل آداب الخلايا السرطانية جنبا إلى جنب مع miRNAs لا تزال غير معروفة إلى حد كبير.
وقد أظهرت الأدلة المتراكمة أن الأدوار الأورامأو المثبطة للأورام من miRNAs تعتمد على أنواع السرطانات. على سبيل المثال، من خلال استهداف مربع forkhead o3 (FOXO3)، miR-155 يعزز انتشار الخلايا، والنقي، والمقاومة الكيميائية لسرطان القولون والمستقيم7،8. وعلى النقيض من ذلك، فإن تقييد غزو الخلايا الدبقية يسببه miR-107 عن طريق تنظيم البروتين المتجانس من الدرجة العصبية locus 2 (NOTCH2) التعبير9. تقييم التفاعلات ميرنا الهدف فيما يتعلق وظائف ميرنا هو جزء لا غنى عنه لفهم أفضل كيف miRNAs تنظيم العمليات البيولوجية المختلفة في كل من الدول الصحية والمرضى10. وبالإضافة إلى ذلك، فإن اكتشاف هدف (أهداف) حسن النية من miRNAs يمكن أن توفر كذلك استراتيجية دقيقة لضبطها لعلاج يستند إلى ميرنا مع مختلف الأدوية المضادة للسرطان. ومع ذلك، فإن التحدي الرئيسي في مجال التقييمات الوطنية للجمهورية الإسلامية هو تحديد الأهداف المباشرة للجمهورية. هنا، يتم تقديم أساليب مفصلة كنهج تجريبية قابلة للاستنساخ لتحديد الجينات الهدف ميرنا. وينطوي التصميم التجريبي الناجح لتحديد هدف ميرنا علىخطوات واعتبارات مختلفة (الشكل 1). مقارنة مستويات ميرنا الناضجة في الخلايا السرطانية والخلايا الطبيعية يمكن أن تكون واحدة من الإجراءات الشائعة لاختيار ميرنا ذات الأهمية (الشكل1A). الدراسة الوظيفية لميرنا مختارة للكشف عن آثار ميرنا على انتشار الخلايا من المهم لتضييق قائمة أفضل الأهداف المرشحة المحتملة من ميرنا ذات الفائدة (الشكل1B). واستناداً إلى الوظائف التي تم التحقق من صحتها تجريبياً لـ miRNAs، يلزم إجراء مراجعة منهجية للمؤلفات وقاعدة البيانات في الشركة التي لها برنامج التنبؤ المستهدف بالحمض النووي الريبي (MIRNA) للبحث عن المعلومات الأكثر صلة بوظائف الجينات (الشكل1C). ويمكن تحقيق تحديد الجينات المستهدفة الحقيقية لميرنا ذات الأهمية من خلال تنفيذ تجارب مثل اختبار لوسيفيراز جنبا إلى جنب مع استنساخ 3 'UTR من الجينات، PCR في الوقت الحقيقي، والنشاف الغربي (الشكل1D). والهدف من البروتوكول الحالي هو توفير أساليب شاملة للتجارب الرئيسية، وتفاعل البوليميراز المتسلسل القائم على المسبار في الوقت الحقيقي، والاختبار بسولفورهوامين B (SRB) بعد أن تحاكي ميرنا عمليات الانحراف، وتوليد منحنى الجرعة والاستجابة، و لوسيفيراز اختبار جنبا إلى جنب مع استنساخ 3 'UTR من جين. البروتوكول الحالي يمكن أن تكون مفيدة لفهم أفضل لوظائف miRNAs الفردية والآثار المترتبة على ميرنا في علاج السرطان.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. ناضجة ميكرورنا (ميرنا) تحليل التعبير
-
ناضجة ميرنا الحمض النووي التكميلي (cDNA) التوليف
- إضافة 254 نانوغرام من إجمالي الحمض النووي الريبي و 4.5 ميكرولتر من خليط ديوكسيريبونوكليس الأول (DNase I)، ثم إضافة المياه النقية جداً في أنابيب قطاع PCR لتعويض ما يصل إلى 18 ميكرولتر (الشكل2A). إعداد رد الفعل لكل عينة RNA مجموع تنقية من عدة خطوط الخلية باستخدام كمية كافية من خليط DNase الأول على أساس العدد الإجمالي من ردود الفعل.
ملاحظة: تتكون خلائط DNase I من DNase I (1.8 ميكرولتر)، ومثبط اتّصال الريبونوكليس (0.3 ميكرولتر)، و25 ممغكل2 (2.4 ميكرولتر). ولشراء الحمض النووي الريبي الكلي، تم تطبيق طريقة استخراج تستند إلى العمود بدلاً من استخدام طريقة استخراج تعتمد على الفينول الكلوروفورم. وأفيد بأن غلة استخراج بعض miRNAs يمكن أن تختلف اعتمادا على عدد الخلايا عند استخدام طريقة استخراج الفينول الكلوروفورم القائم11،12. - احتضان الأنابيب في دورة الحرارية. تشغيل الأنابيب لمدة 10 دقيقة في 37 درجة مئوية، وتعطيل الحرارة DNase الأول من قبل 5 دقيقة الحضانة في 90 درجة مئوية. على الفور وضع الأنابيب على الجليد بعد الحضانة.
- نقل 7.1 ميكرولتر من DNase عالجت الجيش الملكي النيبالي الكلي في 2 مجموعات من الأنابيب الجديدة ومن ثم إضافة 1.5 ميكرولتر من التمهيديات المضادة للتحسس لجين غليسيرالدهايد-3-فوسفات dehydrogenase (GAPDH) (الشكل2B).
ملاحظة: يصبح المبلغة من [رنا] إجماليّة ل [كدن] تأليف 100 [نغ] في هذا خطوة. تركيز المخزون من المواد التمهيدية المضادة للحاسة GAPDH هو 10 μM. إضافة المبادئ التمهيدية المضادة للتحسس GAPDH هو لتوليد cDNAs GAPDH باستخدام طريقة التمهيدي الجينات محددة. - احتضان الأنابيب باستخدام دورة الحرارية. تبدأ في 80 درجة مئوية لمدة 5 دقائق تليها رد الفعل في 60 درجة مئوية لمدة 5 دقائق.
- إضافة 3.4 ميكرولتر من النسخ العكسي (RT) خليط إنزيم في كل رد فعل (الشكل2B). تتكون خلائط إنزيم RT من 100 m deoxyribonucleotide triphosphates (0.15 μL)، 10X RT العازلة (1.5 ميكرولتر)، مثبطات الريبونوكليس (0.75 ميكرولتر)، وإنزيم النسخ العكسي (1 ميكرول). إعداد كمية كافية من الخلائط على أساس العدد الإجمالي من ردود الفعل.
- إضافة 3 ميكرولتر من 5X RT التمهيديات لميرنا محددة في كل رد فعل (الشكل2B).
ملاحظة: الحجم الإجمالي هو 15 درجة مئوية لكل رد فعل. - تشغيل الأنابيب باستخدام دورة الحرارية. تبدأ عند 16 درجة مئوية لمدة 30 دقيقة تليها رد الفعل عند 42 درجة مئوية لمدة 30 دقيقة، وأخيراعند 85 درجة مئوية لمدة 5 دقائق. يتم إنشاء cDNAs واحد الذين تقطعت بهم السبل في هذه الخطوة لكل من الجين ميرنا محددة وGAPDH في نفس الأنبوب.
- إضافة 254 نانوغرام من إجمالي الحمض النووي الريبي و 4.5 ميكرولتر من خليط ديوكسيريبونوكليس الأول (DNase I)، ثم إضافة المياه النقية جداً في أنابيب قطاع PCR لتعويض ما يصل إلى 18 ميكرولتر (الشكل2A). إعداد رد الفعل لكل عينة RNA مجموع تنقية من عدة خطوط الخلية باستخدام كمية كافية من خليط DNase الأول على أساس العدد الإجمالي من ردود الفعل.
-
تفاعل البوليميراز المتسلسل في الوقت الحقيقي (PCR) وتحليل البيانات
- تمييع كل cDNA بالماء النقي بنسبة 1:49.
- إعداد مخاليط التفاعل لميرنا محددةوGAPDH (الجدول 1). للكشف عن ميرنا محددة وGAPDH، إعداد ردود الفعل الثلاثية لكل عينة cDNA.
- إجراء تحليل PCR والبيانات في الوقت الحقيقي (الشكل2C). تحليل البيانات باستخدام طريقة CT المقارنة13،14.
2. ميكرورنا (ميرنا) تقليدترانسفيكأيشن
ملاحظة: يتم تحديد miRNA-107 من الخطوة 1. منذ ميرنا-107 هو أسفل تنظيم في الخلايا السرطانية مقارنة مع الخلايا العادية، ويمكن التكهن بأن ميرنا-107 هو ميرنا قمعالورم. في حالة ميرنا التي يتم تنظيمها في الخلايا السرطانية مقارنة مع الخلايا الطبيعية (على سبيل المثال،ميرنا-301) ، يمكن تطبيق oligonucleotides المضادة للتحسس ضد ميرنا-301 للخطوات 2 و 3 و 4.
- عد الخلايا مع جهاز غرفة العد ولوحة الخلايا في لوحة 96 جيدا. كثافة الخلايا هي 2 × 103 خلايا / 100 ميكرولتر لكل بئر. لا تستخدم وسائط ثقافة الخلية التي تحتوي على البنسلين-ستربيماتسين (P/S) لأن P/S يمكن أن تقلل من كفاءة التغوط.
-
إعداد مجموعة من خليط التغوط لنقل الخلايا في عدة تركيزات نهائية من محاكاة التحكم ميرنا ومحاكاة ميرنا-107 في اليوم التالي (الشكل3).
- من المخزون (تركيز 25 درجة مئوية) من محاكاة التحكم ميرنا أو ميرنا-107 تقليد، تخفيف وإضافة كمية المقابلة من محاكاة السيطرة أو ميرنا-107 تقليد في وسائل الإعلام انخفاض المصل جنبا إلى جنب مع كاشف التغوط باستخدام أنابيب الطرد المركزي الدقيقة (الشكل3A). يُمزج القلة التي تحتوي على خلائط بلطف باستخدام الميكرومابيت. يجب أن يكون المبلغ الإجمالي للقلة (ميرنا تقليد السيطرة + ميرنا-107 تقليد) نفسه في كل بئر. وتشمل الآبار الفارغة 100 درجة مئوية من وسائل الإعلام ثقافة الخلية ووسائط المصل المنخفض التي تحتوي على كاشف التغوط دون خلايا.
- بعد 10 دقائق الحضانة في غطاء محرك السيارة ثقافة الخلية، وخلط بلطف oligo تحتوي على المخاليط مرة أخرى ثم إضافة 50 درجة مئوية من المخاليط في كل بئر. الحفاظ على الخلايا المنقولة في حاضنة ثقافة الخلية. استبدال كاشف التغوط التي تحتوي على وسائل الإعلام مع وسائل الإعلام ثقافة الخلية الطازجة التي تحتوي على كل من المصل البقري الجنيني (FBS) و P / S بعد 6-12 ح الحضانة. مزيد من احتضان الخلايا لمدة 72 ساعة. مدة العلاج الإجمالية لتقليد ميرنا هي 96 ساعة.
3- سفورهودومين ب (SRB)
-
تثبيت الخلايا
- إزالة وسائل الإعلام ثقافة الخلية في كل بئر من لوحة وملء على الفور 100 درجة مئوية من حمض ثلاثي كلورو الخليك 10٪ (TCA) في كل بئر. يستنشق بعناية وسائل الإعلام ثقافة الخلية من كل بئر لتجنب أي تلف الخلية والانفصال من القاع.
ملاحظة: إعداد 40٪ TCA عن طريق إضافة 20 غرام من مسحوق TCA في 50 مل من الماء المقطر. من 40٪ TCA، وجعل 10٪ TCA عن طريق تخفيف 40٪ TCA مع الماء المقطر بنسبة تخفيف 1:3. - احتفظ باللوحة التي تحتوي على 10% TCA في ثلاجة (4 درجات مئوية) لمدة ساعة واحدة.
- غسل لوحة عدة مرات عن طريق الغمر في حوض المياه وتجفيفها. إزالة المياه الزائدة من داخل الآبار عن طريق النقر على لوحة حتى لا يكون هناك ماء في الآبار. ترك لوحة على مقاعد البدلاء المختبر لتجفيفه قبل الذهاب إلى الخطوة التالية.
- إزالة وسائل الإعلام ثقافة الخلية في كل بئر من لوحة وملء على الفور 100 درجة مئوية من حمض ثلاثي كلورو الخليك 10٪ (TCA) في كل بئر. يستنشق بعناية وسائل الإعلام ثقافة الخلية من كل بئر لتجنب أي تلف الخلية والانفصال من القاع.
-
تلطيخ الخلايا
- ماصة 50 درجة مئوية من 0.4٪ SRB الحل في كل بئر بما في ذلك الآبار الفارغة. هز الصحن بلطف حتى يغطي حل SRB بنسبة 0.4% باستمرار الجزء السفلي من الآبار.
ملاحظة: إعداد واستخدام 0.4٪ SRB الحل عن طريق إضافة 0.4 غرام من مسحوق SRB في 100 مل من حمض الخليك 1٪. هز الحل بعناية لخلطه. التفاف زجاجة من 0.4٪ حل SRB في مادة واقية خفيفة مثل رقائق الألومنيوم. تخزين 0.4٪ SRB الحل في الثلاجة. - بعد الحضانة لمدة 40 دقيقة إلى 60 دقيقة، وغسل لوحة عن طريق الرينمنة مع 1٪ حمض الخليك. غسل لوحة حتى يتم غسلها تماما صبغة غير منضم بعيدا (الشكل3B).
- ترك لوحة على مقاعد البدلاء المختبر لتجفيفه قبل الذهاب إلى الخطوة التالية.
ملاحظة: يجب أن يتم تجفيف اللوحة بالكامل قبل الذهاب إلى الخطوة 3.3.
- ماصة 50 درجة مئوية من 0.4٪ SRB الحل في كل بئر بما في ذلك الآبار الفارغة. هز الصحن بلطف حتى يغطي حل SRB بنسبة 0.4% باستمرار الجزء السفلي من الآبار.
-
قياس الامتصاص
- ماصة 100 درجة مئوية من حل قاعدة تريس (10 MM) في الآبار المقابلة بما في ذلك الآبار الفارغة. الحفاظ على لوحة على شاكر لمدة 10 دقيقة.
4. توليد منحنى الجرعة والاستجابة
- تحليل بيانات SRB في جدول بيانات. طرح الامتصاص الفارغ من قيم الامتصاص لكل مجموعة وحساب متوسط (AVE) والانحراف المعياري (STD) لقيم الامتصاص لكل مجموعة.
- حساب النسبة المئوية لمتوسط الامتصاص (AVE) والانحراف المعياري (STD)( لكل مجموعة باستخدام قيم امتصاص SRB.
ملاحظة: AVE٪ من التحكم ميرنا تقليد المجموعة المعالجة هو 100٪. حساب STD٪ باستخدام الصيغة التالية: STD٪ = (STD من كل مجموعة / AVE امتصاص السيطرة تقليد المجموعة المعالجة) × 100. - استيراد البيانات الخام بما في ذلك تركيزات العلاج، AVE٪، وSTD٪ في البرنامج عن طريق محاذاة عموديا تلك البيانات. بما أن السجل 0 غير معرّف، قم بتعيين التركيز الأول للمحورX إلى قيمة قريبة من 0 (على سبيل المثال ، 0.01).
- انقر فوق إنشاء علامة التبويب الرسم البياني واختر أشرطة خطأ مبعثر بسيطة. حدد أعمدة ورقة العمل كقيم رموز وانقر فوق التالي. في لوحة تنسيق البيانات، حدد أزواج XY وانقر فوق التالي. حدد أعمدة البيانات المقابلة في لوحة البيانات المحددة. انقر فوق الزر إنهاء لإنشاء المؤامرة.
ملاحظة: يمثل المحور س التركيزات، ويشير محور ص إلى النسبة المئوية لمتوسط امتصاص كل تركيز (AVE)، وتشير أشرطة الخطأ إلى النسبة المئوية للانحراف المعياري لكل تركيز (STD%).. - انقر نقراً مزدوجاً فوق المحور X لتعديل نوع المقياس وتحجيم المحور. تغيير نوع المقياس من خطي إلى سجل. تعديل رقم نطاق البدء والنهاية إلى 0.01 و 200 على التوالي.
-
انقر بزر الماوس الأيمن على أي مؤامرة مبعثر، واختيار منحنى صالح، انتقل إلى الفئة الفرعية المعرفة من قبل المستخدم. حدد منحنى استجابة الجرعة، انقر فوق الأزرار التالية، ثم انقر فوق الزر إنهاء. يتم الآن إنشاء منحنى الجرعة استجابة جنبا إلى جنب مع علامة التبويب تقرير (الشكل4A).
- لإدخال المعادلة 1 في البرنامج لإنشاء منحنى استجابة الجرعة، انقر فوق علامة التبويب تحليل وحدد معالج الانحدار. انتقل إلى معرفة المستخدم في فئة المعادلة ثم انقر فوق الزر جديد. إدراج المعادلة 1 والمتغيرات والمعلمات الأولية والقيود في مربعات فارغة المقابلة (الشكل4B،C). انقر فوق إضافة كزر وتعيين اسم المعادلة كمنحنى استجابة الجرعة. يتم الآن إنشاء اسم المعادلة في الفئة الفرعية المعرفة من قبل المستخدم في فئة المعادلة. (و) يشير إلى النسبة المئوية لصلاحية الخلية (% من صلاحية الخلية) في المعادلة 1.
المعادلة 1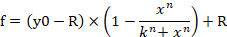
- انتقل إلى علامة التبويب التقرير ثم تحقق من قيم n و k و R.
ملاحظة: يشير y0 إلى قدرة الخلية على البقاء بنسبة 100% من مجموعة محاكاة التحكم في ميرنا، n تشير إلى معامل من نوع التل (منحدر قطعة الأرض)، ك يشير إلى تركيز محاكاة ميرنا-107 التي تنتج 50٪ من تأثير تقليد ميرنا-107 الأقصى (النصف التركيز المثبط القصوى، IC50)،وR يشير إلى الكسر المتبقية غير المتأثرة (كسر المقاومة)15. المعادلة المستخدمة لتوليد منحنى الجرعة استجابة تعترف النطاق من قيمة y0 إلى R (إن وجدت) كما 100٪ (الشكل4A). لذلك، من الضروري الحصول على قيمة k المعدلة (IC50)التي يتم حسابها على أساس النطاق من y0 إلى قيمة صفر (الشكل4A). يمكن الحصول على k المعدلة (IC50)جنبا إلى جنب مع قيم ICx الأخرى (على سبيل المثال، IC10 إلى IC90)باستخدام المعادلة 2، المستمدة من المعادلة 1. وترد اشتقاق المعادلة 2 من المعادلة 1 في الشكل التكميلي1.
المعادلة 2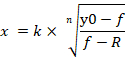
- انقر نقراً مزدوجاً فوق زر الماوس الأيسر على الخلية التي يتم تطبيق المعادلة 2. باستخدام المعادلة 2 والمعلمات من منحنى الجرعة استجابة ولدت، فإنه متاح لحساب القيم المعدلة من ICx، تتراوح بين IC10 إلى IC90 (الشكل4D).
- أدخل علامة المساواة متبوعة بالصيغة التي تبدأ بقوس في الخلية. عند إدخال الصيغة، قم بإصلاح قيمة n و k و R كمراجع مطلقة للخلايا عن طريق إضافة علامة الدولار إلى العمود والصف المقابلين، بحيث لا يتم تغيير هذه القيم الثابتة عند ملء الصيغة تلقائيًا إلى الصفوف (الشكل 4D). بدلاً من ذلك، يمكن حساب القيم المعدلة يدوياً باستخدام المعادلة 2.
ملاحظة: لم يتم تحديد قيمة IC90 لأن قيمة R أكبر من 10. بالإضافة إلى ذلك، إذا كانت قيمة R أعلى من 20، لا يتم تحديد قيمة IC80 (الشكل4D).
5. التحقق من الجين المستهدف المباشر لـ MicroRNA ذات الأهمية
ملاحظة: بعد إجراء التجربة الوظيفية مثل اختبار SRB، يتم تأكيد ميرنا-107 كميرنا قمعية الورم وأنه من الممكن للغاية أن miRNA-107 تستهدف مباشرة oncogenes. تحقق من قائمة جميع الجينات المستهدفة المتوقعة باستخدام برنامج التنبؤ المستهدف miRNA مثل TargetScan (http://www.targetscan.org/vert_71/)، ثم تضييق إلى أهداف المرشحين المحتملة استناداً إلى وظيفة الجين في قواعد البيانات بما في ذلك PubMed و بطاقات الجينات.
- التصميم التمهيدي لاستنساخ 3 'المنطقة غير المترجمة (UTR)
- ضع اسم الجين في بطاقات الجينات (https://www.genecards.org/) وانقر على رمز الجين. تقييم لمتصفح الجينوم Ensembl عن طريق النقر على معرف Ensembl من الجين ثم انقر فوق معرف النصوص في جدول النسخ. بعد ذلك، انقر فوق Exons موجود في قائمة الشاشات المستندة إلى النص على اليسار.
- نسخ تسلسل النيوكليوتيد من UTR 3 'ولصقه في برنامج التصميم التمهيدي. نسخ التسلسلات مرة أخرى من هذا البرنامج ولصقه في معالج النصوص. تحقق من وجود تسلسلات ربط ميرنا وكذلك وجود مواقع إنزيمات تقييدية تستخدم للاستنساخ.
ملاحظة: إذا لم تكن هناك مواقع التعرف على الإنزيمات المقيدة داخل UTR 3'، يمكن استخدام الإنزيمات المقيدة المحددة للاستنساخ للخطوة التالية. - في برنامج التصميم التمهيدي، قبول تسلسل UTR 3 'والبدء في تصميم التمهيديات إلى الأمام وعكس مع الشرط التالي. طول: 20-30 النيوكليوتيدات، Tm: 45-58 درجة مئوية، GC٪: 40-60٪. يجب أن يكون الفرق بين قيم Tm من التمهيديين أقل من 5 درجة مئوية. وترد التسلسلات التمهيدية المستخدمة في هذه الدراسة في الشكل التكميلي2. إضافة تسلسل التعرف على الإنزيم تقييد فضلا عن 4 النيوكليوتيدات عشوائية إلى التمهيديات المصممة.
- PCR متدرجة
- إعداد 25 ميكرولتر من مخاليط تفاعل PCR بما فيذلك المواد التمهيدية المصممة لكل درجة حرارة صلب واحدة (الجدول 2). إعداد كمية كافية من الخلائط على أساس العدد الإجمالي من ردود الفعل. خلط الحل عن طريق الأنابيب وإضافة 25 درجة مئوية من مخاليط رد الفعل في كل أنبوب. طرد مركزي الأنابيب لبضع ثوان.
- تنفيذ 35-40 دورات PCR من خطوة denaturation إلى خطوة التمديد. إعداد دورة PCR على النحو التالي الخطوات التالية: 98 درجة مئوية لمدة 1 دقيقة (1 دورة، خطوة تنشيط البوليمرات)، 95 درجة مئوية لمدة 10 ق (خطوة إزالة النقان)، 45 درجة مئوية-68 درجة مئوية لمدة 30 ق (خطوة الصلب)، 68 درجة مئوية (خطوة التمديد، 10 ق-1 دقيقة لكل 1000 نقطة أساس)، 68 درجة مئوية لمدة 3 دقائق (خطوة إنهاء) ، وأخيراً تبرد إلى 4 درجة مئوية.
- تشغيل منتجات PCR والتحقق من العصابات على هلام أجاروز 1٪ مع سلالم الحمض النووي. العثور على أفضل درجة حرارة الصلب (الشكل5A). تضخيم 3 'UTR من الجين مرة أخرى باستخدام أفضل درجة حرارة الصلب للخطوة التالية.
- الهضم المزدوج
- جعل خليط رد الفعل بما في ذلك اثنين من الإنزيمات تقييد، XhoI (أو AsiSI) ونوتى، في أنبوب (الجدول3). احتضان المخاليط لمدة 3-4 ساعة باستخدام حمام مائي (37 درجة مئوية).
- تشغيل المنتجات هضم مزدوجة على هلام أجاروز 1٪ ومن ثم قطع العصابات تحت ضوء الأشعة فوق البنفسجية. في حالة ناقلات لوسيفيراز، قبل تشغيل على هلام، تتفاعل ناقلات هضم مزدوج ة مع 10 U من الفوسفاتات القلوية لمدة ساعة أخرى لمنع recircularization أثناء خطوة الربط.
- تنقية منتجات PCR مزدوجة هضمها وناقلات لوسيفيراز من العصابات المقتطعة.
- ربط منتجات PCR في ناقلات لوسيفيراز
- جعل 20 درجة مئوية من خلطات رد الفعلالربط بما في ذلك ligase الحمض النووي (الجدول 4).
ملاحظة: يمكن أن تكون نسبة الأضراس من المنتج PCR (إدراج) إلى متجه لوسيفيراز 3:1. 1:1 أو 2:1. - بإيجاز طرد مركزي الأنبوب ل 10-15 s وحضانة في 16 درجة مئوية بين عشية وضحاها باستخدام دورة الحرارية.
ملاحظة: بدلا من ذلك، يمكن احتضان الأنبوب في 4 درجة مئوية لمدة 2-3 أيام للربط. في هذه الخطوة، سيتم استنساخ إدراج PCR في المنطقة المتمركزة في المصب من الجينات مراسل رينيلا (الشكل5B). ربط miRNAs في UTR المستنسخة 3 'من الجينات يمكن أن تنخفض في نشاط رينيلا. اليراعة لوسيفيراز هو لتطبيع مستويات التعبير رينيلا.
- جعل 20 درجة مئوية من خلطات رد الفعلالربط بما في ذلك ligase الحمض النووي (الجدول 4).
- تحويل ومستعمرة [بّر]
- إضافة مخاليط الربط (3-5 درجة مئوية) في الأنبوب الذي يحتوي على الخلايا المختصة. اضغط بلطف على الأنبوب ويبقيه على الجليد (20 دقيقة).
ملاحظة: قم بإلغاء تجميد الخلايا المختصة على الجليد قبل إضافة خلائط الربط. - بسرعة وبلطف نقل الأنبوب إلى كتلة الحرارة. بعد صدمة الحرارة (42 درجة مئوية لمدة 30 ق-1 دقيقة)، وضع الأنبوب على الجليد لمدة 20 دقيقة.
- نشر الخلايا المختصة على لوحة أجار لوريا-بيرتاني (LB). زراعة الخلايا المختصة في حاضنة (37 درجة مئوية) بين عشية وضحاها.
ملاحظة: Ampicillin (50-100 ميكروغرام / مل) موجود في لوحة أجار. - اختيار مستعمرة فردية وإعادة تعليق E. القولونية في واحدة من أنابيب 8-قطاع تحتوي على المياه فائقة النقية. كرر هذه الخطوة لإعادة تعليق E. القولونية من المستعمرات 4-8 مختارة عشوائيا (الشكل5C).
- نقل 25 ميكرولتر من تعليق القولونية E. في مجموعة أخرى من أنابيب 8-قطاع. الآن، هناك 2 مجموعات من أنابيب تعليق القولونية E.
ملاحظة: أنبوب واحد هو لPCR مستعمرة وآخر هو للتلقيح. E. تعليق القولونية للتلقيح يمكن تخزينها مؤقتا في 4 درجة مئوية (الشكل5C). - إجراء PCR المستعمرة باستخدام تعليق القولونية E. هذه الخطوة هي لتحديد ما إذا كانت المستعمرات تحتوي على إدراج. حدد أفضل المستعمرات لتلقيح وعزل ناقلات لوسيفيراز التي تأوي 3 ' UTR من جين (الشكل5C).
ملاحظة: كرر الخطوة 5.1-5.5 لكل 3 'UTR من الجينات المحددة. اتبع حالة تفاعل PCR الموضحة في الجدول 2 عن طريق استبدال الحمض النووي الجينومي بتعليق E. coli.
- إضافة مخاليط الربط (3-5 درجة مئوية) في الأنبوب الذي يحتوي على الخلايا المختصة. اضغط بلطف على الأنبوب ويبقيه على الجليد (20 دقيقة).
Access restricted. Please log in or start a trial to view this content.
النتائج
ومن المهم أن يكون التأكيد الناجح والدقيق لمستويات ميرنا مهماً لتفسير البيانات التي يمكن من خلالها تصنيف الميرنا على أساس الأدوار المتوقعة لـ miRNAs في تطور المرض وتطوره. تم قياس مستويات ميرنا-107 وميرنا-301 في ثلاثة خطوط خلايا البنكرياس باستخدام PCR الكمي القائم على التحقيق. يمكن أن يزيد تركيب cDN...
Access restricted. Please log in or start a trial to view this content.
Discussion
استراتيجيات لتحديد أهداف ميرنا حسن النية مع وظائف ميرنا ذات الأهمية لا غنى عنها لفهم الأدوار المتعددة للميرنا. يمكن أن يكون تحديد الجينات المستهدفة ميرنا مبدأ ً توجيهياً لتفسير أحداث إشارات الخلية التي تم تضمينها بواسطة miRNAs في الخلية. الكشف عن الجينات المستهدفة الهامة وظيفيا من miRNAs يمكن ...
Access restricted. Please log in or start a trial to view this content.
Disclosures
وليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
وقد تم دعم هذه الدراسة من قبل برنامج بحوث العلوم الأساسية من خلال المؤسسة الوطنية للبحوث الكورية (NRF) الممولة من وزارة التعليم (2017R1D1A3B03035662)؛ وصندوق بحوث جامعة هاليم، 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
References
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved