Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un protocolo in vitro para evaluar los niveles, funciones y genes objetivo asociados de MicroRNA en células tumorales
En este artículo
Resumen
Este protocolo utiliza una reacción en cadena de la polimerasa (PCR) basada en sondas en tiempo real, un ensayo de sulforhodamine B (SRB), 3' regiones no traducidas (3' UTR) clonación, y un ensayo de luciferasa para verificar los genes diana de un miRNA de interés y para entender las funciones de los miRNAs.
Resumen
Los MicroRNAs (miRNAs) son pequeños ARN reguladores que se reconocen para modular numerosas vías de señalización intracelular en varias enfermedades, incluidos los cánceres. Estos pequeños ARN reguladores interactúan principalmente con las 3' regiones no traducidas (3' UTR) de sus ARN mensajeros objetivo (RNN) lo que en última instancia resulta en la inhibición de los procesos de decodificación de ARNM y el aumento de las degradaciones de ARNm objetivo. Basados en los niveles de expresión y las funciones intracelulares, los miRNAs son capaces de servir como factores reguladores de los ARNM oncogénicos y supresores tumorales. La identificación de genes diana de buena fe de un miRNA entre cientos o incluso miles de objetivos predicados computacionalmente es un paso crucial para discernir las funciones y los mecanismos moleculares básicos de un miRNA de interés. Varios programas de predicción de objetivos de miRNA están disponibles para buscar posibles interacciones miRNA-mRNA. Sin embargo, la pregunta más difícil es cómo validar los genes diana directos de un miRNA de interés. Este protocolo describe una estrategia reproducible de métodos clave sobre cómo identificar objetivos de miRNA relacionados con la función de un miRNA. Este protocolo presenta una guía práctica sobre los procedimientos paso a paso para descubrir los niveles de miRNA, funciones y ARNM objetivo relacionados utilizando la reacción en cadena de la polimerasa (PCR) basada en la sonda, el ensayo de sulforhodamine B (SRB) después de una imitación de miRNA en la transfección , la generación de curvas de dosis-respuesta y el ensayo de luciferasa junto con la clonación de 3' UTR de un gen, que es necesario para una comprensión adecuada de las funciones de los miRNAs individuales.
Introducción
Los MicroARN (miRNAs) son los pequeños ARN reguladores que modulan principalmente el proceso de traducción y degradación de los ARN mensajeros (ARN) reaccionando a las 3' regiones no traducidas (3' UTR) en genes diana de buena fe1. La expresión de los miRNAs puede ser regulada por mecanismos transcripcionales y post-transcripcionales. El desequilibrio de estos mecanismos reglamentarios aporta niveles de expresión de miRNAs no controlados y distintivos en numerosas enfermedades, incluidos los cánceres2. Un solo miRNA puede tener múltiples interacciones con diversos ARNm. En consecuencia, un ARNm individual puede ser controlado por varios miRNAs. Por lo tanto, las redes de señalización intracelular están intrincadamente influenciadas por miRNA distintivamente expresadas por las cuales se pueden iniciar y deteriorar trastornos fisiológicos y enfermedades2,3,4, 5 , 6. Aunque la expresión alterada de los miRNAs se ha observado en varios tipos de cánceres, los mecanismos moleculares que modulan las formas de las células cancerosas en conjunto con los miRNAs son todavía en gran medida desconocidos.
La acumulación de evidencia ha estado demostrando que los roles oncogénicos o supresores de tumores de los miRNAs dependen de los tipos de cáncer. Por ejemplo, al apuntar a la caja de cabezal de horquilla o3 (FOXO3), miR-155 promueve la proliferación celular, metástasis y quimiorresistencia del cáncer colorrectal7,8. Por el contrario, la restricción de la invasión de células de glioma es inducida por miR-107 a través de la regulación de la locus de locus homolog proteína 2 (NOTCH2) expresión9. La evaluación de las interacciones miRNA-objetivo en relación con las funciones de miRNA es una parte indispensable para entender mejor cómo los miRNAs regulan varios procesos biológicos tanto en estados sanos como enfermos10. Además, el descubrimiento de objetivos de buena fe de los miRNAs puede proporcionar aún más una estrategia ajustada para una terapia basada en miRNA con varios medicamentos contra el cáncer. Sin embargo, el principal desafío en el campo de los miRNAs es la identificación de objetivos directos de los miRNAs. Aquí, los métodos detallados se presentan como enfoques experimentales reproducibles para la determinación del gen objetivo del miRNA. El diseño experimental exitoso para la identificación del objetivo miRNA implica varios pasos y consideraciones (Figura1). La comparación de los niveles de miRNA maduros en células tumorales y células normales puede ser uno de los procedimientos comunes para seleccionar un miRNA de interés (Figura1A). El estudio funcional de un miRNA seleccionado para detectar los efectos de un miRNA en la proliferación celular es importante para reducir la lista de los mejores objetivos candidatos potenciales de un miRNA de interés (Figura1B). Sobre la base de las funciones validadas experimentalmente de los miRNAs, se requiere una revisión sistemática de la literatura y la base de datos en compañía con un programa de predicción de objetivos de miRNA para buscar la información más relevante sobre las funciones genéticas (Figura1C). La identificación de genes diana reales de un miRNA de interés se puede lograr mediante la implementación de experimentos como el ensayo de luciferasa junto con la clonación de 3' UTR de un gen, PCR en tiempo real, y la hincha occidental (Figura1D). El objetivo del protocolo actual es proporcionar métodos integrales de experimentos clave, la reacción en cadena de la polimerasa en tiempo real basada en sondas (PCR), el ensayo de sulforhodamine B (SRB) después de una transfección mimimetial, la generación de curvas de dosis-respuesta y un ensayo de imitación de miRNA, la generación de curvas de dosis-respuesta y ensayo de luciferasa junto con la clonación de 3' UTR de un gen. El protocolo actual puede ser útil para una mejor comprensión de las funciones de los miRNAs individuales y la implicación de un miRNA en la terapia contra el cáncer.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Análisis de expresión de microARN maduro (miRNA)
-
Síntesis madura de ADN complementario de miRNA (ADNc)
- Añadir 254 ng de ARN total y 4,5 l de mezclas de desoxirribonucleasa I (DNase I), y luego añadir agua ultrapura en tubos de tiras PCR para hacer hasta 18 l (Figura2A). Preparar la reacción para cada muestra total de ARN purificada a partir de varias líneas celulares utilizando la cantidad suficiente de mezclas DNase I basadas en el número total de reacciones.
NOTA: Las mezclas de DNase I se componen de DNase I (1,8 l), inhibidor de ribonucleasa (0,3 l) y 25 mM de MgCl2 (2,4 l). Para obtener arnal total de forma reproducible, se aplica un método de extracción basado en columnas en lugar de utilizar un método de extracción basado en fenol-cloroformo. Se informó que el rendimiento de extracción de algunos miRNAs puede variar dependiendo del número de células cuando se utiliza un método de extracción basado en fenol-cloroformo11,12. - Incubar los tubos en un ciclor térmico. Ejecuta los tubos durante 10 min a 37oC, y desactiva la DNase I por 5 min de incubación a 90oC. Coloque inmediatamente los tubos en hielo después de la incubación.
- Transferir 7,1 l de DNase I trataron el ARN total en 2 conjuntos de tubos nuevos y luego añadir 1,5 ml de imprimaciones antisentido para el gen de gliceraldehído-3-fosfato deshidrogenasa (GAPDH) (Figura2B).
NOTA: La cantidad de ARN total para la síntesis de ADNc se convierte en 100 ng en este paso. La concentración de stock de imprimaciones antisentido GAPDH es de 10 M. La adición de imprimaciones antisentido GAPDH es para la generación de cDNAS GAPDH utilizando un método de imprimación específico del género. - Incubar los tubos utilizando un ciclor térmico. Comience a 80 oC durante 5 min seguido de la reacción a 60 oC durante 5 min. Coloque inmediatamente los tubos sobre hielo después de la incubación.
- Añadir 3,4 l de mezclas enzimáticas de transcripción inversa (RT) en cada reacción (Figura2B). Las mezclas de enzimas RT se componen de trifosfatos de desoxirribonucleótido de 100 mM (0,15 l), tampón rt de 10 x (1,5 ml), inhibidor de ribonucleasa (0,75 l) y enzima de transcripción inversa (1 l). Preparar suficiente cantidad de mezclas en función del número total de reacciones.
- Añadir 3 sl de imprimaciones RT de 5x para un miRNA específico en cada reacción (Figura2B).
NOTA: El volumen total es de 15 s para cada reacción. - Ejecute los tubos con un ciclor térmico. Comience a 16 oC durante 30 minutos seguido de la reacción a 42 oC durante 30 min, y finalmente a 85 oC durante 5 min. Mantenga a 4 oC durante el tiempo restante (Figura2B). Los CDNA de una sola cadena se generan en este paso para un miRNA específico y un gen GAPDH en el mismo tubo.
- Añadir 254 ng de ARN total y 4,5 l de mezclas de desoxirribonucleasa I (DNase I), y luego añadir agua ultrapura en tubos de tiras PCR para hacer hasta 18 l (Figura2A). Preparar la reacción para cada muestra total de ARN purificada a partir de varias líneas celulares utilizando la cantidad suficiente de mezclas DNase I basadas en el número total de reacciones.
-
Reacción en cadena de la polimerasa en tiempo real (PCR) y análisis de datos
- Diluir cada ADNc con agua ultrapura en una proporción de 1:49.
- Preparar las mezclas de reacción para un miRNA y GAPDH específicos (Tabla1). Para la detección de un miRNA y GAPDH específicos, configure reacciones triplicados para cada muestra de ADNc.
- Realice el PCR en tiempo real y el análisis de datos (Figura2C). Analizar datos utilizando el método cT comparativo13,14.
2. MicroRNA (miRNA) MimicTransfection
NOTA: miRNA-107 se selecciona en el paso 1. Dado que el miRNA-107 está regulado hacia abajo en las células tumorales en comparación con las células normales, se puede especular que el miRNA-107 es un miRNA supresor tumoral. En el caso de un miRNA que está regulado en célulastumorales en comparación con las células normales (por ejemplo, miRNA-301), los oligonucleótidos antisentido contra el miRNA-301 se pueden aplicar para los pasos 2, 3 y 4.
- Cuente las células con un dispositivo de cámara de conteo y placa reline las células en una placa de 96 pocillos. La densidad celular es de 2 x 103 celdas/100 l para cada pocil. No utilice medios de cultivo celular que contengan penicilina-estreptomicina (P/S) porque P/S puede reducir la eficiencia de transfección.
-
Preparar un conjunto de mezclas de transfección para transfectar las células en varias concentraciones finales de imitación de control de miRNA y miRNA-107 al día siguiente (Figura3).
- A partir de la acción (concentración de 25 m) de imitación de control de miRNA o miRNA-107, diluir y añadir la cantidad correspondiente de imitación de control o miRNA-107 imitación en los medios de suero reducido junto con un reactivo de transfección utilizando tubos de microcentrífuga (Figura 3A). Mezcle suavemente el oligo que contiene las mezclas con un micropipeta. La cantidad total de oligos (miRNA imita el control + miRNA-107 imitación) debe ser la misma en cada pozo. Los pozos en blanco incluyen 100 s de medios de cultivo celular y medios de suero reducido que contienen un reactivo de transfección sin células.
- Después de una incubación de 10 minutos en una campana de cultivo celular, mezcle suavemente el oligo que contiene mezclas de nuevo y luego agregue 50 s de las mezclas en cada poca. Mantener las células transfectóte en una incubadora de cultivo celular. Sustituya el reactivo de transfección que contiene medios por los medios de cultivo celular fresco que contienen tanto suero bovino fetal (FBS) como P/S después de 6-12 h de incubación. Incubar más las células durante 72 h. La duración total del tratamiento de miRNA mimic es de 96 h.
3. Ensayo de Sulforhodamine B (SRB)
-
Fijación celular
- Retire los medios de cultivo celular en cada pocal de la placa y llene rápidamente 100 éL de ácido tricloroacético (TCA) al 10% en cada pocal. Aspirar cuidadosamente los medios de cultivo celular de cada pozo para evitar cualquier daño celular y desprendimiento de la parte inferior.
NOTA: Preparar 40% TCA añadiendo 20 g de polvo tCA en 50 ml de agua destilada. A partir de 40% TCA, hacer 10% TCA mediante la dilución 40% TCA con agua destilada en una relación de dilución 1:3. - Conservar la placa que contiene 10% de TCA en nevera (4oC) durante 1 h.
- Lave el plato varias veces sumergiendo en la bañera de agua y séquelo. Retire el exceso de agua del interior de los pozos tocando la placa hasta que no quede agua en los pozos. Deje la placa en un banco de laboratorio para secarla antes de ir al siguiente paso.
- Retire los medios de cultivo celular en cada pocal de la placa y llene rápidamente 100 éL de ácido tricloroacético (TCA) al 10% en cada pocal. Aspirar cuidadosamente los medios de cultivo celular de cada pozo para evitar cualquier daño celular y desprendimiento de la parte inferior.
-
Tinción celular
- Pipetear 50 l de solución SRB del 0,4% en cada pozo, incluidos los pozos en blanco. Agitar suavemente el plato hasta que la solución SRB 0.4% cubra constantemente la parte inferior de los pozos.
NOTA: Preparar y utilizar 0.4% SRB solución mediante la adición de 0.4 g de polvo SRB en 100 mL de 1% ácido acético. Agitar la solución cuidadosamente para mezclarla. Envuelva la botella de solución SRB al 0,4% en un material de protección ligera como papel de aluminio. Almacene 0.4% Solución SRB en nevera. - Después de la incubación durante 40 min a 60 min, lavar el plato ensimándolo con 1% de ácido acético. Lave el plato hasta que el tinte sin encuaderbrar esté totalmente lavado (Figura3B).
- Deje la placa en un banco de laboratorio para secarla antes de ir al siguiente paso.
NOTA: La placa debe secarse por completo antes de ir al paso 3.3.
- Pipetear 50 l de solución SRB del 0,4% en cada pozo, incluidos los pozos en blanco. Agitar suavemente el plato hasta que la solución SRB 0.4% cubra constantemente la parte inferior de los pozos.
-
Medición de la absorbancia
- Pipetear 100 l de solución base Tris (10 mM) en los pozos correspondientes, incluidos los pozos en blanco. Conservar la placa en una coctelera durante 10 minutos.
4. Generación de una curva de dosis-respuesta
- Analice los datos del ensayo SRB en una hoja de cálculo. Restar la absorbancia en blanco de los valores de absorbancia de cada grupo y calcular el promedio (AVE) y la desviación estándar (STD) de los valores de absorbancia de cada grupo.
- Calcular el porcentaje de absorbancia media (AVE%) y la de la desviación estándar (STD%) de cada grupo utilizando los valores de absorbancia del ensayo sRB.
NOTA: El AVE% del grupo tratado de imitación de control de miRNA es 100%. Calcular el STD% utilizando la siguiente fórmula: STD% ( STD de cada grupo / AVE absorbancia de control imitar grupo tratado) x 100. - Importe los datos sin procesar, incluidas las concentraciones de tratamiento, AVE% y STD% en el software alineando verticalmente esos datos. Puesto que el registro 0 no está definido, fije la primera concentración del eje X a un valor que esté cerca de 0 (por ejemplo,., 0.01).
- Haga clic en la pestaña Crear gráfico y elija Barras de error de dispersiónsimple. Seleccione Columnas de hoja de trabajo como valores de símbolo y haga clic en Siguiente. En el panel de formato de datos, seleccione pares XY y haga clic en Siguiente. Seleccione las columnas de datos correspondientes en el panel de datos seleccionado. Haga clic en el botón Finalizar para crear el trazado.
NOTA: El eje X representa las concentraciones, el eje Y indica el porcentaje de absorbancia media de cada concentración (AVE%) y las barras de error señalan el porcentaje de desviación estándar de cada concentración (STD%). - Haga doble clic en el eje X para modificar el tipo de escala y la escala del eje. Cambie el tipo de escala de lineal a registro. Modifique el número de rango inicial y final a 0.01 y 200, respectivamente.
-
Haga clic con el botón derecho en cualquier gráfico de dispersión, elija Ajuste de curva y vaya a la subcategoría Definido por el usuario. Seleccione Curva de dosis-respuesta, haga clic en Siguiente botones y, a continuación, haga clic en el botón Finalizar. La curva dosis-respuesta se genera ahora junto con una pestaña de informe (Figura4A).
- Para introducir la ecuación 1 en el software para la generación de una curva dosis-respuesta, haga clic en la ficha Análisis y seleccione Asistente de regresión. Vaya a Definido por el usuario en la categoría de ecuación y, a continuación, haga clic en el botón Nuevo. Inserte la ecuación 1, las variables, los parámetros iniciales y las restricciones en los cuadros en blanco correspondientes (figura4B,C). Haga clic en el botón Agregar como y establezca el nombre de la ecuación como Curva dosis-respuesta. El nombre de la ecuación se genera ahora en la subcategoría Definido por el usuario en la categoría de ecuación. f indica el porcentaje de viabilidad celular (% viabilidad celular) en la Ecuación 1.
Ecuación 1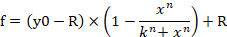
- Vaya a la pestaña de informe y, a continuación, compruebe los valores n, k y R.
NOTA: y0 indica 100% viabilidad celular del control de miRNA imita el grupo tratado, n indica el coeficiente de tipo Hill (la pendiente de una gráfica), k indica la concentración de miRNA-107 imitación que produce un 50% del miRNA-107 imitación efecto máximo (la mitad concentración inhibitoria máxima, IC50) y R indica la fracción residual no afectada (la fracción de resistencia)15. La ecuación utilizada para generar una curva dosis-respuesta reconoce el rango de valor y0 a R (si existe) como 100% (Figura4A). Por lo tanto, es necesario adquirir el valor k ajustado (IC50) que se calcula en función del rango de y0 a un valor de cero (Figura4A). El k ajustado (IC50) junto con otros valores ICx (por ejemplo,., IC10 a IC90)se pueden obtener utilizando la Ecuación 2, que se deriva de la Ecuación 1. La derivación de la Ecuación 2 de la Ecuación 1 se indica en la Figura Suplementaria 1.
Ecuación 2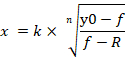
- Haga doble clic en el botón izquierdo del ratón en la celda en la que se aplica la Ecuación 2. Usando la Ecuación 2 y los parámetros de la curva dosis-respuesta generada, está disponible para calcular los valores ajustados de ICx, que van desde IC10 a IC90 (Figura4D).
- Introduzca el signo igual seguido de la fórmula que comienza con un corchete en la celda. Al introducir la fórmula, fije el valor de n, k y R como las referencias de celda absoluta agregando el signo de dólar a la columna y fila correspondientes, de modo que estos valores fijos no se cambiarán al rellenar automáticamente la fórmula en las filas (Figura4D). Alternativamente, los valores ajustados se pueden calcular manualmente usando la Ecuación 2.
NOTA: El valorIC 90 no se determina porque el valor R es mayor que 10. Además, si el valor R es superior a 20, tampoco se determina el valor de IC80 (Figura4D).
5. Verificación del gen objetivo directo de un MicroRNA de interés
NOTA: Después de realizar el experimento funcional como el ensayo SRB, miRNA-107 se confirma como un miRNA supresor tumoral y es altamente factible que miRNA-107 se dirija directamente a los oncogenes. Compruebe la lista de todos los genes diana previstos utilizando un programa de predicción de objetivos de miRNA como TargetScan (http://www.targetscan.org/vert_71/), y luego reduzca a los objetivos candidatos potenciales en función de la función de un gen en bases de datos como PubMed y GeneCards.
- Diseño de imprimación para la clonación de 3' región no traducida (UTR)
- Ponga el nombre de un gen en GeneCards (https://www.genecards.org/) y haga clic en Símbolo de un gen. Evalúe al navegador del genoma de Ensembl haciendo clic en Ensembl ID de un gen y, a continuación, haga clic en Id de transcripciones en la tabla de transcripción. Después de eso, haga clic en Exons existió en la lista de pantallas basadas en transcripción de la izquierda.
- Copie las secuencias de nucleótidos del UTR de 3' y péguelo en el programa de diseño de imprimación. Copie las secuencias de nuevo de este programa y péguelas en un procesador de textos. Compruebe la presencia de secuencias de unión de miRNA, así como la presencia de sitios de enzimas de restricción utilizados para la clonación.
NOTA: Si no hay sitios de reconocimiento de enzimas de restricción dentro del UTR de 3', las enzimas de restricción seleccionadas para la clonación se pueden utilizar para el siguiente paso. - En el programa de diseño de imprimación, acepte las secuencias UTR de 3' y comience a diseñar las imprimaciones delanteras e inversas con la siguiente condición. Longitud: 20-30 nucleótidos, Tm: 45-58 oC, GC%: 40-60%. La diferencia entre los valores Tm de las dos imprimaciones debe ser inferior a 5 oC. Las secuencias de imprimación utilizadas en este estudio se proporcionan en la Figura Complementaria 2. Añadir secuencias de reconocimiento de enzimas de restricción, así como 4 nucleótidos aleatorios a las imprimaciones diseñadas.
- Gradient e ITP
- Preparar 25 l de mezclas de reacción de PCR, incluidas las imprimaciones diseñadas por una temperatura de recocido (Tabla2). Preparar suficiente cantidad de mezclas en función del número total de reacciones. Mezcle la solución pipeteando y agregue 25 ml de mezclas de reacción en cada tubo. Centrifugar los tubos durante unos segundos.
- Realice 35-40 ciclos de PCR desde el paso de desnaturalización hasta el paso de extensión. Configurar el ciclo de PCR como los siguientes pasos: 98 oC para 1 min (1 ciclo, paso de activación de la polimerasa), 95 oC para 10 s (paso de desnaturalización), 45 oC-68 oC para 30 s (paso de recocido), 68 oC (paso de extensión, 10 s-1 min por 1000 bp), 68 oC para 3 min (paso de terminación) , y finalmente enfriar a 4 oC.
- Ejecuta los productos PCR y revisa las bandas en un gel de agarosa del 1% con escaleras de ADN. Encuentra la mejor temperatura de recocido (Figura 5A). Amplifique 3' UTR de un gen de nuevo utilizando la mejor temperatura de recocido para el siguiente paso.
- Doble digestión
- Haga las mezclas de reacción, incluidas dos enzimas de restricción, XhoI (o AsiSI) y NotI, en un tubo (Tabla 3). Incubar las mezclas durante 3-4 h utilizando un baño de agua (37 oC).
- Ejecuta los productos de doble digestión en un gel de agarosa del 1% y luego corta las bandas bajo luz UV. En el caso de los vectores de luciferasa, antes de correr con un gel, reaccionar vectores de doble digestión con 10 U de fosfatasas alcalinas para otra 1 h para evitar una recircularización durante el paso de ligadura.
- Purificar los productos de PCR de doble digestión y los vectores de luciferasa de las bandas extirpadas.
- Ligadura de productos PCR en los vectores de luciferasa
- Hacer 20 l de mezclas de reacción de ligadura, incluida la ligasa de ADN (Tabla4).
NOTA: La relación molar del producto PCR (inserción) al vector de luciferasa puede ser 3:1. 1:1 o 2:1. - Centrifugar brevemente el tubo durante 10-15 s e incubar a 16 oC durante la noche utilizando un ciclor térmico.
NOTA: Alternativamente, el tubo se puede incubar a 4 oC durante 2-3 días para la ligadura. En este paso, el inserto de PCR se clonará en la región posicionada aguas abajo de un gen de reportero de renilla (Figura5B). La unión de miRNAs en el UTR clonado de 3' de un gen puede disminuir en la actividad de la renilla. La luciferasa de la luciérnaga es para la normalización de los niveles de expresión de renilla.
- Hacer 20 l de mezclas de reacción de ligadura, incluida la ligasa de ADN (Tabla4).
- Transformación y PCR de colonias
- Agregue las mezclas de ligadura (3-5 l) en el tubo que contiene células competentes. Toque suavemente el tubo y manténgalo en hielo (20 min).
NOTA: Descongele las células competentes sobre hielo antes de añadir las mezclas de ligadura. - Transfiera el tubo a un bloque de calor de forma rápida y suave. Después de un choque térmico (42 oC durante 30 s-1 min), coloque el tubo en hielo durante 20 minutos.
- Extienda las células competentes en la placa de agar Luria-Bertani (LB). Cultivar células competentes en una incubadora (37 oC) durante la noche.
NOTA: La ampicilina (50-100 g/ml) se encuentra en la placa de agar. - Escoge una colonia individual y resuspende E. coli en uno de los tubos de 8 tiras que contienen agua ultrapura. Repita este paso para resuspender E. coli de 4-8 colonias seleccionadas al azar (Figura5C).
- Transfiera 25 ml de suspensión de E. coli a otro conjunto de tubos de 8 tiras. Ahora, hay 2 juegos de tubos de suspensión de E. coli.
NOTA: Un tubo es para la PCR de la colonia y otro es para la inoculación. La suspensión de E. coli para la inoculación se puede almacenar temporalmente a 4 oC (figura5C). - Realice la PCR de colonia utilizando la suspensión De. coli. Este paso es para determinar si las colonias contienen una inserción. Seleccione las mejores colonias para inocular y aislar vectores de luciferasa que albergan 3' UTR de un gen (Figura 5C).
NOTA: Repita el paso 5.1-5.5 para cada UTR de 3' de genes seleccionados. Siga la condición de la reacción de PCR que se muestra en la Tabla 2 reemplazando el ADN genómico por la suspensión de E. coli.
- Agregue las mezclas de ligadura (3-5 l) en el tubo que contiene células competentes. Toque suavemente el tubo y manténgalo en hielo (20 min).
- Ensayo de Luciferase
- Prepare un plato de 24 pocillos. Utilice 1-2 x 104 celdas en medios de cultivo celular de 500 ml para cada poc. No utilice medios de cultivo celular que contengan P/S para la transfección, ya que el uso de P/S puede reducir la eficiencia de la transfección.
- Transfect 50 ng de vectores de luciferasa en las células con control imitación o una imitación de miRNA específica utilizando un reactivo de transfección (Figura 5D). Si el cribado de los efectos de un miRNA específico imita a más de una concentración, mantenga la cantidad total de oligos igual en cada pocal (ver paso 2).
- Lave el interior de los pozos dos veces usando solución salina tamponada de fosfato (PBS) al día siguiente.
- Aplicar 200 l de reactivo de lisis en los pozos y llevar a cabo suficientemente la lisis celular antes de medir la actividad de la luciferasa.
NOTA: Mantenga la placa en una placa temblorosa al menos 15 minutos. - Transfiera 5-10 l de lisado celular en el nuevo tubo y agregue 100 l de reactivo I. Mezcle inmediatamente la solución pipeteando y lea la actividad luciferasa de la luciérnaga utilizando un luminómetro.
NOTA: Lea la actividad de la luciferasa de luciérnaga durante 10-15 s. - Añadir 100 l de reactivo II en el mismo tubo, y luego mezclar pipeteando dos veces. Lea la actividad de la renilla luciferasa durante 10-15 s utilizando un luminómetro. Repita los pasos 5.6.5 y 5.6.6 para cada muestra.
- Calcular la relación entre renilla y luciérnaga (Figura5E).
NOTA: La actividad de la luciérnaga representa la eficiencia de la transfección de las construcciones de luciferasa en las células.
Access restricted. Please log in or start a trial to view this content.
Resultados
La confirmación exitosa y precisa de los niveles de miRNA es importante para la interpretación de los datos mediante los cuales la clasificación de los miRNAs es posible sobre la base de las funciones previstas de los miRNAs en el desarrollo y progresión de una enfermedad. Los niveles de miRNA-107 y miRNA-301 se midieron en tres líneas celulares de páncreas utilizando el PCR cuantitativo basado en sondas. La síntesis de cDNAs de un miRNA específico y un gen de referencia en la misma reacción puede aumentar la re...
Access restricted. Please log in or start a trial to view this content.
Discusión
Las estrategias para la determinación de objetivos de miRNA de buena fe con las funciones de un miRNA de interés son indispensables para la comprensión de múltiples funciones de los miRNAs. La identificación de genes diana de miRNA puede ser una guía para interpretar los eventos de señalización celular modulados por los miRNAs en una célula. Una revelación de genes diana funcionalmente importantes de los miRNAs puede proporcionar el conocimiento fundamental para desarrollar una terapia basada en miRNA en el cá...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este estudio fue apoyado por el Programa de Investigación de Ciencia Sacial A través de la Fundación Nacional de Investigación de Corea (NRF) financiado por el Ministerio de Educación (2017R1D1A3B03035662); y el Fondo de Investigación de la Universidad de Hallym, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
Referencias
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados