É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Protocolo in vitro para avaliação de níveis de MicroRNA, funções e genes alvo associados em células tumorais
Neste Artigo
Resumo
Este protocolo utiliza uma reação em cadeia da polimerase em tempo real (PCR), um ensaio de sulforhodamina B (SRB), 3 ' regiões não traduzidas (3 ' UTR) clonagem e um ensaio de luciferase para verificar os genes alvo de um miRNA de interesse e para compreender as funções de miRNAs.
Resumo
MicroRNAs (miRNAs) são pequenas RNAs regulatórias que são reconhecidas para modular inúmeras vias de sinalização intracelular em várias doenças, incluindo cânceres. Esses pequenos RNAs regulatórios interagem principalmente com as 3 ' regiões não traduzidas (3 ' UTR) de seus RNAs de mensageiro alvo (mRNAs), resultando na inibição dos processos de decodificação de mRNAs e no aumento das degradações de mRNA alvo. Com base nos níveis de expressão e nas funções intracelulares, os miRNAs são capazes de servir como fatores regulatórios de mRNAs oncogênicas e supressoras de tumores. A identificação de genes-alvo de boa-fé de um miRNA entre centenas ou mesmo milhares de alvos computacionalmente previstos é um passo crucial para discernir os papéis e os mecanismos moleculares básicos de um miRNA de interesse. Vários programas de predição de alvo de miRNA estão disponíveis para pesquisar possíveis interações de miRNA-mRNA. No entanto, a questão mais desafiadora é como validar genes alvo diretos de um miRNA de interesse. Este protocolo descreve uma estratégia reprodutível de métodos chaves em como identificar alvos de miRNA relativos à função de um miRNA. Este protocolo apresenta um guia prático em procedimentos passo a passo para descobrir níveis de miRNA, funções e mRNAs alvo relacionados usando a reação em cadeia da polimerase em tempo real baseada em sonda (PCR), o ensaio de sulforhodamina B (SRB) após uma transfecção mímica de miRNA , geração da curva da dose-resposta, e ensaio do luciferase junto com a clonagem de 3 ' UTR de um gene, que seja necessário para a compreensão apropriada dos papéis de miRNAs individuais.
Introdução
MicroRNAs (miRNAs) são os pequenos RNAs regulatórios que modulam principalmente o processo de tradução e degradação de RNAs mensageiro (mRNAs), reagindo às 3 ' regiões não traduzidas (3 ' UTR) em genes alvo de boa-fé1. A expressão de miRNAs pode ser regulada por mecanismos transcricional e pós-transcricional. O desequilíbrio desses mecanismos reguladores traz níveis de expressão miRNAs descontrolados e distintos em inúmeras doenças, incluindo os cânceres2. Um único miRNA pode ter interações múltiplas com mRNAs diversos. Correspondentemente, um mRNA individual pode ser controlado por vários miRNAs. Portanto, as redes de sinalização intracelular são intrinsecamente influenciadas por miRNAs distintivamente expressas pelas quais distúrbios fisiológicos e doenças podem ser iniciados e deteriorados2,3,4 , 5. º , 6. embora a expressão alterada de miRNAs tenha sido observada em vários tipos de cânceres, os mecanismos moleculares que modulam os modos de células cancerosas em conjunto com miRNAs ainda são largamente desconhecidos.
A acumulação de evidências tem sido evidenciando que os papéis oncogênicos ou supressores de tumores de miRNAs dependem dos tipos de cânceres. Por exemplo, ao segmentar a caixa de os O3 (FOXO3), o miR-155 promove a proliferação celular, metástase e quimiorresistência do câncer colorretal7,8. Ao contrário, a limitação da invasão da pilha do glioma é induzida por miR-107 através da regulação da expressão9da proteína 2 do homólogo do entalhe do locus neurogênica (NOTCH2). A avaliação das interações de miRNA-Target em conexão com as funções de miRNA é uma parte indispensável para entender melhor como os miRNAs regulam vários processos biológicos em Estados saudáveis e doentes10. Além disso, a descoberta de alvo (s) de bona fide de miRNAs pode ainda fornecer uma estratégia aperfeiçoada para uma terapia baseada em miRNA com vários medicamentos anticancerígenos. No entanto, o principal desafio no campo dos miRNAs é a identificação de alvos diretos de miRNAs. Aqui, métodos detalhados são apresentados como abordagens experimentais reprodutíveis para a determinação do gene alvo de miRNA. O projeto experimental bem-sucedido para a identificação do alvo de miRNA envolve várias etapas e considerações (Figura 1). A comparação dos níveis de miRNA maduros nas células tumorais e nas células normais pode ser um dos procedimentos comuns para selecionar um miRNA de interesse (Figura 1a). O estudo funcional de um miRNA selecionado para detectar os efeitos de um miRNA na proliferação celular é importante para estreitar a lista dos melhores alvos potenciais candidatos de um miRNA de interesse (Figura 1b). Com base nas funções experimentalmente validadas de miRNAs, é necessária uma revisão sistemática da literatura e da base de dados em empresa com um programa de predição de alvo de miRNA para pesquisar as informações mais relevantes sobre as funções genéticas (Figura 1C). A identificação de genes alvo reais de um miRNA do interesse pode ser conseguida implementando experiências tais como o ensaio do luciferase junto com a clonagem de 3 ' UTR de um gene, PCR do tempo real, e mancha ocidental (Figura 1D). O objetivo do protocolo atual é fornecer métodos abrangentes de experimentos principais, a reação em cadeia da polimerase em tempo real baseada em sonda (PCR), o ensaio de sulforhodamina B (SRB) após uma transfecção mímica de miRNA, geração de curva de dose-resposta e o ensaio de luciferase juntamente com a clonagem de 3 ' UTR de um gene. O protocolo atual pode ser útil para uma compreensão melhor das funções de miRNAs individuais e da implicação de um miRNA na terapia de cancro.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. maduro MicroRNA (miRNA) análise de expressão
-
Síntese de DNA complementar maduro de miRNA (cDNA)
- Adicionar 254 ng de RNA total e 4,5 μL de misturas de desoxirribonuclease I (DNase I) e, em seguida, adicionar água ultrapura em tubos de tiras de PCR para fazer até 18 μL (Figura 2a). Prepare a reação para cada amostra de RNA total purificada de várias linhas celulares usando quantidade suficiente de misturas de DNase I com base no número total de reações.
Nota: As misturas de DNase I são compostas por DNase I (1,8 μL), inibidor da ribonuclease (0,3 μL) e 25 mM MgCl2 (2,4 μL). Para obter o RNA total de forma revisível, um método de extração baseado em coluna foi aplicado em vez de usar um método de extração com base em fenol-clorofórmio. Foi relatado que o rendimento de extração de alguns miRNAs pode ser variado dependendo do número de células ao usar um método de extração baseado em fenol-clorofórmio11,12. - Incubar os tubos em um ciclador térmico. Executar os tubos por 10 min a 37 ° c, e calor-inactivate DNase I por 5 min incubação em 90 ° c. Coloque imediatamente os tubos no gelo após a incubação.
- Transferência 7,1 μL de DNase eu tratei o RNA total em 2 jogos de tubos novos e adiciono então 1,5 μL de primers antisense para o gene do desidrogenase do glyceraldehyde-3-fosfato (GAPDH) (Figura 2b).
Nota: A quantidade de RNA total para a síntese do cDNA transforma-se 100 ng nesta etapa. A concentração de estoque de primers antisense GAPDH é de 10 μM. a adição de primers antisense GAPDH é para a geração de cDNAs GAPDH usando um método de primer específico para genes. - Incubar os tubos usando um termociclador. Começar a 80 ° c durante 5 min seguido da reacção a 60 ° c durante 5 min. Coloque imediatamente os tubos no gelo após a incubação.
- Adicionar 3,4 μL de misturas de enzimas de transcrição reversa (RT) em cada reação (Figura 2b). As misturas enzimáticas RT são compostas por 100 mM de desoxirribonucleotídeo trifosfatos (0,15 μL), tampão 10x RT (1,5 μL), inibidor de ribonuclease (0,75 μL) e enzima de transcrição reversa (1 μL). Prepare quantidade suficiente de misturas com base no número total de reações.
- Adicionar 3 μL de primers de 5x RT para um miRNA específico em cada reação (Figura 2b).
Nota: O volume total é de 15 μL para cada reação. - Executar os tubos usando um termociclador. Começar a 16 ° c durante 30 min seguido da reacção a 42 ° c durante 30 min e, finalmente, a 85 ° c durante 5 min. Segure a 4 ° c para qualquer tempo restante (Figura 2b). Os cDNAs único-encalhado são gerados nesta etapa para um miRNA específico e um gene de GAPDH no mesmo tubo.
- Adicionar 254 ng de RNA total e 4,5 μL de misturas de desoxirribonuclease I (DNase I) e, em seguida, adicionar água ultrapura em tubos de tiras de PCR para fazer até 18 μL (Figura 2a). Prepare a reação para cada amostra de RNA total purificada de várias linhas celulares usando quantidade suficiente de misturas de DNase I com base no número total de reações.
-
Reação em cadeia da polimerase em tempo real (PCR) e análise de dados
- Diluir cada cDNA com água ultrapura na relação 1:49.
- Prepare as misturas de reacção para um miRNA específico e GAPDH (tabela 1). Para a detecção de um determinado miRNA e GAPDH, configurar reações triplicado para cada amostra de cDNA.
- Realize a PCR em tempo real e a análise de dados (Figura 2C). Analise os dados utilizando o método comparativo CT 13,14.
2. MicroRNA (miRNA) MimicTransfection
Nota: o miRNA-107 é seleccionado a partir do passo 1. Desde que miRNA-107 é para baixo-regulado em pilhas do tumor comparado com as pilhas normais, pode-se especficar que miRNA-107 é um miRNA supressor do tumor. No caso de um Mirna que seja regulado em pilhas do tumor comparado com as pilhas normais (por exemplo, Mirna-301), os oligonucleotídeos antisense de encontro a Mirna-301 podem ser aplicados para as etapas 2, 3, e 4.
- Conte as células com um dispositivo de câmara de contagem e placa as células em uma placa de 96 bem. A densidade celular é de 2 x 103 células/100 μl para cada poço. Não use meios de cultura celular contendo penicilina-estreptomicina (P/S) porque P/S pode reduzir a eficiência do transfection.
-
Prepare um conjunto de misturas de transfecção para transfecção as células em várias concentrações finais de mímica de controle de Mirna e mímica de Mirna-107 no dia seguinte (Figura 3).
- Do estoque (concentração de 25 μM) do mímico do controle de miRNA ou do mímico de miRNA-107, diluir e adicionar a quantidade correspondente de imitações do controle ou de miRNA-107 imitam nos meios de soro reduzidos junto com um reagente do transfection usando os tubos do microcentrifugador (Figura 3a). Misture suavemente o oligo contendo misturas usando uma micropipeta. A quantidade total de oligos (controle mímico de miRNA + mímica de miRNA-107) deve ser mesma em cada poço. Os poços em branco incluem 100 μL de meios de cultura celular e meios séricos reduzidos contendo um reagente de transfecção sem células.
- Depois de uma incubação de 10 min em uma capa de cultura celular, misture suavemente o oligo contendo misturas novamente e, em seguida, adicione 50 μL das misturas em cada poço. Mantenha as células transfectadas em uma incubadora de cultura celular. Substitua o reagente do transfection que contem meios com os meios de cultura frescos da pilha que contêm o soro fetal bovino (FBS) e o P/S após a incubação de 6-12 h. Incubar ainda mais as células para 72 h. A duração total do tratamento do mímico de miRNA é 96 h.
3. ensaio de sulforhodamina B (SRB)
-
Fixação celular
- Retire o meio de cultura celular em cada poço da placa e preencha prontamente 100 μL de ácido tricloroacético a 10% (TCA) em cada poço. Aspirar cuidadosamente os meios de cultura celular de cada poço para evitar qualquer dano celular e descolamento do fundo.
Nota: Prepare 40% TCA adicionando 20 g de pó de TCA em 50 mL de água destilada. De 40% de TCA, faça 10% TCA diluindo 40% TCA com água destilada em uma relação da diluição 1:3. - Mantenha a placa contendo 10% de TCA num frigorífico (4 ° c) durante 1 h.
- Lave a placa várias vezes submergindo na cuba de água e seque-a. Retire o excesso de água do interior dos poços tocando na placa até que não haja água deixada em poços. Deixe a placa em um banco do laboratório para secá-la antes de ir à etapa seguinte.
- Retire o meio de cultura celular em cada poço da placa e preencha prontamente 100 μL de ácido tricloroacético a 10% (TCA) em cada poço. Aspirar cuidadosamente os meios de cultura celular de cada poço para evitar qualquer dano celular e descolamento do fundo.
-
Coloração celular
- Pipeta 50 μL de 0,4% de solução de SRB em cada poço, incluindo poços em branco. Agitar suavemente a placa até 0,4% SRB solução consistentemente cobre a parte inferior dos poços.
Nota: Prepare e use 0,4% de solução de SRB adicionando 0,4 g de pó de SRB em 100 mL de ácido acético a 1%. Agitar a solução cuidadosamente para misturá-lo. Enrole o frasco de 0,4% de solução de SRB em um material protetor claro tal como a folha de alumínio. Armazene 0,4% de solução de SRB em um refrigerador. - Após a incubação para 40 min a 60 min, lave a placa enxaguando-a com 1% de ácido acético. Lave a placa até que o corante não acoplado seja totalmente lavado (Figura 3B).
- Deixe a placa em um banco do laboratório para secá-la antes de ir à etapa seguinte.
Nota: A placa deve ser completamente seca antes de ir para o passo 3,3.
- Pipeta 50 μL de 0,4% de solução de SRB em cada poço, incluindo poços em branco. Agitar suavemente a placa até 0,4% SRB solução consistentemente cobre a parte inferior dos poços.
-
Medição de absorvância
- Pipete 100 μL de solução base tris (10 mM) para os poços correspondentes, incluindo poços em branco. Mantenha a placa em uma coqueteleira por 10 min. Meça a absorvância em 492 nm.
4. geração de uma curva dose-resposta
- Analise os dados do ensaio SRB em uma planilha. Subtrair a absorvência em branco dos valores de absorvância de cada grupo e calcular a média (AVE) e o desvio padrão (STD) dos valores de absorvência de cada grupo.
- Calcular a percentagem de absorvância média (AVE%) e a do desvio padrão (STD%) de cada grupo utilizando valores de absorvância do ensaio SRB.
Nota: O grupo tratado mímico de controle AVE% de miRNA é de 100%. Calcule o STD% usando a seguinte fórmula: STD% = (STD de cada grupo/AVE absorbância de controle imitado grupo tratado) x 100. - Importe os dados brutos, incluindo as concentrações de tratamento, AVE% e STD% para o software, alinhando verticalmente esses dados. Como o log 0 não está definido, defina a primeira concentração do eixo X para um valor que está próximo de 0 (por exemplo, 0, 1).
- Clique em criar guia de gráfico e escolha barras de erro de dispersão simples. Selecione colunas de planilha como valores de símbolo e clique em Avançar. No painel de formato de dados, selecione pares XY e clique em Avançar. Selecione as colunas de dados correspondentes no painel de dados selecionado. Clique no botão concluir para criar o gráfico.
Nota: O eixo X representa as concentrações, o eixo Y indica o percentual de absorbância média de cada concentração (AVE%), e as barras de erro apontam a porcentagem de desvio padrão de cada concentração (STD%). - Clique duas vezes no eixo X para modificar o tipo de escala e o dimensionamento do eixo. Altere o tipo de escala de linear para log. Modifique o número do intervalo de início e término para 0, 1 e 200, respectivamente.
-
Clique com o botão direito do mouse em qualquer gráfico de dispersão, escolha ajuste de curva e vá para a subcategoria definida pelo usuário. Selecione curva dose-resposta, clique em Avançar botões e, em seguida, clique em Concluir botão. A curva dose-resposta é gerada agora junto com uma aba do relatório (figura 4a).
- Para inserir a equação 1 no software para a geração de uma curva de dose-resposta, clique na guia análise e selecione Assistente de regressão. Vá para definido pelo usuário na categoria de equação e, em seguida, clique no botão novo . Insira a equação 1, variáveis, parâmetros iniciais e restrições nas caixas em branco correspondentes (Figura 4B, C). Clique em Adicionar como botão e defina o nome da equação como curva dose-resposta. O nome da equação agora é gerado na subcategoria usuário-definido na categoria de equação. f indica a porcentagem de viabilidade celular (% de viabilidade celular) na equação 1.
Equação 1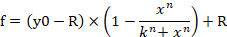
- Vá para a guia relatório e, em seguida, verifique os valores n, k e R.
Nota: y0 indica 100% de viabilidade celular do grupo tratado mímico de controle de Mirna, n indica o coeficiente do tipo colina (a inclinação de um gráfico), k indica a concentração de mímica de mirna-107 que produz um 50% do efeito máximo do mímico de mirna-107 (a metade concentração inibitória máxima, IC50), e R indica a fração residual não afetada (a fração de resistência)15. A equação utilizada para gerar uma curva dose-resposta reconhece o intervalo de y0 a R (se houver) como 100% (figura 4a). Portanto, é necessário adquirir o valor k (IC50) ajustado que é calculado com base no intervalo de y0 para um valor de zero (figura 4a). K ajustado (IC50) juntamente com outros valores de icx (por exemplo, IC10 através de IC90) pode ser obtido usando a equação 2, que é derivada da equação 1. A derivação da equação 2 da equação 1 é indicada na Figura 1 suplementar.
Equação 2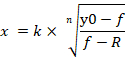
- Clique duas vezes no botão esquerdo do mouse na célula na qual a equação 2 é aplicada. Usando a equação 2 e os parâmetros da curva dose-resposta gerada, está disponível para calcular os valores ajustados de ICx, variando de IC10 a IC90 (Figura 4D).
- Insira o sinal de igual seguido pela fórmula começando com um colchete na célula. Ao inserir a fórmula, corrija o valor de n, k e R como as referências de célula absoluta adicionando o sinal de cifrão à coluna e linha correspondentes, para que esses valores fixos não sejam alterados ao preencher automaticamente a fórmula até as linhas (Figura 4D). Alternativamente, os valores ajustados podem ser calculados manualmente usando a equação 2.
Nota: IC90 valor não é determinado porque o valor de R é maior que 10. Além disso, se o valor R for superior a 20, o valor de IC80 também não é determinado (Figura 4D).
5. verificação do gene alvo direto de um MicroRNA de interesse
Nota: Após a realização do experimento funcional, como o ensaio SRB, o miRNA-107 é confirmado como um tumor supressor de miRNA e é altamente viável que o miRNA-107 atinge diretamente os oncogenes. Verifique a lista de todos os genes-alvo previstos usando um programa de previsão de destino miRNA, como o TargetScan (http://www.targetscan.org/vert_71/), e, em seguida, restrinja-se a potenciais alvos candidatos com base na função de um gene em bancos de dados, incluindo PubMed e Para.
- Primer design para a clonagem de 3 ' região não traduzida (UTR)
- Coloque o nome de um gene em GeneCards (https://www.genecards.org/) e clique no símbolo de um gene. Avalie o navegador do genoma do Ensemble, clicando em Ensemble ID de um gene e, em seguida, clique em transcripts ID na tabela de transcrição. Depois disso, clique em exons existia na lista de exibições baseadas em transcrição à esquerda.
- Copie as sequências de nucleotídeo do 3 ' UTR e cole-o no programa de design da cartilha. Copie as sequências novamente deste programa e cole-a em um processador de texto. Verificar a presença de seqüências de ligação de miRNA, bem como a presença de enzimas de restrição locais utilizados para a clonagem.
Nota: Se não existirem sítios de reconhecimento enzimático de restrição dentro do 3 ' UTR, as enzimas de restrição seleccionadas para a clonagem podem ser utilizadas para a próxima etapa. - No programa do projeto da primeira demão, aceite as 3 ' seqüências de UTR e comece projetar os primers para diante e reverso com a seguinte circunstância. Comprimento: 20-30 nucleotides, TM: 45-58 ° c, GC%: 40-60%. A diferença entre os valores de TM dos dois primers deve ser inferior a 5 ° c. As sequências de primer utilizadas neste estudo são fornecidas na Figura 2 suplementar. Adicionar seqüências de reconhecimento de enzima de restrição, bem como 4 nucleotídeos aleatórios para os primers projetados.
- PCR do inclinação
- Prepare 25 μL de misturas de reação de PCR, incluindo primers projetados por uma temperatura de recozimento (tabela 2). Prepare quantidade suficiente de misturas com base no número total de reações. Misture a solução introduzindo pipetagem e adicione 25 μL de misturas de reacção em cada tubo. Centrifugue os tubos por alguns segundos.
- Execute 35-40 ciclos do PCR da etapa da desnaturação à etapa da extensão. Configurar o ciclo de PCR como as seguintes etapas: 98 ° c para 1 minuto (1 ciclo, etapa da ativação do polymerase), 95 ° c para 10 s (etapa da desnaturação), 45 ° c-68 ° c para 30 s (etapa do recozimento), 68 ° c (etapa da extensão, 10 s-1 minuto por 1000 BP), 68 e, finalmente, arrefecer até 4 ° c.
- Execute os produtos do PCR e verifique as faixas em um gel do agarose de 1% com escadas do ADN. Encontre a melhor temperatura de recozimento (Figura 5a). Amplificar 3 ' UTR de um gene novamente usando a melhor temperatura de recozimento para a próxima etapa.
- Digestão dobro
- Faça as misturas de reação, incluindo duas enzimas de restrição, XhoI (ou AsiSI) e NotI, em um tubo (tabela 3). Incubar as misturas para 3-4 h usando um banho de água (37 ° c).
- Execute os produtos digeridos duplos em um gel de agarose de 1% e, em seguida, corte as bandas luz UV. No caso de vectores de luciferase, antes de funcionar em gel, reaja vetores digeridos duplos com 10 U de fosfatases alcalinas por mais 1 h para evitar a reconstituição durante a etapa de ligadura.
- Purify os produtos digeridos dobro do PCR e os vetores do luciferase das bandas extirpada.
- Ligadura de produtos do PCR nos vetores do luciferase
- Fazer 20 μL de misturas de reacção de ligadura, incluindo a ligase de ADN (tabela 4).
Nota: A relação molar do produto do PCR (inserção) ao vetor do luciferase pode ser 3:1. 1:1 ou 2:1. - Centrifugue momentaneamente o tubo para 10-15 s e incubar em 16 ° c durante a noite usando um Thermal cycler.
Nota: Alternativamente, o tubo pode ser incubado a 4 ° c por 2-3 dias para a ligadura. Nesta etapa, o inserto de PCR será clonado na região posicionada a jusante de um gene repórter de renilla (Figura 5b). A ligação de miRNAs para o clonado 3 ' UTR de um gene pode diminuir na atividade de renilla. Firefly luciferase é para a normalização dos níveis de expressão de renilla.
- Fazer 20 μL de misturas de reacção de ligadura, incluindo a ligase de ADN (tabela 4).
- Transformação e PCR da colônia
- Adicione as misturas de ligadura (3-5 μL) no tubo que contém as células competentes. Bata suavemente no tubo e mantenha-o no gelo (20 min).
Nota: descongelar as células competentes no gelo antes de adicionar as misturas de ligadura. - Transfira rápida e suavemente o tubo para um bloco de calor. Após um choque térmico (42 ° c por 30 s-1 min), coloque o tubo no gelo por 20 min.
- Espalhe as células competentes na placa de agar Luria-Bertani (LB). Cresça pilhas competentes em uma incubadora (° c 37) durante a noite.
Nota: A ampicilina (50-100 μg/mL) está contida na placa de agar. - Escolha uma colônia individual e ressuscitem e . coli em um dos tubos de 8 tiras contendo água ultrapura. Repita este passo para ressuscidar E. coli de 4-8 colônias selecionadas aleatoriamente (Figura 5C).
- Transfira 25 μL de suspensão de E. coli para outro conjunto de tubos de 8 tiras. Agora, existem 2 conjuntos de tubos de E. coli suspensão.
Nota: Um tubo é para a colônia PCR e outro é para inoculação. A suspensão de E. coli para inoculação pode ser temporariamente armazenada a 4 ° c (Figura 5C). - Realize o PCR da colônia usando a suspensão de E. coli . Esta etapa é determinar se as colônias contêm uma inserção. Selecione as melhores colônias para inocular e isolar vetores de luciferase abrigando 3 ' UTR de um gene (Figura 5C).
Nota: Repita o passo 5.1-5.5 para cada 3 ' UTR de genes seleccionados. Siga a condição da reação do PCR mostrada na tabela 2 substituindo o ADN genomic com suspensão de E. coli .
- Adicione as misturas de ligadura (3-5 μL) no tubo que contém as células competentes. Bata suavemente no tubo e mantenha-o no gelo (20 min).
- Ensaio de luciferase
- Prepare uma placa de 24 poços. Use 1-2 x 104 células em 500 μL de mídia de cultura de células para cada poço. Não use meios de cultura da pilha que contêm P/S para o transfection porque usar P/S pode reduzir a eficiência do transfection.
- Transfect 50 ng de vetores de luciferase nas células com mímica de controle ou um mímico específico de miRNA usando um reagente de transfecção (Figura 5D). Se a triagem dos efeitos de uma mímica específica de miRNA em mais de uma concentração, manter a quantidade total de oligos mesmo em cada poço (ver passo 2).
- Lave o interior de poços duas vezes usando fosfato tampão salina (PBS) no dia seguinte.
- Aplique 200 μL de reagente de Lise nos poços e realize suficientemente a lise celular antes de medir a atividade da luciferase.
Nota: Mantenha a placa em uma placa de agitação pelo menos 15 min. - Transferir 5-10 μL de lisado de células para o novo tubo e adicionar 100 μL de reagente I. misture imediatamente a solução introduzindo pipetagem e leia a atividade da luciferase do vagalume utilizando um luminômetro.
Nota: Leia a atividade luciferase Firefly para 10-15 s. - Adicionar 100 μL de reagente II no mesmo tubo e, em seguida, misturar por pipetagem duas vezes. Leia a atividade de renilla luciferase para 10-15 s usando um luminômetro. Repita a etapa 5.6.5 e 5.6.6 para cada amostra.
- Calcule a proporção de renilla para vaga-lume (Figura 5E).
Nota: A atividade do Firefly representa a eficiência do transfection de construções do luciferase nas pilhas.
Access restricted. Please log in or start a trial to view this content.
Resultados
A confirmação bem-sucedida e precisa dos níveis de miRNA é importante para a interpretação dos dados pelos quais a classificação de miRNAs é possível com base nos papéis esperados de miRNAs no desenvolvimento e progressão de uma doença. Os níveis de miRNA-107 e miRNA-301 foram medidos em três linhagens de células pancreáticas utilizando a PCR quantitativa baseada em sonda. A síntese de cDNAs de ambos um miRNA específico e um gene de referência na mesma reação pode aumentar a reprodutibilidade dos da...
Access restricted. Please log in or start a trial to view this content.
Discussão
Estratégias para a determinação de alvos de miRNA de bona fide com as funções de um miRNA de interesse são indispensáveis para a compreensão de múltiplos papéis de miRNAs. A identificação de genes alvo de miRNA pode ser uma diretriz para interpretar os eventos de sinalização celular modulados por miRNAs em uma célula. Um desvelamento de genes-alvo funcionalmente importantes de miRNAs pode fornecer o conhecimento fundamental para desenvolver uma terapia baseada em miRNA no câncer.
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este estudo foi apoiado pelo programa de pesquisa de ciência básica através da Fundação Nacional de pesquisa da Coréia (NRF) financiado pelo Ministério da educação (2017R1D1A3B03035662); e fundo de pesquisa da Universidade Hallym, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
Referências
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados