このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
腫瘍細胞におけるマイクロRNAレベル、機能、および関連する標的遺伝子を評価するためのインビトロプロトコル
要約
このプロトコルは、プローブベースのリアルタイムポリメラーゼ連鎖反応(PCR)、スルフォルホダミンB(SRB)アッセイ、3'未翻訳領域(3'UTR)クローニング、および目的のmiRNAの標的遺伝子を検証し、miRNAの機能を理解するためにルシフェラーゼアッセイを使用します。
要約
マイクロRNA(miRNA)は、癌を含むいくつかの疾患において多数の細胞内シグナル伝達経路を調節することが認識されている小さな調節RNAです。これらの小さな調節RNAは、主に標的メッセンジャーRNA(mRNA)の3'非翻訳領域(3'UTR)と相互作用し、最終的にはmRNAの復号プロセスの阻害および標的mRNA分解の増強をもたらす。発現レベルおよび細胞内機能に基づいて、miRNAは発癌性および腫瘍抑制性mRNAの調節因子として機能することができる。計算的に予測された数百または数千の標的の中でmiRNAのボナフィード標的遺伝子を同定することは、目的とするmiRNAの役割と基本的な分子機構を識別するための重要なステップです。可能なmiRNA-mRNA相互作用を検索するために、様々なmiRNA標的予測プログラムが利用可能です。しかし、最も困難な問題は、目的とするmiRNAの直接標的遺伝子を検証する方法です。このプロトコルは、miRNAの機能に関連するmiRNA標的を同定する方法に関する主要な方法の再現可能な戦略を説明する。このプロトコルは、プローブベースのリアルタイムポリメラーゼ連鎖反応(PCR)、スルフォルホダミンB(SRB)アッセイを用いてmiRNAレベル、機能、および関連する標的mRNAを明らかにするステップバイステップの手順に関する実用的なガイドを提示します。、用量応答曲線生成、およびルシフェラーゼアッセイは、個々のmiRNAの役割を適切に理解するために必要な遺伝子の3'UTRのクローニングと共に。
概要
マイクロRNA(miRNA)は、主にボナフィード標的遺伝子1の3'非翻訳領域(3'UTR)に反応することにより、メッセンジャーRNA(mRNA)の翻訳および分解のプロセスを調節する小さな調節RNAです。miRNAの発現は、転写および転写後のメカニズムによって調節することができる。このような調節機構の不均衡は、癌2を含む多数の疾患において制御不能で特徴的なmiRNA発現レベルをもたらす。単一のmiRNAは、多様なmRNAとの複数の相互作用を持つことができます。それに応じて、個々のmRNAは様々なmiRNAによって制御することができる。したがって、細胞内シグナル伝達ネットワークは、生理的障害および疾患が開始され、悪化することができる特徴的に発現したmiRNAによって複雑に影響され、2、3、4、5,6.miRNAの発現の変化は様々な種類の癌で観察されているが、miRNAと共に癌細胞のマナーを調節する分子機構は依然としてほとんど知られていない。
蓄積された証拠は、miRNAの発癌または腫瘍抑制の役割が癌の種類に依存することを示している。例えば、フォークヘッドボックスo3(FOXO3)を標的とすることにより、miR-155は、大腸癌7、8の細胞増殖、転移、および化学耐性を促進する。対照的に、神経膠腫細胞侵襲の制限は、神経原性遺伝子座ノッチホモログタンパク質2(NOTCH2)発現9の調節を介してmiR-107によって誘導される。miRNA機能に関連するmiRNA標的相互作用の評価は、miRNAが健康な状態と病気状態の両方で様々な生物学的プロセスを調節する方法をよりよく理解するために不可欠な部分です 10.さらに、miRNAのボナ・フィデス・ターゲットの発見は、様々な抗癌剤を用いてmiRNAベースの治療に対する微調整された戦略をさらに提供することができる。しかし、miRNAの分野における主な課題は、miRNAの直接標的の同定です。ここで、miRNA標的遺伝子決定に対する再現可能な実験的アプローチとして詳細な方法を提示する。miRNA標的同定のための実験設計の成功には、様々なステップと考慮事項が含まれます(図1)。腫瘍細胞および正常細胞における成熟したmiRNAレベルの比較は、目的のmiRNAを選択する一般的な手順の1つでありうっていう(図1A)。細胞増殖に対するmiRNAの効果を検出するために選択されたmiRNAの機能的研究は、目的とするmiRNAの最良の候補標的のリストを絞り込む必要がある(図1B)。miRNAの実験的に検証された機能に基づいて、miRNA標的予測プログラムを用いた文献とデータベースの系統的レビューが、遺伝子機能に関する最も関連性の高い情報を検索する必要がある(図1C)。目的とするmiRNAの実際の標的遺伝子の同定は、遺伝子の3'UTRのクローニング、リアルタイムPCR、およびウェスタンブロッティング(図1D)と共にルシフェラーゼアッセイなどの実験を実施することによって達成することができる。現在のプロトコルの目的は、主要な実験の包括的な方法を提供することです, プローブベースのリアルタイムポリメラーゼ連鎖反応 (PCR), スルフォルホダミンB (SRB) アッセイは、miRNA模倣トランスフェクションに続く, 用量応答曲線生成,遺伝子の3'UTRのクローニングと共にルシフェラーゼアッセイ。現在のプロトコルは、個々のmiRNAの機能と癌治療におけるmiRNAの意味をより良く理解するのに役立ちます。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 成熟マイクロRNA(miRNA)発現解析
-
成熟したmiRNA相補DNA(cDNA)合成
- 総RNAの254ngとデオキシリボヌクレアーゼI(DNase I)混合物の4.5μLを加え、PCRストリップチューブに超純水を加えて最大18μL(図2A)を作ります。反応の総数に基づいて十分な量のDNase I混合物を用いて複数の細胞株から精製した各全RNA試料に対する反応を調出す。
注:DNase I混合物は、DNase I(1.8 μL)、リボヌクレアーゼ阻害剤(0.3μL)、および25mM MgCl 2(2.4 μL)で構成されています。全RNAを再現可能に調達するために、フェノールクロロホルム系抽出法を用いた代わりにカラムベースの抽出法を適用した。フェノールクロロホルム系抽出法11,12を用いるとき、一部のmiRNAの抽出収率は細胞数に応じて変化させることができることが報告された。 - 熱サイクラーでチューブをインキュベートします。37°Cで10分間チューブを実行し、90°Cで5分のインキュベーションでDNase Iを熱不活性化します。インキュベーション後すぐにチューブを氷の上に置きます。
- DNaseの7.1 μLを2組の新しいチューブに分けて処理し、グリセラルデヒド-3-リン酸デヒドロゲナーゼ(GAPDH)遺伝子に1.5μLのアンチセンスプライマーを加えました(図2B)。
注:cDNA合成のための総RNAの量は、このステップで100ngになります。GAPDHアンチセンスプライマーのストック濃度は10μMです。 - サーマルサイクラーを使用してチューブをインキュベートします。80°Cで5分間開始し、その後60°Cで5分間反応を行い、インキュベーション後すぐにチューブを氷の上に置きます。
- 各反応に3.4 μLの逆転転写(RT)酵素混合物を加加える(図2B)。RT酵素混合物は、100mMデオキシリボヌクレオチドトリリン酸塩(0.15μL)、10x RTバッファー(1.5μL)、リボヌクレアーゼ阻害剤(0.75μL)、および逆転写酵素(1μL)で構成されています。反応の総数に基づいて混合物の十分な量を準備します。
- 各反応における特定のmiRNAに対して5x RTプライマーの3 μLを添加する(図2B)。
注:総体積は、各反応につき15μLです。 - サーマルサイクラーを使用してチューブを実行します。16 °C で 30 分間開始し、その後 42 °C で 30 分間反応を開始し、最後に 85 °C で 5 分間 5 分間保持し、残りの時間を 4 °C で保持します (図 2B)。このステップでは、同じチューブ内の特定のmiRNA遺伝子とGAPDH遺伝子の両方に対して、一本鎖cDNが生成されます。
- 総RNAの254ngとデオキシリボヌクレアーゼI(DNase I)混合物の4.5μLを加え、PCRストリップチューブに超純水を加えて最大18μL(図2A)を作ります。反応の総数に基づいて十分な量のDNase I混合物を用いて複数の細胞株から精製した各全RNA試料に対する反応を調出す。
-
リアルタイムポリメラーゼ連鎖反応(PCR)とデータ解析
- 各cDNAを1:49の比率で超純水で希釈します。
- 特定のmiRNAおよびGAPDHに対する反応混合物を調出す(表1)。特定のmiRNAおよびGAPDHの検出のために、各cDNAサンプルに対して三重反応を設定する。
- リアルタイム PCR およびデータ分析を実行します (図 2C)。比較CT法13,14を用いてデータを分析する。
2. マイクロRNA(miRNA)ミミックトランスフェクション
注:miRNA-107はステップ1から選択される。miRNA-107は正常細胞と比較して腫瘍細胞においてダウンカワヤされるので、miRNA-107は腫瘍抑制ミルナであると推測できる。正常細胞と比較して腫瘍細胞でアップレギュレートされたmiRNAの場合(例えば、miRNA-301)、miRNA-301に対するアンチセンスオリゴヌクレオチドは、ステップ2、3、および4に適用することができる。
- 計数室装置で細胞を数え、96ウェルプレートに細胞をプレートします。細胞密度は、各ウェルに対して2 x 103セル/100 μLです。P/Sはトランスフェクション効率を低下させることができるので、ペニシリン連鎖マイシン(P/S)を含む細胞培養培養培養剤を使用しないでください。
-
翌日にmiRNA対照模倣およびmiRNA-107模倣のいくつかの最終濃度で細胞をトランスフェクトするトランスフェクション混合物のセットを調製する(図3)。
- miRNA制御模倣またはmiRNA-107模倣のストック(25μM濃度)から、微小遠心管を用いてトランスフェクション試薬と共に還元血清培中に、対応する量の制御模倣またはmiRNA-107模倣を加えて添加する(図3A)。マイクロピペットを使用して混合物を含むオリゴを穏やかに混合します。オリゴの総量(miRNA模倣対照+miRNA-107模倣)は、各ウェルで同じであるべきである。ブランクウェルには、細胞培養培養培養剤の100μLと細胞のないトランスフェクション試薬を含む還元血清培中が含まれる。
- 細胞培養フードに10分のインキュベーションを行った後、再び混合物を含むオリゴを穏やかに混合し、各ウェルに混合物の50 μLを追加します。トランスフェクトされた細胞を細胞培養インキュベーターに保管してください。6-12 h インキュベーション後に、培温剤を含むトランスフェクション試薬を、胎児ウシ血清(FBS)およびP/Sの両方を含む新鮮な細胞培養培養培養剤に置き換える。さらに72時間細胞をインキュベートする。miRNA模倣の総治療持続時間は96hである。
3. スルフォルホダミンB(SRB)アッセイ
-
細胞固定
- プレートの各ウェルの細胞培養培地を取り出し、10%トリクロロ酢酸(TCA)の100μLを各ウェルに速やかに充填します。細胞の損傷や下部からの剥離を避けるために、各ウェルから細胞培養培養培養剤を慎重に吸引します。
注:蒸留水の50 mLにTCA粉末の20グラムを追加することにより、40%のTCAを調調します。40% TCA から、蒸留水で 40% TCA を 1:3 希釈比で希釈して 10% TCA を作成します。 - 10%TCAを含むプレートを冷蔵庫(4°C)に1時間保管してください。
- 水槽に浸してプレートを数回洗い、乾燥させます。井戸に水が残らないまでプレートをタップして、井戸の内部から余分な水を取り除きます。次のステップに進む前に、実験室のベンチにプレートを残して乾燥させます。
- プレートの各ウェルの細胞培養培地を取り出し、10%トリクロロ酢酸(TCA)の100μLを各ウェルに速やかに充填します。細胞の損傷や下部からの剥離を避けるために、各ウェルから細胞培養培養培養剤を慎重に吸引します。
-
細胞染色
- 0.4%SRB溶液のピペット50 μLをブランクウェルを含む各ウェルに入れます。0.4%のSRB溶液が一貫して井戸の底を覆うまで、プレートをゆっくりと振ります。
注:1%酢酸の100 mLにSRB粉末の0.4gを添加することにより、0.4%SRB溶液を調味し、使用します。溶液を慎重に振って混ぜます。0.4%SRB溶液のボトルをアルミ箔などの軽い保護材料に包みます。0.4%のSRB溶液を冷蔵庫に保管してください。 - 40分~60分のインキュベーション後、酢酸1%で洗い流してプレートを洗います。バインドされていない色素が完全に洗い流されるまでプレートを洗います(図3B)。
- 次のステップに進む前に、実験室のベンチにプレートを残して乾燥させます。
注:ステップ3.3に行く前に、プレートを完全に乾燥させる必要があります。
- 0.4%SRB溶液のピペット50 μLをブランクウェルを含む各ウェルに入れます。0.4%のSRB溶液が一貫して井戸の底を覆うまで、プレートをゆっくりと振ります。
-
吸光度測定
- トリス塩基溶液(10mM)のピペット100 μLを、ブランクウェルを含む対応するウェルに入れます。プレートをシェーカーに10分間置き、492nmで吸光度を測定します。
4. 線量応答曲線の生成
- スプレッドシート内の SRB アッセイ データを分析します。各グループの吸光度値からブランク吸光度を減算し、各グループの吸光度値の平均(AVE)と標準偏差(STD)を計算します。
- 平均吸光度のパーセンテージ (AVE%)標準偏差(STD%)SRBアッセイの吸光度値を使用する各グループの。
注:miRNA対照模倣処理群のAVE%は100%である。STD%を次の式で計算する:STD%=(各群のSTD/AVE吸収性模倣処理群)x 100。 - 処理濃度、AVE%、STD% などの生データを垂直に整列させることで、ソフトウェアにインポートします。ログ 0 は定義されていないため、X 軸の最初の濃度を 0 に近い値 (0.01 など) に設定します。
- [グラフを作成] タブをクリックし、[単純な散布区間誤差バー] を選択します。シンボル値として[ワークシートの列]を選択し、[次へ]をクリックします。データ形式パネルで、[XYペア] を選択し、[次へ]をクリックします。選択データ パネルで対応するデータ列を選択します。[完了]ボタンをクリックして、プロットを作成します。
注:X軸は濃度を表し、Y軸は各濃度の平均吸光度(AVE%)のパーセンテージを示し、誤差バーは各濃度の標準偏差(STD%)のパーセンテージを示します。 - X 軸をダブルクリックして、軸のスケールの種類とスケーリングを変更します。縮尺の種類を線形からログに変更します。開始範囲と終了範囲番号をそれぞれ 0.01 と 200 に変更します。
-
散布図を右クリックし、[カーブフィット]を選択し、ユーザー定義のサブカテゴリに移動します。線量応答曲線を選択し、[次へ] ボタンをクリックし、[完了] ボタンをクリックします。線量応答曲線がレポートタブと共に生成されるようになりました(図4A)。
- 線量応答曲線の生成のためにソフトウェアで式 1 を入力するには、[分析]タブをクリックし、[回帰ウィザード]を選択します。数式カテゴリのユーザー定義に移動し、[新規]ボタンをクリックします。方程式 1、変数、初期パラメータ、および制約を対応するブランク ボックス (図 4B、C)に挿入します。[追加]ボタンをクリックし、方程式の名前を線量応答曲線として設定します。これで、方程式名が、数式カテゴリのユーザー定義サブカテゴリで生成されるようになりました。fは、式 1 における細胞生存率(細胞生存率%)のパーセンテージを示します。
方程式 1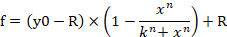
- [レポート] タブに移動し、n、k、および R の値を確認します。
注:y0はmiRNA対照模倣処理群の100%細胞生存率を示し、nはヒル型係数(プロットの傾き)を示し、kはmiRNA-107模倣の最大効果の50%を生み出すmiRNA-107模倣の濃度を示す(半分)最大阻害濃度、IC50、及びRは、残留非影響画分(抵抗画分)15を示す。線量応答曲線の生成に使用される式は、y0 から R 値 (ある場合) までの範囲を 100% (図 4A)として認識します。したがって、y0からゼロの値(図4A)の範囲に基づいて計算される調整されたk(IC50)値を取得する必要があります。調整されたk(IC50)は、他のICx値(例えば、IC10からIC90)と共に、式1から導出される式2を用いて得ることができる。 式1からの方程式2の導出は補足図1に示される。
方程式 2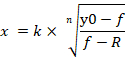
- 数式 2 が適用されているセルのマウスの左ボタンをダブルクリックします。方程式2と生成された線量応答曲線からのパラメータを使用して、IC10からIC90(図4D)までのICxの調整値を計算することができます。
- 等号を入力し、その後にセル内の角かっこで始まる数式を入力します。数式を入力するときは、対応する列と行にドル記号を追加して絶対セル参照として n、k、および R の値を修正し、数式を行に自動入力するときにこれらの固定値が変更しないようにします (図 4D)。または、調整された値は、方程式 2 を使用して手動で計算できます。
注:R値が10より大きいため、IC90値は判定されません。また、R値が20を超えると、IC80の値も判定されない(図4D)。
5. 目的のマイクロRNAの直接標的遺伝子の検証
注:SRBアッセイなどの機能実験を行った後、miRNA-107は腫瘍抑制ミルナとして確認され、miRNA-107が腫瘍遺伝子を直接標的とすることが非常に実現可能である。TargetScan(http://www.targetscan.org/vert_71/などのmiRNA標的予測プログラムを使用して予測されたすべての標的遺伝子のリストを確認し、PubMedを含むデータベース内の遺伝子の機能に基づいて候補ターゲットに絞り込み、ジーンカード
- 3' 未翻訳領域 (UTR) のクローン作成のためのプライマー設計
- GeneCards (https://www.genecards.org/) に遺伝子の名前を入れ、[遺伝子のシンボル]をクリックします。遺伝子のEnsembl IDをクリックしてEnsemblゲノムブラウザに評価し、トランスクリプトテーブルのトランスクリプトIDをクリックします。その後、左側のトランスクリプトベースのディスプレイリストに[Exons]が表示されているのをクリックします。
- 3'UTRのヌクレオチド配列をコピーし、プライマー設計プログラムに貼り付けます。このプログラムからシーケンスをもう一度コピーし、ワード プロセッサに貼り付けます。miRNA結合配列の存在とクローニングに使用される制限酵素部位の存在を確認します。
注:3'UTR内に制限酵素認識部位がない場合は、クローニングに選択された制限酵素を次のステップに使用できます。 - プライマー設計プログラムでは、3' UTR シーケンスを受け入れ、次の条件で前方および逆のプライマーの設計を開始します。長さ: 20-30 ヌクレオチド, Tm: 45-58 °C, GC%: 40-60%.2つのプライマーのTm値の差は5°C未満でなければなりません。本研究で使用されるプライマー配列は、補足図2に記載されている。制限酵素認識配列と4つのランダムヌクレオチドを設計されたプライマーに追加します。
- グラデーション PCR
- 1つのアニーリング温度あたりの設計されたプライマーを含むPCR反応混合物の25μLを調出す(表2)。反応の総数に基づいて混合物の十分な量を準備します。ピペッティングで溶液を混合し、各チューブに反応混合物の25 μLを追加します。チューブを数秒間遠心分離します。
- 脱生ステップから拡張ステップまで35~40個のPCRサイクルを実行します。PCRサイクルを次のステップとして設定する:98 °C 1分(1サイクル、ポリメラーゼ活性化ステップ)、95°C(脱生ステップ)、45°C-68°C 30s(アニーリングステップ)、68°C(延長ステップ、1000bpあたり10s-1分)、3分間68°C(3分間)そして最後に4°Cまで冷却します。
- PCR製品を実行し、DNAはしごを持つ1%アガロースゲル上のバンドをチェックします。最高のアニーリング温度を見つける (図 5A)次のステップのための最高のアニーリング温度を使用して再び遺伝子の3'UTRを増幅する。
- 二重消化
- 2つの制限酵素、XhoI(またはAsiSI)およびNotIを含む反応混合物をチューブ(表3)に作る。水浴(37°C)を使用して3〜4時間の混合物をインキュベートします。
- 1%のアガロースゲルで二重消化製品を実行し、紫外線の下でバンドをカットします。ルシフェラーゼベクターの場合、ゲル上で実行する前に、ライゲーション工程中の再循環を防ぐために、さらに1時間1時間アルカリホスファターゼの10Uで二重消化ベクターを反応させる。
- 二重消化PCR産物とルシフェラーゼベクターを取り除いたバンドから浄化します。
- ルシフェラーゼベクターへのPCR産物のライゲーション
- DNAリガーゼを含むライゲーション反応混合物の20 μLを作る(表4)。
注:ルシフェラーゼクターに対するPCR産物(インサート)のモル比は3:1とすることができる。1:1 または 2:1 - 簡単に10-15 sのためのチューブを遠心分離し、熱サイクラーを使用して一晩16 °Cでインキュベートします。
注:あるいは、チューブは、ライゲーションのために2〜3日間4°Cでインキュベートすることができる。このステップでは、PCRインサートは、レニラレポーター遺伝子の下流に位置する領域にクローニングされます(図5B)。遺伝子のクローン化された3'UTRへのmiRNAの結合は、レニラ活性において減少しうる。ホタルルシフェラーゼは、レニラ発現レベルの正規化のためです。
- DNAリガーゼを含むライゲーション反応混合物の20 μLを作る(表4)。
- 変換とコロニー PCR
- 有能な細胞を含むチューブにライゲーション混合物(3-5 μL)を追加します。チューブをそっとタップし、氷の上に置きます(20分)。
注:ライゲーション混合物を追加する前に、氷上の有能な細胞を凍結解除します。 - チューブを熱ブロックに素早くゆっくりと移します。ヒートショック(30 s-1分間42°C)の後、20分間氷の上にチューブを置きます。
- ルリア・ベルタニ(LB)寒天プレートに有能な細胞を広げます。インキュベーター(37°C)で一晩で有能な細胞を成長させる。
注:アンピシリン(50-100 μg/mL)は寒天プレートに含まれています。 - 個々のコロニーを選び、超純水を含む8ストリップチューブの1つで大腸菌を再中断します。この手順を繰り返して、ランダムに選択された 4-8 コロニーから大腸菌を再中断します (図 5C)。
- 大腸菌懸濁液の25 μLを別の8ストリップチューブに移します。今、大腸菌懸濁液のチューブの2セットがあります。
注:1つのチューブはコロニーPCR用で、もう1つは接種用です。接種用大腸菌懸濁液は、4°C(図5C)で一時的に貯蔵することができる。 - 大腸菌懸濁液を用いてコロニーPCRを行う。この手順では、コロニーに挿入が含まれているかどうかを確認します。遺伝子の3'UTRを収容するルシフェラーゼベクターを接種し、単離するための最良のコロニーを選択します(図5C)。
注:選択した遺伝子の3'UTRごとにステップ5.1-5.5を繰り返します。表2に示すPCR反応の状態に従って、ゲノムDNAを大腸菌懸濁液に置き換える。
- 有能な細胞を含むチューブにライゲーション混合物(3-5 μL)を追加します。チューブをそっとタップし、氷の上に置きます(20分)。
- ルシフェラーゼアッセイ
- 24ウェルプレートを準備します。各ウェルの500 μL細胞培養培養培養培養培養培養培養に1-2 x 104細胞を使用します。P/S を使用するとトランスフェクション効率を低下させる可能性があるため、トランスフェクションに P/S を含む細胞培養培養培養剤を使用しないでください。
- トランスフェクセラーゼベクターのトランスフェクト50 ngは、トランスフェクション試薬を用いて対照模倣または特定のmiRNA模倣を用いて細胞内に入る(図5D)。特定のmiRNAの効果を複数の濃度でスクリーニングする場合は、各ウェルでオリゴの総量を同じにしておいてください(ステップ2参照)。
- 翌日にリン酸緩衝生理食生(PBS)を用いて井戸の内部を2回洗う。
- 200 μLのリシス試薬をウェルに塗布し、ルシフェラーゼ活性を測定する前に十分に細胞リシスを行います。
注:プレートを振るプレートに少なくとも15分置いてください。 - 5-10 μLの細胞溶解物を新しいチューブに移し、100 μLの試薬Iを追加します。
注:10-15 sのためのホタルルシフェラーゼの活動を読んでください。 - 同じチューブに100μLの試薬IIを加え、2回ピペッティングして混ぜます。ルミノメーターを使用して10-15sのレニラルシフェラーゼ活性を読んでください。サンプルごとに手順 5.6.5 と 5.6.6 を繰り返します。
- ホタルに対するレニラの比率を計算します(図5E)。
注:ホタルの活性は、細胞内に構築されるルシフェラーゼのトランスフェクション効率を表す。
Access restricted. Please log in or start a trial to view this content.
結果
miRNAレベルの正常かつ正確な確認は、疾患の発症および進行におけるmiRNAの予想される役割に基づいてmiRNAの分類が可能なデータの解釈にとって重要である。miRNA-107およびmiRNA-301のレベルを、プローブベースの定量PCRを用いて3つの膵臓細胞株で測定した。特定のmiRNAと同じ反応における参照遺伝子の両方のcDNの合成は、データの再現性を高めることができる。PANC-1およびCAPAN-1はヒト膵管腺癌細...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
miRNAの複数の役割を理解するためには、関心のあるmiRNAの機能を有するボナ・フィドmiRNA標的の決定のための戦略が不可欠である。miRNA標的遺伝子の同定は、細胞内のmiRNAによって調節された細胞シグナル伝達事象を解釈するためのガイドラインでありうる。miRNAの機能的に重要な標的遺伝子の発表は、がんにおけるmiRNAベースの治療法を開発するための基礎知識を提供することができます。
...Access restricted. Please log in or start a trial to view this content.
開示事項
著者は何も開示していない。
謝辞
本研究は、文部科学省の助成を受けた韓国国立研究財団(NRF)を通じて基礎科学研究プログラム(2017R1D1A3B03035662)の支援を受けた。とハリム大学研究基金, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
参考文献
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved