Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Tümör hücrelerinde MicroRNA seviyeleri, fonksiyonları ve Ilişkili hedef genler değerlendirmek için bir In vitro protokol
Bu Makalede
Özet
Bu protokol bir prob tabanlı gerçek zamanlı polimeraz zincir reaksiyonu (PCR), bir sülforhodamin içinde B (SRB) tahlil, 3 ' tercüme edilmemiş bölgeler (3 ' UTR) klonlama ve bir Lusiferaz tahlil ilgi bir Mirna hedef genler doğrulamak ve mirnas işlevlerini anlamak için kullanır.
Özet
MicroRNAs (miRNAs), kanserler de dahil olmak üzere çeşitli hastalıklarda çok sayıda hücre içi sinyalizasyon yollarını modülasyon olarak tanınan küçük düzenleyici RNAs 'lardır. Bu küçük düzenleyici RNAs ağırlıklı olarak 3 ' tercüme edilmemiş bölgeler (3 ' UTR) hedef haberci RNAs (mRNAs) ile etkileşim sonuçta mRNAs çözme süreçlerinin inhibisyonu ve hedef mRNA degradations büyütme sonuçlanan. MiRNAs, ifade düzeylerine ve hücre içi işlevlere bağlı olarak Onkojenik ve tümör bastırıcı MRI 'nin düzenleyici faktörler olarak hizmet verebilir. Yüzlerce ve hatta binlerce hesaplamalarıyla öngörülen hedefler arasında bir miRNA 'nın iyi niyetli hedef genlerinin tanımlanması, bir miRNA 'nın rol ve temel moleküler mekanizmalarını ayırt etmek için önemli bir adımdır. Çeşitli miRNA hedef tahmin programları olası miRNA-mRNA etkileşimlerini aramak için kullanılabilir. Ancak, en zorlu soru nasıl bir miRNA ilgi doğrudan hedef genleri doğrulamak için. Bu protokol bir miRNA işlevi ile ilgili miRNA hedeflerini tanımlamak nasıl anahtar yöntemleri tekrarlanabilir bir strateji açıklanır. Bu protokol, Mirna seviyeleri, fonksiyonları ve ilgili hedef mrnas 'ı Probe tabanlı gerçek zamanlı polimeraz zincir reaksiyonu (PCR), sülforhodamin içinde B (SRB) tahlil bir Mirna taklit transfeksiyon aşağıdaki kullanarak ortaya çıkarmak için adım adım prosedürler üzerinde pratik bir kılavuz sunar , doz-yanıt eğrisi oluşturma, ve Lusiferaz tahlil birlikte klonlama ile 3 ' UTR bir gen, hangi bireysel mirnas rollerini doğru anlayış için gerekli olan.
Giriş
MicroRNAs (miRNAs), özellikle haberci RNAs 'ın (mRNAs) tercüme ve bozulma sürecini, gerçek hedef genler1' de 3 ' tercüme edilmemiş bölgeye (3 ' UTR) tepki vererek modüle eden küçük düzenleyici RK 'lerdir. Mirnas 'ın ifadesi transkripsiyon ve post-transkripsiyonel mekanizmalar tarafından düzenlenebilir. Bu tür Düzenleyici mekanizmaların dengesizliği, kanserler de dahil olmak üzere sayısız hastalıklarda kontrolsüz ve ayırt edici miRNAs ifade düzeylerini getiriyor2. Tek bir miRNA çeşitli MRI 'lar ile birden fazla etkileşime sahip olabilir. Buna karşılık, bireysel mRNA çeşitli miRNAs tarafından kontrol edilebilir. Bu nedenle, hücre içi sinyalizasyon ağları, fizyolojik bozuklukların ve hastalıkların başlatılacağı ve bozulacağı2,3,4, 5 , 6. mirnas 'ın değiştirilmiş ifadesi çeşitli kanserlerde gözlenen olsa da, mirnas ile birlikte kanser hücrelerinin davranışlarını modüle eden moleküler mekanizmalar hala büyük ölçüde bilinmiyor.
Biriken kanıtlar, miRNAs 'ın Onkojenik veya tümör bastırıcı rollerini kanserlerin türlerine bağlı olduğunu gösteriyor. Örneğin, Forkhead kutusu O3 (FOXO3) hedefleyerek, Mir-155, hücre proliferasyonu, metasoz ve kolorektal kanser kemoterapi yükseltir7,8. Buna karşılık, glioma hücre invazyonu kısıtlama nörojenik Locus çentik homolog protein 2 (NOTCH2) ifade9düzenlenmesi yoluyla miR-107 tarafından indüklenir. Mirna işlevleriyle bağlantılı mirla-Target etkileşimlerinin değerlendirilmesi, miRNAs 'ın hem sağlıklı hem de hastalıklı Devletler10' da çeşitli biyolojik süreçleri nasıl düzenlemesini daha iyi anlamak için vazgeçilmez bir parçasıdır. Buna ek olarak, miRNAs 'ın iyi niyetli hedef (ler) keşfi, çeşitli anti-kanser ilaçları ile miRNA tabanlı bir terapi için ince ayarlı bir strateji sağlayabilir. Ancak, miRNAs alanındaki ana meydan okuma, miRNAs 'ın doğrudan hedeflerini tanımlamadır. Burada, Mirna hedef gen belirlenmesi için tekrarlanabilir deneysel yaklaşımlar olarak ayrıntılı yöntemler sunulmaktadır. MiRNA hedef tanımlaması için başarılı deneysel tasarım çeşitli adımlar ve hususlar içerir (Şekil 1). Tümör hücrelerinde ve normal hücrelerde olgun miRNA düzeylerinin karşılaştırılması, ilgi bir miRNA seçmek için ortak prosedürlerden biri olabilir (Şekil 1a). Bir Mirna 'nın hücre proliferasyonu üzerindeki etkilerini algılamak için seçilen miRNA 'nın fonksiyonel çalışması, bir miRNA 'nın en iyi potansiyel aday hedefleri listesini daraltmak için önemlidir (Şekil 1B). MiRNAs 'ın deneysel olarak doğrulanmış işlevlerine dayanarak, gen işlevleriyle ilgili en alakalı bilgileri aramak için miRNA hedef tahmin programı ile birlikte literatür ve veritabanı alanında sistematik bir inceleme gereklidir (Şekil 1C). Bir Mirna 'nın gerçek hedef genlerinin tanımlanması, Lusiferaz tahlil gibi bir genin 3 ' UTR, gerçek zamanlı PCR ve Batı blomlama (Şekil 1D) Klonlamayla birlikte deneyler uygulayarak elde edilebilir. Geçerli protokolün amacı, önemli deneylerin kapsamlı yöntemlerini, prob bazlı gerçek zamanlı polimeraz zincir reaksiyonu (PCR), miRNA mimik transfeksiyonunu takiben sülforhodamin B (SRB) tahlil, doz-yanıt eğrisi üretimi ve bir gen 3 ' UTR klonlama ile birlikte Lusiferaz tahlil. Geçerli protokol bireysel miRNAs fonksiyonları daha iyi bir anlayış ve kanser tedavisinde bir miRNA dolaylı için yararlı olabilir.
Access restricted. Please log in or start a trial to view this content.
Protokol
1. olgun MicroRNA (miRNA) Ifade Analizi
-
Olgun miRNA tamamlayıcı DNA (cDNA) sentezini
- 254 ng toplam RNA ve 4,5 μL deoksirribonuclease ı (DNase ı) karışımları ekleyin ve sonra 18 μL (Şekil 2a) yapmak için PCR Strip-tüpler içine ultra saf su ekleyin. Toplam reaksiyon sayısına dayalı olarak yeterli sayıda DNase ı karışımı kullanarak çeşitli hücre hatlarından arındırılmış her toplam RNA örneği için reaksiyonu hazırlayın.
Not: DNase ı karışımları DNase ı (1,8 μL), ribonükleaz inhibitörü (0,3 μL) ve 25 mm MgCl2 (2,4 μL) ' den oluşur. Toplam RNA 'nın yeniden üretilebilirliği için, bir fenol-klorform bazlı ekstraksiyon yöntemi kullanmak yerine sütun bazlı bir ekstraksiyon yöntemi uygulanmıştı. Bazı mirnas ekstraksiyon verimi, bir fenol-klorform bazlı ekstraksiyon yöntemi11,12kullanırken hücre sayısına bağlı olarak değişebilir bildirilmiştir. - Tüplerin bir termal bisikletçinde kulyın. 37 °C ' de 10 dakika için tüpleri çalıştırın ve 90 °C ' de 5 dakika kuluçdak ile ısı-inactivate DNase ı. Hemen sonra kuluçza tüplerini buzun üzerine yerleştirin.
- Transfer 7,1 μL DNase ben toplam RNA içine 2 yeni tüpler setleri ve sonra eklemek 1,5 μL için antisens astar gliseraldehit-3-fosfat dehidrojenaz (gapdh) geni (Şekil 2B).
Not: CDNA sentezi için toplam RNA miktarı bu adımda 100 ng olur. Gapdh antisens astarların stok konsantrasyonu 10 μM ' dir. gapdh antisens astar ekleme, gen spesifik primer yöntemi kullanarak gapdh cdnas 'ın üretimi içindir. - Tüplerin bir termal bisikletçi kullanarak kulyın. 80 °C ' de 5 dakika boyunca başlayan reaksiyon, 60 °C ' de 5 dakika boyunca reaksiyonu takip eder. kuluçza sonrası boruları hemen buz üzerine yerleştirin.
- Her reaksiyona 3,4 μL ters Transkripsiyon (RT) enzim karışımları ekleyin (Şekil 2B). RT enzim karışımları 100 mm deoksirribonucleotid trifosfat (0,15 μL), 10X RT tampon (1,5 μL), ribonükleaz inhibitörü (0,75 μL) ve Ters transkripsiyon enzim (1 μL) ' den oluşur. Toplam reaksiyon sayısına göre yeterli miktarda karışım hazırlayın.
- Her reaksiyonda belirli bir miRNA için 3 μL 5x RT astar ekleyin (Şekil 2B).
Not: Her reaksiyon için toplam hacim 15 μL 'dir. - Boruları termal bisikletçi ile çalıştırın. 16 °C ' de 30 dakika boyunca, 42 °C ' de 30 dakika ve son olarak 85 °C ' de 5 dakika boyunca tepki olarak başlayın. kalan süre için 4 °C ' de tutun (Şekil 2B). Tek telli cDNAs, aynı tüpteki belirli bir miRNA ve GAPDH geni için bu adımda üretilir.
- 254 ng toplam RNA ve 4,5 μL deoksirribonuclease ı (DNase ı) karışımları ekleyin ve sonra 18 μL (Şekil 2a) yapmak için PCR Strip-tüpler içine ultra saf su ekleyin. Toplam reaksiyon sayısına dayalı olarak yeterli sayıda DNase ı karışımı kullanarak çeşitli hücre hatlarından arındırılmış her toplam RNA örneği için reaksiyonu hazırlayın.
-
Gerçek zamanlı polimeraz zincir reaksiyonu (PCR) ve veri analizi
- 1:49 oranında ultra saf su ile her cDNA seyreltin.
- Belirli bir miRNA ve GAPDH (Tablo 1) için reaksiyon karışımlarının hazırlanması. Belirli bir miRNA ve GAPDH algılanması için her cDNA örnek için triplicate reaksiyonlar ayarlayın.
- Gerçek zamanlı PCR ve veri analizini gerçekleştirin (Şekil 2C). Karşılaştırmalı CT yöntemini kullanarak verileri analiz edin13,14.
2. MicroRNA (miRNA) MimicTransfection
Not: Mirna-107, adım 1 ' den seçilir. MiRNA-107 normal hücrelerle karşılaştırıldığında tümör hücrelerinde aşağı düzenlenmiş olduğundan, miRNA-107 bir tümör bastırıcı miRNA olduğunu spekülasyon olabilir. Tümör hücrelerinde normal hücrelerle karşılaştırıldığında (örneğin, Mirna-301), Mirna-301 karşıantisens oligonükleotidler, 2, 3 ve 4 basamakları için uygulanabilir.
- Sayım odası cihazı ile hücreleri saymak ve 96-Well plaka hücreleri plaka. Hücre yoğunluğu 2 x 103 hücreler/100 μL her kuyu için. Penikilin-streptomisin (P/S) içeren hücre kültürü ortamını kullanmayın, çünkü P/S transfeksiyon verimliliğini azaltabilir.
-
Birkaç son konsantrasyonda miRNA kontrol mimik ve miRNA-107 taklit etmek için bir dizi transfeksiyon karışımı hazırlayın (Şekil 3).
- MiRNA kontrol mimik veya miRNA-107 mimik (25 μM konsantrasyonundan), mikrosantrifüjli borular (Şekil 3A) kullanarak transfeksiyon reakajıyla birlikte azaltılmış serum medyasında uygun miktarda kontrol taklit veya mirna-107 mimik ekleyin. Bir micropipet kullanarak karışımlar içeren oligo hafifçe karıştırın. Oligos toplam miktarı (miRNA mimik kontrol + miRNA-107 mimik) her kuyunda aynı olmalıdır. Boş kuyular 100 μL hücre kültürü ortamını ve hücreler olmadan transfeksiyon reaktif içeren azaltılmış serum medyasını içerir.
- Bir hücre kültürü kaputu içinde 10 dakika kuluçkından sonra, hafifçe tekrar karışımlar içeren oligo karıştırın ve sonra her kuyu içine karışımları 50 μL ekleyin. Nakil hücrelerini bir hücre kültürü kuluçorda tutun. 6-12 h inkübasyon sonrası hem fetal sığır serumu (FBS) hem de P/S içeren yeni hücre kültürü medyasıyla medyayı içeren transfeksiyon reakajının yerini alır. Daha fazla 72 h için hücreleri inkübe. MiRNA mimik toplam tedavi süresi 96 h 'dir.
3. sulforhodamine B (SRB) tahlil
-
Hücre fikreme
- Plaka her iyi hücre kültürü medyayı çıkarın ve hemen her iyi içine% 10 Trikloroasetik asit (TCA) 100 μL doldurun. Dikkatle herhangi bir hücre hasarı ve alt dekolmanı önlemek için her iyi hücre kültürü medya Aspire.
Not: Hazırlamak 40% TCA ekleyerek 20 g TCA toz içine 50 mL damıtılmış su. 40% TCA 'dan itibaren% 10 TCA 'yı 1:3 seyreltme oranındaki distile suyla% 40 TCA seyreltilerek yapın. - 1 h için bir buzdolabında (4 °C)% 10 TCA içeren plaka tutun.
- Su küvetine batarak plakayı birkaç kez yıkayın ve kurutun. Kuyuların içine su bırakılıncaya kadar plakaya dokunarak kuyuların içinden fazla suyu çıkarın. Bir sonraki adıma geçmeden önce kuru bir laboratuar tezgah üzerinde plaka bırakın.
- Plaka her iyi hücre kültürü medyayı çıkarın ve hemen her iyi içine% 10 Trikloroasetik asit (TCA) 100 μL doldurun. Dikkatle herhangi bir hücre hasarı ve alt dekolmanı önlemek için her iyi hücre kültürü medya Aspire.
-
Hücre boyama
- Pipette 50 μL,% 0,4 SRB çözeltisi, boş kuyular dahil her bir kuyu içine. 0,4% SRB solüsyonu sürekli olarak kuyuların alt kısmına kadar yavaşça plakayı sallayın.
Not: Hazırlamak ve 0,4% SRB çözeltisi 100 mL 1% asetik asit içine SRB toz 0,4 g ekleyerek kullanın. Karıştırmak için dikkatle çözümü sallayın. 0,4% SRB çözeltisi şişesini alüminyum folyo gibi hafif koruyucu bir malzemeye sarın. Mağaza 0,4% SRB solüsyonu bir buzdolabında. - 40 dk ila 60 dakika boyunca kuluçken sonra,% 1 asetik asit ile durulama ile plaka yıkayın. İlişkisiz boya tamamen yıkılana kadar plakayı yıkayın (Şekil 3B).
- Bir sonraki adıma geçmeden önce kuru bir laboratuar tezgah üzerinde plaka bırakın.
Not: Plaka tamamen 3,3 adıma gitmeden önce kurutulmalıdır.
- Pipette 50 μL,% 0,4 SRB çözeltisi, boş kuyular dahil her bir kuyu içine. 0,4% SRB solüsyonu sürekli olarak kuyuların alt kısmına kadar yavaşça plakayı sallayın.
-
Absorbans ölçümü
- Pipet 100 μL Tris taban çözeltisi (10 mM), boş kuyular da dahil olmak üzere ilgili kuyuların içine. 10 dakika boyunca bir shaker üzerinde plaka tutun. 492 nm de emici ölçün.
4. doz-reaksiyon eğrisi üretimi
- Bir elektronik tablodaki SRB tahlil verilerini analiz edin. Her grubun emici değerlerden boş emici çıkarın ve her grubun ortalama (AVE) ve standart sapma (STD) emilme değerlerinin hesaplamak.
- Ortalama emilme yüzdesini hesaplayın (AVE%) ve standart sapma (STD%) SRB tahlil absorbans değerleri kullanarak her grubun.
Not: MiRNA kontrolünün AVE% tedavi grubu taklit% 100 olduğunu. Aşağıdaki formülü kullanarak STD% hesaplayın: STD% = (her grup/AVE kontrol mimik tedavi grubu Emil emilme STD) x 100. - Tedavi konsantrasyonları, AVE% ve STD% dahil olmak üzere ham verileri, bu verileri dikey olarak hizalayarak yazılım içine alın. Günlük 0 tanımlanmamış olduğundan, X ekseni ilk konsantrasyonu 0 ' a yakın bir değere ayarlayın (örn., 0,01).
- Grafik sekmesi oluştur 'a tıklayın ve basit dağılım-hata çubukları'nı seçin. Çalışma sayfası sütunlarını simge değerleri olarak seçin ve İleri'yi tıklatın. Veri biçimi panelinde, XY çiftleri seçin ve İleri'yi tıklatın. Seçme veri panelinde ilgili veri sütunlarını seçin. Çizimi oluşturmak için son düğmesini tıklatın.
Not: X ekseni konsantrasyonları temsil eder, Y ekseni her konsantrasyonun ortalama emici yüzdesini gösterir (AVE%), ve hata çubukları her konsantrasyon standart sapması yüzdesini işaret (STD%). - Ölçek türünü ve eksen ölçeklendirmesini değiştirmek için X eksenini çift tıklatın. Ölçek türünü doğrusal olarak günlüğe değiştirin. Başlangıç ve bitiş aralığı numarasını 0,01 ve 200, sırasıyla değiştirin.
-
Herhangi bir Dağılım grafiğine sağ tıklayın, eğri Sığdır 'ı seçin ve Kullanıcı tanımlı alt kategoriye gidin. Doz yanıtı eğrisi 'ni seçin, sonraki düğmeler 'i ve ardından son düğmesini tıklatın. Doz-yanıt eğrisi artık bir rapor sekmesiyle birlikte oluşturulur (Şekil 4A).
- Denklem 1 ' i bir doz yanıt eğrisi oluşturmak için yazılımda girmek için analiz sekmesini tıklatın ve regresyon sihirbazınıseçin. Denklem kategorisinde Kullanıcı tanımlı gidin ve sonra Yeni düğmesini tıklatın. Denklem 1, değişkenler, başlangıç parametreleri ve kısıtlamaları karşılık gelen boş kutulara ekleyin (Şekil 4b, C). Düğme olarak ekle 'yi tıklatın ve denklemin adını doz-yanıt eğrisiolarak ayarlayın. Denklem adı artık denklem kategorisinde Kullanıcı tanımlı alt kategoride oluşturulur. f denklem 1 ' de hücre özelliği (% Cell vability) yüzdesini gösterir.
Denklem 1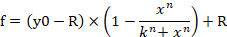
- Rapor sekmesine gidin ve sonra n, k ve R değerlerini kontrol edin.
Not: y0 gösterir 100% hücre canlılığı Mirna kontrol taklit tedavi grubu, n gösterir Hill-tipi katsayısı (bir arsa eğimi), k Mirna konsantrasyonu gösterir-107 taklit bir üretir 50% Mirna-107 taklit maksimum etkisi (yarım maksimal inhibitör konsantrasyonu, ıC50), ve R kalan etkilenmeyen fraksiyonu gösterir (direnç fraksiyonu)15. Bir doz-yanıt eğrisi oluşturmak için kullanılan denklem aralığı y0 tarafından R değerine (varsa)% 100 (Şekil 4A) olarak algılar. Bu nedenle, y0 değerinden sıfır (Şekil 4A) değerine göre hesaplanan ayarlanan k (IC50) değerini elde etmek gereklidir. Ayarlanmış k (ıC50) ile birlikte diğer ICX değerleri (örn., ıc10 Through IC90) denklem 1 ' den türetilmiştir denklemi 2 kullanılarak elde edilebilir. Denklem 2 ' den denklem 1 ' in türetme ek Şekil 1' de belirtilir.
Denklem 2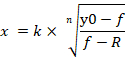
- Denklem 2 ' nin uygulandığı hücrede sol fare düğmesini çift tıklatın. Oluşturulan doz-yanıt eğrisinden denklem 2 ve parametrelerini kullanarak, ıC10 ile IC90 (Şekil 4d) arasında değişen ICX 'in düzeltilmiş değerlerini hesaplamak için kullanılabilir.
- Hücre içinde bir köşeli ayraç ile başlayan formülün ardından eşittir işaretini girin. Formülü girdiğinizde, n, k ve R değerini karşılık gelen sütuna ve satıra dolar işaretini ekleyerek mutlak hücre başvuruları olarak düzeltin, böylece formülü otomatik olarak satırlara doldururken bu sabit değerler değiştirilmez (Şekil 4d). Alternatif olarak, ayarlanan değerler denklem 2 kullanılarak el ile hesaplanabilir.
Not: IC90 değeri, R değeri 10 ' dan büyük olduğundan belirlenmez. Ayrıca, R değeri 20 ' nin üstünde ise, ıC80 değeri de belirlenmemiştir (Şekil 4d).
5. faiz MicroRNA doğrudan hedef geni doğrulama
Not: SRB tahlil gibi fonksiyonel deney yaptıktan sonra, miRNA-107 bir tümör bastırıcı miRNA olarak teyit edilir ve miRNA-107 doğrudan onkogenler hedefleyen son derece uygulanabilir olduğunu. TargetScan (http://www.targetscan.org/vert_71/) gibi bir miRNA hedef tahmin programı kullanarak tüm öngörülen hedef genlerin listesini denetleyin ve sonra PubMed ve dahil veritabanları içinde bir geni işlevine dayalı olası aday hedefleri daraltmak GeneCards.
- 3 ' tercüme edilmemiş bölgenin (UTR) klonlanması için primer tasarım
- GeneCards (https://www.genecards.org/) bir gen adını koyun ve bir geni simgesini tıklatın. Bir geni TOPLULUKBL ID 'yi tıklatarak ve sonra transkriptler kimliği döküm tablosunda tıklatın, toplulukbl genom tarayıcıya değerlendir. Bundan sonra sol tarafta transkript tabanlı görüntüler listesinde exons var 'ı tıklatın.
- 3 ' UTR 'nin nükliotit dizilerini kopyalayın ve primer tasarım programına yapıştırın. Dizileri bu programdan yeniden kopyalayın ve bir sözcük işlemcisine yapıştırın. MiRNA bağlama dizileri varlığı yanı sıra kısıtlama enzimleri siteleri klonlama için kullanılan varlığı kontrol edin.
Not: 3 ' UTR içinde hiçbir kısıtlama enzim tanıma siteleri varsa, klonlama için seçilen kısıtlama enzimleri sonraki adım için kullanılabilir. - Primer tasarım programında, 3 ' UTR dizileri kabul edin ve aşağıdaki koşulla ileri ve ters astar tasarımı başlar. Uzunluk: 20-30 nükleotidler, TM: 45-58 °C, GC%: 40-60%. İki astarın TM değerleri arasındaki fark 5 °C ' den az olmalıdır. Bu çalışmada kullanılan astar dizileri ek Şekil 2' de sağlanmaktadır. Kısıtlama enzim tanıma dizileri yanı sıra 4 rasgele nükleotid tasarlanmış astar ekleyin.
- Degrade PCR
- Tek bir tavlama sıcaklığı (Tablo 2) başına tasarlanan astar dahil 25 ΜL PCR reaksiyon karışımlarının hazırlanması. Toplam reaksiyon sayısına göre yeterli miktarda karışım hazırlayın. Solüsyonu pipetleme ile karıştırın ve her tüpte 25 μL reaksiyon karışımı ekleyin. Tüpleri birkaç saniye santrifüjün.
- 35-40 PCR döngüsü denatürasyon adım uzatma adıma gerçekleştirin. PCR döngüsünü aşağıdaki adımlar olarak ayarlayın: 98 °c için 1 dakika (1 döngü, polimeraz aktivasyon adım), 95 °c için 10 s (denaturation adım), 45 °c-68 °c için 30 s (tavlama adım), 68 °c (uzatma adım, 10 s-1 dk başına 1000 BP), 68 °c 3 dakika (sonlandırma adım) ve son olarak 4 °C ' ye kadar soğutur.
- PCR ürünlerini çalıştırın ve DNA merdivenler ile% 1 agaroz jel üzerinde kontrol bantları. En iyi tavlama sıcaklığını bulun (Şekil 5A). Bir sonraki adım için en iyi tavlama sıcaklığını kullanarak bir genin 3 ' UTR 'i tekrar arttırın.
- Çift sindirimi
- İki kısıtlama enzimleri, XhoI (veya AsiSI) ve NotI dahil olmak üzere bir tüpte (Tablo 3) reaksiyon karışımlarına sahip olun. 3-4 h için bir su banyosu (37 °C) kullanarak karışımlar inküye.
- 1% agaroz jel çift sindirilmiş ürünleri çalıştırın ve sonra UV ışığı altında bantları kesti. Lusiferaz vektörler durumunda, bir jel üzerinde çalıştırmadan önce, iki çift sindirilmiş vektörler tepki 10 U alkalin fosfatazlar için başka bir 1 h ligasyon adım sırasında bir reirkülarizasyon önlemek için.
- Çift sindirilmiş PCR ürünlerini ve Lusiferaz vektörler ile eksiz bantlardan arındırın.
- PCR ürünlerinin Lusiferaz vektörler içine ligasyonu
- DNA ligaz (Tablo 4) dahil olmak üzere 20 μl ligasyon reaksiyonu karışımı yapın.
Not: PCR ürününün molar oranı (Ekle) Lusiferaz vektörüne 3:1 olabilir. 1:1 veya 2:1. - Tüp 10-15 s için kısa bir şekilde santrifüjler ve 16 °C ' de bir termal bisikletçi kullanarak inküye yapın.
Not: Alternatif olarak, tüp ligasyon için 2-3 gün boyunca 4 °C ' de inkübasyon yapılabilir. Bu adımda, PCR kesici uç bir Renilla muhabiri geni (Şekil 5B) aşağı doğru konumlandırılmış bölgeye klonlanır. MiRNAs 'ın klonlanmış 3 ' UTR 'ye bağlanması Renilla aktivitesinde azalabilir. Firefly Lusiferaz Renilla ifade seviyeleri normalleştirme içindir.
- DNA ligaz (Tablo 4) dahil olmak üzere 20 μl ligasyon reaksiyonu karışımı yapın.
- Dönüşüm ve koloni PCR
- Ligasyon karışımlarının (3-5 μL) yetkili hücreler içeren tüpüne ekleyin. Hafifçe tüp dokunun ve buz üzerinde tutun (20 dakika).
Not: ligasyon karışımları eklemeden önce buz üzerinde yetkili hücreler Unfreeze. - Tüpü hızlı ve yavaşça bir ısı bloğuna aktarın. Bir ısı şokunun ardından (42 °C için 30 s-1 dak), tüpü 20 dakika boyunca buzun üzerine yerleştirin.
- Luria-Bertani (LB) agar plakasına yetkili hücreleri yayın. Bir kuluçte (37 °C) bir gecede yetkin hücreler büyütün.
Not: Ampisilin (50-100 μg/mL) agar plakasında yer almaktadır. - Ultra saf su içeren 8-Strip tüpler birinde tek bir koloni ve pelletini E. coli seçin. Rastgele seçilmiş 4-8 kolonilerden E. coli pelletini için bu adımı tekrarlayın (Şekil 5c).
- 25 μL E. coli süspansiyon 8-Strip tüpler başka bir set içine aktarın. Şimdi, E. coli süspansiyon tüpler 2 setleri vardır.
Not: Bir tüp koloni PCR için ve başka bir inoculation içindir. E. coli aşı için süspansiyon geçici olarak 4 °c (Şekil 5c) depolanabilir. - E. coli süspansiyon kullanarak koloni PCR gerçekleştirin. Bu adım kolonilerin bir kesici uç içerdiğini belirlemektir. Aşılamak ve bir geni 3 ' UTR (Şekil 5c) barındıran Lusiferaz vektörler yalıtmak için en iyi kolonileri seçin.
Not: Seçilen genlerin her 3 ' UTR için 5.1-5.5 adımını yineleyin. E. coli süspansiyonu ile genomik DNA 'Yı değiştirerek Tablo 2 ' de gösterilen PCR reaksiyonu durumunu takip edin.
- Ligasyon karışımlarının (3-5 μL) yetkili hücreler içeren tüpüne ekleyin. Hafifçe tüp dokunun ve buz üzerinde tutun (20 dakika).
- Luciferase tahlil
- 24-kuyu tabağı hazırlayın. Her bir kuyu için 500 μL hücre kültürü medyasında 1-2 x 104 hücre kullanın. P/s kullanarak transfeksiyon verimliliğini azaltabilir çünkü transfeksiyon için p/s içeren hücre kültürü medya kullanmayın.
- Bir transfeksiyon reakajı (Şekil 5D) kullanarak kontrol mimik veya belirli bir Mirna taklit hücrelerine Lusiferaz vektörler 50 ng transfect. Belirli bir miRNA 'yı birden fazla konsantrasyonda taklit eden efektler taranırsa, toplam oligos miktarını her bir kuyunda aynı tutun (bkz. Adım 2).
- Ertesi gün fosfat tamponlu tuz (PBS) kullanarak iki kez kuyuların içini yıkayın.
- 200 μL lizis reakajının kuyulara uygulanması ve Lusiferaz aktivitesini ölçmeden önce yeterince hücre Lizi yürütmek.
Not: En az 15 dakika bir sallama plaka üzerinde plaka tutun. - Transfer 5-10 μL hücre lysate yeni tüp içine ve eklemek 100 μL reaktif ı. hemen pipetleme ile çözüm karıştırın ve bir Luminometer kullanarak Firefly Lusiferaz etkinliğini okuyun.
Not: 10-15 s için Firefly Lusiferaz etkinliğini okuyun. - Aynı tüpte 100 μL reaktif II ekleyin ve sonra pipetleme ile iki kez karıştırın. Bir Luminometer kullanarak 10-15 s için Renilla Lusiferaz etkinliğini okuyun. Her örnek için 5.6.5 ve 5.6.6 adım yineleyin.
- Renilla 'nın Ateşböceği oranını hesaplayın (Şekil 5e).
Not: Firefly aktivitesinin hücrelerde Lusiferaz yapıları transfeksiyon verimliliği temsil eder.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
MiRNA 'nın sınıflandırılmasının, bir hastalığın gelişimi ve ilerlemesinde miRNAs 'ın beklenen rollerini temel alan verilerin yorumlanması için başarılı ve doğru bir şekilde onaylaması önemlidir. MiRNA-107 ve miRNA-301 seviyeleri, prob bazlı niceliksel PCR kullanılarak üç pankreas hücresi çizginde ölçülmüştür. Hem belirli bir miRNA 'nın hem de aynı reaksiyonda bir referans geni olan cDNAs sentezini veri yeniden üretilebilirliğini artırabilir. Panc-1 ve capan-1 insan pankreatik duktal ...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
MiRNA 'nın bir miRNA 'nın fonksiyonları ile en uygun şekilde belirlenmesine yönelik stratejiler, Mırnas 'ın birden çok rolünün anlaşılması için vazgeçilmez bir unsurdır. MiRNA hedef genlerinin tanımlanması, bir hücrede miRNAs tarafından modüle edilen hücre sinyalizasyon olaylarını yorumlamak için bir kılavuz olabilir. MiRNAs 'ın işlevsel olarak önemli hedef genlerinin bir açıklanması, kanserde miRNA tabanlı bir tedavi geliştirmek için temel bilgi sağlayabilir.
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarların ifşa etmesi gereken hiçbir şey yok.
Teşekkürler
Bu çalışmada, Eğitim Bakanlığı (2017R1D1A3B03035662) tarafından finanse edilen Kore Ulusal Araştırma Vakfı (NRF) aracılığıyla temel bilim araştırma programı tarafından destekleniyordu; ve Hallym Üniversitesi Araştırma Fonu, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
Referanslar
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır