Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un protocole in vitro pour évaluer les niveaux de microARN, les fonctions et les gènes cibles associés dans les cellules tumorales
Dans cet article
Résumé
Ce protocole utilise une réaction en chaîne de polymérase en temps réel (PCR) basée sur la sonde, un prédisme de sulforhodamine B (SRB), 3' régions non traduites (3' UTR) et un résultat de luciferase pour vérifier les gènes cibles d'un miRNA d'intérêt et pour comprendre les fonctions des miRNAs.
Résumé
Les microARN (miARN) sont de petits ARN régulateurs qui sont reconnus pour moduler de nombreuses voies de signalisation intracellulaire dans plusieurs maladies, y compris les cancers. Ces petits ARN régulateurs interagissent principalement avec les 3' régions non traduites (3' UTR) de leurs ARN messagers cibles (ARNM) aboutissant finalement à l'inhibition des processus de décodage des ARNm et à l'augmentation des dégradations de l'ARNm cible. Basé sur les niveaux d'expression et les fonctions intracellulaires, les miRNAs peuvent servir de facteurs régulateurs des ARNm oncogènes et suppresseurs de tumeurs. L'identification des gènes cibles de bonne foi d'un miRNA parmi des centaines, voire des milliers de cibles prédites par le calcul est une étape cruciale pour discerner les rôles et les mécanismes moléculaires de base d'un miRNA d'intérêt. Divers programmes de prédiction cible miRNA sont disponibles pour rechercher les interactions possibles miRNA-mRNA. Cependant, la question la plus difficile est de savoir comment valider les gènes cibles directs d'un miRNA d'intérêt. Ce protocole décrit une stratégie reproductible des méthodes clés sur la façon d'identifier les cibles miRNA liées à la fonction d'un miRNA. Ce protocole présente un guide pratique sur les procédures étape par étape pour découvrir les niveaux de miRNA, les fonctions et les ARNc cibles connexes à l'aide de la réaction en chaîne de polymérase en temps réel (PCR) à base de sonde, d'un test de sulforhodamine B (SRB) à la suite d'une transfétion miRNA imitant la transfection , génération de courbe dose-réponse, et test de luciferase avec le clonage de 3' UTR d'un gène, qui est nécessaire pour une bonne compréhension des rôles des miRNAs individuels.
Introduction
Les microARN (miARN) sont les petites ARN réglementaires qui modulent principalement le processus de traduction et de dégradation des ARN messagers (ARM) en réagissant aux 3' régions non traduites (3' UTR) dans les gènes cibles de bonne foi1. L'expression des miARN peut être réglée par des mécanismes transcriptionnels et post-transcriptionnels. Le déséquilibre de ces mécanismes de réglementation apporte des niveaux d'expression miRNAs incontrôlés et distinctifs dans de nombreuses maladies, y compris les cancers2. Un seul miRNA peut avoir de multiples interactions avec divers ARNm. En conséquence, un ARNm individuel peut être contrôlé par divers miRNAs. Par conséquent, les réseaux de signalisation intracellulaire sont indirectement influencés par des miARN exprimés de façon distinctive par lesquels les troubles physiologiques et les maladies peuvent être initiés et détériorés2,3,4, 5 Annonces , 6. Bien que l'expression altérée des miARN ait été observée dans divers types de cancers, les mécanismes moléculaires qui modulent les manières des cellules cancéreuses en conjonction avec des miARN sont encore largement inconnus.
L'accumulation des preuves a montré que les rôles oncogènes ou tumorales-suppresseurs des miARN dépendent des types de cancers. Par exemple, en ciblant la boîte de tête de fourche o3 (FOXO3), miR-155 favorise la prolifération cellulaire, la métastasie et la chimiorésistance du cancer colorectal7,8. En revanche, la restriction de l'invasion de cellules de glioma est induite par miR-107 parl'intermédiaire de la régulation de la protéine d'homologaire neurogène d'entaille de locus 2 (NOTCH2) expression 9. L'évaluation des interactions miRNA-cible en relation avec les fonctions miRNA est une partie indispensable pour mieux comprendre comment les miARN régulent divers processus biologiques dans les états sains et malades10. En outre, la découverte de cibles de bonne foi (s) de miARN peut en outre fournir une stratégie affinée pour une thérapie miRNA-basée avec divers médicaments anticancéreux. Cependant, le principal défi dans le domaine des miRNAs est l'identification de cibles directes des miRNAs. Ici, des méthodes détaillées sont présentées comme des approches expérimentales reproductibles pour la détermination du gène cible miRNA. La conception expérimentale réussie de l'identification cible miRNA comporte diverses étapes et considérations (figure 1). La comparaison des niveaux mûrs de miRNA dans les cellules de tumeur et les cellules normales peut être l'une des procédures communes pour sélectionner un miRNA d'intérêt (Figure 1A). L'étude fonctionnelle d'un miRNA sélectionné pour détecter les effets d'un miRNA sur la prolifération cellulaire est importante pour réduire la liste des meilleures cibles candidates potentielles d'un miRNA d'intérêt (Figure 1B). Sur la base des fonctions expérimentalement validées des miARN, un examen systématique de la littérature et de la base de données en compagnie d'un programme de prédiction cible miRNA est nécessaire pour rechercher les informations les plus pertinentes sur les fonctions génétiques (figure 1C). L'identification de gènes cibles réels d'un miRNA d'intérêt peut être réalisée en mettant en œuvre des expériences telles que l'analyse de la luciferase avec le clonage de 3' UTR d'un gène, PCR en temps réel, et le ballonnement occidental (Figure 1D). L'objectif du protocole actuel est de fournir des méthodes complètes d'expériences clés, la réaction en chaîne de polymérase en temps réel (PCR) basée sur la sonde, l'analyse de sulforhodamine B (SRB) suivant un miRNA imitant la transfection, la génération de courbes dose-réponse, et luciferase avec le clonage de 3' UTR d'un gène. Le protocole actuel peut être utile pour une meilleure compréhension des fonctions des miARN individuels et de l'implication d'un miRNA dans la thérapie contre le cancer.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Analyse d'expression de MicroRNA mature (miRNA)
-
Synthèse d'ADN complémentaire de miRNA mûr (cDNA)
- Ajouter 254 ng d'ARN total et 4,5 l de mélanges de désoxyribonuclease I (DNase I), puis ajouter de l'eau ultrapure dans des tubes à bande PCR pour faire jusqu'à 18 l (Figure 2A). Préparer la réaction pour chaque échantillon total d'ARN purifié à partir de plusieurs lignées cellulaires en utilisant une quantité suffisante de mélanges DNase I basé sur le nombre total de réactions.
REMARQUE: Les mélanges DNase I sont composés de DNase I (1,8 l), d'inhibiteur de la ribonuclène (0,3 l) et de 25 mM MgCl2 (2,4 l). Pour obtenir de façon reproductible l'ARN total, une méthode d'extraction à base de colonnes a été applicée au lieu d'utiliser une méthode d'extraction à base de phénol-chloroforme. Il a été signalé que le rendement d'extraction de certaines miARN peut être varié en fonction du nombre de cellules lors de l'utilisation d'une méthode d'extraction à base de phénol-chloroforme11,12. - Incuber les tubes dans un cycleur thermique. Faites fonctionner les tubes pendant 10 min à 37 oC, et inactivez la chaleur DNase I par 5 min d'incubation à 90 oC. Placez immédiatement les tubes sur la glace après l'incubation.
- Transférer 7,1 L de DNase J'ai traité l'ARN total dans 2 ensembles de nouveaux tubes, puis ajouter 1,5 L d'amorces antisens pour le gène de la déshydrogénase glycéraldéhyde-3-phosphate (GAPDH) (Figure 2B).
REMARQUE: La quantité d'ARN total pour la synthèse de l'ADNc devient 100 ng à cette étape. La concentration en stock des amorces antisens GAPDH est de 10 M. L'ajout d'amorces antisens GAPDH est pour la génération de cDNA GAPDH utilisant une méthode d'amorce spécifique aux gènes. - Incuber les tubes à l'aide d'un cycleur thermique. Commencer à 80 oC pendant 5 min, puis à 60 oC pendant 5 min. Placez immédiatement les tubes sur la glace après l'incubation.
- Ajouter 3,4 l de mélanges d'enzymes de transcription inversée (RT) dans chaque réaction (Figure 2B). Les mélanges d'enzymes RT sont composés de 100 mM de triphosphates de désoxyribonucléotide (0,15 l), de tampon stéphanois 10x RT (1,5 l), d'inhibiteur de la ribonuclée (0,75 l) et d'enzyme de transcription inversée (1 l). Préparer suffisamment de mélanges en fonction du nombre total de réactions.
- Ajouter 3 apprêt srt de 5x pour un miRNA spécifique dans chaque réaction (Figure 2B).
REMARQUE: Le volume total est de 15 L pour chaque réaction. - Exécutez les tubes à l'aide d'un cycleur thermique. Commencer à 16 oC pendant 30 min, suivi de la réaction à 42 oC pendant 30 min, et enfin à 85 oC pendant 5 min. Tenir à 4 oC pour tout temps restant (figure2B). Des cDNA à brin unique sont générés dans cette étape pour un gène miRNA spécifique et un gène GAPDH dans le même tube.
- Ajouter 254 ng d'ARN total et 4,5 l de mélanges de désoxyribonuclease I (DNase I), puis ajouter de l'eau ultrapure dans des tubes à bande PCR pour faire jusqu'à 18 l (Figure 2A). Préparer la réaction pour chaque échantillon total d'ARN purifié à partir de plusieurs lignées cellulaires en utilisant une quantité suffisante de mélanges DNase I basé sur le nombre total de réactions.
-
Réaction en chaîne de polymérase en temps réel (PCR) et analyse des données
- Diluer chaque cDNA avec de l'eau ultrapure à 1:49 ratio.
- Préparer les mélanges de réaction pour un miRNA spécifique et GAPDH (tableau 1). Pour la détection d'un miRNA et d'un GAPDH spécifiques, configurez des réactions tripliques pour chaque échantillon d'ADNc.
- Effectuer l'analyse en temps réel du PCR et des données (Figure 2C). Analyser les données à l'aide de la méthode comparative CT 13,14.
2. MicroRNA (miRNA) MimicTransfection
REMARQUE : le miRNA-107 est sélectionné à partir de l'étape 1. Puisque miRNA-107 est down-regulated dans les cellules de tumeur comparées aux cellules normales, on peut supposer que miRNA-107 est un miRNA suppressif de tumeur. Dans le cas d'un miRNA qui est jusqu'à-régulé dans les cellules tumorales par rapport aux cellules normales (par exemple,miRNA-301), oligonucléotides antisens contre miRNA-301 peut être appliqué pour les étapes 2, 3, et 4.
- Comptez les cellules à l'abri d'un dispositif de chambre de comptage et plaquez les cellules dans une plaque de 96 puits. La densité cellulaire est de 2 x 103 cellules/100 L pour chaque puits. N'utilisez pas de support de culture cellulaire contenant de la pénicilline-streptomycine (P/S) parce que le P/S peut réduire l'efficacité de la transfection.
-
Préparer un ensemble de mélanges de transfection pour transfect les cellules à plusieurs concentrations finales de miRNA contrôle imiter et miRNA-107 imiter le lendemain (Figure 3).
- À partir du stock (25 m concentration) de miRNA control imiter ou miRNA-107 imiter, diluer et ajouter la quantité correspondante de contrôle imiter ou miRNA-107 imiter dans le milieu à sérum réduit avec un réactif de transfection en utilisant des tubes microcentrifugeur (Figure 3A). Mélanger délicatement les mélanges contenant de l'oligo à l'aide d'une micropipette. La quantité totale d'oligos (miRNA imiter le contrôle miRNA-107 imiter) devrait être la même dans chaque puits. Les puits vierges comprennent 100 l de supports de culture cellulaire et de médias à sérum réduit contenant un réactif de transfection sans cellules.
- Après une incubation de 10 min dans un capuchon de culture cellulaire, mélanger délicatement l'oligo contenant à nouveau les mélanges, puis ajouter 50 l de mélanges dans chaque puits. Gardez les cellules transfectées dans un incubateur de culture cellulaire. Remplacer le réactif transfection contenant des supports par le support de culture cellulaire frais contenant à la fois du sérum bovin fœtal (SGF) et du P/S après l'incubation de 6 à 12 h. Incuber davantage les cellules pendant 72 h. La durée totale du traitement de miRNA imite est de 96 h.
3. Sulforhodamine B (SRB) Etoiles
-
Fixation cellulaire
- Retirez le support de culture cellulaire dans chaque puits de la plaque et remplissez rapidement 100 L d'acide trichloroacétique (TCA) de 10 % dans chaque puits. Aspirez soigneusement le support de culture cellulaire de chaque puits pour éviter tout dommage cellulaire et le détachement du fond.
REMARQUE: Préparer 40% de TCA en ajoutant 20 g de poudre de TCA dans 50 ml d'eau distillée. À partir de 40 % de TCA, faites 10 % de TCA en diluant 40 % de TCA à l'eau distillée à un taux de dilution de 1:3. - Conserver l'assiette contenant 10 % de TCA au réfrigérateur (4 oC) pendant 1 h.
- Laver la plaque plusieurs fois en plongeant dans le bain d'eau et le sécher. Retirer l'excès d'eau de l'intérieur des puits en tapant sur la plaque jusqu'à ce qu'il n'y ait plus d'eau dans les puits. Laissez la plaque sur un banc de laboratoire pour la sécher avant d'aller à l'étape suivante.
- Retirez le support de culture cellulaire dans chaque puits de la plaque et remplissez rapidement 100 L d'acide trichloroacétique (TCA) de 10 % dans chaque puits. Aspirez soigneusement le support de culture cellulaire de chaque puits pour éviter tout dommage cellulaire et le détachement du fond.
-
Coloration cellulaire
- Pipette 50 'L de 0.4% solution SRB dans chaque puits comprenant des puits vides. Secouez doucement la plaque jusqu'à ce que la solution SRB de 0,4 % recouvre systématiquement le fond des puits.
REMARQUE: Préparer et utiliser la solution SRB de 0,4 % en ajoutant 0,4 g de poudre de SRB dans 100 ml d'acide acétique de 1 %. Secouez soigneusement la solution pour la mélanger. Enveloppez la bouteille de la solution SRB de 0,4 % dans un matériau de protection léger comme le papier d'aluminium. Conserver la solution SRB de 0,4 % dans un réfrigérateur. - Après l'incubation de 40 min à 60 min, laver l'assiette en la rinçant à 1 % d'acide acétique. Laver la plaque jusqu'à ce que le colorant non lié soit totalement lavé (Figure 3B).
- Laissez la plaque sur un banc de laboratoire pour la sécher avant d'aller à l'étape suivante.
REMARQUE: La plaque doit être entièrement séchée avant d'aller à l'étape 3.3.
- Pipette 50 'L de 0.4% solution SRB dans chaque puits comprenant des puits vides. Secouez doucement la plaque jusqu'à ce que la solution SRB de 0,4 % recouvre systématiquement le fond des puits.
-
Mesure de l'absorption
- Pipette 100 l de solution de base Tris (10 mM) dans les puits correspondants, y compris les puits blancs. Garder la plaque sur un shaker pendant 10 min. Mesurer l'absorption à 492 nm.
4. Génération d'une courbe dose-réponse
- Analyser les données d'analyse SRB dans une feuille de calcul. Soustrayez l'absorption vierge des valeurs d'absorption de chaque groupe et calculez l'écart moyen (AVE) et l'écart type (MST) des valeurs d'absorption de chaque groupe.
- Calculer le pourcentage d'absorption moyenne (AVE%) et celle de l'écart type (STD%) de chaque groupe en utilisant les valeurs d'absorption de l'assay SRB.
REMARQUE: Le groupe traité par AVE% de contrôle de miRNA est de 100%. Calculez le % des MST à l'aide de la formule suivante : MST % (MST de chaque groupe / absorption aVE du groupe traité par imitation de contrôle) x 100. - Importer les données brutes, y compris les concentrations de traitement, AVE%, et MST% dans le logiciel en alignant verticalement ces données. Étant donné que le journal 0 n'est pas défini, définiz la première concentration de l'axe X à une valeur proche de 0 (par exemple,0,01).
- Cliquez sur Créer l'onglet graphique et choisissez des barres d'erreur de dispersion simples. Sélectionnez les colonnes de feuille de travail en tant que valeurs de symbole et cliquez sur Suivant. Dans le panneau de format de données, sélectionnez paires XY et cliquez sur Suivant. Sélectionnez les colonnes de données correspondantes dans le panneau de données sélectionné. Cliquez sur le bouton Terminer pour créer l'intrigue.
REMARQUE: L'axe X représente les concentrations, l'axe Y indique le pourcentage d'absorption moyenne de chaque concentration (AVE%), et les barres d'erreur indiquent le pourcentage d'écart standard de chaque concentration (STD%). - Double-cliquez sur l'axe X pour modifier le type d'échelle et la mise à l'échelle de l'axe. Changez le type d'échelle du linéaire au journal. Modifier le nombre de plage de départ et d'extrémité à 0,01 et 200, respectivement.
-
Cliquez à droite sur n'importe quelle parcelle de diffusion, choisissez Curve fit, et allez à la sous-catégorie Utilisateur défini. Sélectionnez la courbe Dose-réponse, cliquez sur les boutons Suivant, puis cliquez sur Bouton Finition. La courbe dose-réponse est maintenant générée avec un onglet de rapport (Figure 4A).
- Pour entrer l'équation 1 dans le logiciel pour la génération d'une courbe dose-réponse, cliquez sur l'onglet Analyse et sélectionnez Assistant de Régression. Passez à la catégorie d'équation définie par l'utilisateur, puis cliquez sur le nouveau bouton. Insérer l'équation 1, les variables, les paramètres initiaux et les contraintes dans les cases vierges correspondantes (Figure 4B, C). Cliquez sur Ajouter comme bouton et définir le nom de l'équation comme dose-réponse courbe. Le nom de l'équation est maintenant généré dans la sous-catégorie définie par l'utilisateur dans la catégorie d'équation. f indique le pourcentage de viabilité cellulaire (% de viabilité cellulaire) dans l'équation 1.
Équation 1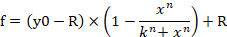
- Allez à l'onglet rapport, puis vérifiez les valeurs n, k et R.
REMARQUE : y0 indique la viabilité cellulaire à 100 % du groupe traité par miRNA control, n indique le coefficient de type Hill (la pente d'une parcelle), k indique la concentration de miRNA-107 imiter qui produit un 50% de l'effet maximal de l'imitation miRNA-107 (la moitié concentration inhibitrice maximale, IC50), et R indique la fraction résiduelle inchangée (la fraction de résistance)15. L'équation utilisée pour générer une courbe dose-réponse reconnaît la fourchette de y0 à la valeur R (le cas échéant) comme 100% (Figure 4A). Par conséquent, il est nécessaire d'acquérir la valeur rajustée k (IC50) qui est calculée sur la base de la fourchette de y0 à une valeur de zéro (Figure 4A). Le k ajusté (IC50) avec d'autres valeurs ICx (par exemple,IC10 à IC90) peut être obtenu en utilisant l'équation 2, qui est dérivée de l'équation 1. La dérivation de l'équation 2 de l'équation 1 est indiquée dans la figure supplémentaire 1.
Équation 2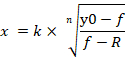
- Double-cliquez sur le bouton de la souris gauche sur la cellule dans laquelle l'équation 2 est appliquée. En utilisant l'équation 2 et les paramètres de la courbe dose-réponse générée, il est disponible pour calculer les valeurs ajustées d'ICx, allant de IC10 à IC90 (Figure 4D).
- Entrez le signe égal suivi de la formule commençant par un support dans la cellule. Lors de l'entrée de la formule, fixer la valeur de n, k, et R que les références de cellule absolue en ajoutant le signe dollar à la colonne correspondante et la ligne, de sorte que ces valeurs fixes ne seront pas modifiées lorsque l'auto-remplissage de la formule vers le bas pour les lignes (Figure 4D). Alternativement, les valeurs ajustées peuvent être calculées manuellement à l'aide de l'équation 2.
REMARQUE: La valeur IC90 n'est pas déterminée parce que la valeur R est supérieure à 10. De plus, si la valeur R est supérieure à 20, la valeur d'IC80 n'est pas non plus déterminée (figure 4D).
5. Vérification du gène cible directe d'un MicroARN d'intérêt
REMARQUE: Après avoir effectué l'expérience fonctionnelle telle que l'analyse de SRB, miRNA-107 est confirmé comme miRNA suppressif de tumeur et il est fortement faisable que miRNA-107 cible directement des oncogenes. Vérifiez la liste de tous les gènes cibles prévus à l'aide d'un programme de prévision cible miRNA comme TargetScan (http://www.targetscan.org/vert_71/), puis réduisez-les aux cibles candidates potentielles en fonction de la fonction d'un gène dans les bases de données, y compris PubMed et Cartes génétiques.
- Conception d'apprêt pour le clonage de la région non traduite de 3' (UTR)
- Mettez le nom d'un gène dans GeneCards (https://www.genecards.org/) et cliquez sur Symbole d'un gène. Évaluez le navigateur du génome Ensembl en cliquant sur Ensembl ID d'un gène, puis cliquez sur Transcriptions ID dans le tableau de transcription. Après cela, cliquez sur Exons existé dans la liste d'affichages basé sur transcription sur la gauche.
- Copiez les séquences de nucléotides de l'UTR 3' et collez-les dans le programme de conception d'amorce. Copiez à nouveau les séquences de ce programme et collez-les dans un traitement de texte. Vérifiez la présence de séquences de liaison miRNA ainsi que la présence de sites d'enzymes de restriction utilisés pour le clonage.
REMARQUE: S'il n'y a aucun site de reconnaissance d'enzyme de restriction dans le 3' UTR, les enzymes de restriction sélectionnées pour le clonage peuvent être employées pour l'étape suivante. - Dans le programme de conception d'amorce, acceptez les séquences UTR de 3' et commencez à concevoir les amorces avant et inverses avec la condition suivante. Longueur : 20-30 nucléotides, Tm : 45-58 oC, GC % : 40-60%. La différence entre les valeurs Tm des deux amorces doit être inférieure à 5 oC. Les séquences d'amorce utilisées dans cette étude sont fournies dans la figure supplémentaire 2. Ajoutez des séquences de reconnaissance enzymatiquede de restriction ainsi que 4 nucléotides aléatoires aux amorces conçues.
- PcR Gradient
- Préparer 25 l de mélanges de réaction PCR, y compris les amorces conçues par température annealing (tableau2). Préparer suffisamment de mélanges en fonction du nombre total de réactions. Mélanger la solution par pipetting et ajouter 25 l de mélanges de réaction dans chaque tube. Centrifuger les tubes pendant quelques secondes.
- Effectuez 35-40 cycles PCR de l'étape de dénaturation à l'étape d'extension. Configurer le cycle PCR comme les étapes suivantes : 98 oC pour 1 min (1 cycle, étape d'activation de la polymérase), 95 oC pour 10 s (étape de dénaturation), 45 oC-68 oC pour 30 s (étape d'annealing), 68 oC (étape d'extension, 10 s-1 min par 1000 bp), 68 oC pour 3 min (étape de terminaison) , et enfin refroidir à 4 oC.
- Exécutez les produits PCR et vérifiez les bandes sur un gel agarose de 1% avec des échelles d'ADN. Trouvez la meilleure température d'annealing (Figure 5A). Amplifiez 3' UTR d'un gène à nouveau en utilisant la meilleure température d'annealing pour l'étape suivante.
- Double digestion
- Faire les mélanges de réaction, y compris deux enzymes de restriction, XhoI (ou AsiSI) et NotI, dans un tube (tableau 3). Incuber les mélanges de 3 à 4 h à l'aide d'un bain d'eau (37 oC).
- Exécuter les produits à double digestion sur un gel agarose 1%, puis couper les bandes sous la lumière UV. Dans le cas des vecteurs de luciferase, avant de fonctionner sur un gel, réagissez à des vecteurs doublement digérés avec 10 U de phosphatases alcaline pendant 1 h pour éviter une recircularisation pendant l'étape de ligature.
- Purifie les produits PCR doublement digérés et les vecteurs de luciferase des bandes excisées.
- Ligation des produits PCR dans les vecteurs de luciférase
- Faire 20 l de mélanges de réaction de ligature, y compris la ligase d'ADN (tableau 4).
REMARQUE: Le rapport molaire du produit PCR (insérer) au vecteur de luciferase peut être 3:1. 1:1 ou 2:1. - Centrifugez brièvement le tube pendant 10-15 s et incuber à 16 oC pendant la nuit à l'aide d'un cycleur thermique.
REMARQUE: Alternativement, le tube peut être incubé à 4 oC pendant 2-3 jours pour la ligature. Dans cette étape, l'insertion PCR sera clonée dans la région positionnée en aval d'un gène de journaliste renilla (Figure 5B). La liaison des miARN dans l'UTR cloné de 3' d'un gène peut diminuer dans l'activité de renilla. La luciferase firefly est pour la normalisation des niveaux d'expression de renilla.
- Faire 20 l de mélanges de réaction de ligature, y compris la ligase d'ADN (tableau 4).
- Transformation et colonie PCR
- Ajouter les mélanges de ligature (3-5 l) dans le tube contenant des cellules compétentes. Appuyez doucement sur le tube et gardez-le sur la glace (20 min).
REMARQUE : Dégelez les cellules compétentes sur la glace avant d'ajouter les mélanges de ligature. - Transférer rapidement et doucement le tube dans un bloc thermique. À la suite d'un choc thermique (42 oC pour 30 s-1 min), placez le tube sur la glace pendant 20 min.
- Étendre les cellules compétentes sur la plaque d'agar Luria-Bertani (LB). Cultivez des cellules compétentes dans un incubateur (37 oC) pendant la nuit.
REMARQUE: L'ampicilline (50-100 g/mL) est contenue dans la plaque d'agar. - Choisissez une colonie individuelle et suspendez E. coli dans l'un des tubes à 8 bandes contenant de l'eau ultrapure. Répétez cette étape pour resuspendre E. coli de 4 à 8 colonies choisies au hasard (figure 5C).
- Transférer 25 lde de suspension E. coli dans un autre ensemble de tubes à 8 bandes. Maintenant, il ya 2 ensembles de tubes de suspension E. coli.
REMARQUE: Un tube est pour la colonie PCR et un autre est pour l'inoculation. La suspension e. coli pour l'inoculation peut être stockée temporairement à 4 oC (figure5C). - Effectuer la colonie PCR en utilisant la suspension E. coli. Cette étape consiste à déterminer si les colonies contiennent un encart. Sélectionnez les meilleures colonies pour inoculer et isoler les vecteurs de luciferase abritant 3' UTR d'un gène (Figure 5C).
REMARQUE: Répétez l'étape 5.1-5.5 pour chaque 3' UTR de gènes sélectionnés. Suivez l'état de réaction de PCR montré dans le tableau 2 en remplaçant l'ADN génomique par la suspension d'E. coli.
- Ajouter les mélanges de ligature (3-5 l) dans le tube contenant des cellules compétentes. Appuyez doucement sur le tube et gardez-le sur la glace (20 min).
- Luciferase assay
- Préparer une assiette de 24 puits. Utilisez 1-2 x 104 cellules dans 500 'L de culture cellulaire pour chaque puits. N'utilisez pas de support de culture cellulaire contenant du P/S pour la transfection, car l'utilisation de P/S peut réduire l'efficacité de la transfection.
- Transfect 50 ng de vecteurs de luciferase dans les cellules avec l'imitation de contrôle ou un miRNA spécifique imitent à l'aide d'un réactif de transfection (Figure 5D). Si le dépistage des effets d'un miRNA spécifique imite à plus d'une concentration, gardez la quantité totale d'oligos même dans chaque puits (voir l'étape 2).
- Laver l'intérieur des puits deux fois à l'aide de phosphate tamponné salin (PBS) le lendemain.
- Appliquer 200 l de lyse réagent dans les puits et effectuer suffisamment de lyse cellulaire avant de mesurer l'activité de la luciferase.
REMARQUE: Garder la plaque sur une plaque tremblante au moins 15 min. - Transférer 5 à 10 l de lysate cellulaire dans le nouveau tube et ajouter 100 l de réactif I. Mélangez immédiatement la solution par pipetage et lisez l'activité de luciferase de luciferase de luciferase de luciferase de luciferase de luciferase de luciferase de luciferase de luciferase de luciferase à l'aide d'un luminomètre.
REMARQUE: Lisez l'activité luciferase luciferase luciferase de luciferase de luciferase de luciferase pour 10-15 s. - Ajouter 100 l de réactif II dans le même tube, puis mélanger par pipetting deux fois. Lisez l'activité de la reniférase pour 10-15 s à l'aide d'un luminomètre. Répétez l'étape 5.6.5 et 5.6.6 pour chaque échantillon.
- Calculer le rapport de la renille à la luciole (figure 5E).
REMARQUE: L'activité de la luciole représente l'efficacité de transfection des constructions de luciferase dans les cellules.
Access restricted. Please log in or start a trial to view this content.
Résultats
La confirmation réussie et précise des niveaux de miRNA est importante pour l'interprétation des données par lesquelles la classification des miARN est possible basée sur les rôles prévus des miRNAs dans le développement et la progression d'une maladie. Les niveaux de miRNA-107 et de miRNA-301 ont été mesurés dans trois lignes de cellules de pancréas utilisant le PCR quantitatif basé sur sonde. La synthèse des ADNC d'un miRNA spécifique et d'un gène de référence dans la même réaction peut augmenter la...
Access restricted. Please log in or start a trial to view this content.
Discussion
Les stratégies pour la détermination des cibles de miRNA de bonne foi avec les fonctions d'un miRNA d'intérêt sont indispensables pour la compréhension des rôles multiples des miRNAs. L'identification des gènes cibles miRNA peut être une ligne directrice pour interpréter les événements de signalisation cellulaire modulées par des miARN dans une cellule. Un dévoilement des gènes cibles fonctionnellement importants des miARN peut fournir les connaissances fondamentales pour développer une thérapie miRNA-bas...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Cette étude a été soutenue par le Programme de recherche en sciences fondamentales par l'intermédiaire de la National Research Foundation of Korea (NRF) financée par le ministère de l'Éducation (2017R1D1A3B03035662); et le Fonds de recherche universitaire Hallym, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
Références
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon