Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein In-Vitro-Protokoll zur Bewertung von MicroRNA-Spiegeln, -Funktionen und assoziierten Zielgenen in Tumorzellen
In diesem Artikel
Zusammenfassung
Dieses Protokoll verwendet eine sondenbasierte Echtzeit-Polymerase-Kettenreaktion (PCR), einen Sulforhodamin-B-Assay (SRB), 3' unübersetztes Klonen (3' UTR) und einen Luziferase-Assay, um die Zielgene einer miRNA von Interesse zu verifizieren und die Funktionen von miRNAs zu verstehen.
Zusammenfassung
MicroRNAs (miRNAs) sind kleine regulatorische RNAs, die anerkannt sind, um zahlreiche intrazelluläre Signalwege bei mehreren Krankheiten, einschließlich Krebserkrankungen, zu modulieren. Diese kleinen regulatorischen RNAs interagieren hauptsächlich mit den 3' unübersetzten Regionen (3' UTR) ihrer Zielboten-RNAs (mRNAs), was letztlich zur Hemmung von Dekodierungsprozessen von mRNAs und zur Erweiterung von Ziel-mRNA-Abbauarten führt. Basierend auf den Expressionsniveaus und intrazellulären Funktionen sind miRNAs in der Lage, als regulatorische Faktoren für onkogene und tumorsuppressive mRNAs zu dienen. Die Identifizierung von gutgläubigen Zielgenen einer miRNA unter Hunderten oder sogar Tausenden von rechnerisch vorhergesagten Zielen ist ein entscheidender Schritt, um die Rollen und grundlegenden molekularen Mechanismen einer miRNA von Interesse zu erkennen. Verschiedene miRNA-Zielvorhersageprogramme stehen zur Verfügung, um mögliche miRNA-mRNA-Interaktionen zu suchen. Die schwierigste Frage ist jedoch, wie direkte Zielgene einer miRNA von Interesse validiert werden können. Dieses Protokoll beschreibt eine reproduzierbare Strategie von Schlüsselmethoden zur Identifizierung von miRNA-Zielen im Zusammenhang mit der Funktion einer miRNA. Dieses Protokoll enthält eine praktische Anleitung zu Schritt-für-Schritt-Verfahren zur Aufdeckung von miRNA-Spiegeln, -Funktionen und zugehörigen Ziel-mRNAs unter Verwendung der sondenbasierten Echtzeit-Polymerase-Kettenreaktion (PCR), Sulforhodamin B (SRB)-Assay nach einer miRNA-Mimiktransfektion , Dosis-Wirkungs-Kurvengenerierung und Luziferase-Assay zusammen mit dem Klonen von 3' UTR eines Gens, das für ein richtiges Verständnis der Rollen einzelner miRNAs notwendig ist.
Einleitung
MicroRNAs (miRNAs) sind die kleinen regulatorischen RNAs, die hauptsächlich den Prozess der Übersetzung und des Abbaus von Messenger RNAs (mRNAs) modulieren, indem sie auf die 3' unübersetzten Regionen (3' UTR) in gutgläubigen Zielgenen1reagieren. Die Expression von miRNAs kann durch transkriptionelle und posttranskriptionelle Mechanismen reguliert werden. Das Ungleichgewicht solcher Regulierungsmechanismen führt zu unkontrollierten und ausgeprägten miRNAs Expressionsniveaus bei zahlreichen Krankheiten, einschließlich Krebsarten2. Eine einzelne miRNA kann mehrere Wechselwirkungen mit verschiedenen mRNAs haben. Entsprechend kann eine einzelne mRNA durch verschiedene miRNAs gesteuert werden. Daher werden intrazelluläre Signalnetze aufwendig durch markant exprimierte miRNAs beeinflusst, durch die physiologische Störungen und Krankheiten initiiert und verschlechtert werden können2,3,4, 5 , 6. Obwohl die veränderte Expression von miRNAs bei verschiedenen Krebsarten beobachtet wurde, sind die molekularen Mechanismen, die die Manieren von Krebszellen in Verbindung mit miRNAs modulieren, noch weitgehend unbekannt.
Die Akkumulierenden Beweise zeigen, dass die onkogenen oder tumorsuppressiven Rollen von miRNAs von den Krebsarten abhängen. Zum Beispiel, indem for targeting Gabelkopf-Box o3 (FOXO3), miR-155 fördert die Zellproliferation, Metastasierung, und Chemoresistenz von Dickdarmkrebs7,8. Im Gegensatz dazu wird die Restriktion der Gliomzellinvasion durch miR-107 durch die Regulation der neurogenen Locus Kerch Homolog Protein 2 (NOTCH2) Expression9induziert. Die Bewertung von miRNA-Ziel-Wechselwirkungen im Zusammenhang mit miRNA-Funktionen ist ein unverzichtbarer Bestandteil, um besser zu verstehen, wie miRNAs verschiedene biologische Prozesse sowohl in gesunden als auch in kranken Zuständen regulieren10. Darüber hinaus kann die Entdeckung von gutgläubigen Zielen von miRNAs eine fein abgestimmte Strategie für eine miRNA-basierte Therapie mit verschiedenen Krebsmedikamenten bieten. Die größte Herausforderung im Bereich der miRNAs ist jedoch die Identifizierung direkter Ziele von miRNAs. Hier werden detaillierte Methoden als reproduzierbare experimentelle Ansätze für die miRNA-Zielgenbestimmung präsentiert. Erfolgreiches experimentelles Design für die miRNA-Zielidentifikation umfasst verschiedene Schritte und Überlegungen (Abbildung 1). Der Vergleich der reifen miRNA-Spiegel in Tumorzellen und normalen Zellen kann eine der üblichen Verfahren zur Auswahl einer miRNA von Interesse sein (Abbildung 1A). Die funktionelle Studie einer ausgewählten miRNA zum Nachweis der Auswirkungen einer miRNA auf die Zellproliferation ist wichtig, um die Liste der besten potenziellen Kandidatenziele einer miRNA von Interesse einzugrenzen (Abbildung 1B). Basierend auf den experimentell validierten Funktionen von miRNAs ist eine systematische Überprüfung von Literatur und Datenbank in Unternehmen mit einem miRNA-Zielvorhersageprogramm erforderlich, um die relevantesten Informationen über Genfunktionen zu durchsuchen (Abbildung 1C). Die Identifizierung von realen Zielgenen einer miRNA von Interesse kann durch die Durchführung von Experimenten wie dem Luziferase-Assay zusammen mit dem Klonen von 3' UTR eines Gens, Echtzeit-PCR und Western Blotting (Abbildung 1D) erreicht werden. Ziel des aktuellen Protokolls ist es, umfassende Methoden für Schlüsselexperimente, die sonsonbasierte Echtzeit-Polymerase-Kettenreaktion (PCR), Sulforhodamin B (SRB) nach einer miRNA-Mimiktransfektion, Dosis-Wirkungs-Kurvengenerierung und luziferase-Assay zusammen mit dem Klonen von 3' UTR eines Gens. Das aktuelle Protokoll kann für ein besseres Verständnis der Funktionen einzelner miRNAs und die Implikation einer miRNA in der Krebstherapie nützlich sein.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Reife MicroRNA (miRNA) Expressionsanalyse
-
Reife miRNA-Komplementär-DNA-Synthese (cDNA)
- Fügen Sie 254 ng der gesamten RNA und 4,5 l Desoxyribonuklease I (DNase I) Gemische hinzu, und fügen Sie dann Reinstwasser in PCR-Streifenröhren ein, um bis zu 18 l zu bilden (Abbildung 2A). Bereiten Sie die Reaktion für jede gesamte RNA-Probe vor, die aus mehreren Zelllinien mit genügend DNase-I-Mischungen gereinigt wird, basierend auf der Gesamtzahl der Reaktionen.
HINWEIS: DNase-I-Mischungen bestehen aus DNase I (1,8 l), Ribonuklease-Inhibitor (0,3 l) und 25 mM MgCl2 (2,4 l). Um die gesamte RNA reproduzierbar zu beschaffen, wurde eine säulenbasierte Extraktionsmethode anstelle einer phenol-chlorformbasierten Extraktionsmethode angewandt. Es wurde berichtet, dass die Extraktionsausbeute einiger miRNAs je nach Anzahl der Zellen variiert werden kann, wenn eine Phenol-Chlorform-basierte Extraktionsmethode11,12verwendet wird. - Inkubieren Sie die Rohre in einem thermischen Cycler. Führen Sie die Rohre 10 min bei 37 °C und wärmeinaktivierend DNase I durch 5 min Inkubation bei 90 °C. Legen Sie die Rohre nach der Inkubation sofort auf Eis.
- Übertragen Sie die gesamte RNA in 2 Sätze neuer Röhrchen und fügen Sie dann 1,5 l Antisense-Primer für das Glyceraldehyd-3-Phosphat-Dehydrogenase (GAPDH)-Gen (GAPDH) hinzu (Abbildung 2B).
HINWEIS: Die Gesamt-RNA-Menge für die cDNA-Synthese wird in diesem Schritt zu 100 ng. Die Lagerkonzentration von GAPDH Antisense Primern beträgt 10 M. Das Hinzufügen von GAPDH Antisense Primern ist für die Erzeugung von GAPDH-CDNAs mit einer genspezifischen Primermethode. - Inkubieren Sie die Rohre mit einem thermischen Cycler. Beginnen Sie bei 80 °C für 5 min, gefolgt von der Reaktion bei 60 °C für 5 min. Legen Sie die Rohre nach der Inkubation sofort auf Eis.
- Fügen Sie in jeder Reaktion 3,4 l Reverse-Transkriptions-Enzymgemische (RT) hinzu (Abbildung 2B). RT-Enzymgemische bestehen aus 100 mM Desoxyribonukleotidtriphosphaten (0,15 l), 10x RT-Puffer (1,5 l), Ribonuklease-Inhibitor (0,75 l) und Reverse-Transkriptionsenzym (1 l). Bereiten Sie genügend Mischungen basierend auf der Gesamtzahl der Reaktionen vor.
- Fügen Sie in jeder Reaktion 3 l 5x RT-Primer für eine bestimmte miRNA hinzu (Abbildung 2B).
HINWEIS: Das Gesamtvolumen beträgt 15 L pro Reaktion. - Führen Sie die Rohre mit einem thermischen Cycler. Beginnen Sie bei 16 °C für 30 min, gefolgt von der Reaktion bei 42 °C für 30 min, und schließlich bei 85 °C für 5 min. Halten Sie bei 4 °C für die verbleibende Zeit (Abbildung2B). In diesem Schritt werden sowohl für ein spezifisches miRNA- als auch für ein GAPDH-Gen in derselben Röhre einsträngige cDNAs erzeugt.
- Fügen Sie 254 ng der gesamten RNA und 4,5 l Desoxyribonuklease I (DNase I) Gemische hinzu, und fügen Sie dann Reinstwasser in PCR-Streifenröhren ein, um bis zu 18 l zu bilden (Abbildung 2A). Bereiten Sie die Reaktion für jede gesamte RNA-Probe vor, die aus mehreren Zelllinien mit genügend DNase-I-Mischungen gereinigt wird, basierend auf der Gesamtzahl der Reaktionen.
-
Echtzeit-Polymerase-Kettenreaktion (PCR) und Datenanalyse
- Verdünnen Sie jede cDNA mit Reinstwasser im Verhältnis 1:49.
- Bereiten Sie die Reaktionsgemische für eine bestimmte miRNA und GAPDH vor (Tabelle 1). Für den Nachweis einer spezifischen miRNA und GAPDH, stellen Sie dreifache Reaktionen für jede cDNA-Probe ein.
- Führen Sie die Echtzeit-PCR- und Datenanalyse durch (Abbildung 2C). Analysieren von Daten mit der vergleichenden C T-Methode13,14.
2. MicroRNA (miRNA) MimicTransfektion
HINWEIS: miRNA-107 wird aus Schritt 1 ausgewählt. Da miRNA-107 in Tumorzellen im Vergleich zu normalen Zellen down-reguliert ist, kann spekuliert werden, dass miRNA-107 eine tumorsuppressive miRNA ist. Bei einer miRNA, die in Tumorzellen im Vergleich zu normalenZellen (z.B. , miRNA-301) hochreguliert ist, können Antisense-Oligonukleotide gegen miRNA-301 für die Schritte 2, 3 und 4 angewendet werden.
- Zählen Sie die Zellen mit einem Zählkammergerät und verkleben Sie die Zellen in einer 96-Well-Platte. Die Zelldichte beträgt 2 x 103 Zellen/100 l für jeden Brunnen. Verwenden Sie keine Zellkulturmedien, die Penicillin-Streptomycin (P/S) enthalten, da P/S die Transfektionseffizienz reduzieren kann.
-
Bereiten Sie eine Reihe von Transfektionsmischungen vor, um die Zellen in mehreren endlichen Konzentrationen von miRNA-Kontrollmimik und miRNA-107 Mimik am nächsten Tag zu transfekieren (Abbildung 3).
- Aus dem Bestand (25 M-Konzentration) der miRNA-Steuerung imitieren oder miRNA-107 imitieren, verdünnen und fügen Sie entsprechende Menge an Kontrollmimik oder miRNA-107 Mimik in den reduzierten Serummedien zusammen mit einem Transfektionsreagenz mit Mikrozentrifugenröhren hinzu (Abbildung 3A). Mischen Sie die Oligo enthaltenden Mischungen vorsichtig mit einer Mikropipette. Die Gesamtmenge der Oligos (miRNA mimisch control + miRNA-107 mimic) sollte in jedem Brunnen gleich sein. Leere Brunnen umfassen 100 L Zellkulturmedien und reduzierte Serummedien, die ein Transfektionsreagenz ohne Zellen enthalten.
- Nach einer 10 min Inkubation in einer Zellkulturhaube die Oligo-haltigen Mischungen vorsichtig wieder mischen und dann 50 l der Mischungen in jeden Brunnen geben. Bewahren Sie die transfizierten Zellen in einem Zellkultur-Inkubator auf. Ersetzen Sie das medienhaltige Transfektionsreagenz durch die Frischzellkulturmedien, die sowohl fetales Rinderserum (FBS) als auch P/S nach 6-12 h Inkubation enthalten. Weitere inkubieren die Zellen für 72 h. Die Gesamtbehandlungsdauer von miRNA mimimimiiert beträgt 96 h.
3. Sulforhodamin B (SRB) Assay
-
Zellfixierung
- Entfernen Sie die Zellkulturmedien in jedem Brunnen der Platte und füllen Sie sofort 100 l 10% Trichloressigsäure (TCA) in jeden Brunnen. Saugen Sie vorsichtig die Zellkulturmedien von jedem Brunnen, um Zellschäden und Ablösung von unten zu vermeiden.
HINWEIS: Bereiten Sie 40% TCA vor, indem Sie 20 g TCA-Pulver in 50 ml destilliertes Wasser geben. Von 40% TCA, machen 10% TCA durch Verdünnung 40% TCA mit destilliertem Wasser bei einem Verdünnungsverhältnis von 1:3. - Bewahren Sie die Platte mit 10% TCA im Kühlschrank (4 °C) für 1 h auf.
- Waschen Sie die Platte mehrmals, indem Sie in die Wasserwanne eintauchen und trocknen. Entfernen Sie überschüssiges Wasser aus dem Inneren der Brunnen, indem Sie die Platte anzapfen, bis kein Wasser mehr in den Brunnen vorhanden ist. Lassen Sie die Platte auf einer Laborbank, um sie zu trocknen, bevor Sie zum nächsten Schritt gehen.
- Entfernen Sie die Zellkulturmedien in jedem Brunnen der Platte und füllen Sie sofort 100 l 10% Trichloressigsäure (TCA) in jeden Brunnen. Saugen Sie vorsichtig die Zellkulturmedien von jedem Brunnen, um Zellschäden und Ablösung von unten zu vermeiden.
-
Zellfärbung
- Pipette 50 l 0,4% SRB-Lösung in jeden Brunnen einschließlich Leerbohrungen. Schütteln Sie die Platte vorsichtig, bis 0,4% SRB-Lösung konsequent den Boden der Brunnen bedeckt.
HINWEIS: Bereiten Sie 0,4% SRB-Lösung vor und verwenden Sie 0,4 g SRB-Pulver in 100 ml 1% Essigsäure. Schütteln Sie die Lösung sorgfältig, um sie zu mischen. Wickeln Sie die Flasche mit 0,4% SRB-Lösung in ein leichtes Schutzmaterial wie Aluminiumfolie. 0,4% SRB-Lösung im Kühlschrank aufbewahren. - Nach der Inkubation für 40 min bis 60 min die Platte waschen, indem Sie sie mit 1% Essigsäure abspülen. Waschen Sie die Platte, bis der ungebundene Farbstoff vollständig weggespült wird (Abbildung 3B).
- Lassen Sie die Platte auf einer Laborbank, um sie zu trocknen, bevor Sie zum nächsten Schritt gehen.
HINWEIS: Die Platte sollte vollständig getrocknet werden, bevor Sie zu Schritt 3.3 gehen.
- Pipette 50 l 0,4% SRB-Lösung in jeden Brunnen einschließlich Leerbohrungen. Schütteln Sie die Platte vorsichtig, bis 0,4% SRB-Lösung konsequent den Boden der Brunnen bedeckt.
-
Absorptionsmessung
- Pipette 100 l Tris Basislösung (10 mM) in die entsprechenden Brunnen einschließlich Leerbrunnen. Halten Sie die Platte 10 min auf einem Shaker. Messen Sie die Absorption bei 492 nm.
4. Erzeugung einer Dosis-Wirkungs-Kurve
- Analysieren Sie die SRB-Assay-Daten in einer Kalkulationstabelle. Subtrahieren Sie die Leerabsorption von den Absorptionswerten jeder Gruppe und berechnen Sie den Durchschnitt (AVE) und die Standardabweichung (STD) der Absorptionswerte jeder Gruppe.
- Berechnen des Prozentsatzes der durchschnittlichen Absorption (AVE%) und die der Standardabweichung (STD%) jeder Gruppe unter Verwendung von Absorptionswerten des SRB-Assays.
HINWEIS: Die AVE% der miRNA-Kontroll-Mimik-behandelten Gruppe beträgt 100%. Berechnen Sie die STD% mit der folgenden Formel: STD% = (STD jeder Gruppe / AVE-Absorption der Kontrollmimik behandelt enikuliert) x 100. - Importieren Sie die Rohdaten einschließlich Behandlungskonzentrationen, AVE% und STD% in die Software, indem Sie diese Daten vertikal ausrichten. Da Log 0 nicht definiert ist, legen Sie die erste Konzentration der X-Achse auf einen Wert nahe 0 fest (z. B.., 0,01).
- Klicken Sie auf Die Registerkarte Diagramm erstellen, und wählen Sie Einfache Streufehlerbalkenaus. Wählen Sie Arbeitsblattspalten als Symbolwerte aus, und klicken Sie auf Weiter. Wählen Sie im Datenformatbedienfeld XY-Paare aus, und klicken Sie auf Weiter. Wählen Sie die entsprechenden Datenspalten im Auswahldatenbedienfeld aus. Klicken Sie auf Fertig stellen, um das Diagramm zu erstellen.
HINWEIS: Die X-Achse stellt die Konzentrationen dar, die Y-Achse gibt den Prozentsatz der durchschnittlichen Absorption jeder Konzentration (AVE) an, und die Fehlerbalken weisen auf den Prozentsatz der Standardabweichung jeder Konzentration (STD) hin. - Doppelklicken Sie auf die X-Achse, um den Maßstabstyp und die Skalierung der Achse zu ändern. Ändern Sie den Maßstabstyp von linear in protokollieren. Ändern Sie die Start- und Endbereichsnummer auf 0,01 bzw. 200.
-
Klicken Sie mit der rechten Maustaste auf ein beliebiges Streudiagramm, wählen Sie Kurve passen, und gehen Sie zur Unterkategorie Benutzerdefiniert. Wählen Sie Dosis-Antwort-Kurve, klicken Sie auf Weiter und dann auf Fertig stellen. Die Dosis-Wirkungs-Kurve wird nun zusammen mit einer Berichtsregisterkarte generiert (Abbildung 4A).
- Um Gleichung 1 in die Software für die Generierung einer Dosis-Wirkungs-Kurve einzugeben, klicken Sie auf die Registerkarte Analyse, und wählen Sie Regressions-Assistentaus. Wechseln Sie zu Benutzerdefiniert in der Gleichungskategorie, und klicken Sie dann auf die Schaltfläche Neu. Fügen Sie Gleichung 1, Variablen, Anfangsparameter und Abhängigkeiten in die entsprechenden leeren Felder ein (Abbildung 4B, C). Klicken Sie auf Als hinzufügen, und legen Sie den Namen der Gleichung als Dosis-Antwort-Kurvefest. Der Gleichungsname wird nun in der Unterkategorie Benutzerdefiniert in der Gleichungskategorie generiert. f gibt den Prozentsatz der Zelllebensfähigkeit (% Zelllebensfähigkeit) in Gleichung 1 an.
Gleichung 1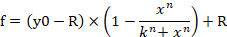
- Wechseln Sie zur Registerkarte Bericht, und überprüfen Sie dann die N-, K- und R-Werte.
ANMERKUNG: y0 zeigt 100% Zelllebensfähigkeit der miRNA-Kontroll-Mimik-behandelten Gruppe an, n zeigt den Hill-Typ-Koeffizienten (die Steigung eines Diagramms), k zeigt die Konzentration von miRNA-107 Mimik an, die eine 50% der maximalen Wirkung der miRNA-107 Mimik erzeugt (die Hälfte maximale hemmende Konzentration, IC50), und R zeigt die verbleibende nicht betroffene Fraktion (die Widerstandsfraktion)15an. Die Gleichung, die zum Generieren einer Dosis-Wirkungs-Kurve verwendet wird, erkennt den Bereich von y0 bis R-Wert (falls vorhanden) als 100 % (Abbildung 4A). Daher ist es notwendig, den angepassten k (IC50) Wert zu erfassen, der auf der Grundlage des Bereichs von y0 bis zu einem Wert von Null berechnet wird (Abbildung 4A). Angepasst k (IC50) zusammen mit anderenICx-Werten (z.B. , IC10 bis IC90) kann mit Gleichung 2 ermittelt werden, die aus Gleichung 1 abgeleitet ist. Die Ableitung von Gleichung 2 aus Gleichung 1 ist in der Ergänzenden Abbildung 1angegeben.
Gleichung 2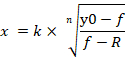
- Doppelklicken Sie auf die linke Maustaste auf die Zelle, in der Gleichung 2 angewendet wird. Unter Verwendung von Gleichung 2 und Parametern aus der generierten Dosis-Wirkungs-Kurve steht es zur Verfügung, um die angepassten Werte von ICx von IC10 bis IC90 zu berechnen (Abbildung 4D).
- Geben Sie das Gleichheitszeichen gefolgt von der Formel ein, die mit einer Klammer in der Zelle beginnt. Legen Sie bei der Eingabe der Formel den Wert von n, k und R als absolute Zellbezüge fest, indem Sie das Dollarzeichen zur entsprechenden Spalte und Zeile hinzufügen, damit diese festen Werte nicht beim automatischen Ausfüllen der Formel bis zu den Zeilen geändert werden (Abbildung 4D). Alternativ können angepasste Werte manuell mit Gleichung 2 berechnet werden.
HINWEIS: Der IC 90-Wert wird nicht ermittelt, da der R-Wert größer als 10 ist. Wenn der R-Wert über 20 liegt, wird auch der Wert von IC80 nicht ermittelt (Abbildung 4D).
5. Überprüfung des direkten Zielgens einer MicroRNA von Interesse
HINWEIS: Nach der Durchführung des funktionellen Experiments wie dem SRB-Assay wird miRNA-107 als tumorsuppressive miRNA bestätigt und es ist durchaus möglich, dass miRNA-107 direkt auf Onkogene abzielt. Überprüfen Sie die Liste aller vorhergesagten Zielgene mit einem miRNA-Zielvorhersageprogramm wie TargetScan (http://www.targetscan.org/vert_71/), und schränken Sie sie dann auf potenzielle Kandidatenziele ein, basierend auf der Funktion eines Gens in Datenbanken wie PubMed und GeneCards.
- Primer-Design zum Klonen von 3' unübersetzten Regionen (UTR)
- Setzen Sie den Namen eines Gens in GeneCards (https://www.genecards.org/) ein, und klicken Sie auf Symbol eines Gens. Bewerten Sie den Ensembl-Genombrowser, indem Sie auf Ensembl ID eines Gens klicken und dann in der Transkriptiontabelle auf Transkripts-ID klicken. Danach wurde in der Liste Transkript-basierte Anzeigen auf der linken Seite auf Exons geklickt.
- Kopieren Sie die Nukleotidsequenzen des 3' UTR und fügen Sie es in das Primer-Design-Programm ein. Kopieren Sie die Sequenzen aus diesem Programm erneut, und fügen Sie sie in eine Textverarbeitung ein. Überprüfen Sie das Vorhandensein von miRNA-Bindungssequenzen sowie das Vorhandensein von Restriktionsenzymen, die für das Klonen verwendet werden.
HINWEIS: Wenn es innerhalb der 3' UTR keine Beschränkungs-Enzymerkennungsstellen gibt, können die für das Klonen ausgewählten Restriktionsenzyme für den nächsten Schritt verwendet werden. - Akzeptieren Sie im Primer-Design-Programm die 3' UTR-Sequenzen und beginnen Sie, die Vorwärts- und Rückwärtsgrundierung mit der folgenden Bedingung zu entwerfen. Länge: 20-30 Nukleotide, Tm: 45-58 °C, GC%: 40-60%. Die Differenz zwischen den Tm-Werten der beiden Primer sollte weniger als 5 °C betragen. Die in dieser Studie verwendeten Primersequenzen sind in der ergänzenden Abbildung 2dargestellt. Fügen Sie den entworfenen Primern Restriktionsenzymerkennungssequenzen sowie 4 zufällige Nukleotide hinzu.
- GradientPCR
- Bereiten Sie 25 L PCR-Reaktionsgemische vor, einschließlich entworfener Primer pro Glühtemperatur (Tabelle 2). Bereiten Sie genügend Mischungen basierend auf der Gesamtzahl der Reaktionen vor. Mischen Sie die Lösung durch Pipettieren und fügen Sie 25 L Reaktionsgemische in jedes Rohr. Zentrifugieren Sie die Rohre für einige Sekunden.
- Führen Sie 35-40 PCR-Zyklen von Denaturierungsschritt zu Erweiterungsschritt durch. Einrichten des PCR-Zyklus in folgenden Schritten: 98 °C für 1 min (1 Zyklus, Polymerase-Aktivierungsschritt), 95 °C für 10 s (Denaturierungsschritt), 45 °C-68 °C für 30 s (Glühschritt), 68 °C (Verlängerungsschritt, 10 s-1 min pro 1000 bp), 68 °C für 3 min (Beendigungsschritt) und abkühlen auf 4 °C.
- Führen Sie die PCR-Produkte und überprüfen Sie Bänder auf einem 1% Agarose-Gel mit DNA-Leitern. Finden Sie die beste Glühtemperatur (Abbildung 5A). Verstärken Sie 3' UTR eines Gens wieder mit der besten Glühtemperatur für den nächsten Schritt.
- Doppelte Verdauung
- Machen Sie die Reaktionsgemische einschließlich zweier Restriktionsenzyme, XhoI (oder AsiSI) und NotI, in einer Röhre (Tabelle 3). Inkubieren Sie die Mischungen für 3-4 h mit einem Wasserbad (37 °C).
- Führen Sie die doppelt verdauten Produkte auf einem 1% Agarose-Gel und schneiden Sie dann die Bänder unter UV-Licht. Im Falle von Luziferase-Vektoren, vor dem Laufen auf einem Gel, reagieren doppelverdaute Vektoren mit 10 U alkalische Phosphatasen für weitere 1 h, um eine Rezirkulierung während des Ligationsschritts zu verhindern.
- Reinigen Sie die doppelt verdauten PCR-Produkte und Luziferase-Vektoren aus den ausgeschnittenen Bändern.
- Ligation von PCR-Produkten in die Luziferase-Vektoren
- Machen Sie 20 l Ligationsreaktionsgemische einschließlich der DNA-Ligase (Tabelle 4).
HINWEIS: Das Molverhältnis von PCR-Produkt (Insert) zu Luziferase-Vektor kann 3:1 sein. 1:1 oder 2:1. - Kurz zentrifugieren Sie das Rohr für 10-15 s und inkubieren Sie bei 16 °C über Nacht mit einem thermischen Cycler.
HINWEIS: Alternativ kann das Rohr bei 4 °C für 2-3 Tage für die Ligation inkubiert werden. In diesem Schritt wird der PCR-Einsatz in den Bereich geklont, der nach einem Renilla-Reporter-Gen positioniert ist (Abbildung 5B). Die Bindung von miRNAs in die geklonte 3' UTR eines Gens kann die Renilla-Aktivität verringern. Firefly luziferase ist für die Normalisierung der Renilla Ausdruck Ebenen.
- Machen Sie 20 l Ligationsreaktionsgemische einschließlich der DNA-Ligase (Tabelle 4).
- Transformation und Kolonie PCR
- Fügen Sie die Ligationsgemische (3-5 l) in das Rohr mit kompetenten Zellen. Tippen Sie vorsichtig auf die Röhre und halten Sie sie auf Eis (20 min).
HINWEIS: Entfrieren Sie kompetente Zellen auf Eis, bevor Sie die Ligationsmischungen hinzufügen. - Übertragen Sie das Rohr schnell und vorsichtig auf einen Wärmeblock. Nach einem Hitzeschock (42 °C für 30 s-1 min) die Röhre 20 min auf Eis legen.
- Verteilen Sie kompetente Zellen auf der Luria-Bertani (LB) Agarplatte. Wachsen Sie kompetente Zellen in einem Inkubator (37 °C) über Nacht.
HINWEIS: Ampicillin (50-100 g/ml) ist in der Agarplatte enthalten. - Wählen Sie eine einzelne Kolonie aus und setzen Sie E. coli in einem der 8-Streifen-Rohre, die Reinstwasser enthalten, wieder aus. Wiederholen Sie diesen Schritt, um E. coli aus zufällig ausgewählten 4-8 Kolonien wieder auszusetzen (Abbildung 5C).
- Übertragen Sie 25 l E. coli Suspension in einen anderen Satz von 8-Streifen-Röhren. Jetzt gibt es 2 Sätze von Röhren von E. coli Suspension.
HINWEIS: Ein Rohr ist für Kolonie PCR und eine andere ist für die Impfung. E. coli Suspension zur Impfung kann vorübergehend bei 4 °C gelagert werden (Abbildung 5C). - Führen Sie die Kolonie PCR mit E. coli Suspension. In diesem Schritt soll festgestellt werden, ob die Kolonien einen Einsatz enthalten. Wählen Sie die besten Kolonien aus, um Luziferasevektoren zu impfen und zu isolieren, die 3' UTR eines Gens beherbergen (Abbildung 5C).
HINWEIS: Wiederholen Sie Schritt 5.1-5.5 für jede 3' UTR ausgewählter Gene. Folgen Sie dem Zustand der PCR-Reaktion in Tabelle 2, indem Sie die genomische DNA durch E. coli Suspension ersetzen.
- Fügen Sie die Ligationsgemische (3-5 l) in das Rohr mit kompetenten Zellen. Tippen Sie vorsichtig auf die Röhre und halten Sie sie auf Eis (20 min).
- Luziferase-Assay
- Bereiten Sie eine 24-Well-Platte vor. Verwenden Sie 1-2 x 104 Zellen in 500 L Zellkulturmedien für jeden Brunnen. Verwenden Sie für die Transfektion keine Zellkulturmedien, die P/S enthalten, da die Verwendung von P/S die Transfektionseffizienz reduzieren kann.
- Transfekte 50 ng Luziferase-Vektoren in die Zellen mit Kontrollmimik oder einer spezifischen miRNA-Mimik mit einem Transfektionsreagenz (Abbildung 5D). Wenn das Screening der Wirkungen einer bestimmten miRNA in mehr als einer Konzentration imitiert, halten Sie die Gesamtmenge der Oligos in jedem Brunnen gleich (siehe Schritt 2).
- Waschen Sie das Innere der Brunnen zweimal mit Phosphat gepufferter Saline (PBS) am nächsten Tag.
- Tragen Sie 200 l Lysereagenz in die Brunnen auf und führen Sie eine Zelllyse ausreichend durch, bevor Sie die Luziferaseaktivität messen.
HINWEIS: Halten Sie die Platte mindestens 15 min auf einer Schüttelplatte. - Übertragen Sie 5-10 l Zelllysat in das neue Rohr und fügen Sie 100 l Reagenz I hinzu. Mischen Sie die Lösung sofort durch Pipettieren und lesen Sie die Galleifegauchaktivität mit einem Luminometer.
HINWEIS: Lesen Sie die Galleife-Aktivität für 10-15 s. - Fügen Sie 100 l Reagenz II in das gleiche Rohr, und dann mischen, indem Sie zweimal pipetieren. Lesen Sie die Renilla-Luziferase-Aktivität für 10-15 s mit einem Luminometer. Wiederholen Sie Schritt 5.6.5 und 5.6.6 für jede Probe.
- Berechnen Sie das Verhältnis von Renilla zu Gunen (Abbildung 5E).
HINWEIS: Die Aktivität von Firefly stellt die Transfektionseffizienz von Luziferasekonstrukten in die Zellen dar.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Eine erfolgreiche und genaue Bestätigung des miRNA-Spiegels ist wichtig für die Interpretation von Daten, durch die die Klassifizierung von miRNAs auf der Grundlage der erwarteten Rolle von miRNAs bei der Entwicklung und dem Fortschreiten einer Krankheit möglich ist. Die Konzentrationen von miRNA-107 und miRNA-301 wurden in drei Pankreaszelllinien mit der sondenbasierten quantitativen PCR gemessen. Die Synthese von cDNAs sowohl einer spezifischen miRNA als auch eines Referenzgens in derselben Reaktion kann die Reprodu...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Strategien zur Bestimmung von gutgläubigen miRNA-Zielen mit den Funktionen einer miRNA von Interesse sind für das Verständnis mehrerer Rollen von miRNAs unverzichtbar. Die Identifizierung von miRNA-Zielgenen kann eine Richtschnur für die Interpretation der von miRNAs in einer Zelle modulierten Zellsignalereignisse sein. Eine Enthüllung funktionell wichtiger Zielgene von miRNAs kann das grundlegende Wissen zur Entwicklung einer miRNA-basierten Therapie bei Krebs liefern.
Mehrere Methoden w...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Studie wurde vom Basic Science Research Program von der National Research Foundation of Korea (NRF) unterstützt, die vom Bildungsministerium (2017R1D1A3B03035662) gefördert wurde. und Hallym University Research Fund, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
Referenzen
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten