È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un protocollo in vitro per la valutazione dei livelli, delle funzioni e dei geni target associati nelle cellule tumorali
In questo articolo
Riepilogo
Questo protocollo utilizza una reazione a catena di polimerasi in tempo reale basata su sonda (PCR), un saggio SRB (sulforhodamine B), una clonazione di 3 regioni non tradotte (3' UTR) e un analisi luciferasi per verificare i geni bersaglio di un miRNA di interesse e per comprendere le funzioni dei miRNA.
Abstract
I microRNA (miRNA) sono piccoli RNA regolatori che sono riconosciuti per modulare numerose vie di segnalazione intracellulare in diverse malattie tra cui i tumori. Questi piccoli RNA normativi interagiscono principalmente con le regioni non tradotte 3' dei loro RNA messaggeri bersaglio (mRNA) con conseguente inibizione dei processi di decodifica degli mRNA e l'aumento delle degradazioni di mRNA bersaglio. Sulla base dei livelli di espressione e delle funzioni intracellulari, i miRNA sono in grado di fungere da fattori regolatori di mRNA oncogeni e soppressivi. L'identificazione dei geni bersaglio in buona fede di un miRNA tra centinaia o addirittura migliaia di bersagli computazionalmente previsti è un passo cruciale per discernere i ruoli e i meccanismi molecolari di base di un miRNA di interesse. Sono disponibili vari programmi di previsione mirati a miRNA per cercare possibili interazioni miRNA-mRNA. Tuttavia, la domanda più impegnativa è come convalidare i geni bersaglio diretti di un miRNA di interesse. Questo protocollo descrive una strategia riproducibile dei metodi chiave su come identificare gli obiettivi di miRNA correlati alla funzione di un miRNA. Questo protocollo presenta una guida pratica sulle procedure passo-passo per scoprire i livelli di miRNA, le funzioni e gli mRNA bersaglio correlati utilizzando la reazione a catena di polimerasi in tempo reale basata su sonda (PCR), il saggio sulla sulforhodamina B (SRB) a seguito di una trasfezione mimica miRNA , generazione di curve dose-risposta, e l'assaggio di luciferasi insieme alla clonazione di 3' UTR di un gene, che è necessario per una corretta comprensione dei ruoli dei singoli miRNA.
Introduzione
I microRNA (miRNA) sono i piccoli RNA regolatori che modulano principalmente il processo di traduzione e degrado degli RNA messaggeri (mRNA) reagendo alle regioni 3 ' non tradotte (3' UTR) nei geni bersaglio in buona fede1. L'espressione dei miRNA può essere regolata da meccanismi trascrizionali e post-trascrizione. Lo squilibrio di tali meccanismi normativi porta livelli di espressione incontrollati e distintivi dei miRNA in numerose malattie, tra cui i tumori2. Un singolo miRNA può avere più interazioni con diversi mRNA. Di conseguenza, un singolo mRNA può essere controllato da vari miRNA. Pertanto, le reti di segnalazione intracellulare sono influenzate da miRNA espressi in modo distintivo attraverso i quali i disturbi fisiologici e le malattie possono essere avviati e deteriorati2,3,4, 5 Del numero 3( , 6. Sebbene l'espressione alterata dei miRNA sia stata osservata in vari tipi di cancro, i meccanismi molecolari che modulano le maniere delle cellule tumorali in combinazione con i miRNA sono ancora in gran parte sconosciuti.
Sempre più prove hanno dimostrato che i ruoli oncogeni o soppressivi dei miRNA dipendono dai tipi di cancro. Ad esempio, prendendo di mira la box forcella o3 (FOXO3), miR-155 promuove la proliferazione cellulare, la metastasi e la chemoresistance del cancro colorettale7,8. Al contrario, la restrizione dell'invasione delle cellule glioma è indotta dal miR-107 attraverso la regolazione dell'espressione neurogenica di locus notch omolog proteina 2 (NOTCH2)9. La valutazione delle interazioni miRNA-bersaglio in relazione alle funzioni di miRNA è una parte indispensabile per comprendere meglio come i miRNA regolano i vari processi biologici in stati sani e malati10. Inoltre, la scoperta di bersagli in buona fede dei miRNA può fornire ulteriormente una strategia messa a punto per una terapia basata su miRNA con vari farmaci anti-cancro. Tuttavia, la sfida principale nel campo dei miRNA è l'identificazione di obiettivi diretti di miRNA. Qui, i metodi dettagliati sono presentati come approcci sperimentali riproducibili per la determinazione del gene bersaglio miRNA. Il successo della progettazione sperimentale per l'identificazione del bersaglio del miRNA comporta vari passaggi e considerazioni (Figura 1). Il confronto dei livelli di miRNA maturi nelle cellule tumorali e nelle cellule normali può essere una delle procedure comuni per selezionare un miRNA di interesse (Figura 1A). Lo studio funzionale di un miRNA selezionato per rilevare gli effetti di un miRNA sulla proliferazione cellulare è importante per restringere l'elenco dei migliori potenziali bersagli candidati di un miRNA di interesse (Figura 1B). Sulla base delle funzioni sperimentalmente convalidate dei miRNA, è necessaria una revisione sistematica della letteratura e del database in azienda con un programma di previsione del target di miRNA per cercare le informazioni più rilevanti sulle funzioni geniche (Figura 1C). L'identificazione dei geni bersaglio reali di un miRNA di interesse può essere ottenuta implementando esperimenti come il saggio luciferasi insieme alla clonazione di 3' UTR di un gene, PCR in tempo reale e gonfiore occidentale (Figura 1D). L'obiettivo dell'attuale protocollo è quello di fornire metodi completi di esperimenti chiave, la reazione a catena di polimerasi in tempo reale basata su sonda (PCR), il saggio sulla sulforhodamina B (SRB) in seguito a una trasfezione mirnamista, generazione di curve dose-risposta e lucidferasi insieme alla clonazione di 3' UTR di un gene. Il protocollo attuale può essere utile per una migliore comprensione delle funzioni dei singoli miRNA e dell'implicazione di un miRNA nella terapia del cancro.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Analisi dell'espressione del MicroRNA maturo (miRNA)
-
Sintesi del DNA complementare (cDNA) del miRNA maturo
- Aggiungere 254 ng di RNA totale e 4,5 ll di miscele deoxyribonuclease I (DNase I), quindi aggiungere acqua ultrapura nei tubi a strisce PCR per fare fino a 18 l (Figura 2A). Preparare la reazione per ogni campione totale di RNA purificato da diverse linee cellulari utilizzando una quantità sufficiente di miscele di DNase I in base al numero totale di reazioni.
NOT: Le miscele DNase I sono composte da DNase I (1,8 gradi centigradi), inibitore di ribonuclease (0,3 l) e 25 mM MgCl2 (2,4 l). Per procurarsi l'RNA totale, è stato applicato un metodo di estrazione basato su colonne invece di utilizzare un metodo di estrazione basato sul fenloroforo. È stato riferito che il rendimento di estrazione di alcuni miRNA può essere variato a seconda del numero di cellule quando si utilizza un metodo di estrazione basato sul fenolo11,12. - Incubare i tubi in un ciclore termico. Far eseguire i tubi per 10 minuti a 37 gradi centigradi, e inattivare il calore DNase I di 5 minuti di incubazione a 90 gradi centigradi. Posizionare immediatamente i tubi sul ghiaccio dopo l'incubazione.
- Trasferire 7,1 - L di DNase I ha trattato l'RNA totale in 2 serie di nuovi tubi e quindi aggiungere 1,5 - L di primer antisenso per il gene di dehydrogenasi di gliceraldeide-3-fosfato (GAPDH) (Figura 2B).
NOT: La quantità di RNA totale per la sintesi cDNA diventa 100 ng in questa fase. La concentrazione di stock di primer antisenso GAPDH è di 10 -M. L'aggiunta di primer antisense GAPDH è per la generazione di cDNA GAPDH utilizzando un metodo di primer gene-specifico. - Incubare i tubi utilizzando un ciclore termico. Iniziare a 80 gradi centigradi per 5 min seguiti dalla reazione a 60 gradi centigradi per 5 min.
- Aggiungere 3,4 l di miscele di enzimi di trascrizione inversa (RT) in ogni reazione (Figura 2B). Le miscele di enzimi RT sono composte da trestide deoxyribonucleotide da 100 mM (0,15 lgg, 10x RT buffer (1,5 l), inibitore di ribonuclease (0,75L) e enzimi di trascrizione inversa (1 llà). Preparare una quantità sufficiente di miscele in base al numero totale di reazioni.
- Aggiungete 3 primer di 5x RT per un miRNA specifico in ogni reazione (Figura 2B).
NOT: Il volume totale è di 15 l per ogni reazione. - Eseguire i tubi utilizzando un ciclore termico. Iniziare a 16 gradi centigradi per 30 minuti seguito dalla reazione a 42 gradi centigradi per 30 minuti, e infine a 85 gradi centigradi per 5 minuti Conservare a 4 gradi centigradi per qualsiasi tempo rimanente (Figura 2B). In questo passaggio vengono generati cDNA a filamento singolo sia per un miRNA specifico che per un gene GAPDH nello stesso tubo.
- Aggiungere 254 ng di RNA totale e 4,5 ll di miscele deoxyribonuclease I (DNase I), quindi aggiungere acqua ultrapura nei tubi a strisce PCR per fare fino a 18 l (Figura 2A). Preparare la reazione per ogni campione totale di RNA purificato da diverse linee cellulari utilizzando una quantità sufficiente di miscele di DNase I in base al numero totale di reazioni.
-
Reazione della catena di polimerasi in tempo reale (PCR) e analisi dei dati
- Diluire ogni cDNA con acqua ultrapura a un rapporto 1:49.
- Preparare le miscele di reazione per un miRNA specifico e GAPDH (Tabella 1). Per la rilevazione di un miRNA e GAPDH specifici, impostare reazioni triplicate per ogni campione di cDNA.
- Eseguire la PCR in tempo reale e l'analisi dei dati (Figura 2C). Analizzare i dati utilizzando il metodo CT comparativo13,14.
2. MicroRNA (miRNA) MimicTransfection
NOTA: miRNA-107 viene selezionato al punto 1. Poiché il miRNA-107 è down-regolato nelle cellule tumorali rispetto alle cellule normali, si può ipotizzare che il miRNA-107 sia un miRNA soppressivo tumorale. Nel caso di un miRNA che è up-regolato nelle celluletumorali rispetto alle cellule normali (ad esempio, miRNA-301), gli oligonucleotidi antisenso contro il miRNA-301 possono essere applicati per i passi 2, 3 e 4.
- Contare le celle con un dispositivo di conteggio camera e piastrare le cellule in una piastra di 96 pozze. La densità cellulare è 2 x 103 celle/100 -L per ogni pozzo. Non utilizzare mezzi di coltura cellulare contenenti penicillina-streptomicina (P/S) perché P/S può ridurre l'efficienza della trasfezione.
-
Preparare una serie di miscele di trasfezione per trasfecare le cellule a diverse concentrazioni finali di imitazione del controllo mimetica e imitare il giorno successivo (Figura 3).
- Dallo stock (concentrazione di 25 M) di mimimi o miRNA-107 mimica di miRNA, diluire e aggiungere la corrispondente quantità di controllo mimi o miRNA-107 nel supporto a ridotto siero insieme a un reagente di trasfezione utilizzando tubi di microcentrifuga (Figura 3A). Mescolare delicatamente l'oligo contenente miscele utilizzando una micropipetta. La quantità totale di oligos (controllo mimetico miRNA - miRNA-107 imitare) dovrebbe essere la stessa in ogni pozzo. I pozze vuoti includono 100 l di supporti di coltura cellulare e supporti a ridotto siero contenenti un reagente di trasfezione senza cellule.
- Dopo un'incubazione di 10 min in una cappa cellulare, mescolare delicatamente l'oligo contenente miscele di nuovo e quindi aggiungere 50 .L delle miscele in ogni pozzo. Mantenere le cellule trasfette in un'incubatrice di coltura cellulare. Sostituire il reagente di trasfezione contenente i supporti con i mezzi di coltura delle cellule fresche contenenti sia il siero bovino fetale fetale (FBS) che la P/S dopo l'incubazione di 6-12 h. Incubano ulteriormente le cellule per 72 h. La durata totale del trattamento del miRNA mirna è di 96 h.
3. Sulforhodamine B (SRB) Assay
-
Fissazione delle celle
- Rimuovere il supporto di coltura cellulare in ogni pozzetto della piastra e riempire prontamente 100 L di 10% di acido tricloroacetico (TCA) in ogni pozzo. Aspirare con attenzione i mezzi di coltura cellulare da ogni pozzo per evitare danni alle cellule e distacco dal basso.
NOT: Preparare il 40% di TCA aggiungendo 20 g di polvere TCA in 50 mL di acqua distillata. Dal 40% di TCA, fai il 10% di TCA diluindo il 40% di TCA con acqua distillata a un rapporto di diluizione 1:3. - Conservare la piastra contenente il 10% di TCA in frigorifero (4 gradi centigradi) per 1 ora.
- Lavare il piatto più volte immergendosi nella vasca d'acqua e asciugarlo. Rimuovere l'acqua in eccesso dall'interno dei pozzi toccando la piastra fino a quando non c'è più acqua nei pozzi. Lasciare il piatto su un banco di laboratorio per asciugarlo prima di andare al passo successivo.
- Rimuovere il supporto di coltura cellulare in ogni pozzetto della piastra e riempire prontamente 100 L di 10% di acido tricloroacetico (TCA) in ogni pozzo. Aspirare con attenzione i mezzi di coltura cellulare da ogni pozzo per evitare danni alle cellule e distacco dal basso.
-
Colorazione cellulare
- Pipetta 50 -L di 0,4% soluzione SRB in ogni pozzo compresi i pozze vuote. Agitare delicatamente la piastra fino a quando la soluzione SRB 0,4% copre costantemente il fondo dei pozze.
NOT: Preparare e utilizzare la soluzione SRB 0,4% aggiungendo 0,4 g di polvere SRB in 100 mL di acido acetico dell'1%. Agitare la soluzione con attenzione per mescolarla. Avvolgere la bottiglia di 0,4% soluzione SRB in un materiale protettivo leggero come la mina di alluminio. Conservare lo 0,4% della soluzione SRB in frigorifero. - Dopo l'incubazione per 40 min a 60 min, lavare la piastra risciacquandola con l'1% di acido acetico. Lavare la piastra fino a quando il tinrinono non legato è completamente lavato via (Figura 3B).
- Lasciare il piatto su un banco di laboratorio per asciugarlo prima di andare al passo successivo.
NOT: La piastra deve essere completamente essiccata prima di andare al passaggio 3.3.
- Pipetta 50 -L di 0,4% soluzione SRB in ogni pozzo compresi i pozze vuote. Agitare delicatamente la piastra fino a quando la soluzione SRB 0,4% copre costantemente il fondo dei pozze.
-
Misurazione dell'assorbimento
- Pipette 100 L di soluzione di base Tris (10 mM) nei pozzi corrispondenti, compresi i pozze vuote. Mantenere la piastra su uno shaker per 10 min. Misurare l'assorbimento a 492 nm.
4. Generazione di una curva dose-risposta
- Analizzare i dati di analisi SRB in un foglio di calcolo. Sottrarre l'assorbimento vuoto dai valori di assorbimento di ciascun gruppo e calcolare la media (AVE) e la deviazione standard (STD) dei valori di assorbimento di ciascun gruppo.
- Calcolare la percentuale di assorbimento medio (AVE%) e quella della deviazione standard (STD%) di ogni gruppo utilizzando i valori di assorbimento del saggio SRB.
NOT: Il aVE% del gruppo trattato con controllo miRNA è del 100%. Calcolare la STD% utilizzando la seguente formula: STD% - (STD di ogni gruppo / assorbimento AVE del controllo imitare il gruppo trattato) x 100. - Importa i dati grezzi, incluse le concentrazioni di trattamento, AVE% e STD% nel software allineando verticalmente tali dati. Poiché il Log 0 non è definito, impostare la prima concentrazione dell'asse X su un valore vicino a 0 (ad esempio,0,01).
- Fare clic sulla scheda Crea grafico e scegliere Barre di errore a dispersione semplice. Selezionare Le colonne del foglio di lavoro come valori di simbolo e fare clic su Avanti. Nel pannello Formato dati, selezionare coppie XY e fare clic su Avanti. Selezionare le colonne di dati corrispondenti nel pannello dati di selezione. Fare clic sul pulsante Fine per creare il grafico.
NOT: L'asse X rappresenta le concentrazioni, l'asse Y indica la percentuale di assorbimento medio di ogni concentrazione (AVE%) e le barre di errore indicano la percentuale di deviazione standard di ogni concentrazione (STD%). - Fare doppio clic sull'asse X per modificare il tipo di scala e il ridimensionamento dell'asse. Modificare il tipo di scala da lineare a log. Modificare il numero di intervallo iniziale e finale rispettivamente su 0,01 e 200.
-
Fare clic con il pulsante destro del mouse su qualsiasi grafico a dispersione, scegliere Adattamento curva e passare alla sottocategoria Definita dall'utente. Selezionare Curva dose-risposta, fare clic su Pulsanti Avanti e quindi fare clic su Fine pulsante. La curva dose-risposta viene ora generata insieme a una scheda del report (Figura 4A).
- Per immettere l'equazione 1 nel software per la generazione di una curva dose-risposta, fare clic sulla scheda Analisi e selezionare Procedura guidata di regressione. Passare a Definito dall'utente nella categoria di equazioni e quindi fare clic sul pulsante Nuovo. Inserire l'equazione 1, le variabili, i parametri iniziali e i vincoli nelle caselle vuote corrispondenti (Figura 4B, C). Fare clic sul pulsante Aggiungi come e impostare il nome dell'equazione come Curva dose-risposta. Il nome dell'equazione viene ora generato nella sottocategoria Definita dall'utente nella categoria di equazioni. f indica la percentuale di fattibilità cellulare (% di fattibilità cellulare) nell'equazione 1.
Equazione 1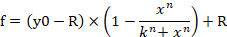
- Passare alla scheda del report e quindi controllare i valori n, k e R.
NOTA: y0 indica la vitalità cellulare al 100% del gruppo trattato con miRNA, n indica il coefficiente di tipo Hill (la pendenza di un grafico), k indica la concentrazione di miRNA-107 imitache che produce un 50% dell'effetto massimo del miRNA-107 (la metà concentrazione inibitoria massima, IC50), e R indica la frazione residua inalterata (la frazione di resistenza)15. L'equazione utilizzata per generare una curva dose-risposta riconosce l'intervallo da y0 a R valore (se presente) come 100% (Figura 4A). Pertanto, è necessario acquisire il valore k (IC50) rettificato calcolato in base all'intervallo da y0 a un valore pari a zero (Figura 4A). Il k regolato (IC50) insieme ad altri valori ICx (ad esempio., da ICda 10 a IC90) può essere ottenuto utilizzando l'equazione 2, derivata dall'equazione 1. La derivazione dell'equazione 2 dall'equazione 1 è indicata nella figura 1 supplementare.
Equazione 2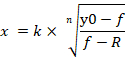
- Fare doppio clic con il pulsante sinistro del mouse sulla cella in cui viene applicata l'equazione 2. Utilizzando l'equazione 2 e i parametri della curva dose-risposta generata, è possibile calcolare i valori rettificati di ICx, che vanno da IC10 a IC90 (Figura 4D).
- Immettere il segno di uguale seguito dalla formula che inizia con una parentesi nella cella. Quando si immette la formula, correggere il valore di n, k e R come riferimenti di cella assoluti aggiungendo il simbolo del dollaro alla colonna e alla riga corrispondenti, in modo che questi valori fissi non vengano modificati quando la formula viene riempita automaticamente nelle righe (Figura 4D). In alternativa, i valori rettificati possono essere calcolati manualmente utilizzando l'equazione 2.
NOT: Valore IC90 non è determinato perché il valore R è maggiore di 10. Inoltre, se il valore di R è superiore a 20, non viene determinato anche il valore di IC80 (Figura 4D).
5. Verifica del gene bersaglio diretto di un microRNA di interesse
NOT: Dopo aver eseguito l'esperimento funzionale come il saggio SRB, il miRNA-107 è confermato come un miRNA soppressivo del tumore ed è altamente fattibile che miRNA-107 si riperdi direttamente agli oncogeni. Controllare l'elenco di tutti i geni bersaglio previsti utilizzando un programma di previsione del bersaglio miRNA come TargetScan (http://www.targetscan.org/vert_71/), quindi restringersi ai potenziali bersagli candidati in base alla funzione di un gene in database, tra cui PubMed e GeneCards.
- Primer design per la clonazione della regione non tradotta a 3' (UTR)
- Inserire il nome di un gene in GeneCards (https://www.genecards.org/) e fare clic su Simbolo di un gene. Valuta il browser del genoma di Ensembl facendo clic su Ensembl ID di un gene e quindi fai clic su Id trascrizione nella tabella delle trascrizioni. Dopo di che, fare clic su Exons esistiti nell'elenco basato su trascrizione viene visualizzato a sinistra.
- Copiare le sequenze nucleotidiche dell'UTR da 3' e incollarlo nel programma di progettazione del primer. Copiare nuovamente le sequenze da questo programma e incollarle in un elaboratore di testi. Controllare la presenza di sequenze di legame miRNA e la presenza di siti di enzimi di restrizione utilizzati per la clonazione.
NOT: Se non ci sono siti di riconoscimento degli enzimi di restrizione all'interno dell'UTR 3', gli enzimi di restrizione selezionati per la clonazione possono essere utilizzati per la fase successiva. - Nel programma di progettazione dei primer, accettare le sequenze UTR 3' e iniziare a progettare i primer avanti e indietro con la seguente condizione. Lunghezza: 20-30 nucleotidi, Tm: 45-58 SC, GC%: 40-60%. La differenza tra i valori Tm dei due primer deve essere inferiore a 5 . Le sequenze di primer utilizzate in questo studio sono fornite nella Figura 2 supplementare. Aggiungi sequenze di riconoscimento degli enzimi di restrizione e 4 nucleotidi casuali ai primer progettati.
- Sfumatura PCR
- Preparare 25 ll l di miscele di reazione PCR tra cui primer progettati per una temperatura di annealing (Tabella 2). Preparare una quantità sufficiente di miscele in base al numero totale di reazioni. Mescolare la soluzione con la pipettatura e aggiungere 25 - L di miscele di reazione in ogni tubo. Centrifugare i tubi per alcuni secondi.
- Eseguire 35-40 cicli PCR dalla fase di denaturazione alla fase di estensione. Impostare il ciclo pcR come segue: 98 gradi centigradi per 1 ciclo, fase di attivazione della polimerasi), 95 C per 10 s (fase di denaturazione), 45 C-68 C per 30 s (passo di irrigazione), 68 gradi (passo di estensione, 10 s-1 min per 1000 bp), 68 c per 3 min (passaggio di interruzione) , e infine raffreddare fino a 4 gradi centigradi.
- Eseguire i prodotti PCR e controllare le bande su un gel di agarose 1% con scale di DNA. Trovare la migliore temperatura di annessione (Figura 5A). Amplifica 3' UTR di un gene utilizzando di nuovo la migliore temperatura di annealing per il passo successivo.
- Doppia digestione
- Fare le miscele di reazione tra cui due enzimi di restrizione, XhoI (o AsiSI) e NotI, in un tubo (Tabella 3). Incubare le miscele per 3-4 h con un bagno d'acqua (37 gradi centigradi).
- Eseguire i prodotti doppi digeriti su un gel di agarose 1% e poi tagliare le bande sotto la luce UV. Nel caso dei vettori luciferasi, prima di essere eseguito su un gel, reagiscono a i vettori a doppio digerimento con 10 U di fosfani alcalini per un altro 1 h per evitare una ricircolazione durante la fase di legatura.
- Purificare i prodotti PCR a doppia digestione e i vettori luciferasi dalle bande eccitate.
- Ligazione dei prodotti PCR nei vettori luciferasi
- Fare 20 - L di miscele di reazione di legatura tra cui il DNA ligase (tabella 4).
NOT: Il rapporto molare tra il prodotto PCR (inserto) e il vettore luciferasi può essere 3:1. 1:1 o 2:1. - Centrifugare brevemente il tubo per 10-15 s e incubare a 16 gradi durante la notte utilizzando un ciclore termico.
NOT: In alternativa, il tubo può essere incubato a 4 gradi centigradi per 2-3 giorni per la legatura. In questa fase, l'inserto PCR verrà clonato nella regione posizionata a valle di un gene di reporter renilla (Figura 5B). Il legame dei miRNA nell'UTR clonato 3' di un gene può diminuire nell'attività della renilla. Firefly luciferase è per la normalizzazione dei livelli di espressione della renilla.
- Fare 20 - L di miscele di reazione di legatura tra cui il DNA ligase (tabella 4).
- Trasformazione e colonia PCR
- Aggiungere le miscele di legatura (3-5 gradi centigradi) nel tubo contenente celle competenti. Toccare delicatamente il tubo e tenerlo sul ghiaccio (20 min).
NOTA: Sbloccare le celle competenti sul ghiaccio prima di aggiungere le miscele di legatura. - Trasferire rapidamente e delicatamente il tubo in un blocco termico. Dopo un'ammortizzatore di calore (42 gradi centigradi per 30 s-1 min), posizionare il tubo sul ghiaccio per 20 minuti.
- Stendere celle competenti sulla piastra di agar Luria-Bertani (LB). Coltivare cellule competenti in un'incubatrice (37 gradi centigradi) durante la notte.
NOT: L'ampicillina è contenuta nella piastra dell'agar. - Scegli una colonia individuale e risospendi nuovamente E. coli in uno dei tubi a 8 strisce contenenti acqua ultrapura. Ripetere questo passaggio per risospendere E. coli da colonie 4-8 selezionate casualmente (Figura 5C).
- Trasferire 25 l di sospensione E. coli in un altro set di tubi a 8 strisce. Ora, ci sono 2 serie di tubi di sospensione E. coli.
NOT: Un tubo è per colonia PCR e un altro è per l'inoculazione. La sospensione E. coli per l'inoculazione può essere temporaneamente conservata a 4 gradi centigradi (Figura5C). - Eseguire la colonia PCR utilizzando la sospensione E. coli. Questo passaggio consente di determinare se le colonie contengono un inserto. Selezionare le migliori colonie per inoculare e isolare vettori luciferasi che ospitano 3' UTR di un gene (Figura 5C).
NOT: Ripetere il passaggio 5.1-5.5 per ogni UTR 3' dei geni selezionati. Seguire le condizioni della reazione PCR mostrate nella Tabella 2 sostituendo il DNA genomico con la sospensione E. coli.
- Aggiungere le miscele di legatura (3-5 gradi centigradi) nel tubo contenente celle competenti. Toccare delicatamente il tubo e tenerlo sul ghiaccio (20 min).
- Luciferasi assay
- Preparare un piatto di 24 pozze. Utilizzare 1-2 x 104 celle in 500 : supporti di coltura cellulare per ogni bene. Non utilizzare supporti di coltura cellulare contenenti P/S per la trasfezione perché l'utilizzo di P/S può ridurre l'efficienza della trasfezione.
- Transfect 50 ng di vettori luciferasi nelle cellule con controllo mimi o un mimio miRNA specifico utilizzando un reagente di trasfezione (Figura 5D). Se lo screening degli effetti di un miRNA specifico imitare a più di una concentrazione, mantenere la quantità totale di oligos stessa in ogni pozzo (vedi passo 2).
- Lavare l'interno dei pozzi due volte utilizzando la salina tampone di fosfato (PBS) il giorno successivo.
- Applicare 200 l di lisi reagente nei pozzi e svolgere sufficientemente la lisi cellulare prima di misurare l'attività di luciferasi.
NOT: Mantenere la piastra su una piastra tremante almeno 15 min. - Trasferire 5-10 luny di cella nel nuovo tubo e aggiungere 100 l of reagent I. Mescolare immediatamente la soluzione pipettando e leggere l'attività di luciferasi della lucciola utilizzando un luminometro.
NOT: Leggi l'attività di luciferasi della lucciola per 10-15 s. - Aggiungere 100 l di reagente II nello stesso tubo, quindi mescolare pipettando due volte. Leggi l'attività di renilla luciferate per 10-15 s utilizzando un luminometro. Ripetere i passaggi 5.6.5 e 5.6.6 per ogni campione.
- Calcolare il rapporto tra renilla e lucciola (Figura 5E).
NOT: L'attività della lucciola rappresenta l'efficienza di trasfezione dei costrutti luciferasi nelle cellule.
Access restricted. Please log in or start a trial to view this content.
Risultati
Una conferma efficace e accurata dei livelli di miRNA è importante per l'interpretazione dei dati con cui la classificazione dei miRNA è possibile sulla base dei ruoli previsti dei miRNA nello sviluppo e nella progressione di una malattia. I livelli di miRNA-107 e miRNA-301 sono stati misurati in tre linee cellulari del pancreas utilizzando la PCR quantitativa basata sulla sonda. La sintesi di cDNA sia di un miRNA specifico che di un gene di riferimento nella stessa reazione può aumentare la riproducibilità dei dati....
Access restricted. Please log in or start a trial to view this content.
Discussione
Le strategie per la determinazione dei bersagli del miRNA in buona fede con le funzioni di un miRNA di interesse sono indispensabili per la comprensione di più ruoli dei miRNA. L'identificazione dei geni bersaglio di miRNA può essere una linea guida per interpretare gli eventi di segnalazione cellulare modulati dai miRNA in una cellula. Una presentazione di geni bersaglio funzionalmente importanti dei miRNA può fornire le conoscenze fondamentali per sviluppare una terapia basata su miRNA nel cancro.
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo studio è stato sostenuto dal Basic Science Research Program attraverso la National Research Foundation of Korea (NRF) finanziata dal Ministero dell'Istruzione (2017R1D1A3B03035662); e Hallym University Research Fund, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
Riferimenti
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon