Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Протокол In Vitro для оценки уровней, функций и связанных с ними целевых генов в опухолевых клетках
В этой статье
Резюме
Этот протокол использует зонд на основе в режиме реального времени полимеразной цепной реакции (PCR), сульфорходамин B (SRB) обзор, 3'непереведенных регионов (3' UTR) клонирования, и luciferase обзор для проверки целевых генов miRNA интереса и понять функции miRNAs.
Аннотация
МикроРНК (миРНК) представляют собой небольшие регулятивные РНК, которые, как признается, модулируют многочисленные внутриклеточные сигнальные пути при нескольких заболеваниях, включая рак. Эти небольшие регулятивные РНК в основном взаимодействуют с 3''e'e'e'd (3' UTR) их целевых РНК-мессенджера (mRNAs), что в конечном итоге приводит к ингибированию процессов декодирования мРНК и увеличению целевых деградаций мРНК. Основываясь на уровнях экспрессии и внутриклеточных функциях, миРНК могут служить регуляторными факторами онкогенных и опухолевых мРНК. Выявление добросовестных целевых генов miRNA среди сотен или даже тысяч вычислительно прогнозируемых целей является важным шагом для определения роли и основных молекулярных механизмов miRNA интереса. Для поиска возможных взаимодействий miRNA-mRNA доступны различные целевые программы miRNA. Однако, самый сложный вопрос заключается в том, как проверить прямые гены цели miRNA интереса. Этот протокол описывает воспроизводимую стратегию ключевых методов по определению целей miRNA, связанных с функцией miRNA. Этот протокол представляет практическое руководство по пошаговым процедурам, чтобы раскрыть уровни miRNA, функции, и связанные с ними целевые мРНК с использованием зонда на основе полимеразной цепной реакции (PCR), сульфорходамин B (SRB) обзор после miRNA имитировать трансфекции , генерация кривой дозы-реакции, и luciferase асссее наряду с клонированием 3' UTR гена, который необходим для правильного понимания ролей отдельных miRNAs.
Введение
МикроРНК (miRNAs) являются небольшие нормативные РНК, которые в основном модулировать процесс перевода и деградации посланникРН (мРНК) реагируя на 3''непереведенных регионов (3' UTR) в добросовестных целевых генов1. Выражение миРНК может регулироваться транскрипционными и посттранскрипционными механизмами. Дисбаланс таких механизмов регулирования приносит неконтролируемые и отличительные уровни выражения miRNAs во многих заболеваниях, включая рак2. Одна miRNA может иметь несколько взаимодействий с различными мРНК. Соответственно, индивидуальная мРНК может контролироваться различными миРНК. Таким образом, внутриклеточные сигнальные сети замысловато влияют на четко выраженные миРНКи, с помощью которых физиологические расстройства и заболевания могут быть инициированы и ухудшились2,3,4, 5 , 6. Хотя измененное выражение miRNAs наблюдалось в различных типах рака, молекулярные механизмы которые модулируют образы раковых клеток в сочетании с miRNAs все еще больш неизвестны.
Накопление доказательств показывает, что онкогенные или опухолевые роли миРНК зависят от видов рака. Например, таргетинг вилочного ящика o3 (FOXO3), miR-155 способствует пролиферации клеток, метастазам и химиорезу колоректального рака7,8. В отличие от этого, ограничение вторжения клеток глиомы индуцируется miR-107 через регуляцию нейрогенного локуса выемки омологического белка 2 (NOTCH2) выражение9. Оценка miRNA-целевых взаимодействий в связи с функциями miRNA является неотъемлемой частью для лучшего понимания того, как miRNAs регулировать различные биологические процессы как в здоровых, так и в больных государствах10. Кроме того, открытие добросовестных целей (ы) miRNAs может дополнительно обеспечить доработанную стратегию для miRNA основе терапии с различными противораковыми препаратами. Однако основной проблемой в области миРНК является выявление прямых целей миРНК. Здесь подробные методы представлены в качестве воспроизводимых экспериментальных подходов к определению гена миРНК. Успешный экспериментальный дизайн для идентификации цели miRNA включает в себя различные шаги и соображения(рисунок 1). Сравнение зрелых уровней миРНК в опухолевых клетках и нормальных клетках может быть одной из распространенных процедур для выбора миРНК интереса(рисунок 1A). Функциональное исследование выбранной miRNA для обнаружения влияния miRNA на пролиферацию клеток важно сузить список лучших потенциальных целей кандидата на miRNA интереса(рисунок 1B). На основе экспериментально подтвержденных функций miRNAs, систематический обзор литературы и базы данных в компании с miRNA целевой программы прогнозирования требуется для поиска наиболее релевантной информации о функциях генов(рисунок 1C). Идентификация реальных генов цели miRNA интереса может быть достигнута путем осуществления экспериментов, таких как luciferase асссе наряду с клонированием 3' UTR гена, в режиме реального времени ПЦР, и западной blotting(Рисунок 1D). Целью текущего протокола является обеспечение комплексных методов ключевых экспериментов, зонд на основе полимеразной цепной реакции (ПЦР), сульфорходамин B (SRB) после мирны имитировать трансфекции, доза-реакция кривой поколения, и люцифераза асссис вместе с клонированием 3' UTR гена. Нынешний протокол может быть полезен для лучшего понимания функций отдельных miRNAs и последствия miRNA в терапии рака.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Зрелый анализ экспрессии микроРНК (miRNA)
-
Зрелый синтез комплементарной ДНК (кДНК) miRNA
- Добавить 254 нг общей РНК и 4,5 л дезоксирибонулеi I (DNase I) смеси, а затем добавить ультрачистую воду в ПЦР полос-труб, чтобы сделать до 18 qL(Рисунок 2A). Подготовьте реакцию для каждого общего образца РНК, очищенного от нескольких клеточных линий, используя достаточное количество смесей DNase I на основе общего количества реакций.
ПРИМЕЧАНИЕ: Смеси DNase I состоят из DNase I (1,8 л), ингибитора рибонуклеаза (0,3 л) и 25 мМ MgCl2 (2,4 л). Для воспроизвоявления для получения общей РНК вместо использования метода извлечения на основе фенола-хлороформа был привит метод извлечения на основе половой РНК. Сообщалось, что выход из визания некоторых миРНК может быть изменен в зависимости от количества клеток при использовании метода экстракции на основе фенола11,12. - Инкубировать трубки в тепловом цикле. Выполнить трубки в течение 10 минут при температуре 37 градусов по Цельсию, и тепло-инактивировать DNase I на 5 мин инкубации при 90 градусов по Цельсию. Немедленно поместите трубки на лед после инкубации.
- Передача 7,1 Л DNase я лечила общую РНК в 2 комплекта новых труб, а затем добавить 1,5 злитромыантийных грунтов к глицеальдегиду-3-фосфатдегеназы (GAPDH) гену(рисунок 2B).
ПРИМЕЧАНИЕ: Сумма общего РНК для синтеза кДНК на этом этапе становится 100 нг. Концентрация запасов праймеров GAPDH антисмыслов составляет 10 км. Добавление антисмысловых праймеров GAPDH предназначено для генерации GAPDH cDNA с использованием генно-специфического метода праймера. - Инкубировать трубки с помощью теплового велосипеда. Начните с 80 градусов по Цельсию в течение 5 минут, а затем реакция на 60 градусов по Цельсию в течение 5 мин. Немедленно поместите трубки на лед после инкубации.
- Добавьте 3,4 л ферментных смесей (RT) в каждой реакции(рисунок 2B). Ферментные смеси RT состоят из 100 мМ дезоксирибонуклеотидных трифосфатов (0,15 л), 10-x RT-буфера (1,5 л), ингибитора рибонуклеаза (0,75 л) и фермента обратной транскрипции (1 кЛ). Приготовьте достаточное количество смесей на основе общего количества реакций.
- Добавьте 3 ЗЛ 5x RT праймеры для конкретной miRNA в каждой реакции(рисунок 2B).
ПРИМЕЧАНИЕ: Общий объем составляет 15 л для каждой реакции. - Выполнить трубки с помощью теплового велосипеда. Начните с 16 градусов по Цельсию в течение 30 мин с последующей реакцией при 42 градусах по Цельсию в течение 30 мин, и, наконец, при 85 градусах по Цельсию в течение 5 мин. Держите при 4 кв. м в течение оставшегося времени(рисунок 2B). Одноцепочечные cDNA генерируются на этом этапе как для конкретного гена miRNA, так и для гена GAPDH в одной трубке.
- Добавить 254 нг общей РНК и 4,5 л дезоксирибонулеi I (DNase I) смеси, а затем добавить ультрачистую воду в ПЦР полос-труб, чтобы сделать до 18 qL(Рисунок 2A). Подготовьте реакцию для каждого общего образца РНК, очищенного от нескольких клеточных линий, используя достаточное количество смесей DNase I на основе общего количества реакций.
-
Цепная реакция полимеразы в реальном времени (ПЦР) и анализ данных
- Разбавить каждую кДНК ультрачистой водой при соотношении 1:49.
- Подготовка реакционных смесей для конкретной miRNA и GAPDH(Таблица 1). Для обнаружения специфической miRNA и GAPDH, установите тройные реакции для каждого образца кДНК.
- Выполните в режиме реального времени ПЦР и анализ данных(рисунок 2C). Анализ данных с помощью сравнительного метода CT 13,14.
2. МимикаТрансфекция микроРНК (miRNA)
ПРИМЕЧАНИЕ: miRNA-107 выбирается из шага 1. Так как miRNA-107 регулируется в опухолевых клетках по сравнению с нормальными клетками, можно предположить, что miRNA-107 является опухолевой супрессивной миРНК. В случае миРНК, которая регулируется в опухолевых клетках посравнению с нормальными клетками (например, miRNA-301), антисмысловые олигонуклеотиды против miRNA-301 могут быть применены для шагов 2, 3 и 4.
- Подсчитайте клетки с прибором счет-камеры и плиту клетки в плите 96-наилучшим образом. Плотность клеток составляет 2 х 103 ячейки/100 Л для каждой скважины. Не используйте среду клеток, содержащий пенициллин-стрептомицин (P/S), потому что P/S может снизить эффективность трансфекции.
-
Подготовьте набор трансфекционных смесей для трансфекционного переноса клеток в нескольких конечных концентрациях мимилиты мирнки и мимилимит мимля-107 на следующий день(рисунок3).
- Из запаса (25 мМ концентрации) мирны управления имитировать или miRNA-107 имитировать, разбавить и добавить соответствующее количество контроля имитировать или miRNA-107 имитировать в сокращенной сыворотки средств наряду с трансфекционным реагентом с использованием микроцентрифуговых труб (Рисунок 3A). Аккуратно смешайте олиго, содержащие смеси, используя микропипет. Общее количество олиго (miRNA имитировать контроль miRNA-107 имитировать) должно быть одинаковым в каждом колодце. Пустые скважины включают 100 зЛ носителей клеточной культуры и сывороточный носитель, содержащий трансфекционный реагент без клеток.
- После 10 мин инкубации в клеточной культуре капот, осторожно смешать олиго, содержащих смеси снова, а затем добавить 50 зл и смесей в каждом колодце. Храните трансинфицированные клетки в инкубаторе клеточной культуры. Замените трансфекционный реагент, содержащий средства массовой информации, свежими средствами культуры клеток, содержащими как сыворотку крупного рогатого скота (FBS), так и P/S после инкубации 6-12 ч. Далее насигните клетки на 72 ч. Общая продолжительность лечения миРНК-мимики составляет 96 ч.
3. Суфоходамин B (SRB) Ассай
-
Фиксация ячеек
- Удалите клеточные культуры носителя в каждой скважине пластины и быстро заполнить 100 л 10% трихлороацетической кислоты (TCA) в каждом колодце. Тщательно аспирировать средства массовой информации клеточной культуры от каждого колодца, чтобы избежать каких-либо повреждений клеток и отрешенности от дна.
ПРИМЕЧАНИЕ: Приготовьте 40% TCA, добавив 20 г порошка TCA в 50 мл дистиллированной воды. От 40% TCA, сделать 10% TCA путем разбавления 40% TCA с дистиллированной водой в 1:3 коэффициент разбавления. - Храните тарелку, содержащую 10% TCA, в холодильнике (4 кВ) на 1 ч.
- Вымойте тарелку несколько раз, погрузившись в ванну и высушите ее. Удалите избыток воды изнутри колодцев, нажав на тарелку, пока в колодцах не останется воды. Оставьте тарелку на лабораторной скамейке, чтобы высушить ее, прежде чем перейти к следующему шагу.
- Удалите клеточные культуры носителя в каждой скважине пластины и быстро заполнить 100 л 10% трихлороацетической кислоты (TCA) в каждом колодце. Тщательно аспирировать средства массовой информации клеточной культуры от каждого колодца, чтобы избежать каких-либо повреждений клеток и отрешенности от дна.
-
Окрашивание клеток
- Pipette 50 л 0,4% SRB решение в каждой скважине, включая пустые скважины. Аккуратно встряхните пластину до 0,4% SRB раствор последовательно охватывает дно скважин.
ПРИМЕЧАНИЕ: Подготовка и использование 0,4% SRB раствор, добавив 0,4 г порошка SRB в 100 мл 1% уксусной кислоты. Встряхните раствор тщательно, чтобы смешать его. Оберните бутылку 0,4% SRB раствор в легкий защитный материал, такой как алюминиевая фольга. Храните 0,4% SRB раствор в холодильнике. - После инкубации в течение 40 мин до 60 мин, мыть тарелку, промывего его с 1% уксусной кислоты. Вымойте тарелку до тех пор, пока не связанный краситель полностью смыт(рисунок 3B).
- Оставьте тарелку на лабораторной скамейке, чтобы высушить ее, прежде чем перейти к следующему шагу.
ПРИМЕЧАНИЕ: Пластина должна быть полностью высушена перед шагом 3.3.
- Pipette 50 л 0,4% SRB решение в каждой скважине, включая пустые скважины. Аккуратно встряхните пластину до 0,4% SRB раствор последовательно охватывает дно скважин.
-
Измерение абсорбции
- Pipette 100 л базового раствора Tris (10 мМ) в соответствующие скважины, включая пустые скважины. Держите тарелку на шейкере в течение 10 мин. Измерьте абсорбцию на уровне 492 нм.
4. Поколение кривой доза-реакции
- Проанализируйте данные sRB-анализа в электронной таблице. Вычесть пустое абсорбцию из значений абсорбции каждой группы и вычислить среднее (AVE) и стандартное отклонение (STD) значений абсорбции каждой группы.
- Рассчитайте процент среднего абсорбции (AVE%) и стандартное отклонение (STD%) каждой группы, используюй значения абсорбции sRB.
ПРИМЕЧАНИЕ: AVE% контроля miRNA имитировать обработанной группы составляет 100%. Рассчитайте STD% с помощью следующей формулы: STD% (STD каждой группы / AVE абсорбции управления имитировать обработанную группу) х 100. - Импортней необработанные данные, включая концентрации обработки, AVE% и STD% в программное обеспечение путем вертикального выравнивания этих данных. Поскольку журнал 0 не определен, установите первую концентрацию оси Xк значению, близкому к 0 (например, 0.01).
- Нажмите Создать вкладку графика и выбрать простые панели рассеяния ошибок. Выберите столбцы листа в качестве значений символов и нажмите далее. В панели формата данных выберите пары XY и нажмите Далее. Выберите соответствующие столбцы данных в панели избранных данных. Нажмите кнопку "Закончить", чтобы создать сюжет.
ПРИМЕЧАНИЕ: Ось X представляет концентрации, ось Y указывает процент среднего поглощения каждой концентрации (AVE), а бары ошибок указывают на процент стандартного отклонения каждой концентрации (STD%). - Дважды нажмите на ось X, чтобы изменить тип шкалы и масштабирование оси. Измените тип шкалы от линейного к журналу. Измените число стартового и конечных диапазонов до 0,01 и 200 соответственно.
-
Нажмите правой кнопкой мыши на любой участок рассеяния, выберите кривую подгонку, и перейдите к подкатегории Пользователь-определенный. Выберите кривую дозы ответа, нажмите кнопки «Следующие кнопки», а затем нажмите кнопку «Финиш». Кривая доза-ответ в настоящее время генерируется вместе с вкладкой отчета(рисунок 4A).
- Для ввода Equation 1 в программном обеспечении для генерации кривой дозы-реакции нажмите на вкладку «Анализ» и выберите мастер регрессии. Перейти к пользователь-определенных в категории уравнения, а затем нажмите новую кнопку. Вставьте уравнение 1, переменные, начальные параметры и ограничения в соответствующие пустые ящики(рисунок 4B,C). Нажмите Добавить в качестве кнопки и установить название уравнения, как доза-ответ кривой. Имя уравнения теперь генерируется в подкатегории Пользователь-определяется в категории уравнения. f указывает процент жизнеспособности клеток (% жизнеспособность клеток) в уравнении 1.
Уравнение 1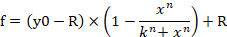
- Перейдите на вкладку отчета, а затем проверьте значения n, k и R.
ПРИМЕЧАНИЕ: y0 указывает 100% жизнеспособность клеток miRNA управления имитировать обработанной группы, n указывает на хилл-типа коэффициент (наклон участка), k указывает на концентрацию miRNA-107 имитировать, что производит 50% от максимального эффекта miRNA-107 (половина максимальная ингибирующая концентрация, IC50) и R указывает на остаточную нетронутую фракцию (фракцию сопротивления)15. Уравнение, используемое для создания кривой дозы-реакции, распознает диапазон от y0 до R значения (если таковые имеется) как 100%(рисунок 4A). Поэтому необходимо приобрести скорректированное значение k (IC50),рассчитанное на основе диапазона от y0 до значения ноль (рисунок4A). Скорректированный k (IC50) наряду с другими значениями ICx (например, IC10 через IC90)можно получить с помощью Equation 2, которое происходит от equation 1. Производные уравнения 2 из уравнения 1 указаны на дополнительной рисунке 1.
Уравнение 2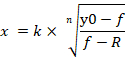
- Дважды щелкните левую кнопку мыши на ячейке, в которой применяется уравнение 2. Используя equation 2 и параметры из сгенерированной кривой доза-реакции, можно вычислить скорректированные значения ICx, начиная от IC10 до IC90 (рисунок4D).
- Ввейте равный знак, за которым следует формула, начинающаяся с кронштейна в ячейке. При вводе формулы, исправить значение n, k, и R в качестве абсолютной ячейки ссылки, добавив знак доллара к соответствующему столбец и строку, так что эти фиксированные значения не будут изменены при автоматическом заполнении формулы до строк (Рисунок 4D). Кроме того, скорректированные значения могут быть вычисляться вручную с помощью Equation 2.
ПРИМЕЧАНИЕ: Значение IC90 не определяется, так как значение R превышает 10. Кроме того, если значение R выше 20, значение IC80 также не определяется(рисунок 4D).
5. Проверка непосредственного целевого гена микроРНК, представляющих интерес
ПРИМЕЧАНИЕ: После выполнения функционального эксперимента, такого как SRB-assay, miRNA-107 подтверждается как подавляющая миРНКа опухоли, и вполне возможно, что miRNA-107 непосредственно нацелен на онкогены. Проверьте список всех прогнозируемых генов-целевых целей, используя программу прогнозирования цели miRNA, такую как TargetScan (http://www.targetscan.org/vert_71/), а затем сузить до потенциальных целей кандидатов на основе функции гена в базах данных, включая PubMed и ДжинКарты.
- Дизайн праймера для клонирования непереведенного региона 3' (UTR)
- Поместите название гена в GeneCards (https://www.genecards.org/) и нажмите Символ гена. Оцените браузер генома Ensembl, нажав На Идентификатор Гена Ensembl, а затем нажмите Transcripts ID в таблице стенограммы. После этого нажмите Exons, существовавший в списке дисплеев на основе стенограммы слева.
- Копируйте нуклеотидные последовательности 3' UTR и вставьте его в программу проектирования грунтовки. Копируйте последовательности снова из этой программы и вставьте его в текстовый процессор. Проверьте наличие последовательности связывания miRNA, а также наличие участков ферментов ограничения, используемых для клонирования.
ПРИМЕЧАНИЕ: Если в 3' UTR нет места распознавания ферментов ограничений, ферменты ограничения, выбранные для клонирования, могут быть использованы для следующего шага. - В программе проектирования грунтовки, принять 3' UTR последовательностей и начать проектировать вперед и обратный грунтовки со следующим условием. Длина: 20-30 нуклеотидов, ТМ: 45-58 градусов по Цельсию, GC%: 40-60%. Разница между значениями ТМ двух грунтовок должна быть меньше 5 градусов по Цельсию. Основные последовательности, используемые в данном исследовании, приведены в дополнительномрисунке 2 . Добавьте в разработанные грунтовки последовательности распознавания ферментов ограничения, а также 4 случайных нуклеотидов.
- Градиент ПЦР
- Приготовьте 25 ЗЛ смесей реакции ПЦР, включая разработанные грунтовки на одну обезвреживание температуры(таблица 2). Приготовьте достаточное количество смесей на основе общего количества реакций. Смешайте раствор путем пипетки и добавьте 25 л реакционных смесей в каждую трубку. Центрифуги труб в течение нескольких секунд.
- Выполните 35-40 циклов ПЦР от шага денатурации до шага расширения. Настройка цикла ПЦР в качестве следующих шагов: 98 c для 1 мин (1 цикл, шаг активации полимераза), 95 C для 10 s (шаг денатурации), 45 C-68 C для 30 s (шаг annealing), 68 C (шаг расширения, 10 s-1 мин на 1000 bp), 68 C для 3 мин прекращения (шаг) , и, наконец, остыть до 4 градусов по Цельсию.
- Запустите продукты ПЦР и проверьте полосы на 1% агарозный гель с ДНК лестницы. Найти лучшие annealing температуры(рисунок 5A). Усиль 3' UTR гена снова, используя лучшую температуру annealing для следующего шага.
- Двойное пищеварение
- Сделать реакции смеси, включая два фермента ограничения, XhoI (или AsiSI) и NotI, в трубке (Таблица 3). Инкубировать смеси в течение 3-4 ч с помощью водяной ванны (37 градусов по Цельсию).
- Запустите двойной переваренных продуктов на 1% агарозный гель, а затем сократить полосы под ультрафиолетовым светом. В случае векторов люциферазы, перед запуском на гель, реагировать двойной переваренных векторов с 10 U щелочных фосфатаз еще 1 ч, чтобы предотвратить реcircularization во время перевязки шаг.
- Очистите двойные переваренные продукты ПЦР и векторы люциферазы из вырезанных полос.
- Лигация продуктов ПЦР в векторы люциферазы
- Сделать 20 qL перевязочной реакции смеси, включая ДНК лигазы (Таблица 4).
ПРИМЕЧАНИЕ: Отношение молярного продукта ПЦР (вставки) к вектору люциферазы может составлять 3:1. 1:1 или 2:1. - Кратко центрифуги трубки для 10-15 s и инкубировать на 16 градусов по Цельсию ночь с помощью теплового велосипедиста.
ПРИМЕЧАНИЕ: Кроме того, трубка может быть инкубирована при 4 градусах По Цельсию в течение 2-3 дней для перевязки. На этом этапе вставка PCR будет клонирована в область, расположенную ниже по течению гена репортера Рениллы(рисунок 5B). Связывание miRNAs в клонированный 3' UTR гена может уменьшить активность ренильи. Светлячок люцифераза для нормализации уровня экспрессии ренилья.
- Сделать 20 qL перевязочной реакции смеси, включая ДНК лигазы (Таблица 4).
- Преобразование и колония PCR
- Добавьте перевязочные смеси (3-5 л) в трубку, содержащую компетентные клетки. Аккуратно коснитесь трубки и держите ее на льду (20 мин).
ПРИМЕЧАНИЕ: Разморозить компетентные клетки на льду перед добавлением перевязочных смесей. - Быстро и аккуратно перенесите трубку в теплоблок. После тепло-шока (42 градусов по Цельсию в течение 30 с-1 мин) поместите трубку на лед в течение 20 мин.
- Распространение компетентных клеток на Лурия-Бертани (LB) агар пластины. Выращивайте компетентные клетки в инкубаторе (37 градусов по Цельсию) в одночасье.
ПРИМЕЧАНИЕ: Ампициллин (50-100 мкг/мл) содержится в агарной пластине. - Выберите индивидуальную колонию и повторно E. coli в одной из 8-полосных труб, содержащих ультрачистую воду. Повторите этот шаг, чтобы повторно e. coli из случайно выбранных 4-8 колоний(рисунок 5C).
- Передача 25 Л подвески кишечной палочки в другой комплект 8-полосных трубок. Теперь, Есть 2 комплекта труб E. coli подвески.
ПРИМЕЧАНИЕ: Одна трубка предназначена для колонии ПЦР, а другая для прививки. E. coli подвеска для прививки может быть временно храниться при 4 C (рисунок5C). - Выполните колонии PCR с помощью E. coli подвески. Этот шаг заключается в том, чтобы определить, если колонии содержат вставку. Выберите лучшие колонии, чтобы привить и изолировать векторы люциферазы укрывательство 3' UTR гена(Рисунок 5C).
ПРИМЕЧАНИЕ: Повторите шаг 5.1-5.5 для каждого 3' UTR выбранных генов. Следуйте состоянию реакции ПЦР, показанной в таблице 2, заменив геномную ДНК суспензией E. coli.
- Добавьте перевязочные смеси (3-5 л) в трубку, содержащую компетентные клетки. Аккуратно коснитесь трубки и держите ее на льду (20 мин).
- Люцифераза ассси
- Приготовьте 24-колодную тарелку. Используйте 1-2 х 104 ячейки в 500 мили культуры клеток для каждого хорошо. Не используйте среду клеточной среды, содержащую P/S для трансфекции, потому что использование P/S может снизить эффективность трансфекции.
- Трансфект 50 нг векторов люциферазы в клетки с контролем имитировать или конкретных miRNA имитировать с помощью трансфекционного реагента(Рисунок 5D). При скрининге эффектов конкретной миРНК имитируют более одной концентрации, сохраняйте общее количество олиго одинаковых в каждом колодце (см. шаг 2).
- Вымойте внутреннюю часть скважин дважды с помощью фосфата буферного сольника (PBS) на следующий день.
- Нанесите 200 л лисисового реагента в скважины и достаточно провести лиза клеток перед измерением активности люциферазы.
ПРИМЕЧАНИЕ: Держите тарелку на встряхиваемая пластине не менее 15 мин. - Передача 5-10 л клеточного лисата в новую трубку и добавить 100 зл реагента I. Немедленно смешать раствор путем пайпетирования и читать светлячок деятельности с помощью светинометра.
ПРИМЕЧАНИЕ: Читайте светлячок люциферазы деятельности для 10-15 с. - Добавить 100 л реагента II в той же трубке, а затем смешать путем пайпеттинг дважды. Прочитайте активность рениллы люциферазы в течение 10-15 с с помощью светинометра. Повторите шаг 5.6.5 и 5.6.6 для каждой выборки.
- Рассчитайте соотношение ренильи к светлячоку(рисунок 5E).
ПРИМЕЧАНИЕ: Активность светлячка представляет трансфекционную эффективность люциферазы, конструируемой в клетки.
Access restricted. Please log in or start a trial to view this content.
Результаты
Успешное и точное подтверждение уровней миРНК имеет важное значение для интерпретации данных, с помощью которых можно классифицировать миРНК на основе ожидаемой роли миРНК в развитии и прогрессировании заболевания. Уровни miRNA-107 и miRNA-301 были измерены в трех линиях клеток поджелудочной...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Стратегии определения добросовестных целей miRNA с функциями miRNA интересов необходимы для понимания нескольких ролей miRNA. Идентификация генов миРНК может быть ориентиром для интерпретации событий сигнализации клеток, модулируемых миРНК в клетке. Открытие функционально важных целевых г...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Это исследование было поддержано Программой фундаментальных научных исследований через Национальный исследовательский фонд Кореи (NRF), финансируемый Министерством образования (2017R1D1A3B03035662); и Исследовательский фонд Галлимского университета, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
Ссылки
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены