A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
פרוטוקול מחוץ לגופית להערכת רמות מיקרוארנה, פונקציות, וגנים היעד הקשורים בתאי הגידול
In This Article
Summary
פרוטוקול זה משתמש ב-החללית מבוסס בזמן אמת בתגובת שרשרת פולימראז (PCR), שיטת sulforhodamine B (SRB), 3 ' אזורים לא מתורגמים (3 ' UTR) שיבוט, ושיטת לוציפראז לאמת את הגנים היעד של miRNA עניין ולהבין את הפונקציות של Mirna.
Abstract
MicroRNAs (miRNAs) הם RNAs הרגולציה קטנים אשר מזוהים לווסת מסלולים מסוימים של איתות תאיים במספר מחלות כולל סרטן. אלה RNAs הרגולציה קטנים בעיקר אינטראקציה עם 3 ' אזורים לא מתורגם (3 ' UTR) של שליח היעד שלהם RNAs (mRNAs) בסופו של דבר וכתוצאה מכך עיכוב תהליכי פענוח של mRNAs ואת הגדלת Mrnas היעד degradations. בהתבסס על רמות הביטוי ופונקציות תאיים, miRNAs מסוגלים לשמש גורמי רגולציה של mRNAs ומדכאים הגידול המדכא. זיהוי של גנים היעד בתום המטרה של miRNA בין מאות או אפילו אלפי מטרות חזוי מבחינה חישובית הוא צעד מכריע כדי להבחין את התפקידים ומנגנונים מולקולאריים בסיסיים של מירנה של עניין. תוכניות מסוימות לחיזוי היעד miRNA זמינים כדי לחפש אינטראקציות miRNA-mRNA אפשרי. עם זאת, השאלה המאתגרים ביותר היא כיצד לאמת גנים מטרה ישירה של miRNA עניין. פרוטוקול זה מתאר אסטרטגיה הניתן לפונקציה של שיטות מפתח כיצד לזהות יעדי miRNA הקשורים לפונקציה miRNA. פרוטוקול זה מציג מדריך מעשי על הליכים צעד אחר צעד כדי לחשוף רמות miRNA, פונקציות, ו-mRNAs הקשורות היעד באמצעות הבדיקה מבוסס בזמן אמת התגובה שרשרת פולימראז (PCR), sulforhodamine B (SRB) שיטת בעקבות מחקה מחקים העברה , מינון התגובה עקומת הדור, ושיטת לוציפראז יחד עם שיבוט של 3 ' UTR של גן, אשר הכרחי להבנה נכונה של התפקידים של miRNAs בודדים.
Introduction
MicroRNAs (miRNAs) הם RNAs הרגולציה קטן כי בעיקר לווסת את תהליך התרגום והשפלה של RNAs שליח (mRNAs) על ידי התגובה ל 3 ' אזורים לא מתורגם (3 ' UTR) בתום היעד לאחד גנים1. ביטוי של miRNAs יכול להיות מוסדר על ידי מנגנונים שלאחר ההמרה. חוסר האיזון של מנגנונים כגון מביא בלתי מבוקרת וייחודית רמות הביטוי miRNAs במחלות רבות כולל סרטן2. ל-miRNA אחת יכול להיות מספר אינטראקציות עם mRNAs מגוונות. בהתאמה, mRNA הפרט יכול להיות נשלט על ידי miRNAs שונים. לכן, רשתות איתות תאיים מושפע באופן מסובכת על ידי ביטוי באופן מתבטא על ידי הפרעות פיסיולוגיים ומחלות ניתן ליזום והידרדר2,3,4, מיכל 5 , 6. למרות הביטוי השונה של mirnas נצפתה סוגים שונים של סרטן, המנגנונים המולקולריים לווסת את הנימוסים של תאים סרטניים בשילוב עם mirnas עדיין לא ידוע ברובו.
צבירת ראיות הראה כי התפקידים אונגניים או מדכאים הגידול של miRNAs תלוי בסוגי סרטן. לדוגמה, על-ידי פילוח forkhead box o3 (FOXO3), מיר-155 מקדם את התפשטות התא, גרורות, ו-cheמורשת של סרטן המעי הגס7,8. לעומת זאת, ההגבלה של הפלישה התאים glioma נגרמת על ידי מיר-107 באמצעות התקנה של חלבון מקור נוירוגניים הומולוג 2 (NOTCH2) ביטוי9. הערכה של האינטראקציות miRNA-target בקשר עם פונקציות miRNA הוא חלק הכרחי כדי להבין טוב יותר כיצד Mirna לווסת תהליכים ביולוגיים שונים במדינות בריאות וחולה10. בנוסף, הגילוי של היעד בתום האדם (s) של miRNAs יכול עוד לספק אסטרטגיה מכוון עדין עבור טיפול מבוסס Mirnas עם תרופות נגד סרטן שונים. עם זאת, האתגר העיקרי בתחום של miRNAs הוא זיהוי של יעדים ישירים של miRNAs. כאן, שיטות מפורטות מוצגים כגישות ניסיוני לצורך הגדרה גנטית היעד miRNA. תכנון ניסיוני מוצלח עבור זיהוי יעד miRNA כרוך בשלבים שונים ושיקולים (איור 1). השוואה של רמות miRNA בוגרת בתאי הגידול תאים נורמליים יכול להיות אחד ההליכים השכיחים כדי לבחור miRNA עניין (איור 1A). המחקר הפונקציונלי של miRNA שנבחרו כדי לזהות את ההשפעות של miRNA על התפשטות התא חשוב לצמצם את רשימת המטרות המועמדים הפוטנציאליים הטובים ביותר של miRNA עניין (איור 1B). מבוסס על פונקציות מאומת הניסויים של miRNAs, סקירה שיטתית של ספרות ומסד נתונים בחברה עם תוכנית חיזוי יעד Mirnas נדרש כדי לחפש את המידע הרלוונטי ביותר על פונקציות גן (איור 1C). הזיהוי של גנים היעד האמיתי של mirna העניין יכול להיות מושגת על ידי יישום ניסויים כגון שיטת ללוציפראז יחד עם שיבוט של 3 ' utr של גן, PCR בזמן אמת, ו בלוק מערבי (איור 1d). המטרה של הפרוטוקול הנוכחי היא לספק שיטות מקיפות של ניסויים מרכזיים, את תגובת שרשרת פולימראז מבוסס בזמן אמת (PCR), sulforhodamine B (SRB) שיטה בעקבות מחקים miRNA מחקה, מינון תגובה הדור עקומת, ו שיטת לוציפראז יחד עם שיבוט של 3 UTR של גן. הפרוטוקול הנוכחי יכול להיות שימושי להבנה טובה יותר של הפונקציות של miRNAs בודדים ואת המשמעות של Mirnas בטיפול בסרטן.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. ניתוח ביטוי מיקרו-מיקרוארנה (מירנא)
-
בוגרת DNA משלימה (cDNA) סינתזה
- להוסיף 254 ng בסך הכל RNA ו 4.5 μl של deoxyribonuclease אני (dnase i) תערובות, ולאחר מכן להוסיף מים אלקטרופורזה ל-PCR רצועת צינורות לפצות עד 18 μl (איור 2a). הכן את התגובה עבור כל מדגם RNA הכולל מטוהרים ממספר קווי תאים באמצעות כמות מספקת של תערובות DNase I מבוסס על מספר כולל של תגובות.
הערה: תערובות dnase אני מורכב dnase i (1.8 μl), מעכב ריבונוקלאז (0.3 μl), ו -25 מ"מ mgcl2 (2.4 μl). כדי להבטיח להשיג RNA מוחלט, שיטת החילוץ המבוססת על עמודה הapplicated במקום להשתמש בשיטת החילוץ המבוססת על פנול-כלורופורם. דווח כי יבול החילוץ של מירנאס מסוימים יכול להיות מגוון בהתאם למספר התאים בעת שימוש בשיטת החילוץ המבוססת על פנול-כלורופורם11,12. - מודפה את הצינורות. בתוך ציקלייט תרמי הפעל את הצינורות במשך 10 דקות ב 37 ° c, וחום להפעיל DNase ב-5 דקות דגירה ב 90 ° c. מיד למקם את הצינורות על הקרח לאחר הדגירה.
- העבר 7.1 μL של DNase התייחס סה כ RNA לתוך 2 סטים של צינורות חדשים ולאחר מכן להוסיף 1.5 μL של התחל אנטי תחושה של גליצראלדהיד-3-פוספט דהידרוגנאז (הגנד) גן (איור 2B).
הערה: כמות ה-RNA הכולל לסינתזה cDNA הופך 100 ng בשלב זה. ריכוז המניה של התחל היסוד של "פרנד" הוא 10 μM. הוספת כדור היסוד של מאגר המידע הנוסף מיועד ליצירת מגוון של שיטות נוספות של שימוש בשיטה הייחודית לגנים. - מודב את הצינורות באמצעות ציקלונט תרמי. התחל ב 80 ° c עבור 5 דקות ואחריו התגובה ב 60 ° c עבור 5 דקות. מיד למקם את הצינורות על הקרח לאחר הדגירה.
- הוסף 3.4 μL של שעתוק הפוכה (RT) תערובות אנזים בכל תגובה (איור 2B). לתערובות אנזים מורכבים מ-100 מילימטר הדלחי triphosphates (0.15 μL), מאגר 10x RT (1.5 μL), מעכב ribonucחכירה (0.75 μL), והפוך האנזים תמלול (1 μL). הכינו כמות מספקת של תערובות בהתבסס על מספר התגובות הכולל.
- הוסף 3 μL של 5x RT התחל עבור miRNA ספציפית בכל תגובה (איור 2B).
הערה: הנפח הכולל הוא 15 μL עבור כל תגובה. - הפעל את הצינורות באמצעות הציקלאה תרמית. התחל ב -16 ° c עבור 30 דקות ואחריו התגובה ב 42 ° c עבור 30 דקות, ולבסוף ב 85 ° c עבור 5 דקות. המתן בארבע מעלות צלזיוס עבור כל זמן שנותר (איור 2B). CDNAs עם שירות יחיד מופקים בשלב זה הן עבור הגן הספציפי של miRNA והן עבור גן מיוחד של מירנד באותה צינורית.
- להוסיף 254 ng בסך הכל RNA ו 4.5 μl של deoxyribonuclease אני (dnase i) תערובות, ולאחר מכן להוסיף מים אלקטרופורזה ל-PCR רצועת צינורות לפצות עד 18 μl (איור 2a). הכן את התגובה עבור כל מדגם RNA הכולל מטוהרים ממספר קווי תאים באמצעות כמות מספקת של תערובות DNase I מבוסס על מספר כולל של תגובות.
-
תגובת שרשרת פולימראז (PCR) בזמן אמת וניתוח נתונים
- לדלל כל cDNA עם מים באולטרסאונד ביחס 1:49.
- הכינו את תערובות התגובה עבור miRNA מסוימת (שולחן 1). לאיתור של miRNA מסוימת ו-"הגנד", הגדר תגובות טרילקאט לכל דגימת cDNA.
- בצע את ה-PCR וניתוח הנתונים בזמן אמת (איור 2C). ניתוח נתונים באמצעות השיטה השוואתית CT 13,14.
2. מיקרוארנה לחיקוי
הערה: mirna-107 נבחרה משלב 1. מאז miRNA-107 הוא למטה מוסדר בתאי הגידול לעומת תאים נורמליים, זה יכול להיות העריך כי miRNA-107 הוא מדכא הגידול miRNA. במקרה של miRNA אשר מוסדר בתאי הגידול בהשוואה לתאים נורמליים (למשל,mirna-301), אנטי תחושה oligונומטר נגד mirna-301 ניתן להחיל על שלבים 2, 3, ו 4.
- לספור את התאים עם המכשיר קאמרית ספירה לוחית את התאים בצלחת 96-היטב. צפיפות התא היא 2 x 103 תאים/100 μl עבור כל באר. אין להשתמש במדיה של תרבות התא המכילה פניצילין-סטרפטומיצין (P/S) מכיוון ש-P/S יכול להפחית את היעילות של התרגום.
-
להכין מערכת תערובות החצייה כדי transfection את התאים בריכוזים הסופי מספר של שליטה miRNA לחקות ו miRNA-107 לחקות ביום למחרת (איור 3).
- מן המניה (25 μM ריכוז) של שליטה miRNA לחקות או miRNA-107 לחקות, לדלל ולהוסיף כמות מתאימה של שליטה לחקות או miRNA-107 לחקות במדיה מופחתת סרום יחד עם מגיב החצייה באמצעות צינורות מיקרוצנטריפוגה (איור 3A). מערבבים בעדינות את האוליגו המכיל תערובות בעזרת מיקרופיפטה. הסכום הכולל של oligos (miRNA לחקות שליטה + miRNA-107 לחקות) צריך להיות זהה בכל טוב. בארות ריקות כוללות 100 μL של מדיית תרבות התא ומדיה בסרום מופחת המכיל מעבר מגיב ללא תאים.
- לאחר הדגירה 10 דקות במכסה התרבות התא, בעדינות לערבב oligo המכילים תערובות שוב ולאחר מכן להוסיף 50 μL של התערובות לתוך כל טוב. שמור את התאים המוגברים. בחממה לתרבית תאים החלף את הגידול מגיב המכיל מדיה עם התקשורת החדשה תרבות התא המכיל שני סרום העוברי (FBS) ו-P/S לאחר הדגירה 6-12 h. עוד המדגירה את התאים עבור 72 h. משך הטיפול הכולל של miRNA מחקה הוא 96 h.
3. שיטת sulforhodamine B (SRB)
-
קיבוע תאים
- הסר את מדיית תרבות התא בכל באר של הצלחת ולמלא מיד 100 μL של 10% חומצה אצטית טריכלור (TCA) לתוך כל באר. מחלק בזהירות את המדיה של תרבות התא מכל באר כדי למנוע נזק לתאים וניתוק מלמטה.
הערה: הכינו 40% TCA על ידי הוספת 20 גרם של אבקת TCA לתוך 50 מ ל של מים מזוקקים. מ 40% TCA, לעשות 10% TCA על ידי דילול 40% TCA עם מים מזוקקים ביחס הדילול של 1:3. - שמור את הצלחת המכילה 10% TCA במקרר (4 ° c) עבור 1 h.
- לשטוף את הצלחת כמה פעמים על ידי מיזוג לתוך אמבט מים ולייבש אותו. מסירים את המים העודפים מתוך הבארות על ידי הקשה על הצלחת עד שלא נשאר מים בבארות. השאירו את הצלחת על ספסל במעבדה כדי לייבש אותו לפני הולך לשלב הבא.
- הסר את מדיית תרבות התא בכל באר של הצלחת ולמלא מיד 100 μL של 10% חומצה אצטית טריכלור (TCA) לתוך כל באר. מחלק בזהירות את המדיה של תרבות התא מכל באר כדי למנוע נזק לתאים וניתוק מלמטה.
-
צביעת תאים
- פיפטה 50 μL של 0.4% SRB פתרון לכל טוב כולל בארות ריקות. לנער בעדינות את הצלחת עד 0.4% SRB פתרון בעקביות מכסה את החלק התחתון של בארות.
הערה: להכין ולהשתמש 0.4% SRB פתרון על ידי הוספת 0.4 g של אבקת SRB לתוך 100 mL של 1% חומצה אצטית. טלטל את הפתרון בקפידה כדי לערבב אותו. לעטוף את הבקבוק של 0.4% SRB פתרון בחומר מגן בהיר כגון רדיד אלומיניום. אחסן 0.4% SRB פתרון במקרר. - לאחר הדגירה של 40 דקות עד 60 דקות, לשטוף את הצלחת על ידי שטיפה אותו עם 1% חומצה אצטית. שטוף את הצלחת עד שצבען לא מאוגד נשטף לחלוטין (איור 3B).
- השאירו את הצלחת על ספסל במעבדה כדי לייבש אותו לפני הולך לשלב הבא.
הערה: הצלחת צריך להיות יבש לחלוטין לפני הולך צעד 3.3.
- פיפטה 50 μL של 0.4% SRB פתרון לכל טוב כולל בארות ריקות. לנער בעדינות את הצלחת עד 0.4% SRB פתרון בעקביות מכסה את החלק התחתון של בארות.
-
מדידת ספיגת הרוויה
- פיפטה 100 μL של פתרון הבסיס של Tris (10 מ"מ) לתוך הבארות המתאימות כולל בארות ריקות. לשמור על הצלחת על שייקר במשך 10 דקות. למדוד את ספיגת ב 492 nm.
4. יצירת מינון-התגובה עקומת
- ניתוח הנתונים של שיטת SRB בגיליון אלקטרוני. הפחת את הספיגה הריקה מערכי הספיגה של כל קבוצה וחשב את הממוצע (AVE) ואת סטיית התקן (STD) של ערכי ספיגה של כל קבוצה.
- חישוב אחוז הספיגה הממוצעת (AVE%) והוא של סטיית התקן (STD%) של כל קבוצה באמצעות ערכי ספיגה של הערך SRB.
הערה: ה-AVE% של הפקד miRNA לחקות הקבוצה מטופלת היא 100%. חשב את ה-STD% באמצעות הנוסחה הבאה: STD% = (STD של כל קבוצה/ספיגת AVE של שליטה בקבוצה מטופלת) x 100. - יבא את הנתונים הגולמיים כולל ריכוזי טיפול, AVE% ו-STD% לתוך התוכנה על-ידי יישור אנכי של נתונים אלה. מכיוון שהיומן 0 אינו מוגדר, הגדר את הריכוז הראשון של ציר X לערך הקרוב ל-0 (לדוגמה, 0.01).
- לחצו על ' צור כרטיסיית גרף ' ובחרו בקווי שגיאת פיזור פשוטים. בחר עמודות גליון עבודה כערכי סמלים ולחץ על הבא. בחלונית ' תבנית נתונים ', בחרו ' זוגות XY ' ולחצו על ' הבא'. בחר את עמודות הנתונים המתאימות בלוח הנתונים הנבחר. לחץ על לחצן סיום כדי ליצור את העלילה.
הערה: ציר ה-X מייצג את הריכוזים, ציר Y מציין את אחוז הספיגה הממוצעת של כל אחד מההתרכזות (AVE%), ומייצגי השגיאות מצביעים על אחוז סטיית התקן של כל ריכוז (STD%). - לחצו פעמיים על ציר X כדי לשנות את סוג הסרגל ואת קנה המידה של הציר. שינוי סוג הקנה מידה ליניארי ליומן. שנה את מספר טווח ההתחלה והסיום ל-0.01 ו-200, בהתאמה.
-
לחצו לחיצה ימנית על כל מגרש פיזור, בחרו ' התאמה עקומה ' ועבור אל קטגוריית המשנה המוגדרת על-ידי המשתמש. בחר בעקומה של תגובת מינון, לחץ על הלחצנים הבא ולאחר מכן לחץ על לחצן סיום. עקומת המינון-תגובה נוצרת כעת יחד עם כרטיסיית דוח (איור 4A).
- כדי להזין משוואה 1 בתוכנה עבור הדור של עקומת תגובת מינון, לחץ על הכרטיסיה ניתוח ובחר אשף רגרסיה. עבור להגדרת המשתמש בקטגוריית המשוואה ולאחר מכן לחץ על הלחצן החדש. הוסף משוואה 1, משתנים, פרמטרים ראשוניים ואילוצים לתוך התיבות הריקות המתאימות (איור 4B, C). לחץ על הוסף כלחצן והגדר את שם המשוואה כעקומת תגובה למנה. שם המשוואה נוצר כעת בקטגוריית המשנה המוגדרת על -ידי המשתמש בקטגוריית המשוואה. f מציין את אחוז הכדאיות של התאים (% הכדאיות של התאים) במשוואה 1.
משוואה 1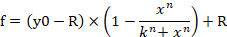
- עבור אל הכרטיסיה ' דוח ' ולאחר מכן בדוק את ערכי n, k ו-R.
הערה: y0 מציין 100% הכדאיות של שליטה mirna בקרת לחקות קבוצה, n מציין את מקדם סוג הגבעה (השיפוע של העלילה), k מציין את הריכוז של mirna-107 לחקות כי מייצרת 50% ההשפעה המקסימלית של mirna-107 (חצי ריכוז מעכבות מקסימלי, IC50) ו-R מציין את השבר השיורי (שבר ההתנגדות)15. המשוואה המשמשת להפקת עקומה ממנה-תגובה מזהה את הטווח מy0 לערך R (אם בכלל) כ-100% (איור 4A). לכן, יש לרכוש ערך k (IC50) מותאם המחושב על בסיס הטווח מy0 לערך אפס (איור 4a). מותאם k (IC50) יחד עם ערכי icx אחרים (למשל, IC10 עד ic90) ניתן להשיג באמצעות משוואה 2, אשר נגזר מהמשוואה 1. הנגזרת של משוואה 2 ממשוואה 1 מצוין באיור המשלים 1.
משוואה 2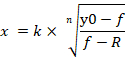
- לחץ פעמיים על לחצן העכבר השמאלי בתא שבו מוחל משוואה 2. באמצעות משוואה 2 ופרמטרים מתוך עקומת תגובה מינון שנוצר, זה זמין כדי לחשב את הערכים מנוכי ICx, החל IC10 כדי ic90 (איור 4d).
- הקלט את סימן השוויון שלאחריו הנוסחה מתחילה בסוגריים מזהים בתא. בעת הזנת הנוסחה, תקן את הערך של n, k ו-R כהפניות לתא מוחלטות על-ידי הוספת סימן הדולר לעמודה ולשורה המתאימות, כך שערכים קבועים אלה לא ישתנו בעת מילוי אוטומטי של הנוסחה לשורות (איור 4D). לחלופין, ניתן לחשב באופן ידני ערכים מותאמים באמצעות משוואה 2.
הערה: IC90 value אינו נקבע מכיוון שהערך R גדול מ-10. בנוסף, אם ערך R הוא מעל 20, הערך של IC80 הוא גם לא נקבע (איור 4d).
5. אימות של גן היעד הישיר של מיקרועם של האדם המעניין
הערה: לאחר ביצוע הניסוי פונקציונלי כגון שיטת SRB, miRNA-107 מאושר כמו מדכאים הגידול miRNA וזה אפשרי מאוד כי miRNA-107 מטרות ישירות אונאוגנים. בדוק את הרשימה של כל הגנים היעד החזוי באמצעות תוכנית חיזוי יעד mirna כגון TargetScan (http://www.targetscan.org/vert_71/), ולאחר מכן לצמצם את מטרות מועמד פוטנציאלי בהתבסס על הפונקציה של גן במסדי נתונים, כולל ה . כרטיסי הג
- פריימר עיצוב עבור שיבוט של 3 ' אזור שלא תורגם (UTR)
- שים את שמו של גן בכרטיסי הג (https://www.genecards.org/) ולחץ על סמל של גן. העריכו את דפדפן Ensembl הגנום על ידי לחיצה על ENSEMBL id של הגן ולאחר מכן לחץ על מזהה תמלילים בטבלה תעתיק. לאחר מכן, לחץ על Exons ברשימת הצגים המבוססים על תעתיק משמאל.
- העתק את רצפי נוקלאוטיד של 3 ' UTR ולהדביק אותו לתוך תוכנית עיצוב פריימר. העתק שוב את הרצפים מתוכנית זו והדבק אותו במעבד תמלילים. בדוק את הנוכחות של רצפי כריכת miRNA, כמו גם נוכחות של אתרי אנזימים הגבלה המשמשים לשכפול.
הערה: אם אין אתרי זיהוי אנזים הגבלה בתוך 3 ' UTR, ניתן להשתמש באנזימי ההגבלה שנבחרו עבור השכפול עבור השלב הבא. - בתוכנית עיצוב פריימר, לקבל את 3 ' רצף UTR ולהתחיל לעצב את התחל קדימה והפוך עם התנאי הבא. אורך: 20-30 נוקלאוטידים, Tm: 45-58 ° c, GC%: 40-60%. ההבדל בין הערכים Tm של שני התחל צריך להיות פחות מ 5 ° c. הרצפים הפריימר המשמשים במחקר זה מסופקים באיור המשלים 2. הוסף רצפי זיהוי אנזים הגבלה, כמו גם 4 נוקלאוטידים אקראיים לצבעי היסוד המיועדים.
- PCR מעבר צבע
- הכינו 25 μL של תערובות התגובה של ה-PCR, כולל העיצוב התחל לאחד (שולחן 2). הכינו כמות מספקת של תערובות בהתבסס על מספר התגובות הכולל. מערבבים את הפתרון על ידי ליטוף ולהוסיף 25 μL של תערובות התגובה לתוך כל צינור. צנטריפוגה את הצינורות. לכמה שניות
- בצע 35-40 מחזורי PCR משלב דנטורציה לשלב ההרחבה. הגדר את מחזור ה-PCR כשלבים הבאים: 98 ° צ' עבור 1 דקות (1 מחזור, הפעלה פולימרואז שלב), 95 ° צ' עבור 10 s (צעד הזמן), 45 ° c-68 ° צ' עבור 30 s (השלב המעיק), 68 ° צ' (שלב הארכה, 10 s-1 דקות לכל 1000 bp), , ולבסוף מצננים עד 4 ° c.
- הפעל את המוצרים PCR ולבדוק להקות על 1% ג'ל צמח עם סולמות DNA. מצא את טמפרטורת הריפוי הטובה ביותר (איור 5A). באמצעות טמפרטורת הריפוי. הטובה ביותר לשלב הבא
- עיכול כפול
- הפוך את תערובות התגובה כולל שתי אנזימי הגבלה, XhoI (או AsiSI) ו NotI, בצינור (שולחן 3). מודב את התערובות עבור 3-4 h באמצעות אמבט מים (37 ° c).
- הפעל את המוצרים מתעכל כפול על 1% צמח ג'ל ולאחר מכן חותכים את הלהקות תחת אור UV. במקרה של וקטורים לוציפראז, לפני הריצה על ג'ל, להגיב וקטורים מתעכל כפול עם 10 U של פוספאצטיות אלקליין עבור אחר 1 h כדי למנוע recircularization במהלך השלב השני.
- לטהר את מוצרי ה-PCR הכפול והוקטורים המאובלים מלהקות החפירה.
- הארכה של מוצרי PCR לתוך וקטורים לוציפראז
- הפוך 20 μl של תערובות תגובה כולל ה-DNA ליגאז (שולחן 4).
הערה: היחס הטוחנת של המוצר PCR (הכנס) כדי וקטור לוציפראז יכול להיות 3:1. 1:1 או 2:1. - בקצרה צנטריפוגה את הצינור עבור 10-15 s ו-הדגירה ב 16 ° צ' לילה באמצעות ציקלונט תרמית.
הערה: לחילופין, ניתן לשלב את הצינורית ב-4 ° c עבור 2-3 ימים לצורך הארכה. בשלב זה, הוספת ה-PCR ישוכפל לאזור הממוקם במורד הזרם של גן עיתונאי renilla (איור 5B). איגוד של miRNAs לתוך המוכפל 3 ' UTR של גן יכול להקטין את פעילות renilla. גחלילית לוציפראז היא לנורמליזציה של רמות ביטוי renilla.
- הפוך 20 μl של תערובות תגובה כולל ה-DNA ליגאז (שולחן 4).
- המרה והמושבה PCR
- הוסף את תערובות הריסוס (3-5 μL) לתוך הצינורית המכילה תאים מוכשרים. הקש בעדינות על השפופרת ושמור אותה על קרח (20 דקות).
הערה: הקפאת תאים מוכשרים בקרח לפני הוספת תערובות החיבור. - העבירו את הצינור במהירות ובעדינות לבלוק חום. בעקבות הלם חום (42 ° c עבור 30 s-1 דקות), מניחים את השפופרת על הקרח 20 דקות.
- הפיצו תאים מוכשרים על צלחת אגר לוריא-ברטני (LB). הגדל תאים מוכשרים באינקובטור (37 ° c) בלילה.
הערה: אמפיצילין (50-100 μg/mL) כלול בצלחת אגר. - בחר מושבה בודדת ולהשעות את ה-E. coli באחד הצינורות 8-הסטריפ המכילים מים באולטרטהורים. חזור על שלב זה כדי להשעות את ה-E. coli מבחירה אקראית 4-8 מושבות (איור 5c).
- העברה 25 μL של E. coli מושעה לתוך קבוצה אחרת של שפופרות 8-רצועה. עכשיו, יש 2 סטים של צינורות. של ההשעיה של אי. קולי
הערה: צינור אחד מיועד למושבה. ועוד אחד לחיסון ניתן לאחסן באופן זמני ב - 4 ° צ' (איור 5c). - לבצע את ה-PCR המושבה באמצעות E. coli השעיה. שלב זה הוא לקבוע אם המושבות מכילות הוספה. בחר את המושבות הטובות ביותר כדי לבודד ומבודדים לוציפראז וקטורים מחסה 3 ' UTR של גן (איור 5C).
הערה: חזור על שלב 5.1-5.5 עבור כל 3 ' UTR של הגנים הנבחרים. בצע את המצב של תגובת PCR המוצגת בטבלה 2 על ידי החלפת ה-DNA גנומית עם השעיית E. coli .
- הוסף את תערובות הריסוס (3-5 μL) לתוך הצינורית המכילה תאים מוכשרים. הקש בעדינות על השפופרת ושמור אותה על קרח (20 דקות).
- שיטת לוציפראז
- הכינו צלחת באורך 24 שעות. השתמש 1-2 x 104 תאים ב 500 μl מדיה תרבות התא עבור כל באר. אין להשתמש במדיה של תרבות התא המכילה את השימוש ב-P/S עבור התרגום מכיוון שהשימוש ב-P/S יכול להפחית את יעילות הגידול.
- Transfect 50 ng של וקטורים לוציפראז לתוך התאים עם שליטה לחקות או לחקות הספציפי miRNA באמצעות מגיב החצייה (איור 5D). אם לסנן את ההשפעות של miRNA מסוים לחקות ביותר מאשר ריכוז אחד, לשמור על הסכום הכולל של oligos זהה בכל טוב (ראה שלב 2).
- רוחצים את החלק הפנימי של הבארות פעמיים בעזרת תמיסת מלח (PBS) ביום שלמחרת.
- החל 200 μL של הליזה מגיב לבארות מספיק לבצע הפירוק התא לפני מדידת פעילות לוציפראז.
הערה: לשמור על צלחת לרעוד לפחות 15 דקות. - העבר 5-10 μL של תא לאחר לתוך הצינור החדש ולהוסיף 100 μL של מגיב I. מיד לערבב את הפתרון על ידי ליטוף ולקרוא את הפעילות לוציה גחלילית באמצעות לומימטר.
הערה: לקרוא את הפעילות הלוצ גחלילית עבור 10-15 s. - הוסף 100 μL של מגיב II באותה צינורית, ולאחר מכן לערבב על ידי ליטוף פעמיים. לקרוא את הפעולה renilla לוציפראז עבור 10-15 s באמצעות לומימטר. חזור על שלב 5.6.5 ו5.6.6 עבור כל דוגמה.
- לחשב את היחס של renilla כדי גחלילית (איור 5E).
הערה: הפעילות של גחלילית מייצגת את היעילות של הגלגול של ללוציפראז לתוך התאים.
Access restricted. Please log in or start a trial to view this content.
תוצאות
אישור מוצלח ומדויק של רמות miRNA חשוב לפרשנות של הנתונים שבהם הסיווג של Mirna אפשרי מבוסס על התפקידים הצפויים של Mirna בפיתוח והתקדמות של מחלה. רמות של miRNA-107 ו miRNA-301 נמדדו שלושה קווי התא הלבלב באמצעות ה-PCR כמותי המבוסס על בדיקה. סינתזה של cDNAs הן של miRNA ספציפית ו גן התייחסות באותה תגובה יכולה להגביר את...
Access restricted. Please log in or start a trial to view this content.
Discussion
אסטרטגיות לקביעת מטרות miRNA בתום ובלבד עם פונקציות של מירנה של עניין הם חיוניים להבנת תפקידים מרובים של Mirna. זיהוי של גנים היעד miRNA יכול להיות מנחה לפענוח התא איתות אירועים מאופנן על ידי Mirna בתא. החשיפה של גנים מטרה חשובה מבחינה פונקציונלית של miRNAs יכול לספק את הידע הבסיסי לפתח טיפול מבוססי Mirna...
Access restricted. Please log in or start a trial to view this content.
Disclosures
. למחברים אין מה לגלות
Acknowledgements
מחקר זה נתמך על ידי תוכנית בסיסית מחקר המדע באמצעות קרן המחקר הלאומי של קוריאה (NRF) ממומן על ידי משרד החינוך (2017R1D1A3B03035662); הקרן לחקר האוניברסיטה, 2017 (HRF-201703-003).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
References
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved