Method Article
فك رموز منظمة كروماتين 3D عالية الدقة عبر التقاط Hi-C
In This Article
Summary
يصف هذا البروتوكول طريقة Capture Hi-C المستخدمة لتوصيف تنظيم 3D للمناطق الجينومية المستهدفة ذات الحجم الضخم بدقة عالية ، بما في ذلك حدود المجالات المرتبطة طوبولوجيا (TADs) وتفاعلات الكروماتين طويلة المدى بين عناصر تسلسل الحمض النووي التنظيمية وغيرها.
Abstract
يساهم التنظيم المكاني للجينوم في وظيفته وتنظيمه في العديد من السياقات ، بما في ذلك النسخ والتكرار وإعادة التركيب والإصلاح. لذلك فإن فهم السببية الدقيقة بين طوبولوجيا الجينوم ووظيفته أمر بالغ الأهمية وموضوع بحث مكثف بشكل متزايد. تسمح تقنيات التقاط تشكل الكروموسوم (3C) باستنتاج البنية ثلاثية الأبعاد للكروماتين عن طريق قياس تواتر التفاعلات بين أي منطقة من الجينوم. نصف هنا بروتوكولا سريعا وبسيطا لأداء Capture Hi-C ، وهي طريقة تخصيب مستهدفة قائمة على 3C تميز التنظيم ثلاثي الأبعاد الخاص بالأليل للأهداف الجينومية الضخمة الحجم بدقة عالية. في Capture Hi-C ، يتم التقاط المناطق المستهدفة بواسطة مجموعة من مجسات البيوتينيل قبل تسلسل عالي الإنتاجية في المصب. وبالتالي ، يتم تحقيق دقة أعلى وخصوصية الأليل مع تحسين فعالية الوقت والقدرة على تحمل تكاليف التكنولوجيا. لإثبات نقاط قوته ، تم تطبيق بروتوكول Capture Hi-C على مركز تعطيل X للماوس (Xic) ، وهو الموضع التنظيمي الرئيسي لتعطيل كروموسوم X (XCI).
Introduction
يحتوي الجينوم الخطي على جميع المعلومات اللازمة للكائن الحي للخضوع للتطور الجنيني والبقاء على قيد الحياة طوال مرحلة البلوغ. ومع ذلك ، فإن توجيه الخلايا المتطابقة وراثيا لأداء وظائف مختلفة أمر أساسي للتحكم بدقة في المعلومات المستخدمة في سياقات محددة ، بما في ذلك الأنسجة المختلفة و / أو مراحل النمو. يعتقد أن التنظيم ثلاثي الأبعاد للجينوم يشارك في هذا التنظيم المكاني الزماني الدقيق لنشاط الجينات من خلال تسهيل أو منع التفاعل المادي بين العناصر التنظيمية التي يمكن فصلها بعدة مئات من الكيلوباسات في الجينوم الخطي (للمراجعات1،2،3). في السنوات ال 20 الماضية ، زاد فهمنا للتفاعل بين طي الجينوم والنشاط بسرعة ، ويرجع ذلك إلى حد كبير إلى تطوير تقنيات التقاط تشكل الكروموسوم (3C) (للمراجعة4،5،6،7). تقيس هذه الطرق تواتر التفاعلات بين أي مناطق من الجينوم وتعتمد على ربط تسلسل الحمض النووي الذي يقع على مقربة من 3D داخل النواة. تبدأ بروتوكولات 3C الأكثر شيوعا بتثبيت مجموعات الخلايا بعامل ربط متقاطع مثل الفورمالديهايد. ثم يتم هضم الكروماتين المتصالب بإنزيم تقييد ، على الرغم من استخدام هضم MNase أيضا 8,9. بعد الهضم ، يتم إعادة ربط نهايات الحمض النووي الحرة على مقربة مكانية ، ويتم عكس الارتباط المتقاطع. تؤدي هذه الخطوة إلى ظهور "مكتبة" أو "قالب" 3C ، وهي مجموعة مختلطة من الأجزاء الهجينة التي يكون فيها للتسلسلات التي كانت قريبة من النواة فرص أكبر للارتباط في نفس جزء الحمض النووي. يتيح القياس الكمي لهذه الأجزاء الهجينة استنتاج التشكل ثلاثي الأبعاد للمناطق الجينومية التي تقع على بعد آلاف أزواج القواعد في الجينوم الخطي ولكنها قد تتفاعل في الفضاء ثلاثي الأبعاد.
تم تطوير العديد من الأساليب المختلفة لتوصيف مكتبة 3C ، والتي تختلف من حيث المجموعات الفرعية من شظايا الربط التي يتم تحليلها والتكنولوجيا المستخدمة في قياسها الكمي. اعتمد بروتوكول 3C الأصلي على اختيار منطقتين من الاهتمام والقياس الكمي لتردد التفاعل "واحد مقابل واحد" بواسطة PCR10,11. يقيس نهج 4C (التقاط التشكل الدائري للكروموسومات) التفاعلات بين موضع اهتمام واحد (أي "وجهة نظر") وبقية الجينوم ("واحد مقابل الكل")12،13،14. في 4C ، تخضع مكتبة 3C لجولة ثانية من الهضم وإعادة الربط لتوليد جزيئات الحمض النووي الدائرية الصغيرة التي يتم تضخيمها بواسطة مشغلات أولية محددة لنقطةالعرض 15. يتيح 5C (نسخة الكربون لالتقاط الكروموسومات) توصيف التفاعلات ثلاثية الأبعاد عبر مناطق الاهتمام الأكبر ، مما يوفر رؤى حول طي الكروماتين عالي الترتيب داخل تلك المنطقة ("كثير مقابل كثير")16. في 5C ، يتم تهجين مكتبة 3C إلى مجموعة من oligonucleotides مواقع تقييد متداخلة يمكن تضخيمها لاحقا بواسطة تفاعل البوليميراز المتسلسل المتعدد مع الاشعال العالمية15. في كل من 4C و 5C ، تم تحديد شظايا الحمض النووي المفيدة في البداية بواسطة المصفوفات الدقيقة وبعد ذلك بواسطة تسلسل الجيل التالي (NGS) 17،18،19. تميز هذه الاستراتيجيات مناطق الاهتمام المستهدفة ولكن لا يمكن تطبيقها لرسم خريطة التفاعلات على مستوى الجينوم. يتم تحقيق هذا الهدف الأخير باستخدام Hi-C ، وهي استراتيجية عالية الإنتاجية قائمة على 3C يسمح فيها التسلسل المتوازي بشكل كبير لقالب 3C بالتوصيف غير المتحيز لطي الكروماتين على مستوى الجينوم ("الكل مقابل الكل") 20. يتضمن بروتوكول Hi-C دمج بقايا البيوتينيل في نهايات الشظايا المهضومة ، والتي يتبعها سحب شظايا الربط مع حبات الستربتافيدين لزيادة استعادة الشظايا المربوطة20.
كشف Hi-C أن جينومات الثدييات منظمة هيكليا على مستويات متعددة في نواة 3D. على نطاق megabase ، ينقسم الجينوم إلى مناطق من الكروماتين النشط وغير النشط ، المقصورات A و B ، على التوالي20,21. كما تبين لاحقا وجود المزيد من الأجزاء الفرعية التي تمثلها حالات مختلفة من الكروماتين والنشاط22. في دقة أعلى ، يتم تقسيم الجينوم إلى مجالات ذاتية التفاعل شبه ضخمة تسمى مجالات الارتباط الطوبولوجي (TADs) ، والتي تم الكشف عنها لأول مرة من خلال تحليل Hi-C و 5C للجينومات البشرية والفأر23,24. على عكس المقصورات التي تختلف بطريقة خاصة بالأنسجة ، تميل TADs إلى أن تكون ثابتة (على الرغم من وجود العديد من الاستثناءات). الأهم من ذلك ، يتم الحفاظ على حدود TAD عبر الأنواع25. في خلايا الثدييات ، غالبا ما تشمل TADs الجينات التي تشترك في نفس المشهد التنظيمي وقد ثبت أنها تمثل إطارا هيكليا يسهل التنظيم المشترك للجينات مع الحد من التفاعلات مع المجالات التنظيمية المجاورة (للمراجعة3،26،27،28). علاوة على ذلك ، داخل TADs ، قد تزيد التفاعلات الناتجة عن مواقع CTCF في قاعدة الحلقات المبثوقة من التماسك من احتمال تفاعلات المروج والمحسن أو المحسن (للمراجعة29).
في Hi-C ، يمكن اكتشاف المقصورات و TADs بدقة 1 ميجا بايت إلى 40 كيلو بايت ، ولكن يمكن تحقيق دقة أعلى لتوصيف جهات الاتصال الأصغر حجما مثل تفاعلات الحلقات بين العناصر البعيدة بمقياس 5-10 كيلو بايت. ومع ذلك ، فإن زيادة الدقة لتكون قادرة على اكتشاف مثل هذه الحلقات بكفاءة بواسطة HiC تتطلب زيادة كبيرة في عمق التسلسل ، وبالتالي تكاليف التسلسل. يتفاقم هذا إذا كان التحليل يحتاج إلى أن يكون خاصا بالأليل. في الواقع ، تتطلب زيادة الدقة بمقدار X أضعاف زيادة X2 في عمق التسلسل ، مما يعني أن الأساليب عالية الدقة والخاصة بالجينوم على مستوى الأليل يمكن أن تكون باهظةالتكلفة 30.
لتحسين فعالية التكلفة والقدرة على تحمل التكاليف مع الحفاظ على دقة عالية ، يمكن سحب المناطق المستهدفة ذات الأهمية فعليا من مكتبات 3C أو Hi-C على مستوى الجينوم بعد تهجينها مع مجسات oligonucleotide التكميلية التي تحمل علامة البيوتين قبل تسلسل المصب. يشار إلى استراتيجيات التخصيب المستهدفة هذه باسم طرق Capture-C وتسمح باستجواب تفاعلات مئات المواقع المستهدفة المنتشرة عبر الجينوم (أي التقاط المروج (PC) Hi-C ؛ الجيل التالي (NG) Capture-C ؛ الإدخال المنخفض (LI) Capture-C ؛ المعايرة النووية (NuTi) Capture-C ؛ Tri-C)31,32,33,34,35,36,37,38,39,40، أو عبر المناطق التي تمتد حتى عدة قواعد ضخمة (أي التقاط HiC؛ HYbrid التقاط Hi-C (Hi-C2) ؛ البلاط-C)41،42،43. يمكن أن يختلف جانبان في الأساليب القائمة على الالتقاط: (1) طبيعة وتصميم قليل النيوكليوتيدات البيوتينيلاتي (أي الحمض النووي الريبي أو الحمض النووي ، أو أوليغوس مفرد يلتقط أهدافا جينومية مشتتة أو أوليغو متعددة تبليط منطقة ذات أهمية) ؛ (2) طبيعة وتصميم أوليغونيوكليوتيدات البيوتينيل (أي الحمض النووي الريبي أو الحمض النووي ، أو أوليغوس مفرد يلتقط أهدافا جينومية مشتتة أو أوليغو متعددة تبليط منطقة ذات أهمية) ؛ (2) طبيعة وتصميم أوليغوكليوتيدات البيوتينيل (أي الحمض النووي الريبي أو الحمض النووي ، أو أوليغوس مفرد يلتقط أهدافا جينومية مشتتة أو أوليغو متعددة تبليط منطقة ذات أهمية) ؛ (2) طبيعة وتصميم أوليغوكليوتيدات البيوتينيل ( و (2) القالب المستخدم لسحب الأهداف التي يمكن أن تكون مكتبة 3C أو Hi-C ، وتتكون الأخيرة من شظايا تقييد البيوتينيلات المسحوبة لأسفل من مكتبة 3C.
هنا ، يتم وصف بروتوكول Capture Hi-C استنادا إلى إثراء جهات الاتصال المستهدفة من مكتبة 3C. يعتمد البروتوكول على تصميم مجموعة تبليط مصممة خصيصا من مجسات الحمض النووي الريبي البيوتينيلاتي ويمكن إجراؤها في أسبوع 1 من إعداد مكتبة 3C إلى تسلسل NGS. البروتوكول سريع وبسيط ويسمح بتوصيف التنظيم ثلاثي الأبعاد الأعلى مرتبة للمناطق ذات الحجم الضخم ذات الأهمية بدقة 5 كيلوبايت مع تحسين فعالية الوقت والقدرة على تحمل التكاليف مقارنة بطرق 3C الأخرى. تم تطبيق بروتوكول Capture Hi-C على الموضع التنظيمي الرئيسي لتعطيل الكروموسوم X (XCI) ، ومركز تعطيل X (Xic) ، الذي يستضيف الحمض النووي الريبي غير المشفر Xist. كان Xic سابقا موضوع تحليلات هيكلية ووظيفية واسعة النطاق (للمراجعة44،45). في الثدييات ، يعوض XCI عن جرعة الجينات المرتبطة بالكروموسوم X بين الإناث (XX) والذكور (XY) ويتضمن إسكات النسخ لكامل أحد كروموسومي X تقريبا في الخلايا الأنثوية. يمثل Xic موقعا قياسيا ذهبيا قويا للدراسات في طوبولوجيا الجينوم 3D والتفاعل مع تنظيم الجينات44. أدى تحليل 5C ل Xic في الخلايا الجذعية الجنينية للفأر (mESCs) إلى اكتشاف وتسمية TADs ، مما يوفر أول رؤى حول الأهمية الوظيفية للتقسيم الطوبولوجي والتنظيم المشترك للجينات24. تبين لاحقا أن التنظيم الطوبولوجي ل Xic متورط بشكل حاسم في التوقيت التنموي المناسب لتنظيم Xist و XCI 46 ، كما تم اكتشاف عناصر تنظيمية غير متوقعة يمكن أن تؤثر على نشاط الجينات داخل وبين TADs مؤخرا داخل Xic47،48،49. يوضح تطبيق Capture Hi-C على 3 ميجا بايت من كروموسوم X للفأر الذي يمتد عبر Xic قوة هذا النهج في تشريح طي الكروماتين على نطاق واسع بدقة عالية. يتم توفير بروتوكول مفصل وسهل المتابعة ، بدءا من تصميم مجموعة مجسات البيوتينيلات عبر كل موقع تقييد DpnII داخل المنطقة ذات الاهتمام إلى إنشاء مكتبة 3C على مستوى الجينوم ، والتهجين والتقاط جهات الاتصال المستهدفة ، وتحليل البيانات النهائية. كما يتضمن لمحة عامة عن ضوابط الجودة المناسبة والنتائج المتوقعة، وتناقش نقاط القوة والقصور في النهج في ضوء الأساليب الحالية المماثلة.
Protocol
تم اشتقاق الخلايا الجذعية الجنينية للفأر (mESCs) المستخدمة في هذه الدراسة من صليب أنثى TX / TX R26 rtTA/ rtTA 50 مع ذكر Mus musculus castaneus وفقا لإرشادات رعاية الحيوان لمعهد كوري (باريس) 51.
1. تصميم التحقيق
- تصميم مجموعة من مجسات البيوتينيل (120-mer RNA oligonucleotides) تغطي المنطقة المستهدفة محل الاهتمام.
- قم بتجانب المنطقة ذات الاهتمام باستخدام قليل النيوكليوتيدات المتداخلة بحيث يتم تغطية كل تسلسل داخل الهدف في المتوسط بمجسين فريدين (تغطية 2x) (الشكل 1).
- استبعاد التسلسلات المتكررة من تغطية المسبار لتجنب إثراء التفاعلات غير المحددة.
ملاحظة: لتحقيق أقصى قدر من إثراء شظايا الربط المعلوماتية ، تم تحديد مناطق تمتد على 300 نقطة أساس في المنبع والمصب لكل موقع تقييد DpnII عبر الهدف (ChrX: 102,475,000-105,475,000) ، وتم تصميم 28,913 مجسا بيوتينيل وفقا لتقنية إثراء هدف SureSelect DNA من خلال منصة Sure Design52. وفقا لهذه الاستراتيجية ، يسمح بحد أقصى 40 قاعدة من التسلسلات المتكررة في كل قليل النوكليوتيد لتقليل إثراء التفاعلات غير المحددة. تم تصنيع مجموعة المسبار بواسطة Agilent. هنا ، يتم استخدام DpnII كإنزيم تقييد لسببين: (1) إنه قاطع رباعي يستخدم بشكل روتيني في العديد من الطرق القائمة على 3C53 ؛ و (2) يزيد من فرص التقاط تعدد أشكال النوكليوتيدات المفردة (SNPs) بالقرب من مواقع القطع مقارنة بإنزيمات التقييد الأخرى التي تم اختبارها في السيليكو في الخطوط الهجينة F1 المستخدمة في هذه الدراسة (C57BL / 6J x CASTEi / J).
2. الإجراء التجريبي
- إعداد الخلية
- قم بزرع العدد المناسب من الخلايا على لوحة واحدة أو عدة لوحات لزراعة الخلايا لتحقيق إجمالي عدد الخلايا ≥ 5 × 107 خلايا في يوم التثبيت.
ملاحظة: تم استخدام الخلايا الجذعية الجنينية للفأر (mESCs) في هذه الدراسة. يتم طلاء mESCs على ألواح زراعة الخلايا الجيلاتينية (0.1٪ جيلاتين في 1x PBS - o / n عند 37 درجة مئوية ، حاضنة CO2 بنسبة 5٪) في وسط mESCs يحتوي على 2i + LIF ومصل عجل الجنين الذي تم اختباره على دفعات (DMEM ، 15٪ FBS ، 0.1 mM β-mercaptoethanol ، 1000 U / mL −1 عامل تثبيط سرطان الدم (LIF) ، CHIR99021 (3 ميكرومتر) ، و PD0325901 (1 ميكرومتر)). بالنسبة لهذا النوع من الخلايا ، تحتوي لوحة واحدة متقاربة بنسبة 80٪ مقاس 10 سم على ما يقرب من 2 × 107 خلايا. - قم بإعداد لوحة زراعة خلية إضافية واحدة لعد الخلايا.
ملاحظة: يمكن استخدام لوحة زراعة خلايا أصغر لتقليل استخدام الوسائط. في هذه الحالة ، يجب تعديل عدد الخلايا المراد زرعها على اللوحة الأصغر وفقا لذلك (على سبيل المثال ، خلايا أقل بمقدار 3 مرات على لوحة 10 سم مقارنة بلوحة 15 سم).
- قم بزرع العدد المناسب من الخلايا على لوحة واحدة أو عدة لوحات لزراعة الخلايا لتحقيق إجمالي عدد الخلايا ≥ 5 × 107 خلايا في يوم التثبيت.
- تثبيت الفورمالديهايد
- تقدير العدد الإجمالي للخلايا المراد ربطها بشكل متبادل.
- قبل البدء في تفاعل الربط المتقاطع ، قم بالتريبسين وعد الخلايا من لوحة التحكم المعدة خصيصا لعد الخلايا باستخدام عداد خلايا آلي وفقا لتعليمات الشركة المصنعة.
- قم بتضمين تلطيخ الجدوى (على سبيل المثال ، Trypan Blue) لتحديد النسبة المئوية للخلايا القابلة للحياة54. من عدد الخلايا هذا ، قم بتقدير العدد الإجمالي للخلايا في اللوحة (اللوحات) المعدة للربط المتقاطع.
- قم بإزالة وسط الاستزراع من الألواح المعدة للربط المتشابك واستبدله بالكمية المناسبة من محلول التثبيت (2٪ فورمالديهايد في وسط زراعة الخلايا). استخدم 10 مل على طبق 10 سم (على سبيل المثال ، ~ 20 مل للوحة 15 سم).
ملاحظة: أضف حجما دقيقا لمحلول التثبيت. إذا كان إصلاح الخلايا الملتصقة غير ممكن ، فيمكن تكييف هذه الخطوة مع الخلايا التربسينية وتنفيذها في 30 مل من محلول التثبيت في أنابيب طرد مركزي مخروطية سعة 50 مل. يجب ألا يزيد عمر الفورمالديهايد عن 1 سنة. يفضل استخدام قوارير تستخدم مرة واحدة. يجب إحضار محلول التثبيت إلى درجة حرارة الغرفة (RT) قبل الاستخدام.
تنبيه: الفورمالديهايد خطير ويجب التعامل معه وفقا للوائح الصحة والسلامة المناسبة. - ثبت لمدة 10 دقائق في RT تحت خلط لطيف على شاكر.
- إخماد تفاعل التثبيت بإضافة 2.5 M جليكاين-1x PBS إلى تركيز نهائي قدره 0.125 M. أضف 530 ميكرولتر من 2.5 M جليكاين-1x PBS إلى 10 مل على لوحة 10 سم (على سبيل المثال ، 1060 ميكرولتر إلى 20 مل على لوحة 15 سم).
ملاحظة: إذا تم تثبيت الخلايا في محلول ، فقم بإخماد تفاعل التثبيت ب 1590 ميكرولتر من 2.5 M جليكاين -1x PBS. - احتضان لمدة 5 دقائق في RT ، واخلطه برفق على شاكر.
- انقل الأطباق إلى الثلج واحتضنها لمدة 15 دقيقة إضافية على الثلج مع الخلط برفق على شاكر.
ملاحظة: من الآن فصاعدا ، يجب أن تبقى الخلايا على الجليد ، ويجب تبريد المخازن المؤقتة مسبقا لتجنب المزيد من الارتباط المتقاطع. انتقل إلى غرفة باردة إذا كانت هناك حاجة إلى معالجة العديد من اللوحات. - قم بإزالة محلول التثبيت من الخلايا عن طريق سكبه في دورق لضمان التعامل السريع.
ملاحظة: تأكد من التخلص من النفايات السائلة المحتوية على الفورمالديهايد وفقا للوائح الصحة والسلامة المناسبة. - اشطف الطبق مقاس 10 سم بسرعة مرتين باستخدام 5 مل من البرد 0.125 M glycine-1x PBS (8 مل للوحة 15 سم) لغسل الحطام والخلايا الميتة. قم بإزالة السائل من اللوحة عن طريق سكبه في دورق لضمان التعامل السريع.
- أضف 5 مل من البرد 0.125 م جليكاين -1x PBS إلى لوحة 10 سم (10 مل للوحة 15 سم) وكشط الخلايا بسرعة من اللوحة باستخدام مكشطة الخلايا البلاستيكية.
- انقل تعليق الخلية إلى أنبوب طرد مركزي مخروطي سعة 50 مل مبرد مسبقا باستخدام ماصة مصلية.
- شطف لوحة مرتين مع 5 مل من البرد 0.125 M الجلايسين -1x PBS وإضافة تعليق الخلية إلى أنبوب الطرد المركزي المخروطي.
- قم بتدويره لأسفل عند 480 × جم لمدة 10 دقائق عند 4 درجات مئوية.
ملاحظة: إذا كانت الخلايا مثبتة في محلول ، انقل الخلية إلى أنبوب طرد مركزي مخروطي مبرد مسبقا وقم بتدويرها لأسفل عند 480 × جم لمدة 10 دقائق عند 4 درجات مئوية. قم بإزالة محلول التثبيت عن طريق سكبه في دورق واغسله ثلاث مرات في 10 مل من البرد 0.125 M الجلايسين -1x PBS. تأكد من إعادة تعليق الخلايا في كل خطوة من خطوات الغسيل. - قم بإزالة المادة الطافية عن طريق الشفط باستخدام نظام شفط منضدة. أعد تعليق الخلايا في 500 ميكرولتر من 1x PBS لكل 1 × 107 خلايا عن طريق السحب بعناية لأعلى ولأسفل باستخدام ماصة P1000. لإعادة تعليق الخلايا بالحجم الدقيق ، راجع تقدير إجمالي عدد الخلايا الذي تم الحصول عليه في 2.2.1.
- القسمة 500 ميكرولتر من تعليق الخلية في العدد المحسوب لأنابيب الطرد المركزي الدقيقة 1.5 مل (1 × 107 خلايا / أنبوب).
- قم بتدويره لأسفل عند 480 × جم لمدة 10 دقائق عند 4 درجات مئوية.
- قم بإزالة المادة الطافية باستخدام نظام شفط منضدة وقم بتجميد كريات الخلية في النيتروجين السائل. قم بتخزين كريات الخلايا الجافة في درجة حرارة -80 درجة مئوية.
ملاحظة: يمكن تخزين العينات لمدة 1 سنة على الأقل.
- تقدير العدد الإجمالي للخلايا المراد ربطها بشكل متبادل.
- تحلل الخلية
- قم بإذابة الحبيبات المجمدة على الجليد.
- تحضير 1.5 مل من المخزن المؤقت للتحلل في H 2 0 لكل عينة: أضف 10 mM Tris-HCl ، ودرجة الحموضة 8.0 ، و 10 mM NaCl ، و0.2٪ NP40.
- أضف 600 ميكرولتر من محلول التحلل البارد وأعد تعليقه جيدا على الثلج.
- احتضان على الجليد لمدة 15 دقيقة للسماح للخلايا تنتفخ.
- قم بتدويرها عند 2655 × جم لمدة 5 دقائق عند 4 درجات مئوية وقم بإزالة المادة الطافية باستخدام نظام شفط فوق الطاولة.
- لإزالة الحطام ، أعد تعليق الحبيبات في 1 مل من المخزن المؤقت للتحلل البارد ، وقم بتدويرها لأسفل عند 2655 × جم لمدة 5 دقائق عند 4 درجات مئوية ، وقم بإزالة المادة الطافية.
- قم بالدوران مرة أخرى لفترة وجيزة عند 2655 × جم و 4 درجات مئوية وقم بإزالة أكبر قدر ممكن من المادة الطافية المتبقية باستخدام نظام شفط منضدة مزود بطرف P200.
- إعادة التعليق في 100 ميكرولتر من 0.5٪ (حجم / حجم) SDS.
- احتضان في خلاط حراري عند 62 درجة مئوية ، والدوران عند 1400 دورة في الدقيقة لمدة 10 دقائق.
- أضف 290 ميكرولتر من H2O + 50 ميكرولتر من 10٪ TritonX-100 واخلطها جيدا ، وتجنب فقاعات الهواء.
- احتضان في خلاط حراري عند 37 درجة مئوية ، والدوران عند 1400 دورة في الدقيقة لمدة 15 دقيقة.
- أضف 50 ميكرولتر من المخزن المؤقت 10x Dpnll واقلب الأنبوب للخلط.
- خذ 50 ميكرولتر من الحمض النووي غير المهضوم لمراقبة الجودة في أنبوب منفصل. لا تنس أن تأخذ عينة التحكم غير المهضومة.
- هضم DpnII
- أضف 10 ميكرولتر من تركيز Dpnll العالي (إجمالي 500 U) واخلطه عن طريق التقليب.
- احتضان العينات والتحكم غير المهضوم في خلاط حراري عند 37 درجة مئوية ، والدوران عند 1400 دورة في الدقيقة لمدة >4 ساعة.
- أضف 10 ميكرولتر من تركيز Dpnll العالي (إجمالي 500 U) في نهاية اليوم.
- احتضان العينات والتحكم غير المهضوم عند 37 درجة مئوية ، والدوران عند 1400 دورة في الدقيقة طوال الليل.
- أضف 10 ميكرولتر من تركيز Dpnll العالي (إجمالي 500 U) في بداية اليوم التالي إلى العينات.
- احتضان العينات والتحكم غير المهضوم في خلاط حراري عند 37 درجة مئوية ، والدوران عند 1400 دورة في الدقيقة لمدة 4 ساعات.
- ربط وعكس الربط المتقاطع
- احتضان الأنابيب عند 65 درجة مئوية لمدة 20 دقيقة عند 1400 دورة في الدقيقة.
ملاحظة: لا تقم بإضافة SDS في هذه المرحلة. الفكرة هي الحفاظ على السلامة النووية ، لذلك يتم الربط داخل النوى ، والتحايل على الحاجة إلى التخفيف الشديد. - قم بتبريد العينات على الثلج لمدة أقصاها 5-10 دقائق. لتجنب هطول الأمطار SDS ، لا تترك العينات على الجليد لفترة أطول من هذا.
- خذ 50 ميكرولتر من الحمض النووي المهضوم غير المرتبط لمراقبة الجودة في أنبوب منفصل. قم بتخزين أدوات التحكم غير المهضومة وغير المربوطة عند -20 درجة مئوية.
ملاحظة: لا تنس أن تأخذ عينة التحكم غير المربوطة. - أضف 800 ميكرولتر من كوكتيل الربط: 122 ميكرولتر من 10x ligase buffer ، 8 μL من T4 ligase (30 U / μL) ، و 670 μL من H20.
- احتضان عند 16 درجة مئوية ، والدوران عند 1000 دورة في الدقيقة بين عشية وضحاها.
- أضف 7.5 ميكرولتر من Proteinase K (20 مجم / مل) إلى العينات و 2 ميكرولتر إلى الضوابط.
- احتضان عند 65 درجة مئوية لمدة 4 ساعات عند 1000 دورة في الدقيقة.
- احتضان الأنابيب عند 65 درجة مئوية لمدة 20 دقيقة عند 1400 دورة في الدقيقة.
- تنقية الحمض النووي
- انقل العينات الموجودة على الثلج إلى أنابيب طرد مركزي مخروطية سعة 15 مل مبردة مسبقا وأضف 2 مل من الماء و 10.5 مل من EtOH المثلج البارد و 583 ميكرولتر من 3 M NaAC.
ملاحظة: تهدف المياه الإضافية إلى منع ترحيل DTT إلى الحبيبات. - أضف 200 ميكرولتر من EtOH البارد المثلج ، و 10.8 ميكرولتر من NaAC ، و 1 ميكرولتر من coprecipitant إلى ضوابط الجودة غير المهضومة وغير المربوطة.
- احتضان في -80 درجة مئوية لمدة 4 ساعات على الأقل حتى بين عشية وضحاها.
- أدر أنابيب 15 مل عند 2200 × جم عند 4 درجات مئوية لمدة 45 دقيقة.
- أدر أنابيب التحكم سعة 1.5 مل عند 20500 × جم عند 4 درجات مئوية لمدة 30 دقيقة.
- اغسل مرة واحدة باستخدام 3 مل (عينات) و 1 مل (ضوابط) من الثلج البارد 70٪ EtOH.
- تدور عند 2200 × جم ( عينات) أو 20500 × جم (ضوابط) عند 4 درجات مئوية لمدة 10 دقائق.
- قم بإزالة EtOH بعناية وجففه في الهواء في RT لمدة 10-15 دقيقة ؛ لا تبالغ.
- أعد تعليق العينات والضوابط في 100 ميكرولتر و 20 ميكرولتر من H20 ، على التوالي.
- أضف 1 ميكرولتر من RNAseA واحتضانها عند 37 درجة مئوية ، مع الدوران عند 1400 دورة في الدقيقة لمدة 30 دقيقة.
- انقل العينات الموجودة على الثلج إلى أنابيب طرد مركزي مخروطية سعة 15 مل مبردة مسبقا وأضف 2 مل من الماء و 10.5 مل من EtOH المثلج البارد و 583 ميكرولتر من 3 M NaAC.
- مراقبة جودة إعداد قالب 3C
- تحديد كمية كل عينة والتحكم باستخدام مجموعة مقياس الفلور لقياسات تركيز الحمض النووي عالية الحساسية.
- قم بتحميل 100-200 نانوغرام من كل عينة وكل عنصر تحكم على 1٪ أغوار / 1x TBE هلام.
- تحقق من أن صورة الهلام تظهر النتيجة المتوقعة من خلال مقارنة الاختلافات في أحجام أجزاء الحمض النووي لعناصر التحكم وقالب 3C كما هو موضح في الشكل 2A.
- قم بتخزين العينات والضوابط في درجة حرارة -20 درجة مئوية.
- التهجين والالتقاط ومعالجة العينات للتسلسل المتعدد
- لتهجين مجموعة مجسات الحمض النووي الريبي البيوتينيل إلى قالب 3C ، والتقاط شظايا الربط المستهدفة ، وإعداد العينات للتسلسل المتعدد وفقا لنظام التخصيب المستهدف المستخدم في هذه الدراسة للتسلسل متعدد الإرسال المزدوج (انظر جدول المواد). اتبع البروتوكول وفقا لتعليمات الشركة المصنعة أثناء إدخال التعديلات الطفيفة التالية:
- القسم 2 من بروتوكول الشركة المصنعة: تحضير العينة
- اتبع التعليمات الخاصة بالتخصيب المستهدف بدءا من 3 ميكروغرام من مدخلات gDNA.
- قص الحمض النووي في صوتي باستخدام المواصفات التالية: دورة عمل 10٪ ، 4 شدة ، 200 cyc / انفجار ، و 130 ثانية. ابدأ ب 4 ميكروغرام من قالب 3C المعاد تعليقه في 130 ميكرولتر من الماء لكل تفاعل التقاط لضمان وجود مادة كافية لمواصلة تحضير العينة ب 3 ميكروغرام من الحمض النووي المنفصم.
- تقييم جودة الحمض النووي المنفصم. قم بتشغيل 1 ميكرولتر من الحمض النووي المقطوع على محلل حيوي للحمض النووي وفقا لبروتوكول الحساسية العالية. توقع توزيع حجم الشظية بين 150-700 نقطة أساس (الشكل 2).
- تنقية العينة باستخدام حبات تثبيت الطور الصلب القابل للانعكاس (SPRI). أضف 124 ميكرولتر من حبات SPRI إلى 124 ميكرولتر من عينة الحمض النووي لإجراء اختيار بحجم الجانب الأيسر 1: 1 وفقا لتعليمات الشركة المصنعة وقم بإذابة 25 ميكرولتر من الماء الخالي من النيوكلياز. ستزيل خطوة التنقية هذه شظايا أقصر لإثراء شظايا تبلغ حوالي 300 نقطة أساس (الشكل 2).
ملاحظة: تأخذ كمية العينات وخرز SPRI المستخدمة في هذه الخطوة في الاعتبار فقدان الحجم الذي حدث أثناء نقل العينات إلى أنابيب جديدة وتشغيل ضوابط الجودة في المحلل الحيوي. يتم تنفيذ جميع خطوات اختيار الحجم اللاحقة وفقا للنسب الموصى بها من قبل بروتوكول الشركة المصنعة. يتم إجراء شطف الحمض النووي من حبات SPRI في RT طوال البروتوكول. - تقييم جودة الحمض النووي المنفصم المختار الحجم. قم بتشغيل 1 ميكرولتر من الحمض النووي المقطوع على محلل الحمض النووي الحيوي وفقا لبروتوكول الحساسية العالية (HS). توقع توزيع أحجام الشظايا بأعلى تخصيب عند 300 نقطة أساس (الشكل 2). المضي قدما في تحديد كمية الحمض النووي المقطوع إذا كان القص ناجحا.
- قم بقياس الحمض النووي المقطوع باستخدام مجموعة مقياس الفلور لقياسات تركيز الحمض النووي HS.
ملاحظة: إذا أدى قص الحمض النووي إلى إنتاج الحمض النووي بمقدار <3 ميكروغرام ، فقم بإجراء جولة ثانية من قص الحمض النووي مع 4 ميكروغرام أخرى من الحمض النووي ودمج عينات الحمض النووي المقطوعة بعد خطوة تنقية حبة SPRI الأولى لتحقيق إجمالي 3 ميكروغرام من الحمض النووي المقطوع. - أضف الماء الخالي من النيوكلياز إلى عينة الحمض النووي النظيفة المختارة بالحجم (إجمالي 3 ميكروغرام) إلى حجم نهائي يبلغ 48 ميكرولتر واستمر في تفاعل الإصلاح النهائي وفقا لبروتوكول الشركة المصنعة.
- بعد ربط المحولات ذات النهاية المزدوجة ، قم بتضخيم المكتبة عن طريق إجراء خمس دورات من تفاعل البوليميراز المتسلسل قبل الالتقاط وفقا لتعليمات الشركة المصنعة (يتم توفير شروط PCR والبادئات في المجموعة).
- القسم 4 من بروتوكول الشركة المصنعة: التهجين والالتقاط
- لتهجين عينات الحمض النووي المحضرة إلى مجسات الحمض النووي الريبي الخاصة بالهدف ، قم بتخفيف 750 نانوغرام من عينات الحمض النووي في حجم نهائي قدره 3.4 ميكرولتر ، مما ينتج عنه تركيز أولي قدره 221 نانوغرام / ميكرولتر. بالنسبة لعينات الحمض النووي المخففة بكميات أكبر ، استخدم مكثف فراغ السرعة لتقليل الحجم النهائي. عادة ما يكون تركيز فراغ السرعة (250 × جم ؛ ≤45 درجة مئوية) لمدة 15-20 دقيقة كافيا للعينات المعاد تعليقها في 10 ميكرولتر. تأكد من أن يكون لديك نفس حجم الإدخال لكل عينة قبل بدء مكثف فراغ السرعة.
- احتضان خليط التهجين لمدة 16-18 ساعة عند 65 درجة مئوية بغطاء ساخن عند 105 درجة مئوية وفقا لتعليمات الشركة الصانعة.
- القسم 5 من بروتوكول الشركة المصنعة: الفهرسة ومعالجة العينات للتسلسل المتعدد
- لتضخيم المكتبات الملتقطة باستخدام بادئات الفهرسة ، قم بإجراء 12 دورة من PCR بعد الالتقاط وفقا لتعليمات الشركة المصنعة (يتم توفير شروط PCR والبادئات في المجموعة).
- القسم 2 من بروتوكول الشركة المصنعة: تحضير العينة
- لتهجين مجموعة مجسات الحمض النووي الريبي البيوتينيل إلى قالب 3C ، والتقاط شظايا الربط المستهدفة ، وإعداد العينات للتسلسل المتعدد وفقا لنظام التخصيب المستهدف المستخدم في هذه الدراسة للتسلسل متعدد الإرسال المزدوج (انظر جدول المواد). اتبع البروتوكول وفقا لتعليمات الشركة المصنعة أثناء إدخال التعديلات الطفيفة التالية:
- تسلسل الجيل التالي
- لتشغيل مكتبات التقاط Hi-C متعددة على نفس خلية التدفق ، قم بإعداد خليط متساوي المولي من مكتبات الالتقاط وتسلسل قراءات 100-120 M لكل مكتبة.
- إذا كانت هناك حاجة إلى تحليل خاص بالأليل ، فقم بتسلسل 150 نقطة أساس مزدوجة لضمان تغطية SNP كافية.
3. تحليل البيانات
- قم بتطبيق خط أنابيب HiC-Pro لإجراء تحليل بيانات CaptureHi-C 55. يوفر HiC-Pro ضوابط الجودة في كل خطوة من خطوات المعالجة ، بما في ذلك (الشكل 3):
(ط) معدل المحاذاة على الجينوم المرجعي الذي يحدد جزء القراءات التي تمتد عبر موقع الربط ، بالإضافة إلى عدد الأزواج والمفردات.
(ثانيا) جزء من منتجات الربط الصالحة وأزواج القراءة غير الإعلامية (النهاية المتدلية ، الربط الذاتي ، إلخ).
(ثالثا) جزء من الاتصالات قصيرة / طويلة المدى وداخل / بين الكروموسومات.
(رابعا) جزء من جهات الاتصال على الهدف لالتقاط Hi-C.
(ت) يقرأ جزء الأليل الخاص إذا تم تحديده.
ملاحظة: يدعم HiC-Pro مجموعة كبيرة من البروتوكولات بما في ذلك Hi-C في الموقع والتقاط Hi-C. في الحالة الأخيرة ، يحتاج المستخدم ببساطة إلى تحديد المنطقة المستهدفة (تنسيق BED) في ملف التكوين. بمجرد معالجة البيانات ، يمكن بسهولة تحويل مخرجات HiC-Pro إلى كائن أكثر برودة لتحليل المصب56. في هذه الخطوة ، يتم تطبيع خرائط الاتصال بدقة مختلفة باستخدام طريقة ICE التي وصفها سابقا Imakaev وزملاؤه57. يمكن بعد ذلك إجراء العديد من التحليلات لاستدعاء مقصورات الكروموسوم أو TADs أو حلقات الكروماتين (للمراجعة58). يظهر سير عمل البروتوكول في الشكل 4. هنا ، يتم تطبيق مجموعة "cooltools" لحساب درجة العزل وحدود TADs ، كما هو موضح في الشكل 5 والشكل 659.
النتائج
يعتمد بروتوكول Capture Hi-C الموصوف على إعداد قالب 3C على مستوى الجينوم باستخدام قاطع رباعي القواعد (DpnII). يتم الحصول على الإثراء اللاحق لشظايا الربط عبر المنطقة الجينومية ذات الأهمية عن طريق تهجين مجموعة من مجسات الحمض النووي الريبي التبليطية والتقاطها القائم على الستربتافيدين وفقا لنظام التخصيب المستهدف المستخدم في هذه الدراسة (الشكل 1). تم اختيار مجسات الحمض النووي الريبي البيوتينيلية لأنها تظهر تقاربا أكثر إحكاما لأهدافها مقارنة بمجسات الحمض النووي52,60. ثم يتم فهرسة المكتبات التي تم التقاطها وتجميعها من أجل تسلسل متعدد الإنتاجية عالي الإنتاجية. يمكن تصور بيانات التقاط Hi-C كخرائط تفاعل Hi-C عالية الدقة ولكن أيضا كخرائط اتصال أحادية نقطة عرض تشبه 4C لتصور تفاعلات التسلسلات الأصغر على وجه التحديد مثل المروجين أو المعززات داخل المنطقة الملتقطة بأكملها. يظهر سير عمل البروتوكول في الشكل 4. يتم عرض ضوابط جودة التسلسل المسبق في الشكل 2 وتشمل تقييم الهضم السليم وإعادة ربط قالب 3C والقص والتنقية الفعالة عبر الخطوات المختلفة للبروتوكول. من المتوقع أن يعمل الحمض النووي لقالب 3C المقطوع بين 150 إلى 700 نقطة أساس ، ولا ينبغي الكشف عن أي إثراء للشظايا >2 كيلو بايت. خلال الخطوات التالية ، يتم تنفيذ العديد من خطوات تنظيف الحمض النووي القائمة على الخرزة واختيار الحجم ، أولا بعد القص ، ثم بعد PCRs قبل الالتقاط وما بعد الالتقاط. تظهر المكتبات التي تم تنظيفها ملف تعريف إثراء شظية مميز كما هو موضح في محلل حيوي عالي الحساسية للحمض النووي (الشكل 2). يزداد متوسط حجم الجزء على مدار إعداد المكتبة بسبب ربط المحولات والتسلسل وفهرسة البادئات. يتم الحصول على ضوابط الجودة بعد التسلسل عبر Hi-C Pro وتظهر في الشكل 3. تم اقتراح العديد من تطبيقات برامج المعلوماتية الحيوية المختلفة لمعالجة وتحليل البيانات الشبيهة ب 3C. من بينها ، يعد خط أنابيب HiC-Pro أحد أكثر الحلول شيوعا ، مما يسمح بمعالجة بيانات التسلسل الخام إلى خرائط الاتصال النهائية بدقةمختلفة 55. يستخدم HiC-Pro استراتيجية رسم خرائط من خطوتين لمحاذاة قراءات التسلسل على الجينوم المرجعي. ثم يتم إعادة بناء منتجات 3C وتصفيتها لإزالة أزواج الاتصال غير المفيدة وإنشاء خرائط الاتصال. بالإضافة إلى ذلك ، فإنه قادر على استخدام قائمة من الأشكال المتعددة المعروفة لإجراء تحليل خاص بالأليل وفصل جهات الاتصال القادمة من الأليلين الأبويين في خرائط اتصال متميزة. في الآونة الأخيرة ، تم تضمين HiC-Pro وتوسيعه في إطار عمل nf-core (nf-core-hic) ، مما يوفر خط أنابيب مدفوعا بالمجتمع قابل للتطوير وقابل للتكراربدرجة كبيرة 61,62.
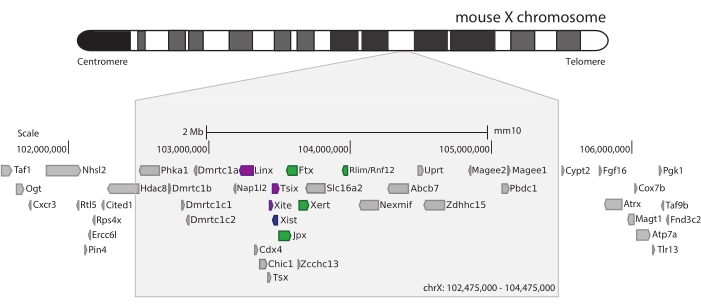
لالتقاط الفأر Xic ، تم تصميم مجموعة من 28,913 مسبار RNA تبليط 3 ميجا بايت من كروموسوم X. تتضمن هذه المنطقة اللاعب الرئيسي في XCI ، الجين الطويل غير المشفر Xist ، والمشهد التنظيمي المعروف ~ 800 كيلو بايت (الشكل 5). تنقسم هذه المنطقة ~ 800 كيلو بايت إلى اثنين من TADs: أحدهما يتضمن مروج Xist ومنظماته الإيجابية المعروفة (أي النصوص غير المشفرة Ftx و Jpx و Xert والجين المشفر للبروتين Rnf12) ، و TAD المجاور الذي يشمل منظمات رابطة الدول المستقلة السلبية ل Xist (أي نسخته المضادة للمعنى Tsix ، وعنصر المحسن Xite ، والنص غير المشفر Linx) (للمراجعة 44 ، 45).
من خلال تطبيق بروتوكول Capture Hi-C الموصوف على Xic ، تم الحصول على التنظيم الطوبولوجي لهذا الموضع بدقة غير مسبوقة (الشكل 6 والشكل 7). هذا واضح بشكل خاص عند مقارنة ملف تعريف Capture Hi-C ب 5C47 المنشور مسبقا (الشكل 6 والشكل 7 ؛ الجدول التكميلي 1) و Hi-C61 (الشكل 6 والشكل 7 ؛ الجدول التكميلي 1) ملفات تعريف. على سبيل المثال ، تكون هياكل TAD الفرعية أكثر وضوحا - من الواضح أن TAD الذي يحتوي على مروج Xist (Xist-TAD) ينقسم إلى مجالين أصغر (الشكل 6A ، رأس السهم الأزرق). في السابق ، كان من الممكن "تخمين" هذا بصريا فقط من ملف تعريف 5C (الشكل 6B) ، وإن كان اكتشاف حدود في هذه المنطقة باستخدام خوارزمية درجة العزل. وبالمثل ، فإن دقة ملف تعريف Capture Hi-C تسمح بتحديد مجالين أصغر في TAD المجاور (الشكل 6A ، B) ، والذي يحتوي على مروج موضع Tsix ( Tsix-TAD ) ؛ لم يتحقق ذلك من قبل مع 5C (الشكل 6B). وتجدر الإشارة إلى أن الحدود الطوبولوجية التي تحددها درجة العزل من بيانات Capture Hi-C و 5C يتم اكتشافها بشكل عام في مواقع مختلفة قليلا وبقوة نسبية مختلفة.
علاوة على ذلك ، فإن هياكل TAD الفرعية الأخرى مثل حلقات الاتصال مرئية بوضوح من بيانات Capture Hi-C ، مثل الحلقة بين Xist و Ftx (الشكل 7A) ، التي تم تحديدها مسبقا مع Capture-C63 ، والحلقة بين Xist و Xert (الشكل 7B) ، تم تحديدها مؤخرا باستخدام بروتوكول مماثل ل Capture Hi-C48. يمكن أيضا تعيين جهات الاتصال الأخرى بشكل أكثر دقة بسبب زيادة دقة ملفات تعريف Capture Hi-C ، مثل تلك التي تشكل نقاط الاتصال المعروفة داخل Tsix-TAD بين مواقع Linx و Chic1 و Xite (الشكل 7A).
بالمقارنة مع بيانات Hi-C الموضحة في الشكل 7 ، سمح Capture Hi-C بزيادة أربعة أضعاف في الدقة ، ومع ذلك فقد تطلب فقط ربع عمق التسلسل (أي 126 M يقرأ مقابل 571 M) (الجدول التكميلي 1). تسمح هذه الزيادة في الدقة باكتشاف subTADs وتفاعلات الحلقات التي لا يمكن اكتشافها بواسطة Hi-C عند عمق التسلسل الموضح في الشكل 6 والشكل 7. وبالتالي ، فإن البروتوكول الموصوف ل Capture Hi-C يسمح بتوصيف أكثر تفصيلا وعالي الدقة لمنطقة جينومية كبيرة ذات أهمية ، مقارنة بالأساليب السابقة.
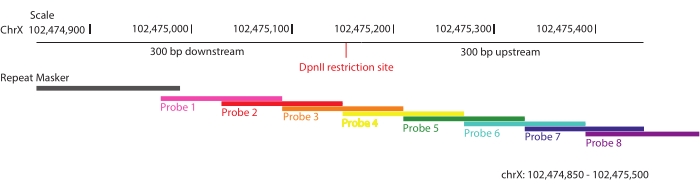
الشكل 1: تصميم المسبار. تمثيل تخطيطي للاستراتيجية المستخدمة لتصميم المسبار. تم اختيار مناطق 300 نقطة أساس في المنبع والمصب لكل موقع تقييد DpnII عبر المنطقة المستهدفة 3 ميجا بايت وتم تبليطها بمجسات الحمض النووي الريبي البيوتينيلية المتداخلة. تظهر إحدى هذه المناطق المحددة، chrX: 102,474,805-102,475,500. لا يسمح بأكثر من 40 قاعدة من التسلسلات المتكررة في كل مسبار. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
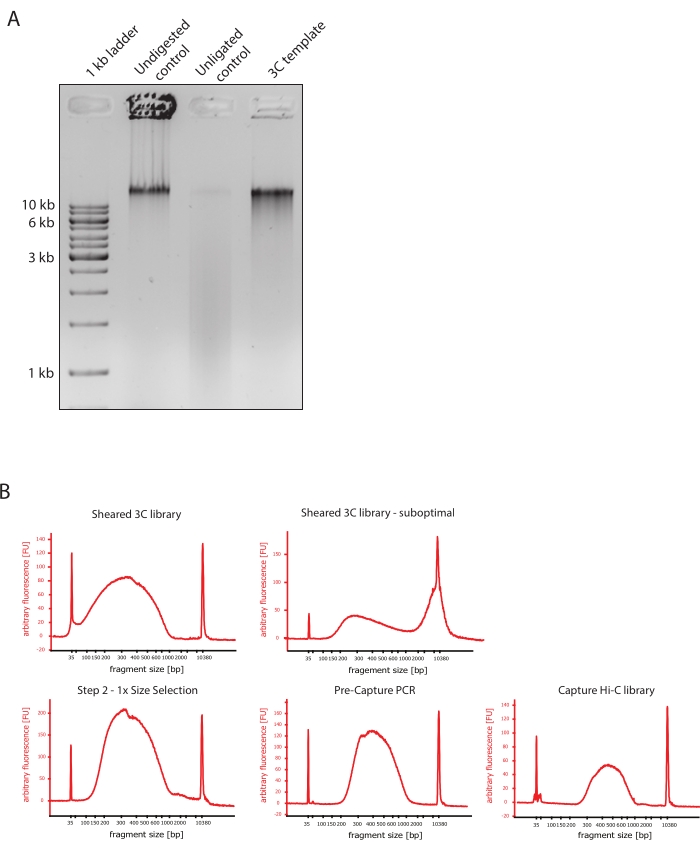
الشكل 2: التقاط ضوابط جودة التسلسل المسبق Hi-C . (أ) مثال تمثيلي لضوابط جودة قالب 3C. تم تحميل 200 نانوغرام من الحمض النووي على هلام أغاروز 1٪. حارة 1: 1 كيلو بايت سلم. الحارة 2: يعمل الكروماتين غير المهضوم والمتشابك والسليم كشريط حاد عند >10 كيلو بايت. الحارة 3: يعمل الكروماتين المتصالب المهضوم DpnII كمسحة يتراوح حجمها بين 1 كيلو بايت إلى 3 كيلو بايت. الحارة 4: مكتبة أو قالب 3C النهائي ؛ يتم إعادة ربط الأطراف الحرة لشظايا الحمض النووي المتشابكة المهضومة. لا يمكن اكتشاف مسحة الحمض النووي ذات الحجم الجزيئي المنخفض تقريبا ، ويتم اكتشاف منتج الربط كنطاق >10 كيلو بايت. (ب) أمثلة تمثيلية لملامح الحمض النووي للمحلل الحيوي عالي الحساسية. أعلى اليسار: مكتبة 3C تم قصها بنجاح تظهر توزيعا لحجم الجزء بين 150 نقطة أساس و 700 نقطة أساس. أعلى اليمين: مكتبة 3C غير مرضية. تم الكشف عن الحمض النووي غير المقطوع على أنه إثراء واسع للشظايا >2 كيلو بايت. (ج) أسفل اليسار: عينة الحمض النووي المنفصمة بعد اختيار حجم الجانب الأيسر 1: 1 باستخدام حبات SPRI. يتم إثراء شظايا ~ 300 bp. الوسط السفلي: ملف تعريف PCR قبل الالتقاط بعد ربط المحولات ذات النهاية المزدوجة وفقا لبروتوكول الشركة المصنعة. أسفل اليمين: مكتبة Capture Hi-C النهائية بما في ذلك المحولات والتسلسل وبادئات الفهرسة للتسلسل متعدد الإرسال. الاختصارات: bp = أزواج القواعد ، FU = وحدة مضان تعسفية. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
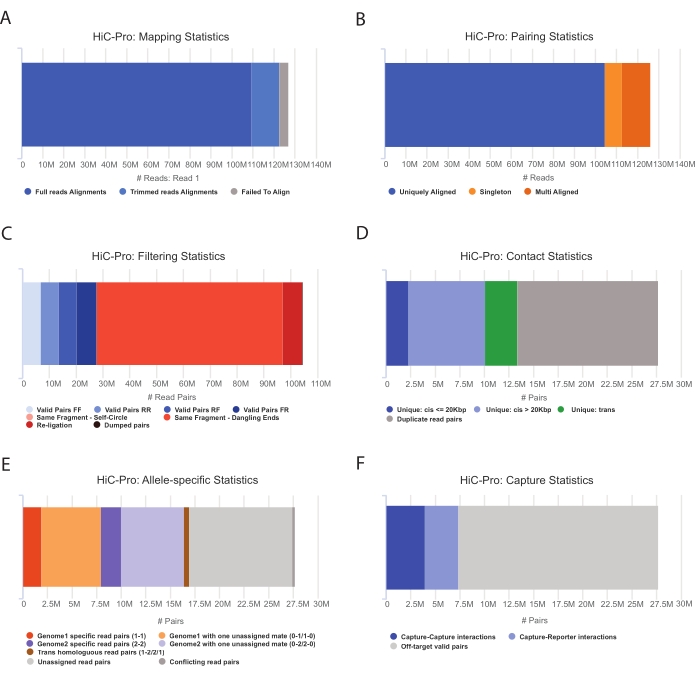
الشكل 3: التقاط ضوابط الجودة بعد التسلسل Hi-C باستخدام HiC-Pro . (أ) مثال على معدل رسم الخرائط على الجينوم المرجعي للرفيق الأول لأزواج التسلسل. يمثل الجزء الأزرق الفاتح القراءات المحاذية بواسطة HiC-Pro وتمتد عبر تقاطع ربط. وبالتالي يمكن استخدام هذا المقياس للتحقق من صحة خطوة الربط التجريبية. (ب) بمجرد محاذاة زملاء التسلسل على الجينوم ، يتم الاحتفاظ بأزواج القراءة المحاذاة بشكل فريد فقط للتحليل. (ج) يتم استبعاد الأزواج غير الصالحة (باللون الأحمر) مثل الطرف المتدلي أو الدائرة الذاتية أو إعادة الربط من التحليل. يعد جزء الأزواج الصالحة مؤشرا جيدا على كفاءة الربط والقائمة المنسدلة. (د) يمكن تقسيم الأزواج الصالحة إلى ملامسات داخل الكروموسومات وبينها وبين ملامسات قصيرة/طويلة المدى. يتم تجاهل أزواج القراءة المكررة التي من المحتمل أن تمثل عناصر تفاعل البوليميراز المتسلسل من التحليل. (ه) بالنسبة للتحليل الخاص بالأليل ، يبلغ HiC-Pro عن عدد القراءات الأليلية التي يدعمها إما واحد أو اثنين من الشركاء لكل جينوم أبوي (أي C57BL / 6J x CASTEi / J). من المتوقع نفس الجزء من القراءات المخصصة لأليل الأم والأب. (F) أخيرا ، يتم تحديد الأزواج الصالحة المتداخلة مع منطقة الالتقاط فقط لإنشاء خرائط الاتصال. تمثل أزواج الالتقاط والالتقاط جهات اتصال داخل المنطقة المستهدفة ، بينما تتضمن أزواج الالتقاط والمراسل التفاعل بين المنطقة المستهدفة والمنطقة خارج الهدف. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
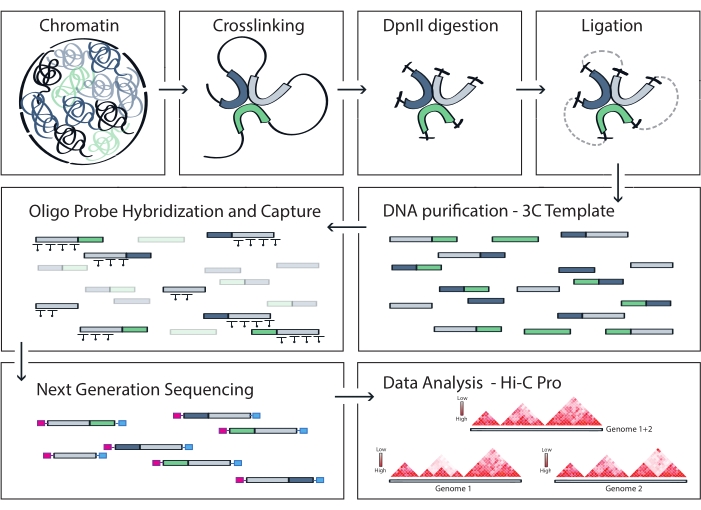
الشكل 4: سير عمل بروتوكول التقاط Hi-C. تمثيل تخطيطي لخطوات البروتوكول المختلفة. لإنشاء قالب 3C على مستوى الجينوم ، يتم ربط الكروماتين أولا بالفورمالديهايد ثم يتم هضمه باستخدام إنزيم تقييد DpnII. ثم يتم إعادة ربط نهايات الحمض النووي الحرة ، ويتم عكس الارتباط المتقاطع ، ويتم تنقية الحمض النووي. لإثراء الشظايا التي تشمل المنطقة المستهدفة ، يتم تهجين مجموعة من مجسات الحمض النووي الريبي البيوتينيلاتي إلى قالب 3C ويتم التقاطها بواسطة السحب بوساطة الستربتافيدين لأسفل. تتم معالجة مكتبات الالتقاط للتسلسل المتعدد ، ويتم تحديد أجزاء الربط الصالحة لاستنتاج تكرار اتصالات الكروماتين عبر الهدف ، والتي يتم تصورها كخرائط تفاعل عالية الدقة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الشكل 5: نظرة عامة على المنطقة التي تضم Xic على كروموسوم X للفأر. تمثيل تخطيطي لكروموسوم الماوس X وتكبير المنطقة الملتقطة 3 ميجا بايت (ChrX: 102,475,000-105,475,000). تتضمن المنطقة المستهدفة ~ 800 كيلو بايت من الحمض النووي المقابل ل Xic ، الموضع التنظيمي الرئيسي ل XCI. يتضمن Xic الجينات الطويلة غير المشفرة ، Xist ، وهو لاعب رئيسي في XCI ، والمشهد التنظيمي. تظهر المنظمات الإيجابية ل Xist باللون الأخضر ، والمنظمين السالبين باللون الأرجواني. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
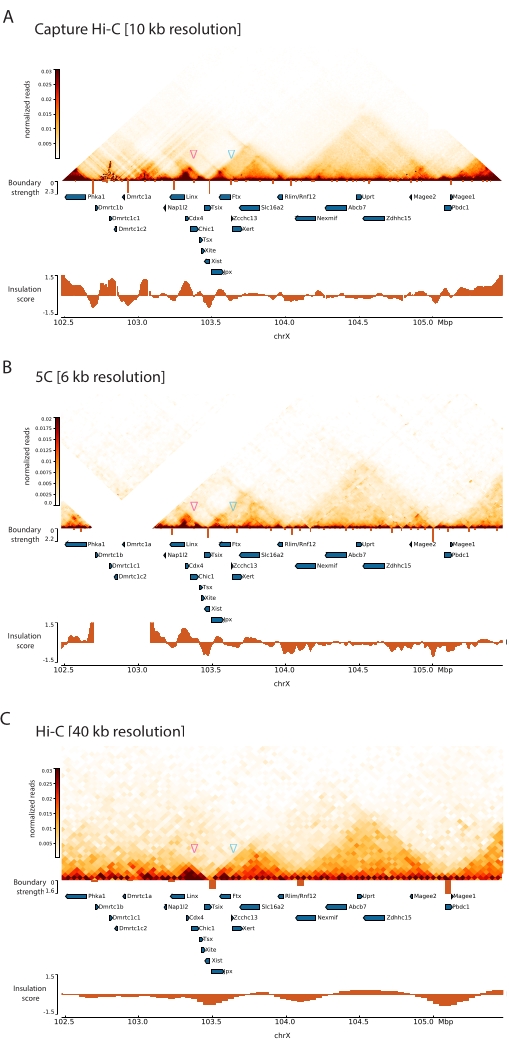
الشكل 6: التقط خرائط تفاعل Hi-C و 5C و Hi-C عبر المنطقة الملتقطة 3 ميجا بايت. (أ) التقاط خريطة تفاعل Hi-C لهدف 3 ميجا بايت يشمل الماوس Xic بدقة 10 كيلو بايت (هذه الدراسة). (B) خريطة تفاعل 5C لنفس المنطقة المستهدفة كما في A بدقة 6 كيلو بايت (البيانات المعاد معالجتها من47). المناطق المتكررة غير المدرجة في التحليلات مقنعة باللون الأبيض. تتطلب بيانات 5C معالجة المعلوماتية الحيوية الخاصة بها (انظر47). بعد التنظيف والمحاذاة ، يتم تثبيت خرائط 5C بدقة التمهيدي باستخدام وسيط قيد التشغيل (نافذة = 30 كيلو بايت ، الخطوة = 5) للوصول إلى دقة نهائية تبلغ 6 كيلوبايت. (C) خريطة تفاعل Hi-C لنفس المنطقة الجينومية كما في A و B بدقة 40 كيلو بايت (البيانات المعاد معالجتها من64). تم إنشاء جميع خرائط التفاعل من ESCs الماوس. تم حساب درجة العزل باستخدام أدوات التبريد ويتم تمثيلها كرسوم بيانية مع الحد الأدنى للعزل عند حدود TAD. تظهر حدود TAD كخطوط عمودية أسفل الخريطة. يشير ارتفاع كل خط إلى قوة الحدود. تظهر الجينات في صورة أسهم تشير إلى اتجاه النسخ. تتم الإشارة إلى حدود TAD الفرعية التي يتم اكتشافها بشكل حصري أو أكثر دقة في خرائط Capture Hi-C بواسطة رؤوس الأسهم الأرجواني والأزرق ل TADs الفرعية في Tsix و Xist TADs ، على التوالي. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
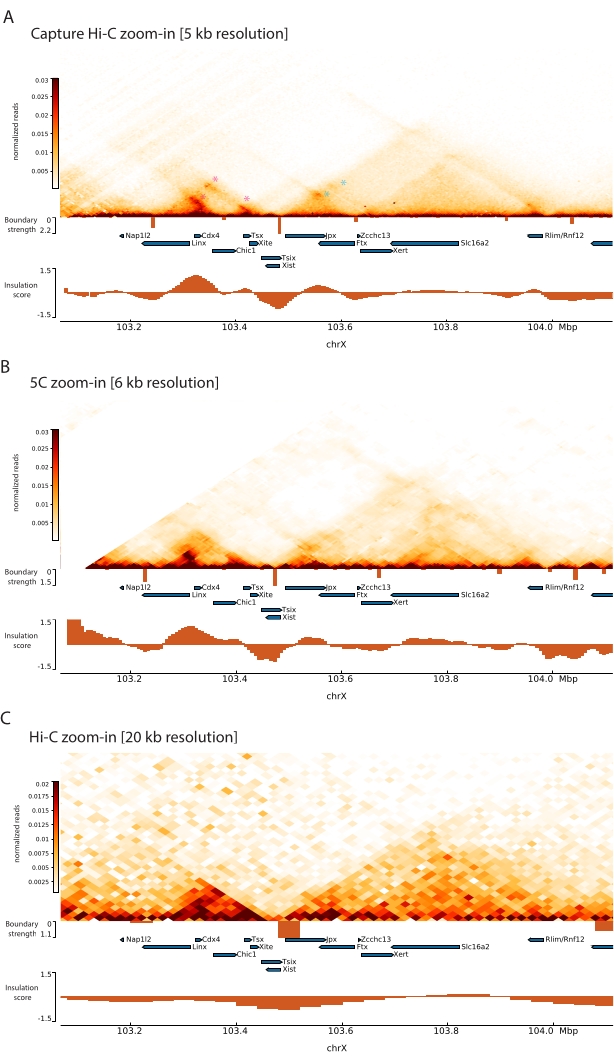
الشكل 7: التقط خرائط تفاعل Hi-C و 5C و Hi-C عبر 1 ميجا بايت داخل المنطقة الملتقطة. (أ) التقاط خريطة تفاعل Hi-C للمنطقة الجينومية 1 ميجا بايت التي تشمل الفأر Xic بدقة 5 كيلو بايت (هذه الدراسة). (ب) خريطة تفاعل 5C لنفس المنطقة الجينومية كما في A. بدقة 6 كيلوبايت (تمت إعادة معالجة البيانات من47). المناطق المتكررة غير المدرجة في التحليلات مقنعة باللون الأبيض. وتجدر الإشارة إلى أن بيانات 5C تتطلب معالجة المعلوماتية الحيوية الخاصة بها (انظر47). بعد التنظيف والمحاذاة ، يتم تثبيت خرائط 5C بدقة التمهيدي باستخدام وسيط قيد التشغيل (نافذة = 30 كيلو بايت ، الخطوة = 5) للوصول إلى دقة نهائية تبلغ 6 كيلوبايت. (C) خريطة تفاعل Hi-C لنفس المنطقة الجينومية كما في A و B من Hi-C بدقة 20 كيلو بايت (البيانات المعاد معالجتها من64). تم إنشاء جميع خرائط التفاعل من mESCs. تم حساب درجة العزل باستخدام أدوات التبريد ويتم تمثيلها كرسوم بيانية مع الحد الأدنى للعزل عند حدود TAD. تظهر حدود TAD كخطوط عمودية أسفل الخريطة. يشير ارتفاع كل خط إلى قوة الحدود. تظهر الجينات في صورة أسهم تشير إلى اتجاه النسخ. يشار إلى حلقات التلامس التي يتم اكتشافها بشكل حصري أو أكثر دقة في Capture Hi-C بعلامات نجمية أرجوانية وزرقاء للحلقات في Tsix و Xist TADs ، على التوالي. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الجدول التكميلي 1: إحصائيات ما بعد التسلسل لمجموعات البيانات المستخدمة في هذه المخطوطة: Capture Hi-C (هذه الدراسة) ، Hi-C64 ، و 5C47. الرجاء الضغط هنا لتنزيل هذا الملف.
Discussion
هنا نصف بروتوكول Capture Hi-C السريع والسهل نسبيا لتوصيف التنظيم الأعلى ترتيبا للمناطق الجينومية ذات الحجم الضخم بدقة 5-10 كيلو بايت. ينتمي Capture Hi-C إلى عائلة تقنيات Capture-C المصممة لإثراء تفاعلات الكروماتين المستهدفة من قوالب 3C أو Hi-C على مستوى الجينوم. حتى الآن ، تم استغلال الغالبية العظمى من تطبيقات Capture-C لرسم خريطة لاتصالات الكروماتين للعناصر التنظيمية الصغيرة نسبيا المنتشرة عبر الجينوم بأكمله. في بروتوكول Capture-C الأول ، تم استخدام مجسات متعددة متداخلة من الحمض النووي الريبي الحيوي لالتقاط >400 تم اختيارهم مسبقا في مكتبات 3C المحضرة من خلايا الكريات الحمر31. تم تحسين نفس الاستراتيجية لاحقا في الجيل التالي (NG) والمعايرة النووية (NuTi) Capture-C لتحقيق ملفات تعريف تفاعل عالية الدقة ل >8000 مروج باستخدام طعوم DNA مفردة 120 bp تغطي مواقع تقييد فردية وجولتين متتاليتين من الالتقاط لزيادة إثراء شظايا الربط المعلوماتية إلى أقصى حد32,40. أدت هذه الاستراتيجيات إلى التشريح الوظيفي للعناصر التي تعمل في رابطة الدول المستقلة في العديد من السياقات المختلفة ، بما في ذلك التطور الجنيني للفأر ، وتمايز الخلايا ، وتعطيل الكروموسوم X ، وسوء تنظيم الجينات في الحالات المرضية 46،63،65،66،67،68،69،70،71.
في Promoter Capture Hi-C (PCHi-C) ، تم سحب > 22000 مشروح يحتوي على شظايا تقييد من مكتبات Hi-C عن طريق تهجين مجسات البيوتينيل أحادية الحمض النووي الريبي 120-mer في أي من طرفي جزء التقييد34,72 أو كلاهما. سمحت هذه الطريقة بتشريح تفاعل الآلاف من المروجين في عدد متزايد بسرعة من أنواع الخلايا ، بما في ذلك الخلايا الجذعية الجنينية للفأر ، وخلايا الكبد الجنينية ، والخلايا الشحمية 34،35،72،73 ، ولكن أيضا خطوط الأرومة اللمفاوية البشرية ، والأسلاف المكونة للدم ، والخلايا الكيراتينية للبشرة ، والخلايا متعددة القدرات37،74،75،76،77.
بالمقارنة مع تقنيات التخصيب المستهدفة هذه ، يستهدف Capture Hi-C المناطق الجينومية المتجاورة حتى النطاق الضخم ، وبالتالي يمتد على واحد أو أكثر من TADs ويشمل المناظر الطبيعية التنظيمية للجينات. يجب أن تكون المنطقة محل الاهتمام بأكملها مبلطة بمجموعة من مجسات البيوتينيلات التي تشمل كل موقع تقييد DpnII داخل الهدف. يتم إجراء تهجين المصفوفة البيوتينيلية إلى قالب 3C ، والتقاطها اللاحق القائم على الستربتافيدين ، ومعالجة التسلسل المتعدد الإرسال باستخدام نظام إثراء مستهدف لتسلسل Illumina Paired-End متعدد الإرسال. البروتوكول بأكمله سريع ، حيث يمكن إجراؤه في أسبوع 1 من إعداد مكتبة 3C إلى تسلسل NGS ، ولا يتطلب سوى تعديلات طفيفة و / أو استكشاف الأخطاء وإصلاحها حسب الطلب.
يوفر البروتوكول أيضا مزايا مقارنة بالطرق الأخرى المستندة إلى 3C. للحصول على خرائط التفاعل بدقة 5-10 كيلو بايت ، قمنا بتسلسل قراءات 100-120 M ذات النهاية الزوجية. على سبيل المقارنة ، استخدمنا هنا مجموعة بيانات Hi-C من 571 مليون قراءة للوصول إلى دقة 20 كيلو بايت64 (GSM2053973) ، وستكون هناك حاجة إلى 1 مليار قراءة على الأقل للوصول إلى دقة 5 كيلو بايت مع Hi-C22 على مستوى الكروموسوم.
يصل التقاط Hi-C كما هو مستخدم في هذه الدراسة إلى دقة أعلى بكثير من 5C المنشور سابقا بناء على إنزيم تقييد قاطع 6-bp47 (الجدول التكميلي 1). الأهم من ذلك ، أن الاستراتيجية المصممة لإثراء وتضخيم التفاعلات المستهدفة في 5C لا تسمح بتحليل خاص بالأليل لتفاعلات الكروماتين. على العكس من ذلك ، يمكن تعيين بيانات Capture Hi-C على وجه التحديد ، مما يسمح بتشريح المناظر الطبيعية الهيكلية ثلاثية الأبعاد لأزواج من الكروموسومات المتماثلة ، على سبيل المثال في الخلايا البشرية أو في خطوط الخلايا الهجينة F1 المشتقة عن طريق عبور سلالات الفئران المختلفةوراثيا 78. لإنشاء خرائط تفاعل Capture Hi-C الخاصة بالأليل بدقة 5 كيلوبايت ، قمنا بتسلسل قراءات طرفية مزدوجة 150 بت لزيادة تغطية SNP. يمكن تطبيق مناهج مماثلة خاصة بالأليل على خطوط الخلايا البشرية ، والتي يتوفر لها شرح SNPs22.
الأهم من ذلك ، على الرغم من أن Capture Hi-C يضمن عموما دقة عالية مع تحسين القدرة على تحمل تكاليف التسلسل ، فإن إنتاج قليل النوكليوتيدات البيوتينيلاتي المصممة خصيصا له تأثير على التكلفة الإجمالية لهذه الطريقة. لذلك ، سيختلف اختيار طريقة 3C الأنسب للتطبيقات المختلفة ، وسيعتمد على السؤال البيولوجي الذي يتم معالجته والقرار المطلوب ، بالإضافة إلى حجم منطقة الاهتمام. تشترك بروتوكولات Capture Hi-C الأخرى التي تم تطويرها في الميزات الرئيسية مع البروتوكول الموضح هنا. على سبيل المثال ، تم تطبيق استراتيجية Capture Hi-C لتوصيف ~ 50 كيلو بايت إلى 1 ميجا بايت من المناطق الجينومية التي تغطي المتغيرات غير المشفرة المرتبطة بخطر الإصابة بسرطان الثدي والقولون والمستقيم. في هذا البروتوكول ، تم سحب المناطق المستهدفة من مكتبات Hi-C عن طريق تهجين طعوم الحمض النووي الريبي 120 مير لتبليط المناطق المستهدفة بتغطية 3x33،38،79. وبالمثل ، تم استخدام HYbrid Capture Hi-C (Hi-C 2) لاستهداف التفاعلات داخل المناطق ذات الاهتمام حتى2 ميجا بايت80. في كلا البروتوكولين ، أدى استخدام قالب Hi-C المخصب لشظايا ربط البيوتين المسحوبة إلى أسفل إلى زيادة النسبة المئوية لإجمالي القراءات الإعلامية مقارنة ببروتوكولنا. على سبيل المثال ، في مجموعة بيانات Hi-C التي استخدمناها هنا للمقارنة64 (GSM2053973) ، فإن النسبة المئوية للأزواج الصالحة بعد إزالة التكرارات أعلى بمقدار 4.8 أضعاف من الأزواج الصالحة التي تم الحصول عليها في Capture Hi-C كما هو موضح في الشكل 3 والجدول التكميلي 1. ومع ذلك ، فإن السحب المتتالي للشظايا المربوطة بالبيوتينيل والمجسات المهجنة يجعل البروتوكول أكثر تعقيدا واستهلاكا للوقت مع تقليل تعقيد المنطقة التي تم التقاطها.
طريقة أخرى متاحة لإثراء قوالب 3C بمجسات التبليط هي Tiled-C ، والتي تم تطبيقها لدراسة بنية الكروماتين بدقة مكانية وزمانية عالية أثناء تمايز الإريثرويدللفأر 43. في Tiled-C ، يتم استخدام لوحة من مجسات البيوتينيل 70 bp لإثراء الاتصالات داخل المناطق واسعة النطاق في جولتين متتاليتين من الالتقاط لإنشاء خرائط عالية الدقة للتفاعلات المستهدفة43,81. كما أن إثراء الالتقاط المزدوج يجعل البروتوكول أطول وأكثر تعقيدا عند مقارنته ب Capture Hi-C. ومع ذلك ، تختلف عن استراتيجيات Capture-C التي تستهدف مواقع تقييد فردية ، في Tiled-C ، لا يبدو أن الجولة الثانية من الالتقاط تزيد بشكل كبير من كفاءة الالتقاط ، وبالتالي يمكن حذفهاعلى الأرجح 43. أخيرا ، تم تطبيق نهج تبليط مماثل يعتمد على نفس استراتيجية التخصيب المستهدفة المستخدمة في هذه الدراسة على تشريح المناظر الطبيعية التنظيمية التي تشمل المتغيرات الهيكلية الموصوفة في المرضى الذين يعانون من تشوهات خلقية وإعادة هندستها في الفئران المعدلة وراثيا41،42. في هذه الحالة ، تم تصميم مجموعة مجسات التجانب عبر الهدف بأكمله بدلا من بالقرب من مواقع تقييد DpnII41. ومع ذلك ، كان هذا العمل أساسيا في تسليط الضوء على حساسية وقوة هذه الاستراتيجية لتحقيق توصيف عالي الدقة للمناطق الجينومية الكبيرة في سياقات مختلفة41،42،48.
في الختام ، يمثل البروتوكول الموصوف هنا استراتيجية سهلة وقوية وقوية لتوصيف 3D عالي الدقة لأي مناطق جينومية ذات أهمية. من المرجح أن يسهل تطبيق هذا النهج على أنظمة النماذج المختلفة ، وأنواع الخلايا ، والمناظر الطبيعية للكروماتين المنظمة تنمويا ، وتنظيم الجينات في الظروف الصحية والمرضية فهمنا للتفاعل والسببية بين طوبولوجيا الجينوم وتنظيم الجينات ، وهو أحد الأسئلة الأساسية المفتوحة في مجال علم التخلق. علاوة على ذلك ، فإن تطبيق Capture Hi-C لرسم خريطة للتفاعلات بعيدة المدى وطي الكروماتين الأعلى مرتبة لمتغيرات المخاطر التي حددتها دراسات GWAS لديه القدرة على الكشف عن الأهمية الوظيفية للمواقع الجينومية غير المشفرة المرتبطة بالأمراض البشرية في سياقات مختلفة ، وبالتالي توفير رؤى جديدة حول العمليات الكامنة الكامنة وراء الإمراض.
Disclosures
كاي هاوشولتز هو عالم التطبيقات الميدانية في Agilent Technologies - مجموعة التشخيص والجينوم. جميع المؤلفين الآخرين يعلنون عدم وجود مصالح متنافسة.
Acknowledgements
تم دعم العمل في مختبر هيرد بجائزة الباحث المتقدم لمجلس البحوث الأوروبي (XPRESS - AdG671027). يتم دعم A.L. من قبل زمالة ماري سكودوفسكا كوري الفردية للاتحاد الأوروبي (IF-838408). يتم دعم A.H. من قبل شبكة ChromDesign المبتكرة والمتعددة التخصصات ITN ، بموجب اتفاقية Marie Skłodowska-Curie Grant 813327. يعرب المؤلفون عن امتنانهم لدانيال إبراهيم (MPI لعلم الوراثة الجزيئية ، برلين) للحصول على المشورة الفنية المفيدة ، ولمنصة NGS في معهد كوري (باريس) ، ولفلاديمير بينيس ومرفق الجينوم الأساسي في EMBL (هايدلبرغ) ، للدعم والمساعدة.
Materials
| Name | Company | Catalog Number | Comments |
| 10x PBS pH 7.4 | Gibco | 10010-023 | |
| 37% (vol/vol) paraformaldehyde solution | Electron Microscopy Sciences | 15686 | single use glass-vials; do not reuse |
| 50 mL PP conical tube | Falcon | 352070 | |
| Agarose | Sigma | A9539-500g | |
| Bioanalyzer | Agilent | G2939BA | |
| Cell Scrapers - 25 cm Handle and 3.0 cm Blade | Falcon | 353089 | |
| CHIR99021 | Axon Medchem BV | Axon 1386 | |
| cOmplete Mini, Protease inhibitor cocktail (EDTA-free) | Merck | 11836170001 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL | Invitrogen | ZGEXSCCOUNTESS2FL | Automated cell counter |
| Covaris S2 | Covaris | 500217 | Sonicator |
| DNA LoBind tube, 1.5 mL | Eppendorf | 30108051 | |
| DpnII (50000 units/mL) | New England Biolabs | R0543M | |
| Dulbecco's Modified Eagle Medium (DMEM) | Merck | D6429 | |
| Ethanol (100%) | Merck | 1.00983.2500 | |
| Fetal Bovine Serum (FBS) | Thermo Scientific | 10270106 | |
| gelatine from porcine skin | Sigma | G1890 | |
| GeneRuler 1 kb Plus DNA Ladder | Thermo Scientific | SM0313 | |
| GlycoBlue | Thermo Scientific | AM9516 | Coprecipitant |
| High-Sensitivity Bioanlayzer chips | Agilent | 5067-4626 | |
| Large Cooling Centrifuge 5920 R | Eppendorf | 5948000018 | |
| leukaemia inhibitory factor (LIF) | Merck | ESG1107 | |
| Liquiport | KNF | NF300 | Benchtop aspiration system |
| Low-binding filter tips | Biozym | VT0260U, VT0240, VT0220, VT0200U | |
| Molecular biology grade water | Merck | W3500-6x500ML | |
| Next Seq 500 | Illumina | SY-415-1001 | |
| Next Seq 500 High Output v2 Kit (300 cycles) | Illumina | FC-404-2004 | |
| Nonidet P40 Substitute (NP40) | Merck | 11332473001 | |
| PD0325901 | Axon Medchem BV | Axon 1408 | |
| Protease inhibitor cocktail (EDTA-free) | Merck | 11873580001 | |
| Proteinase K - recombinant, PCR-grade (20 mg/mL) | Thermo Scientific | EO0491 | |
| Qubit 2.0 | Thermo Scientific | Q32871 | |
| Qubit assay tubes | Thermo Scientific | Q32856 | |
| Qubit dsDNA High Sensitivity kit | Thermo Scientific | Q32851 | |
| RNase A (10 mg/mL) | Thermo Scientific | EN0531 | |
| Sodium acetate pH 5.2 (3M) | Merck | S7899 | |
| speed vacuum concentrator | Eppendorf | EP5305000100-1EA | |
| Agencourt AMPureXP | Beckman Coulter | A63881 | SPRI beads |
| SureSelect Target Enrichment Box 1 | Agilent | 5190-8645 | |
| SureSelect Target Enrichment Kit ILM Indexing Hyb Module Box 2 | Agilent | 5190-4455 | |
| SureSelect XT Library Prep Kit ILM | Agilent | 5500-0132 | |
| T4 ligase (30 units/µL) | Thermo Scientific | EL0013 | |
| table-top Centrifuge 5427 R | Eppendorf | 5409000012 | |
| Triton-X-100 (500 mL) | Merck | X100-500ML | |
| Trypan Blue | Invitrogen | T10282 | |
| Trypsine | Thermo Scientific | 25300054 | |
| UltraPure Glycine | Thermo Scientific | 15527013 | |
| β-mercaptoethanol | Thermo Scientific | 31350010 |
References
- Ibrahim, D. M., Mundlos, S. The role of 3D chromatin domains in gene regulation: a multi-facetted view on genome organization. Current Opinion in Genetics & Development. 61, 1-8 (2020).
- Bolt, C. C., Duboule, D. The regulatory landscapes of developmental genes. Development. 147 (3), (2020).
- Glaser, J., Mundlos, S. 3D or not 3D: Shaping the genome during development. Cold Spring Harbor Perspectives in Biology. 14 (5), 040188(2021).
- Denker, A., De Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Kempfer, R., Pombo, A. Methods for mapping 3D chromosome architecture. Nature Reviews Genetics. 21 (4), 207-226 (2020).
- McCord, R. P., Kaplan, N., Giorgetti, L. Chromosome conformation capture and beyond: Toward an integrative view of chromosome structure and function. Molecular Cell. 77 (4), 688-708 (2020).
- Jerkovic, I., Cavalli, G. Understanding 3D genome organization by multidisciplinary methods. Nature ReviewsMolecular Cell Biology. 22 (8), 511-528 (2021).
- Hsieh, T. -H. S., et al. Mapping nucleosome resolution chromosome folding in yeast by Micro-C. Cell. 162 (1), 108-119 (2015).
- Krietenstein, N., et al. Ultrastructural details of mammalian chromosome architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Naumova, N., Smith, E. M., Zhan, Y., Dekker, J. Analysis of long-range chromatin interactions using Chromosome Conformation Capture. Methods. 58 (3), 192-203 (2012).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Zhao, Z., et al. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra-and interchromosomal interactions. Nature Genetics. 38 (11), 1341-1347 (2006).
- Würtele, H., Chartrand, P. Genome-wide scanning of HoxB1-associated loci in mouse ES cells using an open-ended Chromosome Conformation Capture methodology. Chromosome Research. 14 (5), 477-495 (2006).
- De Wit, E., De Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes & Development. 26 (1), 11-24 (2012).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Splinter, E., et al. The inactive X chromosome adopts a unique three-dimensional conformation that is dependent on Xist RNA. Genes & Development. 25 (13), 1371-1383 (2011).
- Ferraiuolo, M. A., Sanyal, A., Naumova, N., Dekker, J., Dostie, J. From cells to chromatin: capturing snapshots of genome organization with 5C technology. Methods. 58 (3), 255-267 (2012).
- Kim, J. H., et al. 5C-ID: Increased resolution Chromosome-Conformation-Capture-Carbon-Copy with in situ 3C and double alternating primer design. Methods. 142, 39-46 (2018).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Zhang, Y., et al. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations. Cell. 148 (5), 908-921 (2012).
- Rao, S. S. P., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Dixon, J. R., et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485 (7398), 376-380 (2012).
- Nora, E. P., et al. Spatial partitioning of the regulatory landscape of the X-inactivation centre. Nature. 485 (7398), 381-385 (2012).
- Krefting, J., Andrade-Navarro, M. A., Ibn-Salem, J. Evolutionary stability of topologically associating domains is associated with conserved gene regulation. BMC Biology. 16 (1), 87(2018).
- Galupa, R., Heard, E. Topologically associating domains in chromosome architecture and gene regulatory landscapes during development, disease, and evolution. Cold Spring Harbor Symposia on Quantitative Biology. 82, 267-278 (2017).
- Tena, J. J., Santos-Pereira, J. M. Topologically associating domains and regulatory landscapes in development, evolution and disease. Frontiers in Cell and Developmental Biology. 9, 702787(2021).
- Lupiáñez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How alterations of chromatin domains result in disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Davidson, I. F., Peters, J. -M. Genome folding through loop extrusion by SMC complexes. Nature Reviews Molecular Cell Biology. 22 (7), 445-464 (2021).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews Molecular Cell Biology. 17 (12), 743-755 (2016).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Davies, J. O. J., et al. Multiplexed analysis of chromosome conformation at vastly improved sensitivity. Nature Methods. 13 (1), 74-80 (2016).
- Jäger, R., et al. Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci. Nature Communications. 6, 6178(2015).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Sahlén, P., et al. Genome-wide mapping of promoter-anchored interactions with close to single-enhancer resolution. Genome Biology. 16, 156(2015).
- Joshi, O., et al. Dynamic reorganization of extremely long-range promoter-promoter interactions between two states of pluripotency. Cell Stem Cell. 17 (6), 748-757 (2015).
- Mifsud, B., et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nature Genetics. 47 (6), 598-606 (2015).
- Dryden, N. H., et al. Unbiased analysis of potential targets of breast cancer susceptibility loci by Capture Hi-C. Genome Research. 24 (11), 1854-1868 (2014).
- Oudelaar, A. M., Davies, J. O. J., Downes, D. J., Higgs, D. R., Hughes, J. R. Robust detection of chromosomal interactions from small numbers of cells using low-input Capture-C. Nucleic Acids Research. 45 (22), 184(2017).
- Oudelaar, A. M., et al. Single-allele chromatin interactions identify regulatory hubs in dynamic compartmentalized domains. Nature Genetics. 50 (12), 1744-1751 (2018).
- Franke, M., et al. Formation of new chromatin domains determines pathogenicity of genomic duplications. Nature. 538 (7624), 265-269 (2016).
- Despang, A., et al. Functional dissection of the Sox9-Kcnj2 locus identifies nonessential and instructive roles of TAD architecture. Nature Genetics. 51 (8), 1263-1271 (2019).
- Oudelaar, A. M., et al. Dynamics of the 4D genome during in vivo lineage specification and differentiation. Nature Communications. 11 (1), 1-12 (2020).
- Galupa, R., Heard, E. X-chromosome inactivation: A crossroads between chromosome architecture and gene regulation. Annual Review of Genetics. 52, 535-566 (2018).
- Loda, A., Collombet, S., Heard, E. Gene regulation in time and space during X-chromosome inactivation. Nature Reviews. Molecular Cell Biology. 23 (4), 231-249 (2022).
- van Bemmel, J. G., et al. The bipartite TAD organization of the X-inactivation center ensures opposing developmental regulation of Tsix and Xist. Nature Genetics. 51 (6), 1024-1034 (2019).
- Galupa, R., et al. A conserved noncoding locus regulates random monoallelic Xist expression across a topological boundary. Molecular Cell. 77 (2), 352-367 (2020).
- Gjaltema, R. A. F., et al. Distal and proximal cis-regulatory elements sense X chromosome dosage and developmental state at the Xist locus. Molecular Cell. 82 (1), 190-208 (2022).
- Galupa, R., et al. Inversion of a topological domain leads to restricted changes in its gene expression and affects inter-domain communication. Development. 149 (9), (2022).
- Savarese, F., Flahndorfer, K., Jaenisch, R., Busslinger, M., Wutz, A. Hematopoietic precursor cells transiently reestablish permissiveness for X inactivation. Molecular and Cellular Biology. 26 (19), 7167-7177 (2006).
- Schulz, E. G., et al. The two active X chromosomes in female ESCs block exit from the pluripotent state by modulating the ESC signaling network. Cell Stem Cell. 14 (2), 203-216 (2014).
- Gnirke, A., et al. Solution hybrid selection with ultra-long oligonucleotides for massively parallel targeted sequencing. Nature Biotechnology. 27 (2), 182-189 (2009).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Piccinini, F., Tesei, A., Arienti, C., Bevilacqua, A. Cell counting and viability assessment of 2D and 3D Cell cultures: Expected reliability of the trypan blue assay. Biological Procedures Online. 19 (1), 8(2017).
- Servant, N., et al. HiC-Pro: an optimized and flexible pipeline for Hi-C data processing. Genome Biology. 16, 259(2015).
- Abdennur, N., Mirny, L. A. Cooler: scalable storage for Hi-C data and other genomically labeled arrays. Bioinformatics. 36 (1), 311-316 (2020).
- Imakaev, M., et al. Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nature Methods. 9 (10), 999-1003 (2012).
- Forcato, M., et al. Comparison of computational methods for Hi-C data analysis. Nature Methods. 14 (7), 679-685 (2017).
- Venev, S., et al. open2c/cooltools: v0.4.1. , (2021).
- Wages, J. M. NUCLEIC ACIDS | Immunoassays. Encyclopedia of Analytical Science. , Elsevier. 408-417 (2005).
- Ewels, P. A., et al. The nf-core framework for community-curated bioinformatics pipelines. Nature Biotechnology. 38 (3), 276-278 (2020).
- Servant, N., Peltzer, A. nf-core/hic: Initial release of nf-core/hic. Zenodo. , (2019).
- Furlan, G., et al. The Ftx noncoding locus controls X chromosome inactivation independently of its RNA products. Molecular Cell. 70 (3), 462-472 (2018).
- Giorgetti, L., et al. Structural organization of the inactive X chromosome in the mouse. Nature. 535 (7613), 575-579 (2016).
- Simon, C. S., et al. Functional characterisation of cis-regulatory elements governing dynamic Eomes expression in the early mouse embryo. Development. 144 (7), 1249-1260 (2017).
- Williams, R. M., et al. Reconstruction of the global neural crest gene regulatory network in vivo. Developmental Cell. 51 (2), 255-276 (2019).
- Godfrey, L., et al. DOT1L inhibition reveals a distinct subset of enhancers dependent on H3K79 methylation. Nature Communications. 10 (1), 2803(2019).
- Hanssen, L. L. P., et al. Tissue-specific CTCF-cohesin-mediated chromatin architecture delimits enhancer interactions and function in vivo. Nature Cell Biology. 19 (8), 952-961 (2017).
- Larke, M. S. C., et al. Enhancers predominantly regulate gene expression during differentiation via transcription initiation. Molecular Cell. 81 (5), 983-997 (2021).
- Oudelaar, A. M., et al. A revised model for promoter competition based on multi-way chromatin interactions at the α-globin locus. Nature Communications. 10 (1), 5412(2019).
- Long, H. K., et al. Loss of extreme long-range enhancers in human neural crest drives a craniofacial disorder. Cell Stem Cell. 27 (5), 765-783 (2020).
- Schoenfelder, S., Javierre, B. -M., Furlan-Magaril, M., Wingett, S. W., Fraser, P. Promoter Capture Hi-C: High-resolution, genome-wide profiling of promoter interactions. Journal of Visualized Experiments. (136), e57320(2018).
- Siersbæk, R., et al. Dynamic rewiring of promoter-anchored chromatin loops during adipocyte differentiation. Molecular Cell. 66 (3), 420-435 (2017).
- Rubin, A. J., et al. Lineage-specific dynamic and pre-established enhancer-promoter contacts cooperate in terminal differentiation. Nature Genetics. 49 (10), 1522-1528 (2017).
- Freire-Pritchett, P., et al. Global reorganisation of cis-regulatory units upon lineage commitment of human embryonic stem cells. eLife. 6, 21926(2017).
- Javierre, B. M., et al. Lineage-specific genome architecture links enhancers and non-coding disease variants to target gene promoters. Cell. 167 (5), 1369-1384 (2016).
- Miguel-Escalada, I., et al. Human pancreatic islet three-dimensional chromatin architecture provides insights into the genetics of type 2 diabetes. Nature Genetics. 51 (7), 1137-1148 (2019).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Baxter, J. S., et al. Capture Hi-C identifies putative target genes at 33 breast cancer risk loci. Nature Communications. 9 (1), 1028(2018).
- Sanborn, A. L., et al. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes. Proceedings of the National Academy of Sciences. 112 (47), 6456-6465 (2015).
- Owens, D. D. G., et al. Dynamic Runx1 chromatin boundaries affect gene expression in hematopoietic development. Nature Communications. 13 (1), 773(2022).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved