Method Article
פענוח ארגון כרומטין תלת-ממדי ברזולוציה גבוהה באמצעות לכידת Hi-C
In This Article
Summary
פרוטוקול זה מתאר את שיטת Capture Hi-C המשמשת לאפיון הארגון התלת-ממדי של אזורים גנומיים ממוקדים בגודל מגה-מבוסס ברזולוציה גבוהה, כולל גבולות של תחומים משויכים טופולוגית (TADs) ואינטראקציות כרומטין ארוכות טווח בין אלמנטים רגולטוריים ורצפי DNA אחרים.
Abstract
הארגון המרחבי של הגנום תורם לתפקודו ולוויסותו בהקשרים רבים, כולל שעתוק, שכפול, רקומבינציה ותיקון. הבנת הסיבתיות המדויקת בין טופולוגיית הגנום לתפקוד היא אפוא חיונית והופכת יותר ויותר לנושא למחקר אינטנסיבי. טכנולוגיות לכידת קונפורמציה של כרומוזומים (3C) מאפשרות להסיק את המבנה התלת-ממדי של הכרומטין על ידי מדידת תדירות האינטראקציות בין כל אזור בגנום. כאן אנו מתארים פרוטוקול מהיר ופשוט לביצוע Capture Hi-C, שיטת העשרת מטרות מבוססת 3C המאפיינת את הארגון התלת-ממדי הספציפי לאלל של מטרות גנומיות בגודל מגה-מבוסס ברזולוציה גבוהה. ב- Capture Hi-C, אזורי המטרה נלכדים על ידי מערך של גשושיות ביוטיניליות לפני ריצוף במורד הזרם. לפיכך, רזולוציה גבוהה יותר וספציפיות אלל מושגות תוך שיפור יעילות הזמן והמחיר המשתלם של הטכנולוגיה. כדי להדגים את נקודות החוזק שלו, פרוטוקול Capture Hi-C הוחל על מרכז ההפעלה X של העכבר ( Xic), מוקד הבקרה הראשי של השבתת כרומוזום X (XCI).
Introduction
הגנום הליניארי מכיל את כל המידע הדרוש לאורגניזם כדי לעבור התפתחות עוברית ולשרוד לאורך כל הבגרות. עם זאת, הנחיית תאים זהים גנטית לבצע פונקציות שונות היא בסיסית לשליטה מדויקת במידע המשמש בהקשרים ספציפיים, כולל רקמות שונות ו / או שלבי התפתחות. הארגון התלת-ממדי של הגנום נחשב כמשתתף בוויסות מרחבי-זמני מדויק זה של פעילות הגנים על ידי הקלה או מניעה של אינטראקציה פיזית בין אלמנטים רגולטוריים שניתן להפריד על ידי כמה מאות קילו-בסיסים בגנום הליניארי (עבור סקירות 1,2,3). ב-20 השנים האחרונות, הבנתנו את יחסי הגומלין בין קיפול גנום ופעילות גדלה במהירות, בעיקר הודות לפיתוח טכנולוגיות ללכידת קונפורמציה של כרומוזומים (3C) (לסקירה 4,5,6,7). שיטות אלה מודדות את תדירות האינטראקציות בין אזורים כלשהם בגנום ומסתמכות על קשירת רצפי דנ"א הנמצאים בסמיכות תלת-ממדית בתוך הגרעין. פרוטוקולי 3C הנפוצים ביותר מתחילים בקיבוע אוכלוסיות תאים עם סוכן cross-linking כגון פורמלדהיד. הכרומטין הצולב, לאחר מכן מתעכל עם אנזים הגבלה, אם כי עיכול MNase שימש גם 8,9. לאחר העיכול, קצוות הדנ"א החופשי בקרבה מרחבית קרובים נקשרים מחדש, והקישור הצולב. שלב זה מוליד את "הספרייה" או "התבנית" של 3C, מאגר מעורב של מקטעים היברידיים שבו רצפים שהיו בקרבה תלת-ממדית לגרעין הם בעלי סיכוי גבוה יותר להיקשר באותו מקטע DNA. הכימות במורד הזרם של מקטעים היברידיים אלה מאפשר להסיק את הקונפורמציה התלת-ממדית של אזורים גנומיים הממוקמים אלפי זוגות בסיסים זה מזה בגנום הליניארי, אך עשויים לקיים אינטראקציה במרחב התלת-ממדי.
גישות רבות ושונות פותחו כדי לאפיין את ספריית 3C, הנבדלות זו מזו הן מבחינת תת-קבוצות של מקטעי קשירה מנותחות והן מבחינת הטכנולוגיה המשמשת לכימות במורד הזרם. פרוטוקול 3C המקורי הסתמך על בחירת שני אזורי עניין וכימות תדירות האינטראקציה שלהם 'אחד מול אחד' על ידי PCR10,11. גישת 4C (לכידת קונפורמציה מעגלית של כרומוזומים) מודדת את יחסי הגומלין בין מוקד עניין יחיד (כלומר, "נקודת המבט") לבין שאר הגנום ("אחד מול כולם")12,13,14. ב-4C, ספריית 3C עוברת סבב שני של עיכול וקשירה מחדש כדי ליצור מולקולות DNA מעגליות קטנות המועצמות ב-PCR על ידי פריימרים ספציפיים לנקודת מבט15. 5C (Chromosome Conformation Capture Carbon Copy) מאפשר אפיון של אינטראקציות תלת-ממדיות על פני אזורי עניין גדולים יותר, ומספק תובנות לגבי קיפול כרומטין מסדר גבוה יותר בתוך אזור זה ("רבים לעומת רבים")16. ב-5C, ספריית 3C עוברת הכלאה למאגר של אוליגונוקלאוטידים החופפים לאתרי הגבלה שניתן להגביר לאחר מכן על ידי מולטיפלקס PCR עם פריימרים אוניברסליים15. הן ב-4C והן ב-5C, מקטעי הדנ"א האינפורמטיביים כומתו בתחילה על ידי מיקרו-מערכים ומאוחר יותר על ידי ריצוף הדור הבא (NGS)17,18,19. אסטרטגיות אלה מאפיינות אזורי עניין ממוקדים, אך לא ניתן ליישם אותן כדי למפות אינטראקציות ברחבי הגנום. מטרה אחרונה זו מושגת באמצעות Hi-C, אסטרטגיה מבוססת 3C בעלת תפוקה גבוהה שבה ריצוף מקבילי מאסיבי של תבנית 3C מאפשר אפיון בלתי מוטה של קיפול כרומטין ברמת הגנום הרחב ('הכל לעומת כולם')20. פרוטוקול Hi-C כולל שילוב של שאריות ביוטינילציה בקצות השברים המעוכלים, ולאחר מכן משיכה כלפי מטה של מקטעי קשירה עם חרוזי סטרפטווידין כדי להגביר את ההתאוששות של שברים קשורים20.
Hi-C גילה כי גנומים של יונקים מאורגנים באופן מבני בקני מידה מרובים בגרעין התלת-ממדי. בסקאלת המגה-בסיס, הגנום מחולק לאזורים של כרומטין, כרומטין, תאים A ו-B, בהתאמה20,21. קיומם של תת-תאים נוספים המיוצגים על ידי מצבי כרומטין ופעילות שונים הוצג גם הוא לאחר מכן22. ברזולוציה גבוהה יותר, הגנום מחולק עוד יותר לתחומים תת-מגה-בסיסים בעלי אינטראקציה עצמית הנקראים תחומים שיוך טופולוגי (TADs), שנחשפו לראשונה על ידי ניתוח Hi-C ו-5C של גנום האדם והעכבר23,24. בניגוד לתאים המשתנים באופן ספציפי לרקמות, TADs נוטים להיות קבועים (אם כי ישנם יוצאים מן הכלל רבים). חשוב לציין, גבולות TAD נשמרים בין מינים25. בתאי יונקים, TADs כוללים לעתים קרובות גנים החולקים את אותו נוף רגולטורי והוכחו כמייצגים מסגרת מבנית המאפשרת ויסות משותף של גנים תוך הגבלת האינטראקציות עם תחומי בקרה שכנים (לסקירה 3,26,27,28). יתר על כן, בתוך TADs, אינטראקציות עקב אתרי CTCF בבסיס של לולאות cohesin-extruded עשוי להגדיל את ההסתברות של אינטראקציות מקדם-משפר או משפר משפר (לסקירה29).
ב- Hi-C, ניתן לזהות תאים ו- TADs ברזולוציה של 1 Mb עד 40 kb, אך ניתן להשיג רזולוציה גבוהה יותר כדי לאפיין מגעים בקנה מידה קטן יותר כגון אינטראקציות לולאה בין אלמנטים דיסטליים בקנה מידה של 5-10 kb. עם זאת, הגדלת הרזולוציה כדי להיות מסוגל לזהות לולאות כאלה ביעילות על ידי HiC דורש עלייה משמעותית בעומק הרצף, ולכן, עלויות רצף. זה מחמיר אם הניתוח צריך להיות ספציפי אלל. ואכן, עלייה של פי X ברזולוציה דורשת עלייה של X2 בעומק הריצוף, כלומר גישות ברזולוציה גבוהה וספציפיות לאלל יכולות להיות יקרות מאוד30.
כדי לשפר את העלות-תועלת והמחיר המשתלם תוך שמירה על רזולוציה גבוהה, ניתן למשוך פיזית את אזורי היעד המעניינים אותם מספריות 3C או Hi-C רחבות גנום לאחר ההכלאה שלהם עם בדיקות אוליגונוקלאוטידים משלימות המסומנות בביוטין לפני ריצוף במורד הזרם. אסטרטגיות העשרת מטרות אלה מכונות שיטות Capture-C ומאפשרות חקירה של אינטראקציות של מאות מיקומי מטרה הפזורים ברחבי הגנום (כלומר, Promoter Capture (PC) Hi-C; הדור הבא (NG) Capture-C; קלט נמוך (LI) לכידה-C; לכידת טיטרייטד גרעיני (NuTi)-C; Tri-C)31,32,33,34,35,36,37,38,39,40, או על פני אזורים המשתרעים על פני מספר מגה-בסיסים (כלומר, Capture HiC; HYbrid ללכוד Hi-C (Hi-C2); Tiled-C)41,42,43. שני היבטים יכולים להשתנות בשיטות מבוססות לכידה: (1) האופי והעיצוב של אוליגונוקלאוטידים ביוטיניליים (כלומר, RNA או DNA, אוליגוס יחיד לוכד מטרות גנומיות מפוזרות או אוליגוס מרובים המרצפות אזור עניין); ו-(2) התבנית המשמשת למשיכת מטרות כלפי מטה שיכולה להיות ספריית 3C או Hi-C, האחרונה מורכבת מקטעי הגבלה ביוטיניליים שנמשכו מספריית 3C.
כאן מתואר פרוטוקול Capture Hi-C המבוסס על העשרת אנשי קשר יעד מספריית 3C. הפרוטוקול מסתמך על תכנון מערך ריצוף מותאם אישית של בדיקות RNA ביוטינילציה וניתן לבצע אותו תוך שבוע אחד מהכנת ספריית 3C ועד לריצוף NGS. הפרוטוקול מהיר, פשוט, ומאפשר לאפיין ארגון תלת-ממדי מסדר גבוה יותר של אזורי עניין בגודל מגה-בסיס ברזולוציה של 5 קילו-בתים תוך שיפור יעילות הזמן והמחיר המשתלם בהשוואה לשיטות 3C אחרות. פרוטוקול Capture Hi-C הוחל על מוקד הבקרה הראשי של השבתת כרומוזום X (XCI), מרכז X-inactivation (Xic), המארח את ה-RNA הלא מקודד Xist. ה- Xic היה בעבר נושא לניתוחים מבניים ופונקציונליים נרחבים (לסקירה44,45). ביונקים, XCI מפצה על המינון של גנים מקושרי X בין נקבות (XX) לזכרים (XY) וכולל השתקת שעתוק של כמעט כל אחד משני כרומוזומי X בתאי נקבה. ה- Xic ייצג מוקד רב עוצמה ותקן זהב למחקרים בטופולוגיה גנומית תלת-ממדית וליחסי הגומלין עם בקרת גנים44. ניתוח 5C של Xic בתאי גזע עובריים של עכברים (mESCs) הוביל לגילוי ושיום של TADs, וסיפק את התובנות הראשונות לגבי הרלוונטיות התפקודית של חלוקה טופולוגית וויסות משותף של גנים24. הארגון הטופולוגי של ה-Xic הוכח לאחר מכן כמעורב באופן קריטי בתזמון ההתפתחותי המתאים של Xist upregulation ו-XCI 46, ואלמנטים לא חשודים של cis-regulatory שיכולים להשפיע על פעילות גנים בתוך ובין TADs התגלו לאחרונה גם בתוך Xic47,48,49. החלת Capture Hi-C על 3 Mb של כרומוזום X של העכבר המשתרע על פני Xic מדגימה את כוחה של גישה זו בניתוח קיפול כרומטין בקנה מידה גדול ברזולוציה גבוהה. פרוטוקול מפורט וקל למעקב מסופק, החל מתכנון מערך הגשושיות הביוטיניליות בכל אתר הגבלת DpnII באזור העניין ועד ליצירת ספריית 3C רחבת גנום, הכלאה ולכידת אנשי קשר מטרה, וניתוח נתונים במורד הזרם. כמו כן נכללת סקירה כללית של בקרות האיכות המתאימות והתוצאות הצפויות, והן נקודות החוזק והמגבלות של הגישה נדונות לאור שיטות קיימות דומות.
Protocol
תאי הגזע העובריים של העכבר (mESCs) ששימשו במחקר זה נגזרו מצלב של נקבת TX/TX R26 rtTA/rtTA 50 עם זכר Mus musculus castaneus בהתאם להנחיות הטיפול בבעלי חיים של מכון קירי (פריז)51.
1. תכנון בדיקה
- תכנן מערך של בדיקות ביוטינילציה (120-mer RNA oligonucleotides) המכסים את אזור היעד המעניין.
- ארחו את אזור העניין באוליגונוקלאוטידים חופפים כך שבממוצע כל רצף בתוך המטרה יכוסה על-ידי שתי גשושיות ייחודיות (כיסוי פי 2) (איור 1).
- אל תכלול רצפים חוזרים בכיסוי הבדיקה כדי למנוע העשרה של אינטראקציות לא ספציפיות.
הערה: כדי למקסם את ההעשרה של מקטעי קשירה אינפורמטיביים, הוגדרו אזורים המשתרעים על פני 300 bp במעלה ובמורד הזרם של כל אתר הגבלת DpnII ברחבי היעד (ChrX: 102,475,000-105,475,000), ו-28,913 בדיקות ביוטינילציה תוכננו בהתאם לטכנולוגיית העשרת מטרות הדנ"א של SureSelect באמצעות פלטפורמת Sure Design52. על פי אסטרטגיה זו, עד מקסימום של 40 בסיסים של רצפים חוזרים מותרים בכל אוליגונוקלאוטיד כדי למזער את העשרה של אינטראקציות לא ספציפיות. מערך הגשושיות סונתז על ידי Agilent. כאן, DpnII משמש כאנזים הגבלה משתי סיבות: (1) זהו חותך ארבע המשמש באופן שגרתי במספר שיטות מבוססות 3C53; ו-(2) הוא ממקסם את הסיכויים ללכוד פולימורפיזמים אינפורמטיביים של נוקלאוטידים בודדים (SNPs) בקרבת אתרי החיתוך בהשוואה לאנזימי הגבלה אחרים שנבדקו בסיליקו בקווים היברידיים של F1 ששימשו במחקר זה (C57BL/6J x CASTEi/J).
2. הליך ניסיוני
- הכנת תאים
- זרעו את מספר התאים המתאים על צלחת תרבית תאים אחת או יותר כדי להשיג מספר תאים כולל של ≥ 5 x 107 תאים ביום הקיבוע.
הערה: במחקר זה נעשה שימוש בתאי גזע עובריים של עכברים (mESCs). mESCs מצופים על לוחות תרבית תאים מסוג Gelatinized (0.1% ג'לטין ב-1x PBS - o/n ב-37°C, 5% CO2 אינקובטור) בלוחות mESCs בינוניים המכילים 2i + LIF וסרום עגל עוברי שנבדק באצווה (DMEM, 15% FBS, 0.1 mM β-מרקפטואתנול, 1,000 U/mL−1 גורם מעכב לוקמיה (LIF), CHIR99021 (3 מיקרומטר) ו-PD0325901 (1 מיקרומטר)). עבור סוג תא זה, צלחת אחת של 80% בקוטר 10 ס"מ מכילה כ-2 x 107 תאים. - הכינו צלחת תרבית תאים אחת נוספת לספירת תאים.
הערה: ניתן להשתמש בלוח תרבית תאים קטן יותר כדי להפחית את השימוש במדיה. במקרה זה, מספר התאים שיש לזרוע בצלחת הקטנה יותר צריך להיות מותאם בהתאם (למשל, פי 3 פחות תאים על צלחת של 10 ס"מ לעומת צלחת של 15 ס"מ).
- זרעו את מספר התאים המתאים על צלחת תרבית תאים אחת או יותר כדי להשיג מספר תאים כולל של ≥ 5 x 107 תאים ביום הקיבוע.
- קיבוע פורמלדהיד
- הערך את מספר התאים הכולל שיש להצליב.
- לפני תחילת תגובת הקישור הצולב, טריפסין ולספור תאים מלוח הבקרה שהוכן במיוחד לספירת תאים באמצעות מונה תאים אוטומטי בהתאם להוראות היצרן.
- כלול צביעת כדאיות (למשל, כחול טריפאן) כדי לקבוע את אחוז התאים בני קיימא54. מתוך ספירת תאים זו, הערך את המספר הכולל של תאים בלוחות שהוכנו לקישור צולב.
- מוציאים את מדיום התרבית מהלוחות שהוכנו להצלבה ומחליפים אותו בכמות מתאימה של תמיסת קיבוע (2% פורמלדהיד בתווך תרבית תאים). השתמש 10 מ"ל על צלחת 10 ס"מ (למשל, ~ 20 מ"ל עבור צלחת 15 ס"מ).
הערה: הוסף נפח מדויק של תמיסת קיבוע. אם תיקון תאים דבקים אינו אפשרי, שלב זה יכול להיות מותאם לתאים טריפסיניים ולבצע 30 מ"ל של פתרון קיבוע בצינורות צנטריפוגה חרוטית 50 מ"ל. פורמלדהיד לא חייב להיות בן יותר משנה. עדיף להשתמש בקבוקונים חד פעמיים. יש להביא את תמיסת הקיבוע לטמפרטורת החדר (RT) לפני השימוש.
אזהרה: פורמלדהיד הוא מסוכן ויש לטפל בו בהתאם לתקנות הבריאות והבטיחות המתאימות. - תקן במשך 10 דקות ב- RT תחת ערבוב עדין על שייקר.
- להרוות את תגובת הקיבוע על ידי תוספת של 2.5 M גליצין-1x PBS לריכוז סופי של 0.125 M. הוסף 530 μL של 2.5 M גליצין-1x PBS ל 10 מ"ל על צלחת 10 ס"מ (למשל, 1060 μL עד 20 מ"ל על צלחת 15 ס"מ).
הערה: אם התאים היו קבועים בתמיסה, להרוות את תגובת הקיבוע עם 1590 μL של 2.5 M גליצין-1x PBS. - דוגרים במשך 5 דקות ב-RT, מערבבים בעדינות על שייקר.
- מעבירים את הצלחות לקרח ודגרים על קרח במשך 15 דקות נוספות תוך ערבוב עדין על שייקר.
הערה: מעתה ואילך, התאים חייבים להישמר על קרח, ומאגרים חייבים להיות מקוררים מראש כדי למנוע קישורים צולבים נוספים. עברו לחדר קר אם יש צורך לעבד צלחות רבות. - הסר את תמיסת הקיבוע מהתאים על ידי מזיגה לתוך כדי להבטיח טיפול מהיר.
הערה: יש להקפיד להשליך את הפסולת הנוזלית המכילה פורמלדהיד בהתאם לתקנות הבריאות והבטיחות המתאימות. - שטפו את הצלחת בקוטר 10 ס"מ במהירות פעמיים עם 5 מ"ל גליצין-1x PBS קר בקוטר 0.125 מ"ל (8 מ"ל לצלחת בקוטר 15 ס"מ) כדי לשטוף את הלכלוך והתאים המתים. הוציאו את הנוזל מהצלחת על ידי מזיגה לתוך כדי להבטיח טיפול מהיר.
- הוסף 5 מ"ל גליצין-1x PBS קר 0.125 מ"ל לצלחת בקוטר 10 ס"מ (10 מ"ל לצלחת בקוטר 15 ס"מ) וגרדו במהירות את התאים מהצלחת באמצעות מגרד תאי פלסטיק.
- מעבירים את מתלה התא לצינור צנטריפוגה חרוטי מקורר מראש של 50 מ"ל באמצעות פיפטה סרולוגית.
- שטפו את הצלחת פעמיים עם 5 מ"ל של גליצין-1x PBS קר 0.125 M והוסיפו את תרחיף התא לצינור הצנטריפוגה החרוטית.
- סחרור כלפי מטה ב 480 x גרם במשך 10 דקות ב 4 ° C.
הערה: אם התאים היו קבועים בתמיסה, העבר את התא לצינור צנטריפוגה חרוטי מקורר מראש וסובב כלפי מטה ב 480 x גרם במשך 10 דקות ב 4 ° C. הסר את תמיסת הקיבוע על ידי מזיגה לתוך ושטוף שלוש פעמים ב 10 מ"ל של קר 0.125 M גליצין-1x PBS. הקפידו להשהות מחדש את התאים בכל שלב כביסה. - הסר את supernatant על ידי שאיפה עם מערכת שאיפות benchtop. להשהות מחדש את התאים ב 500 μL של 1x PBS לכל 1 x 107 תאים על ידי pipeting בזהירות למעלה ולמטה עם פיפטה P1000. כדי להשעות מחדש תאים באמצעי האחסון המדויק, עיין בהערכת מספר התאים הכולל המתקבל ב- 2.2.1.
- Aliquot 500 μL של תרחיף התא למספר המחושב של 1.5 מ"ל צינורות מיקרוצנטריפוגות (1 x 107 תאים / צינור).
- סחרור כלפי מטה ב 480 x גרם במשך 10 דקות ב 4 ° C.
- מוציאים את הסופרנאטנט עם מערכת שאיפת ספסל ומקפיאים את כדורי התא בחנקן נוזלי. אחסנו את כדורי התא היבש בטמפרטורה של -80°C.
הערה: ניתן לאחסן דוגמאות למשך שנה אחת לפחות.
- הערך את מספר התאים הכולל שיש להצליב.
- ליזה של תאים
- הפשירו את הכדוריות הקפואות על קרח.
- הכן 1.5 מ"ל של חיץ ליזיס ב- H 2 0 לכל דגימה: הוסף 10 mM Tris-HCl, pH 8.0, 10 mM NaCl ו-0.2% NP40.
- מוסיפים 600 μL של חיץ הליזיס הקר ומרחפים היטב על קרח.
- דגרו על קרח במשך 15 דקות כדי לתת לתאים להתנפח.
- יש לסחרר כלפי מטה במהירות של 2655 x גרם למשך 5 דקות ב-4°C ולהסיר את הסופרנאטנט באמצעות מערכת שאיפות ספסל.
- כדי להסיר לכלוך, להשהות מחדש את הגלולה ב 1 מ"ל של חיץ ליזה קר, להסתובב למטה ב 2655 x גרם במשך 5 דקות ב 4 °C, ולהסיר את supernatant.
- סובב שוב תוך זמן קצר במהירות של 2655 x גרם ו -4 ° C והסר כמה שיותר מהסופרנאטנט שנותר באמצעות מערכת שאיפות benchtop המצוידת בקצה P200.
- השהיה מחדש ב-100 μL של 0.5% (vol/vol) SDS.
- דוגרים בתרמומיסר ב-62°C, מסתחררים ב-1400 סל"ד למשך 10 דקות.
- מוסיפים 290 μL של H2O + 50 μL של 10% TritonX-100 ומערבבים היטב, תוך הימנעות מבועות אוויר.
- דוגרים בתרמומיסר ב-37°C, מסתחררים ב-1400 סל"ד למשך 15 דקות.
- הוסף 50 μL של 10x Dpnll buffer והפוך את הצינור כדי לערבב.
- קח 50 μL של DNA מעוכל לבקרת איכות לתוך צינור נפרד. אל תשכח לקחת את דגימת הבקרה מעוכל.
- עיכול DpnII
- מוסיפים 10 μL של Dpnll בריכוז גבוה (500 U בסך הכל) ומערבבים על ידי היפוך.
- דוגרים על הדגימות והבקרה הלא מעוכלת בתרמומיקסר ב-37°C, מסתחררים ב-1400 סל"ד במשך >4 שעות.
- הוסף 10 μL של ריכוז גבוה Dpnll (500 U בסך הכל) בסוף היום.
- לדגור על הדגימות ועל הבקרה הלא מעוכלת ב-37 מעלות צלזיוס, ולהסתחרר ב-1400 סל"ד בן לילה.
- הוסף 10 μL של ריכוז גבוה Dpnll (500 U סה"כ) בתחילת היום הבא לדגימות.
- לדגור את הדגימות ואת הבקרה מעוכל ב thermomixer ב 37 ° C, מסתחרר ב 1400 סל"ד במשך 4 שעות.
- קשירה והיפוך של cross-linking
- לדגור את הצינורות ב 65 ° C במשך 20 דקות ב 1400 סל"ד.
הערה: אל תוסיף SDS בשלב זה. הרעיון הוא לשמור על שלמות הגרעין, כך שהקשירה מתבצעת בתוך הגרעינים, תוך עקיפת הצורך בדילול קיצוני. - מצננים את הדגימות על קרח למשך 5-10 דקות לכל היותר. כדי למנוע משקעים SDS, לא להשאיר את הדגימות על קרח יותר מזה.
- קח 50 μL של DNA מעוכל unligated לבקרת איכות בצינור נפרד. אחסן את הפקדים הלא מעוכלים והלא קשורים בטמפרטורה של -20°C.
הערה: אל תשכח לקחת את דגימת הבקרה הלא קשורה. - הוסף 800 μL של קוקטייל קשירה: 122 μL של 10x ligase buffer, 8 μL של T4 ligase (30 U/μL), ו 670 μL של H20.
- דוגרים ב-16°C, מסתחררים ב-1000 סל"ד במהלך הלילה.
- הוסף 7.5 μL של פרוטאינאז K (20 מ"ג / מ"ל) לדגימות ו -2 μL לבקרות.
- דגירה ב-65°C למשך 4 שעות ב-1000 סל"ד.
- לדגור את הצינורות ב 65 ° C במשך 20 דקות ב 1400 סל"ד.
- טיהור DNA
- העבירו את הדגימות על קרח לצינורות צנטריפוגות חרוטיות מקוררות מראש של 15 מ"ל והוסיפו 2 מ"ל מים, 10.5 מ"ל של EtOH קר כקרח, ו-583 מיקרוליטר של 3 מ"ל NaAC.
הערה: מים נוספים נועדו למנוע נשיאה של DTT לתוך הכדורית. - הוסף 200 μL של EtOH קר כקרח, 10.8 μL של NaAC, ו 1 μL של המשקע לבקרות איכות לא מעוכלות ולא מחויבות.
- יש לדגור בטמפרטורה של -80°C למשך 4 שעות לפחות עד הלילה.
- סובב את צינורות 15 מ"ל ב 2200 x גרם ב 4 ° C במשך 45 דקות.
- סובב את צינורות הבקרה של 1.5 מ"ל ב- 20,500 x גרם ב- 4 ° C למשך 30 דקות.
- יש לשטוף פעם אחת עם 3 מ"ל (דגימות) ו-1 מ"ל (בקרות) של 70% EtOH קר כקרח.
- סבב ב- 2200 x גרם (דגימות) או 20,500 x גרם (פקדים) ב- 4 ° צלזיוס למשך 10 דקות.
- בזהירות להסיר EtOH ולייבש באוויר ב RT במשך 10-15 דקות; אין לייבש יתר על המידה.
- השהה מחדש את הדגימות והבקרות ב- 100 μL ו- 20 μL של H20, בהתאמה.
- הוסף 1 μL של RNAseA ודגור ב 37 ° C, מסתחרר ב 1400 סל"ד במשך 30 דקות.
- העבירו את הדגימות על קרח לצינורות צנטריפוגות חרוטיות מקוררות מראש של 15 מ"ל והוסיפו 2 מ"ל מים, 10.5 מ"ל של EtOH קר כקרח, ו-583 מיקרוליטר של 3 מ"ל NaAC.
- בקרת איכות של הכנת תבנית 3C
- כמת כל דגימה ובקרה באמצעות ערכת פלואורומטר למדידות ריכוז DNA ברגישות גבוהה.
- יש להעמיס 100-200 ננוגרם מכל דגימה וכל בקרה על ג'ל TBE 1% אגרוז/1x.
- ודאו שתמונת הג'ל מציגה את התוצאה הצפויה על-ידי השוואת ההבדלים בגדלים של מקטעי דנ"א של הפקדים ותבנית 3C כפי שמוצג באיור 2A.
- אחסן את הדגימות והפקדים ב -20 °C.
- הכלאה, לכידה ועיבוד דגימה לריצוף מרובה
- כדי להכליא את מערך בדיקות הרנ"א הביוטינילציה לתבנית 3C, ללכוד את מקטעי הקשירה הממוקדים, ולהכין את הדגימות לריצוף מרובב בהתאם למערכת העשרת המטרה המשמשת במחקר זה לריצוף מרובב זוגות (ראו טבלת חומרים). עקוב אחר הפרוטוקול בהתאם להוראות היצרן תוך הצגת השינויים הקלים הבאים:
- סעיף 2 לפרוטוקול היצרן: הכנת דוגמאות
- בצע את ההוראות להעשרת מטרה החל מ- 3 מיקרוגרם של קלט gDNA.
- גזירת הדנ"א בסוניקטור באמצעות המפרטים הבאים: מחזור עבודה של 10%, 4 עוצמה, 200 מחזור/פרץ ו-130 שניות. התחל עם 4 מיקרוגרם של תבנית 3C התלויה מחדש ב -130 מיקרוליטר מים עבור כל תגובת לכידה כדי להבטיח מספיק חומר כדי להמשיך את הכנת הדגימה עם 3 מיקרוגרם של הדנ"א הגזוז.
- להעריך את איכות הדנ"א החתוך. הפעל 1 μL של הדנ"א החתוך על ביואנלייזר DNA בהתאם לפרוטוקול רגישות גבוהה. צפו להתפלגות של גודל המקטע בין 150 ל-700 bp (איור 2).
- לטהר את הדגימה באמצעות חרוזי אימוביליזציה הפיכה בשלב מוצק (SPRI). הוסף 124 μL של חרוזי SPRI ל- 124 μL של דגימת ה- DNA כדי לבצע בחירה בגודל צד שמאל ביחס של 1:1 בהתאם להוראות היצרן ולנטוש 25 μL של מים נטולי נוקלאז. שלב הטיהור הזה יסיר מקטעים קצרים יותר כדי להעשיר שברים של כ-300 bp (איור 2).
הערה: כמות הדגימות וחרוזי SPRI המשמשים בשלב זה לוקחים בחשבון את אובדן הנפח שהתרחש בעת העברת הדגימות לצינורות חדשים והפעלת בקרות האיכות בביואנלייזר. כל שלבי בחירת הגודל הבאים מבוצעים בהתאם ליחסים המומלצים על ידי פרוטוקול היצרן. פליטת DNA מחרוזי SPRI מבוצעת ב-RT לאורך כל הפרוטוקול. - הערך את האיכות של DNA גזוז שנבחר בגודל. הפעל 1 μL של הדנ"א החתוך על הביואנלייזר של הדנ"א בהתאם לפרוטוקול רגישות גבוהה (HS). צפו להתפלגות של גדלי מקטעים עם ההעשרה הגבוהה ביותר של 300 bp (איור 2). המשך לכימות הדנ"א הגזור אם הגזירה הצליחה.
- כמת את הדנ"א החתוך באמצעות ערכת פלואורומטר למדידות ריכוז DNA HS.
הערה: אם גזירת דנ"א מביאה לתפוקת דנ"א של <3 מיקרוגרם, בצע סבב שני של גזירת דנ"א עם עוד 4 מיקרוגרם דנ"א ושלב את דגימות הדנ"א שנחתכו לאחר שלב טיהור חרוזי SPRI הראשון כדי להשיג סך של 3 מיקרוגרם דנ"א גזוז. - הוסף מים נטולי נוקלאז לדגימת ה- DNA הנקי בגודל שנבחר (3 מיקרוגרם בסך הכל) לנפח סופי של 48 μL והמשך בתגובת התיקון הסופית בהתאם לפרוטוקול היצרן.
- לאחר קשירת מתאמים זוגיים, הגבירו את הספרייה על ידי ביצוע חמישה מחזורים של PCR טרום לכידה בהתאם להוראות היצרן (התנאים ל-PCR והפריימרים מסופקים בערכה).
- סעיף 4 לפרוטוקול היצרן: הכלאה ולכידה
- כדי להכליא את דגימות הדנ"א המוכנות לגשושיות הרנ"א הספציפיות למטרה, יש לדלל 750 ננוגרם של דגימות דנ"א בנפח סופי של 3.4 מיקרוליטר, והתוצאה היא ריכוז התחלתי של 221 ננוגרם/מיקרוליטר. עבור דגימות DNA מדוללות בנפחים גדולים יותר, השתמש ברכז ואקום מהיר כדי להפחית לנפח הסופי. ריכוז ואקום מהיר (250 x גרם; ≤45 ° C) למשך 15-20 דקות מספיק בדרך כלל לדגימות המושעות מחדש ב -10 μL. ודא שיש לך נפח קלט זהה עבור כל דגימה לפני הפעלת רכז ואקום מהיר.
- יש לדגור על תערובת ההכלאה למשך 16-18 שעות ב-65°C עם מכסה מחומם ב-105°C בהתאם להוראות היצרן.
- סעיף 5 בפרוטוקול היצרן: יצירת אינדקס ועיבוד דגימות לריצוף מרובה
- כדי להגביר את הספריות שנתפסו עם פריימרים לאינדקס, בצע 12 מחזורים של PCR לאחר לכידה בהתאם להוראות היצרן (התנאים ל- PCR והפריימרים מסופקים בערכה).
- סעיף 2 לפרוטוקול היצרן: הכנת דוגמאות
- כדי להכליא את מערך בדיקות הרנ"א הביוטינילציה לתבנית 3C, ללכוד את מקטעי הקשירה הממוקדים, ולהכין את הדגימות לריצוף מרובב בהתאם למערכת העשרת המטרה המשמשת במחקר זה לריצוף מרובב זוגות (ראו טבלת חומרים). עקוב אחר הפרוטוקול בהתאם להוראות היצרן תוך הצגת השינויים הקלים הבאים:
- ריצוף הדור הבא
- כדי להפעיל ספריות Hi-C מרובות של לכידה על אותו תא זרימה, הכינו תערובת שווה ערך של ספריות הלכידה ורצף קריאות של 100-120 M לכל ספרייה.
- אם יש צורך בניתוח ספציפי לאלל, רצף 150 bp paired-end כדי להבטיח כיסוי SNP מספיק.
3. ניתוח נתונים
- החל את צינור HiC-Pro כדי לבצע ניתוח נתוני Hi-C55. HiC-Pro מספק בקרות איכות בכל שלב של העיבוד, כולל (איור 3):
(ט) קצב היישור בגנום הייחוס המציין את שבר הקריאות המתפרש על פני אתר קשירה, כמו גם את מספר הזוגות והסינגלטונים.
(ב) החלק של מוצרי קשירה תקפים וזוגות קריאה לא אינפורמטיביים (קצה משתלשל, קשירה עצמית וכו ').
(ג) החלק של מגעים קצרים/ארוכי טווח ותוך/בין-כרומוזומליים.
(ד) החלק המרבי של אנשי קשר ממוקדים עבור Capture Hi-C.
(v) החלק של אלל ספציפי קורא אם צוין.
הערה: HiC-Pro תומך במגוון רחב של פרוטוקולים, כולל Hi-C באתרו ו-Capture Hi-C. במקרה האחרון, המשתמש פשוט צריך לציין את אזור היעד (פורמט BED) בקובץ התצורה. לאחר עיבוד הנתונים, ניתן להמיר בקלות את יציאות HiC-Pro לאובייקט קריר יותר לניתוח במורד הזרם56. בשלב זה, מפות אנשי הקשר ברזולוציות שונות מנורמלות באמצעות שיטת ICE שתוארה בעבר על ידי Imakaev ועמיתיו57. לאחר מכן ניתן להריץ מספר ניתוחים כדי לקרוא לתאי כרומוזומים, TADs, או לולאות כרומטין (לסקירה58). זרימת העבודה של הפרוטוקול מוצגת באיור 4. כאן, חבילת 'cooltools' מיושמת כדי לחשב את ציון הבידוד ואת גבולות TADs, כפי שמודגם באיור 5 ובאיור 659.
תוצאות
פרוטוקול Capture Hi-C המתואר מבוסס על הכנת תבנית 3C רחבת גנום באמצעות חותך בעל ארבעה בסיסים (DpnII). העשרה מאוחרת יותר של מקטעי קשירה על פני האזור הגנומי המעניין מתקבלת על-ידי הכלאה של מערך של גשושיות RNA מרוצפות ולכידת הסטרפטווידין שלהן בהתאם למערכת העשרת המטרה ששימשה במחקר זה (איור 1). בדיקות RNA ביוטינילציה נבחרו מכיוון שהן מראות זיקה הדוקה יותר למטרותיהם בהשוואה לגשושיות DNA52,60. לאחר מכן, ספריות שנלכדו נכללות באינדקס ומאוגדות לצורך ריצוף מרובה בתפוקה גבוהה. ניתן להציג באופן חזותי נתוני Hi-C ברזולוציה גבוהה כמפות אינטראקציה Hi-C ברזולוציה גבוהה, אך גם כמפות קשר בודדות דמויות 4C כדי להמחיש באופן ספציפי את האינטראקציות של רצפים קטנים יותר, כגון מקדמים או משפרים בתוך האזור כולו שנלכד. זרימת העבודה של הפרוטוקול מוצגת באיור 4. בקרות איכות קדם-ריצוף מוצגות באיור 2 וכוללות הערכה של עיכול תקין וקשירה מחדש של תבנית 3C וגזירה וטיהור יעילים שלה לאורך השלבים השונים של הפרוטוקול. הדנ"א של תבנית 3C צפוי לרוץ בין 150 ל-700 bp, ואין לזהות העשרה של מקטעים >2 קילובייט. במהלך השלבים הבאים, מבוצעים מספר שלבי ניקוי דנ"א מבוסס חרוזים ובחירת גודל, תחילה לאחר הגזירה, ולאחר מכן לאחר ה-PCR לפני הלכידה ולאחר הלכידה. ספריות מנוקות מציגות פרופיל העשרת מקטעים מובהק כפי שניתן לראות בביואנלייזר דנ"א בעל רגישות גבוהה (איור 2). גודל המקטע הממוצע גדל במהלך הכנת הספרייה עקב קשירת מתאמים, ריצוף ופריימרים לאינדוקס. בקרות איכות לאחר ריצוף מתקבלות באמצעות Hi-C Pro ומוצגות באיור 3. יישומי תוכנה ביואינפורמטיים רבים ושונים הוצעו לעיבוד וניתוח נתונים דמויי 3C. ביניהם, צינור HiC-Pro הוא אחד הפתרונות הפופולריים ביותר, המאפשר עיבוד של נתוני רצף גולמיים למפות הקשר הסופיות ברזולוציות שונות55. HiC-Pro משתמש באסטרטגיית מיפוי דו-שלבית כדי ליישר את קריאות הריצוף בגנום הייחוס. לאחר מכן מוצרי 3C משוחזרים ומסוננים כדי להסיר זוגות אנשי קשר שאינם אינפורמטיביים וליצור את מפות אנשי הקשר. בנוסף, הוא מסוגל להשתמש ברשימה של פולימורפיזמים ידועים כדי לבצע אנליזה ספציפית לאלל ולהפריד את המגעים המגיעים משני האללים ההוריים במפות קשר נפרדות. לאחרונה, HiC-Pro נכלל והורחב למסגרת nf-core (nf-core-hic), ומספק צינור מונחה קהילה61,62 הניתן להרחבה ולשחזור.
כדי ללכוד את העכבר Xic, תוכנן מערך של 28,913 גשושיות RNA המרצפות 3 Mb של כרומוזום X. אזור זה כולל את שחקן המפתח ב-XCI, הגן הארוך שאינו מקודד Xist, ואת הנוף הרגולטורי הידוע שלו ~800 קילו-בתים (איור 5). אזור ~ 800 kb זה מחולק לשני TADs: האחד כולל את מקדם Xist והרגולטורים החיוביים הידועים שלו (כלומר, התעתיקים שאינם מקודדים Ftx, Jpx ו- Xert והגן המקודד לחלבון Rnf12), וה- TAD השכן הכולל את מווסתי cis השליליים של Xist (כלומר, תעתיק האנטיסנס שלו Tsix, אלמנט המשפר Xite, ואת התמליל שאינו מקודד Linx) (לסקירה44, 45).
על ידי יישום פרוטוקול Capture Hi-C המתואר על Xic, הארגון הטופולוגי של מוקד זה התקבל ברזולוציה חסרת תקדים (איור 6 ואיור 7). הדבר בולט במיוחד כאשר משווים את פרופיל Capture Hi-C ל-5C47 שפורסם בעבר (איור 6 ואיור 7; טבלה משלימה 1) ו-Hi-C61 (איור 6 ואיור 7; טבלה משלימה 1) פרופילים. לדוגמה, מבני תת-TAD ברורים יותר – ה-TAD המכיל את מקדם Xist (Xist-TAD) מחולק בבירור לשני תחומים קטנים יותר (איור 6A, ראש חץ כחול). בעבר, ניתן היה "לנחש" זאת באופן חזותי רק מפרופיל 5C (איור 6B), אם כי זיהוי גבול באזור זה באמצעות אלגוריתם ציון הבידוד. כמו כן, הרזולוציה של פרופיל Capture Hi-C מאפשרת זיהוי של שני תחומים קטנים יותר ב-TAD השכן (איור 6A, B), המכיל את מקדם לוקוס Tsix (Tsix-TAD ); זה לא הושג קודם לכן עם 5C (איור 6B). יש לציין כי גבולות טופולוגיים הנקבעים על ידי ציון הבידוד מנתוני Capture Hi-C ו- 5C מזוהים בדרך כלל במקומות שונים במקצת ובעוצמות יחסיות שונות.
יתר על כן, מבנים תת-TAD אחרים כגון לולאות מגע נראים בבירור מנתוני Capture Hi-C, כגון הלולאה בין Xist ו-Ftx (איור 7A), שזוהתה בעבר עם Capture-C63, והלולאה בין Xist ל-Xert (איור 7B), שזוהתה לאחרונה באמצעות פרוטוקול דומה עבור Capture Hi-C48. אנשי קשר אחרים יכולים גם להיות ממופים בצורה מדויקת יותר הודות לרזולוציה המוגברת של פרופילי Capture Hi-C, כגון אלה שיוצרים את נקודות המגע החמות הידועות בתוך Tsix-TAD בין אתרי Linx, Chic1 ו-Xite (איור 7A).
בהשוואה לנתוני Hi-C המוצגים באיור 7, Capture Hi-C איפשר עלייה של פי ארבעה ברזולוציה, אך הוא דרש רק רבע מעומק הרצף (כלומר, 126 M קריאות לעומת 571 M) (טבלה משלימה 1). עלייה זו ברזולוציה מאפשרת זיהוי של תת-TADs ואינטראקציות לולאה שלא ניתן היה לזהות על-ידי Hi-C בעומק הרצף המוצג באיור 6 ובאיור 7. הפרוטוקול המתואר עבור Capture Hi-C מאפשר אפוא אפיון מפורט הרבה יותר, ברזולוציה גבוהה של אזור גנומי גדול של עניין, בהשוואה לגישות קודמות.
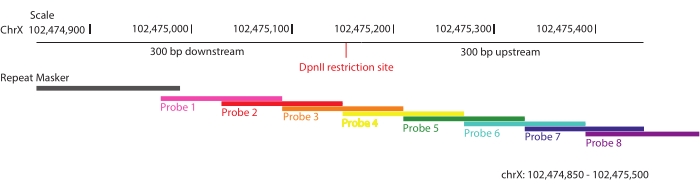
איור 1: תכנון החלושית. ייצוג סכמטי של האסטרטגיה המשמשת לתכנון הגשושית. אזורים של 300 bp במעלה ובמורד הזרם של כל אתר הגבלה DpnII על פני אזור היעד של 3 Mb נבחרו ורוצפו בבדיקות RNA חופפות ביוטינילציה. אחד מהאזורים שנבחרו אלה מוצג, chrX: 102,474,805-102,475,500. לא יותר מ-40 בסיסים של רצפים חוזרים מותרים בכל גשוש. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
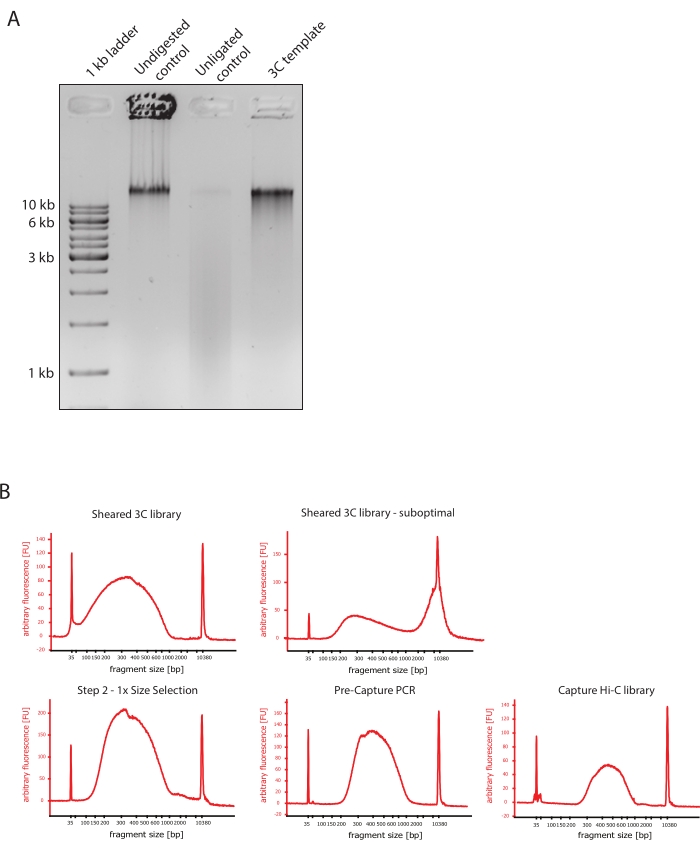
איור 2: לכידת בקרות איכות קדם-ריצוף Hi-C . (A) דוגמה מייצגת לבקרות איכות של תבנית 3C. 200 ננוגרם של DNA הועמסו על ג'ל אגרוז 1%. נתיב 1: סולם 1 קילובייט. נתיב 2: כרומטין לא מעוכל, מוצלב ושלם פועל כרצועה חדה במהירות של >10 קילובייט. נתיב 3: כרומטין צולב מעוכל DpnII פועל ככתם בגודל שבין 1 KB ל -3 kb. נתיב 4: ספריית 3C סופית או תבנית; קצוות חופשיים של מקטעי דנ"א צולבים מעוכלים נקשרו מחדש. כתם הדנ"א בגודל מולקולרי נמוך יותר כמעט בלתי ניתן לגילוי, ותוצר הקשירה מזוהה כרצועה של >10 קילובייט. (B) דוגמאות מייצגות של פרופילי DNA של ביואנלייזר בעל רגישות גבוהה. משמאל למעלה: ספריית 3C שנחתכה בהצלחה ומציגה התפלגות של גודל הקטע בין 150 bp ל- 700 bp. מימין למעלה: ספריית 3C גזוז לא משביעת רצון. DNA לא גזוז מזוהה כהעשרה רחבה של מקטעים >2 קילובייט. (C) משמאל למטה: דגימת דנ"א גזורה לאחר בחירה בגודל צד שמאל ביחס של 1:1 באמצעות חרוזי SPRI. שברים של ~ 300 bp מועשרים. אמצע תחתון: פרופיל PCR טרום לכידה לאחר קשירה של מתאמים משויכים בהתאם לפרוטוקול היצרן. מימין למטה: ספריית Capture Hi-C סופית כולל מתאמים, ריצוף ופריימרים ליצירת אינדקס לריצוף מרובה. קיצורים: bp = זוגות בסיסים, FU = יחידת פלואורסצנטיות שרירותית. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
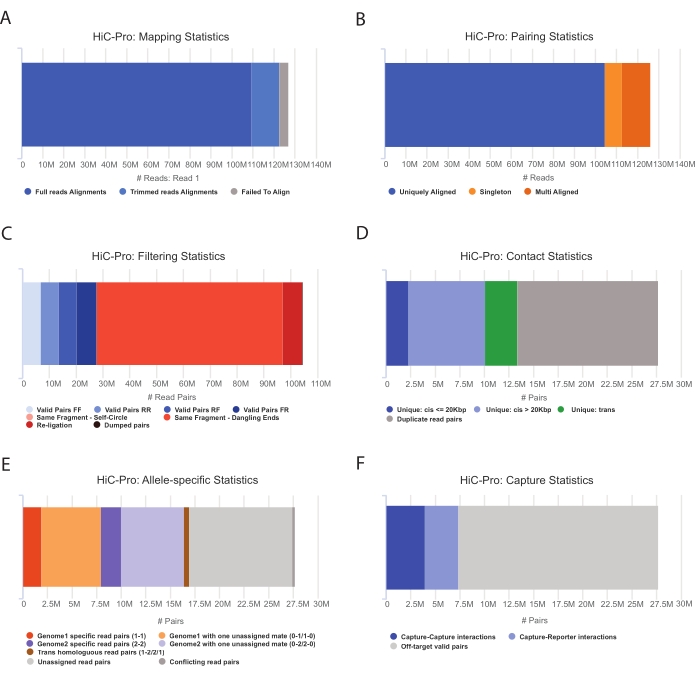
איור 3: לכוד בקרות איכות Hi-C לאחר ריצוף באמצעות HiC-Pro . (A) דוגמה לקצב מיפוי בגנום הייחוס עבור בן הזוג הראשון של זוגות הריצוף. החלק הכחול הבהיר מייצג את הקריאות המיושרות על ידי HiC-Pro ומתפרשות על פני צומת קשירה. לפיכך ניתן להשתמש במדד זה כדי לאמת את שלב הקשירה הניסיוני. (B) ברגע שבני זוג מרוצפים מיושרים על הגנום, רק זוגות קריאה מיושרים באופן ייחודי נשמרים לניתוח. (C) זוגות לא חוקיים (באדום) כגון קצה משתלשל, עיגול עצמי או קשירה מחדש נמחקים מהניתוח. שבר הזוגות החוקיים הוא אינדיקטור טוב ליעילות הקשירה והמשיכה כלפי מטה. (D) ניתן לחלק את הזוגות התקפים למגעים תוך-כרומוזומליים וקצרים/ארוכי טווח. זוגות קריאה משוכפלים שעשויים לייצג תוצרי PCR מושלכים מהניתוח. (E) עבור ניתוח ספציפי לאלל, HiC-Pro מדווח על מספר הקריאות האלליות הנתמכות על ידי בן זוג אחד או שניים עבור כל גנום הורי (כלומר, C57BL/6J x CASTEi/J). אותו חלק מהקריאות המוקצות לאלל האימהי והאבהי צפויות. (F) לבסוף, רק זוגות חוקיים החופפים לאזור הלכידה נבחרים לבניית מפות אנשי הקשר. זוגות לכידה-לכידה מייצגים אנשי קשר בתוך אזור היעד, בעוד שזוגות לכידה-מדווח כוללים אינטראקציה בין אזור היעד לאזור מחוץ ליעד. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
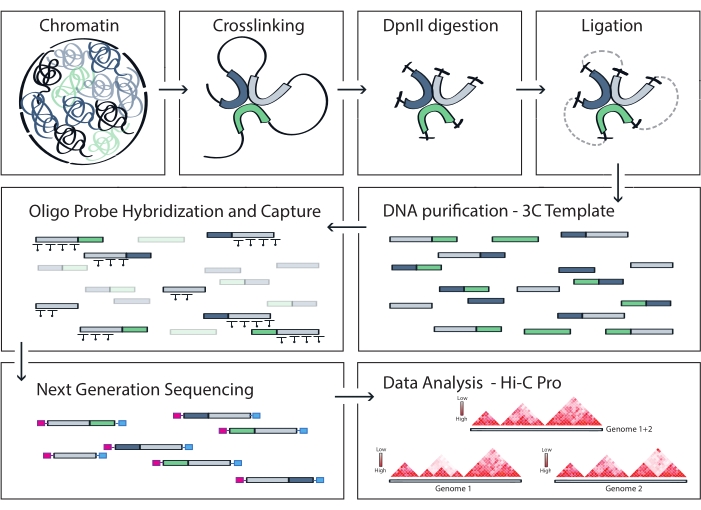
איור 4: זרימת עבודה של פרוטוקול Capture Hi-C. ייצוג סכמטי של שלבי פרוטוקול שונים. כדי ליצור את תבנית 3C רחבת הגנום, הכרומטין מוצלב תחילה עם פורמלדהיד ולאחר מכן מתעכל עם אנזים הגבלת DpnII. לאחר מכן קצוות דנ"א חופשיים נקשרים מחדש, הקישור הצולב מתהפך, והדנ"א מטוהר. כדי להעשיר מקטעים המקיפים את אזור המטרה, מערך של גשושיות RNA ביוטינילציה עובר הכלאה לתבנית 3C ונלכד על ידי משיכה בתיווך סטרפטווידין. ספריות לכידה מעובדות לריצוף מרובה, ומקטעי קשירה חוקיים מכומתים כדי להסיק את התדירות של מגעי הכרומטין על פני המטרה, המוצגים באופן חזותי כמפות אינטראקציה ברזולוציה גבוהה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
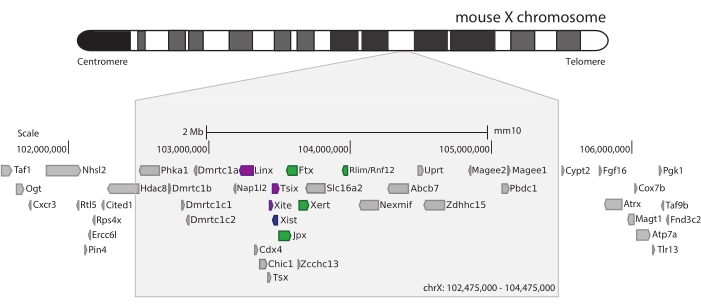
איור 5: סקירה כללית של האזור שמקיף את ה-Xic בכרומוזום X של העכבר. ייצוג סכמטי של כרומוזום X של העכבר והגדלה של האזור שצולם 3 Mb (ChrX: 102,475,000-105,475,000). אזור היעד כולל ~ 800 KB של DNA המתאים ל - Xic, מוקד הבקרה הראשי של XCI. ה-Xic כולל את הגנים הארוכים שאינם מקודדים, את Xist, שחקן מפתח ב-XCI, ואת הנוף הרגולטורי שלו. רגולטורים חיוביים של Xist מוצגים בירוק, ורגולטורים שליליים בסגול. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
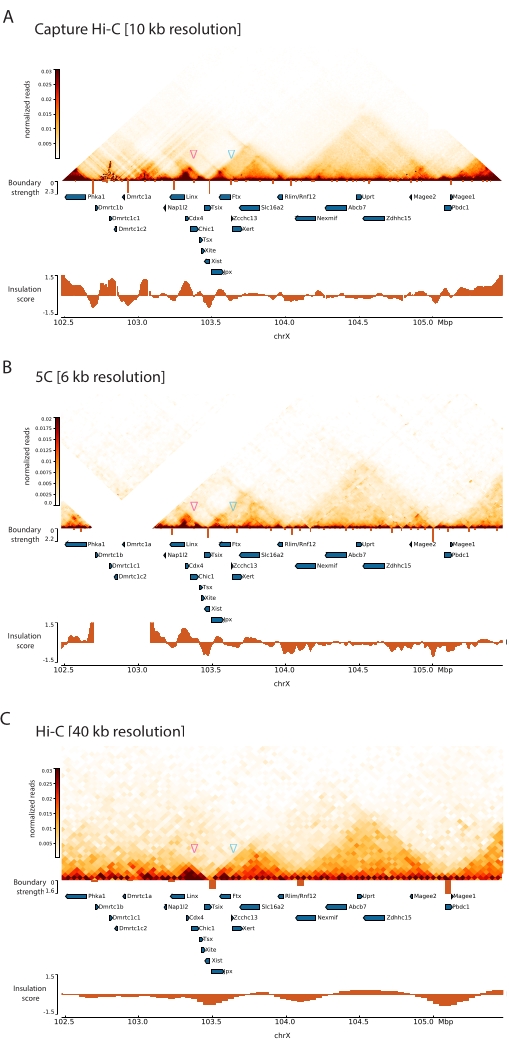
איור 6: לכוד מפות אינטראקציה של Hi-C, 5C ו- Hi-C ברחבי האזור שצולם ב- 3 Mb. (A) צלם מפת אינטראקציה Hi-C של היעד בגודל 3 Mb המקיף את העכבר Xic ברזולוציה של 10 KB (מחקר זה). (B) מפת אינטראקציה 5C של אותו אזור יעד כמו ב- A ברזולוציה של 6 KB (הנתונים עובדו מחדש מ-47). אזורים חוזרים שאינם נכללים בניתוחים מוסווים בלבן. נתוני 5C דורשים עיבוד ביואינפורמטי משלהם (ראה47). לאחר ניקוי ויישור, מפות 5C ברזולוציית פריימר מאוגדות באמצעות חציון פועל (חלון = 30 kb, שלב = 5) כדי להגיע לרזולוציה סופית של 6 kb. (C) מפת אינטראקציה Hi-C של אותו אזור גנומי כמו ב-A וב-B ברזולוציה של 40 KB (הנתונים עובדו מחדשמ-64). כל מפות האינטראקציה נוצרו מתאי גזע עובריים של עכברים. ציון הבידוד חושב באמצעות cooltools והוא מיוצג כהיסטוגרמות עם מינימום בידוד בגבולות TAD. גבולות TAD מוצגים כקווים אנכיים מתחת למפה. גובהו של כל קו מציין חוזק גבול. גנים מוצגים כחצים המצביעים לכיוון השעתוק. גבולות Sub-TAD המזוהים באופן בלעדי או מדויק יותר במפות Capture Hi-C מסומנים על ידי מגנטה וראשי חץ כחולים עבור תת-TADs ב- Tsix ו- Xist TADs, בהתאמה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
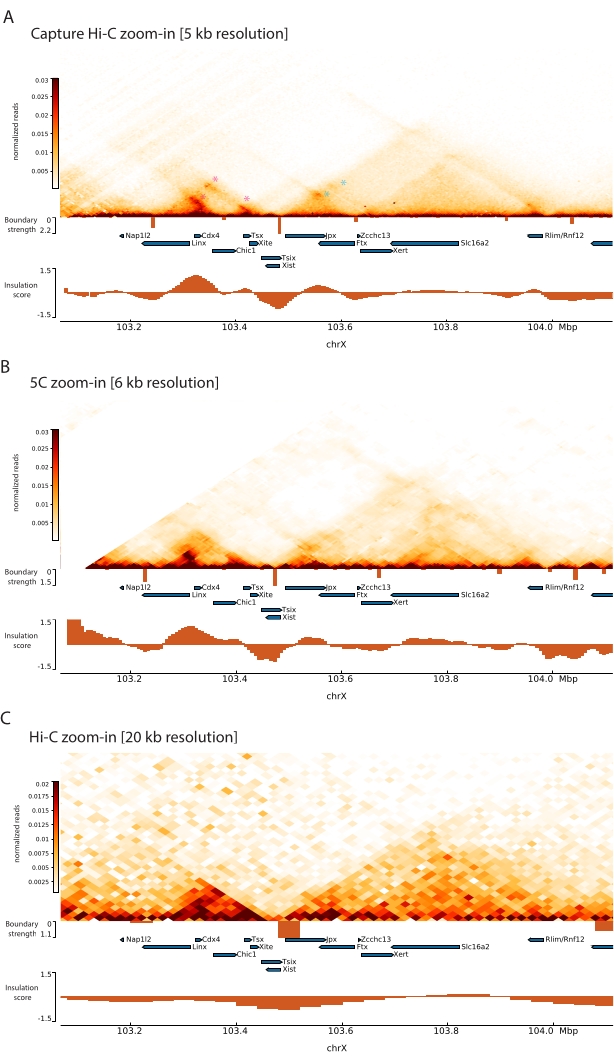
איור 7: לכוד מפות אינטראקציה של Hi-C, 5C ו- Hi-C על פני 1 Mb בתוך האזור שנלכד. (A) לכוד מפת אינטראקציה Hi-C של האזור הגנומי של 1 Mb המקיף את העכבר Xic ברזולוציה של 5 KB (מחקר זה). (B) מפת אינטראקציה 5C של אותו אזור גנומי כמו ב-A. ברזולוציה של 6 KB (הנתונים עובדו מחדש מ-47). אזורים חוזרים שאינם נכללים בניתוחים מוסווים בלבן. יש לציין כי נתוני 5C דורשים עיבוד ביואינפורמטי משלהם (ראה47). לאחר ניקוי ויישור, מפות 5C ברזולוציית פריימר מאוגדות באמצעות חציון פועל (חלון = 30 kb, שלב = 5) כדי להגיע לרזולוציה סופית של 6 kb. (C) מפת אינטראקציה Hi-C של אותו אזור גנומי כמו ב- A ו- B של Hi-C ברזולוציה של 20 KB (הנתונים עובדו מחדש מ-64). כל מפות האינטראקציה נוצרו מ-mESC. ציון הבידוד חושב באמצעות cooltools והוא מיוצג כהיסטוגרמות עם מינימום בידוד בגבולות TAD. גבולות TAD מוצגים כקווים אנכיים מתחת למפה. גובהו של כל קו מציין חוזק גבול. גנים מוצגים כחצים המצביעים על כיוון השעתוק. לולאות מגע המזוהות באופן בלעדי או מדויק יותר ב- Capture Hi-C מסומנות על ידי מגנטה וכוכביות כחולות עבור לולאות ב- Tsix ו- Xist TADs, בהתאמה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
טבלה משלימה 1: סטטיסטיקה לאחר ריצוף עבור מערכי הנתונים המשמשים בכתב יד זה: Capture Hi-C (מחקר זה), Hi-C64 ו-5C47. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
כאן אנו מתארים פרוטוקול Capture Hi-C מהיר וקל יחסית לאפיון ארגון מסדר גבוה יותר של אזורים גנומיים בגודל מגה-בסיס ברזולוציה של 5-10 קילובייט. Capture Hi-C שייך למשפחת טכנולוגיות Capture-C שנועדו להעשיר אינטראקציות כרומטין ממוקדות מתבניות 3C או Hi-C ברחבי הגנום. עד כה, הרוב הגדול של יישומי Capture-C נוצלו למיפוי מגעי כרומטין של אלמנטים רגולטוריים קטנים יחסית המפוזרים על פני הגנום כולו. בפרוטוקול Capture-C הראשון, נעשה שימוש במספר בדיקות RNA ביוטיניליות חופפות כדי ללכוד >400 מקדמים שנבחרו מראש בספריות 3C שהוכנו מתאי אריתרואידים31. אותה אסטרטגיה שופרה לאחר מכן ב-Next Generation (NG) וב-Nuclear Titrated (NuTi) Capture-C כדי להשיג פרופילי אינטראקציה ברזולוציה גבוהה של >8,000 מקדמים באמצעות פיתיונות DNA בודדים של 120 bp המתפרשים על פני אתרי הגבלה בודדים ושני סבבי לכידה עוקבים כדי למקסם את ההעשרה של מקטעי קשירה אינפורמטיביים32,40. אסטרטגיות אלה הובילו לדיסקציה תפקודית של אלמנטים הפועלים על ידי CIS בהקשרים רבים ושונים, כולל התפתחות עוברית עכבר, התמיינות תאים, השבתה של כרומוזום X ובקרה שגויה של גנים בתנאים פתולוגיים 46,63,65,66,67,68,69,70,71.
ב-Promoter Capture Hi-C (PCHi-C), >22,000 מקדמים מבוארים המכילים מקטעי הגבלה נמשכו מספריות Hi-C על ידי הכלאה של גשושיות RNA בודדות 120-mer biotinylated באחד או בשני הקצוות של מקטע ההגבלה 34,72. שיטה זו אפשרה דיסקציה של האינטראקטום של אלפי מקדמים במספר גדל במהירות של סוגי תאים, כולל תאי גזע עובריים של עכברים, תאי כבד עובריים ואדיפוציטים 34,35,72,73, אך גם קווי לימפובלסטואידים אנושיים, אבות המטופויטיים, קרטינוציטים אפידרמיסים ותאים פלוריפוטנטיים 37,74,75,76,77 .
בהשוואה לטכנולוגיות העשרת מטרות אלה, Capture Hi-C מכוון לאזורים גנומיים רציפים עד לקנה מידה מגה-בסיס, ובכך משתרע על פני TAD אחד או יותר ומקיף נופים רגולטוריים של גנים. כל אזור העניין חייב להיות מרוצף עם מערך של בדיקות biotinylated המקיף כל אתר הגבלה DpnII בתוך המטרה. ההכלאה של מערך הביוטינילציה לתבנית 3C, לכידה מבוססת סטרפטווידין לאחר מכן ועיבוד לריצוף מרובב מבוצעים באמצעות מערכת העשרת מטרה עבור ריצוף מרובב אילומינה Paired-End. הפרוטוקול כולו מהיר, מכיוון שניתן לבצע אותו תוך שבוע מהכנת ספריית 3C ועד לריצוף NGS, והוא דורש רק התאמות קלות ו / או פתרון בעיות ספציפי בהתאמה אישית.
הפרוטוקול מספק גם יתרונות בהשוואה לשיטות מבוססות 3C אחרות. כדי לקבל מפות אינטראקציה ברזולוציה של 5-10 קילו-בתים, רצפנו 100-120 M קריאות קצה. לשם השוואה, השתמשנו כאן במערך נתונים Hi-C של 571 M קריאות כדי להגיע לרזולוציה של 20 KB64 (GSM2053973), ולפחות מיליארד קריאות יידרשו כדי להגיע לרזולוציה של 5 KB עם Hi-C22 רחב כרומוזומים.
לכידת Hi-C כפי שנעשה בה שימוש במחקר הנוכחי מגיעה לרזולוציה גבוהה בהרבה מזו של 5C שפורסם בעבר בהתבסס על אנזים הגבלת חותך 6 bp47 (טבלה משלימה 1). חשוב לציין, האסטרטגיה שנועדה להעשיר ולהעצים אינטראקציות ממוקדות ב-5C אינה מאפשרת ניתוח ספציפי לאלל של אינטראקציות כרומטין. נהפוך הוא, ניתן למפות את נתוני Capture Hi-C באופן ספציפי, ולאפשר דיסקציה של נופים מבניים תלת-ממדיים של זוגות כרומוזומים הומולוגיים, למשל בתאים אנושיים או בקווי תאים היברידיים F1 הנגזרים מחציית זני עכברים שונים גנטית78. כדי ליצור מפות אינטראקציה ספציפיות ללכידת Hi-C ברזולוציה של 5 קילו-בתים, ריצפנו קריאות מותאמות של 150 bp כדי להגדיל את כיסוי SNP. גישות ספציפיות לאלל דומות יכולות להיות מיושמות על קווי תאים אנושיים, שעבורם הביאור של SNPs זמין22.
חשוב לציין, למרות ש- Capture Hi-C מבטיח בדרך כלל רזולוציה גבוהה תוך שיפור הסבירות של עלויות ריצוף, לייצור אוליגונוקלאוטידים ביוטינילטים בהתאמה אישית יש השפעה על העלות הכוללת של שיטה זו. לכן, הבחירה של שיטת 3C המתאימה ביותר תהיה שונה עבור יישומים שונים, ויהיה תלוי בשאלה הביולוגית כי הוא מטופל ואת הפתרון הנדרש, כמו גם את גודל האזור של עניין. פרוטוקולי Capture Hi-C אחרים שפותחו חולקים תכונות מפתח עם הפרוטוקול המתואר כאן. לדוגמה, אסטרטגיית Capture Hi-C יושמה כדי לאפיין ~ 50 kb עד 1 Mb אזורים גנומיים המשתרעים על פני וריאנטים לא מקודדים הקשורים לסיכון לסרטן השד וסרטן המעי הגס; בפרוטוקול זה, אזורי המטרה נשלפו מספריות Hi-C על ידי הכלאה של פיתיונות RNA של 120 מר המרצפות את אזורי המטרה בכיסוי פי 3 33,38,79. באופן דומה, HYbrid Capture Hi-C (Hi-C 2) שימש למיקוד אינטראקציות בתוך אזורי עניין של עד2 Mb80. בשני הפרוטוקולים, השימוש בתבנית Hi-C מועשרת עבור מקטעי קשירה שנמשכו כלפי מטה של ביוטין הגדיל את אחוז הקריאות האינפורמטיביות הכולל בהשוואה לפרוטוקול שלנו. לדוגמה, במערך הנתונים Hi-C שבו השתמשנו כאן להשוואה64 (GSM2053973), אחוז הזוגות התקפים לאחר הסרת כפילויות גבוה פי 4.8 מהזוגות החוקיים שהתקבלו ב- Capture Hi-C כמתואר באיור 3 ובטבלה משלימה 1. עם זאת, המשיכה הרצופה של מקטעים קשורים ביוטינילטים וגשושיות היברידיות הופכת את הפרוטוקול למורכב יותר באופן משמעותי וגוזל זמן רב, תוך הפחתת המורכבות של האזור שנלכד.
שיטה זמינה נוספת להעשרת תבניות 3C בבדיקות אריחים היא Tiled-C, אשר יושמה לחקר ארכיטקטורת הכרומטין ברזולוציה מרחבית וטמפורלית גבוהה במהלך התמיינות אריתרואידיםשל עכבר 43. ב-Tiled-C, פאנל של 70 bp biotinylated probes משמש להעשרת מגעים בתוך אזורים בקנה מידה גדול בשני סבבי לכידה רצופים כדי ליצור מפות ברזולוציה גבוהה מאוד של אינטראקציות ממוקדות 43,81. העשרת הלכידה הכפולה גם הופכת את הפרוטוקול לארוך ומורכב יותר בהשוואה ל- Capture Hi-C. עם זאת, בשונה מאסטרטגיות Capture-C המכוונות לאתרי הגבלה בודדים, ב- Tiled-C נראה כי הסבב השני של הלכידה אינו מגדיל באופן משמעותי את יעילות הלכידה, ולכן ניתן להשמיט אותו43. לבסוף, גישת ריצוף דומה המבוססת על אותה אסטרטגיית העשרת מטרה ששימשה במחקר זה יושמה על נתיחת נופים רגולטוריים הכוללים וריאנטים מבניים שתוארו בחולים עם מומים מולדים והונדסו מחדש בעכברים טרנסגניים41,42. במקרה זה, מערך הריצוף של הגשושיות תוכנן על פני המטרה כולה ולא בקרבת אתרי הגבלה DpnII41. אף על פי כן, עבודה זו הייתה מכרעת בהדגשת הרגישות והעוצמה של אסטרטגיה זו להשגת אפיון ברזולוציה גבוהה של אזורים גנומיים גדולים בהקשרים שונים41,42,48.
לסיכום, הפרוטוקול המתואר כאן מייצג אסטרטגיה קלה, חזקה ורבת עוצמה לאפיון תלת-ממדי ברזולוציה גבוהה של כל אזורי העניין הגנומיים. יישום גישה זו על מערכות מודל שונות, סוגי תאים, נופי כרומטינים מווסתים התפתחותית ובקרת גנים בתנאים בריאים ופתולוגיים עשוי להקל על הבנתנו את יחסי הגומלין והסיבתיות בין טופולוגיית הגנום לבין בקרת גנים, אחת השאלות הפתוחות הבסיסיות בתחום האפיגנטיקה. יתר על כן, יישום Capture Hi-C כדי למפות אינטראקציות ארוכות טווח וקיפול כרומטין מסדר גבוה יותר של וריאנטים בסיכון שזוהו על ידי מחקרי GWAS יש פוטנציאל לחשוף את הרלוונטיות הפונקציונלית של אתרים גנומיים לא מקודדים הקשורים למחלות אנושיות בהקשרים שונים, ובכך לספק תובנות חדשות על התהליכים שעשויים להיות בבסיס הפתוגנזה.
Disclosures
קאי האושולץ הוא מדען יישומי שטח בקבוצת Agilent Technologies - Diagnostic and Genomics. כל שאר המחברים מצהירים שאין אינטרסים מתחרים.
Acknowledgements
העבודה במעבדת הרד נתמכה על ידי פרס החוקר המתקדם של מועצת המחקר האירופית (XPRESS - AdG671027). א.ל. נתמכת על ידי מלגת מארי סקלודובסקה-קירי לפעולות אינדיבידואליות של האיחוד האירופי (IF-838408). A.H. נתמכת על ידי הרשת החדשנית והבינתחומית של ITN ChromDesign, במסגרת הסכם מענק מארי סקלודובסקה-קירי 813327. המחברים מודים לדניאל איברהים (MPI לגנטיקה מולקולרית, ברלין) על ייעוץ טכני מועיל, לפלטפורמת NGS במכון קירי (פריז), ולוולדימיר בנס ולמתקן הליבה הגנומית ב- EMBL (היידלברג), על התמיכה והסיוע.
Materials
| Name | Company | Catalog Number | Comments |
| 10x PBS pH 7.4 | Gibco | 10010-023 | |
| 37% (vol/vol) paraformaldehyde solution | Electron Microscopy Sciences | 15686 | single use glass-vials; do not reuse |
| 50 mL PP conical tube | Falcon | 352070 | |
| Agarose | Sigma | A9539-500g | |
| Bioanalyzer | Agilent | G2939BA | |
| Cell Scrapers - 25 cm Handle and 3.0 cm Blade | Falcon | 353089 | |
| CHIR99021 | Axon Medchem BV | Axon 1386 | |
| cOmplete Mini, Protease inhibitor cocktail (EDTA-free) | Merck | 11836170001 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL | Invitrogen | ZGEXSCCOUNTESS2FL | Automated cell counter |
| Covaris S2 | Covaris | 500217 | Sonicator |
| DNA LoBind tube, 1.5 mL | Eppendorf | 30108051 | |
| DpnII (50000 units/mL) | New England Biolabs | R0543M | |
| Dulbecco's Modified Eagle Medium (DMEM) | Merck | D6429 | |
| Ethanol (100%) | Merck | 1.00983.2500 | |
| Fetal Bovine Serum (FBS) | Thermo Scientific | 10270106 | |
| gelatine from porcine skin | Sigma | G1890 | |
| GeneRuler 1 kb Plus DNA Ladder | Thermo Scientific | SM0313 | |
| GlycoBlue | Thermo Scientific | AM9516 | Coprecipitant |
| High-Sensitivity Bioanlayzer chips | Agilent | 5067-4626 | |
| Large Cooling Centrifuge 5920 R | Eppendorf | 5948000018 | |
| leukaemia inhibitory factor (LIF) | Merck | ESG1107 | |
| Liquiport | KNF | NF300 | Benchtop aspiration system |
| Low-binding filter tips | Biozym | VT0260U, VT0240, VT0220, VT0200U | |
| Molecular biology grade water | Merck | W3500-6x500ML | |
| Next Seq 500 | Illumina | SY-415-1001 | |
| Next Seq 500 High Output v2 Kit (300 cycles) | Illumina | FC-404-2004 | |
| Nonidet P40 Substitute (NP40) | Merck | 11332473001 | |
| PD0325901 | Axon Medchem BV | Axon 1408 | |
| Protease inhibitor cocktail (EDTA-free) | Merck | 11873580001 | |
| Proteinase K - recombinant, PCR-grade (20 mg/mL) | Thermo Scientific | EO0491 | |
| Qubit 2.0 | Thermo Scientific | Q32871 | |
| Qubit assay tubes | Thermo Scientific | Q32856 | |
| Qubit dsDNA High Sensitivity kit | Thermo Scientific | Q32851 | |
| RNase A (10 mg/mL) | Thermo Scientific | EN0531 | |
| Sodium acetate pH 5.2 (3M) | Merck | S7899 | |
| speed vacuum concentrator | Eppendorf | EP5305000100-1EA | |
| Agencourt AMPureXP | Beckman Coulter | A63881 | SPRI beads |
| SureSelect Target Enrichment Box 1 | Agilent | 5190-8645 | |
| SureSelect Target Enrichment Kit ILM Indexing Hyb Module Box 2 | Agilent | 5190-4455 | |
| SureSelect XT Library Prep Kit ILM | Agilent | 5500-0132 | |
| T4 ligase (30 units/µL) | Thermo Scientific | EL0013 | |
| table-top Centrifuge 5427 R | Eppendorf | 5409000012 | |
| Triton-X-100 (500 mL) | Merck | X100-500ML | |
| Trypan Blue | Invitrogen | T10282 | |
| Trypsine | Thermo Scientific | 25300054 | |
| UltraPure Glycine | Thermo Scientific | 15527013 | |
| β-mercaptoethanol | Thermo Scientific | 31350010 |
References
- Ibrahim, D. M., Mundlos, S. The role of 3D chromatin domains in gene regulation: a multi-facetted view on genome organization. Current Opinion in Genetics & Development. 61, 1-8 (2020).
- Bolt, C. C., Duboule, D. The regulatory landscapes of developmental genes. Development. 147 (3), (2020).
- Glaser, J., Mundlos, S. 3D or not 3D: Shaping the genome during development. Cold Spring Harbor Perspectives in Biology. 14 (5), 040188(2021).
- Denker, A., De Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Kempfer, R., Pombo, A. Methods for mapping 3D chromosome architecture. Nature Reviews Genetics. 21 (4), 207-226 (2020).
- McCord, R. P., Kaplan, N., Giorgetti, L. Chromosome conformation capture and beyond: Toward an integrative view of chromosome structure and function. Molecular Cell. 77 (4), 688-708 (2020).
- Jerkovic, I., Cavalli, G. Understanding 3D genome organization by multidisciplinary methods. Nature ReviewsMolecular Cell Biology. 22 (8), 511-528 (2021).
- Hsieh, T. -H. S., et al. Mapping nucleosome resolution chromosome folding in yeast by Micro-C. Cell. 162 (1), 108-119 (2015).
- Krietenstein, N., et al. Ultrastructural details of mammalian chromosome architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Naumova, N., Smith, E. M., Zhan, Y., Dekker, J. Analysis of long-range chromatin interactions using Chromosome Conformation Capture. Methods. 58 (3), 192-203 (2012).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Zhao, Z., et al. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra-and interchromosomal interactions. Nature Genetics. 38 (11), 1341-1347 (2006).
- Würtele, H., Chartrand, P. Genome-wide scanning of HoxB1-associated loci in mouse ES cells using an open-ended Chromosome Conformation Capture methodology. Chromosome Research. 14 (5), 477-495 (2006).
- De Wit, E., De Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes & Development. 26 (1), 11-24 (2012).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Splinter, E., et al. The inactive X chromosome adopts a unique three-dimensional conformation that is dependent on Xist RNA. Genes & Development. 25 (13), 1371-1383 (2011).
- Ferraiuolo, M. A., Sanyal, A., Naumova, N., Dekker, J., Dostie, J. From cells to chromatin: capturing snapshots of genome organization with 5C technology. Methods. 58 (3), 255-267 (2012).
- Kim, J. H., et al. 5C-ID: Increased resolution Chromosome-Conformation-Capture-Carbon-Copy with in situ 3C and double alternating primer design. Methods. 142, 39-46 (2018).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Zhang, Y., et al. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations. Cell. 148 (5), 908-921 (2012).
- Rao, S. S. P., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Dixon, J. R., et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485 (7398), 376-380 (2012).
- Nora, E. P., et al. Spatial partitioning of the regulatory landscape of the X-inactivation centre. Nature. 485 (7398), 381-385 (2012).
- Krefting, J., Andrade-Navarro, M. A., Ibn-Salem, J. Evolutionary stability of topologically associating domains is associated with conserved gene regulation. BMC Biology. 16 (1), 87(2018).
- Galupa, R., Heard, E. Topologically associating domains in chromosome architecture and gene regulatory landscapes during development, disease, and evolution. Cold Spring Harbor Symposia on Quantitative Biology. 82, 267-278 (2017).
- Tena, J. J., Santos-Pereira, J. M. Topologically associating domains and regulatory landscapes in development, evolution and disease. Frontiers in Cell and Developmental Biology. 9, 702787(2021).
- Lupiáñez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How alterations of chromatin domains result in disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Davidson, I. F., Peters, J. -M. Genome folding through loop extrusion by SMC complexes. Nature Reviews Molecular Cell Biology. 22 (7), 445-464 (2021).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews Molecular Cell Biology. 17 (12), 743-755 (2016).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Davies, J. O. J., et al. Multiplexed analysis of chromosome conformation at vastly improved sensitivity. Nature Methods. 13 (1), 74-80 (2016).
- Jäger, R., et al. Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci. Nature Communications. 6, 6178(2015).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Sahlén, P., et al. Genome-wide mapping of promoter-anchored interactions with close to single-enhancer resolution. Genome Biology. 16, 156(2015).
- Joshi, O., et al. Dynamic reorganization of extremely long-range promoter-promoter interactions between two states of pluripotency. Cell Stem Cell. 17 (6), 748-757 (2015).
- Mifsud, B., et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nature Genetics. 47 (6), 598-606 (2015).
- Dryden, N. H., et al. Unbiased analysis of potential targets of breast cancer susceptibility loci by Capture Hi-C. Genome Research. 24 (11), 1854-1868 (2014).
- Oudelaar, A. M., Davies, J. O. J., Downes, D. J., Higgs, D. R., Hughes, J. R. Robust detection of chromosomal interactions from small numbers of cells using low-input Capture-C. Nucleic Acids Research. 45 (22), 184(2017).
- Oudelaar, A. M., et al. Single-allele chromatin interactions identify regulatory hubs in dynamic compartmentalized domains. Nature Genetics. 50 (12), 1744-1751 (2018).
- Franke, M., et al. Formation of new chromatin domains determines pathogenicity of genomic duplications. Nature. 538 (7624), 265-269 (2016).
- Despang, A., et al. Functional dissection of the Sox9-Kcnj2 locus identifies nonessential and instructive roles of TAD architecture. Nature Genetics. 51 (8), 1263-1271 (2019).
- Oudelaar, A. M., et al. Dynamics of the 4D genome during in vivo lineage specification and differentiation. Nature Communications. 11 (1), 1-12 (2020).
- Galupa, R., Heard, E. X-chromosome inactivation: A crossroads between chromosome architecture and gene regulation. Annual Review of Genetics. 52, 535-566 (2018).
- Loda, A., Collombet, S., Heard, E. Gene regulation in time and space during X-chromosome inactivation. Nature Reviews. Molecular Cell Biology. 23 (4), 231-249 (2022).
- van Bemmel, J. G., et al. The bipartite TAD organization of the X-inactivation center ensures opposing developmental regulation of Tsix and Xist. Nature Genetics. 51 (6), 1024-1034 (2019).
- Galupa, R., et al. A conserved noncoding locus regulates random monoallelic Xist expression across a topological boundary. Molecular Cell. 77 (2), 352-367 (2020).
- Gjaltema, R. A. F., et al. Distal and proximal cis-regulatory elements sense X chromosome dosage and developmental state at the Xist locus. Molecular Cell. 82 (1), 190-208 (2022).
- Galupa, R., et al. Inversion of a topological domain leads to restricted changes in its gene expression and affects inter-domain communication. Development. 149 (9), (2022).
- Savarese, F., Flahndorfer, K., Jaenisch, R., Busslinger, M., Wutz, A. Hematopoietic precursor cells transiently reestablish permissiveness for X inactivation. Molecular and Cellular Biology. 26 (19), 7167-7177 (2006).
- Schulz, E. G., et al. The two active X chromosomes in female ESCs block exit from the pluripotent state by modulating the ESC signaling network. Cell Stem Cell. 14 (2), 203-216 (2014).
- Gnirke, A., et al. Solution hybrid selection with ultra-long oligonucleotides for massively parallel targeted sequencing. Nature Biotechnology. 27 (2), 182-189 (2009).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Piccinini, F., Tesei, A., Arienti, C., Bevilacqua, A. Cell counting and viability assessment of 2D and 3D Cell cultures: Expected reliability of the trypan blue assay. Biological Procedures Online. 19 (1), 8(2017).
- Servant, N., et al. HiC-Pro: an optimized and flexible pipeline for Hi-C data processing. Genome Biology. 16, 259(2015).
- Abdennur, N., Mirny, L. A. Cooler: scalable storage for Hi-C data and other genomically labeled arrays. Bioinformatics. 36 (1), 311-316 (2020).
- Imakaev, M., et al. Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nature Methods. 9 (10), 999-1003 (2012).
- Forcato, M., et al. Comparison of computational methods for Hi-C data analysis. Nature Methods. 14 (7), 679-685 (2017).
- Venev, S., et al. open2c/cooltools: v0.4.1. , (2021).
- Wages, J. M. NUCLEIC ACIDS | Immunoassays. Encyclopedia of Analytical Science. , Elsevier. 408-417 (2005).
- Ewels, P. A., et al. The nf-core framework for community-curated bioinformatics pipelines. Nature Biotechnology. 38 (3), 276-278 (2020).
- Servant, N., Peltzer, A. nf-core/hic: Initial release of nf-core/hic. Zenodo. , (2019).
- Furlan, G., et al. The Ftx noncoding locus controls X chromosome inactivation independently of its RNA products. Molecular Cell. 70 (3), 462-472 (2018).
- Giorgetti, L., et al. Structural organization of the inactive X chromosome in the mouse. Nature. 535 (7613), 575-579 (2016).
- Simon, C. S., et al. Functional characterisation of cis-regulatory elements governing dynamic Eomes expression in the early mouse embryo. Development. 144 (7), 1249-1260 (2017).
- Williams, R. M., et al. Reconstruction of the global neural crest gene regulatory network in vivo. Developmental Cell. 51 (2), 255-276 (2019).
- Godfrey, L., et al. DOT1L inhibition reveals a distinct subset of enhancers dependent on H3K79 methylation. Nature Communications. 10 (1), 2803(2019).
- Hanssen, L. L. P., et al. Tissue-specific CTCF-cohesin-mediated chromatin architecture delimits enhancer interactions and function in vivo. Nature Cell Biology. 19 (8), 952-961 (2017).
- Larke, M. S. C., et al. Enhancers predominantly regulate gene expression during differentiation via transcription initiation. Molecular Cell. 81 (5), 983-997 (2021).
- Oudelaar, A. M., et al. A revised model for promoter competition based on multi-way chromatin interactions at the α-globin locus. Nature Communications. 10 (1), 5412(2019).
- Long, H. K., et al. Loss of extreme long-range enhancers in human neural crest drives a craniofacial disorder. Cell Stem Cell. 27 (5), 765-783 (2020).
- Schoenfelder, S., Javierre, B. -M., Furlan-Magaril, M., Wingett, S. W., Fraser, P. Promoter Capture Hi-C: High-resolution, genome-wide profiling of promoter interactions. Journal of Visualized Experiments. (136), e57320(2018).
- Siersbæk, R., et al. Dynamic rewiring of promoter-anchored chromatin loops during adipocyte differentiation. Molecular Cell. 66 (3), 420-435 (2017).
- Rubin, A. J., et al. Lineage-specific dynamic and pre-established enhancer-promoter contacts cooperate in terminal differentiation. Nature Genetics. 49 (10), 1522-1528 (2017).
- Freire-Pritchett, P., et al. Global reorganisation of cis-regulatory units upon lineage commitment of human embryonic stem cells. eLife. 6, 21926(2017).
- Javierre, B. M., et al. Lineage-specific genome architecture links enhancers and non-coding disease variants to target gene promoters. Cell. 167 (5), 1369-1384 (2016).
- Miguel-Escalada, I., et al. Human pancreatic islet three-dimensional chromatin architecture provides insights into the genetics of type 2 diabetes. Nature Genetics. 51 (7), 1137-1148 (2019).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Baxter, J. S., et al. Capture Hi-C identifies putative target genes at 33 breast cancer risk loci. Nature Communications. 9 (1), 1028(2018).
- Sanborn, A. L., et al. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes. Proceedings of the National Academy of Sciences. 112 (47), 6456-6465 (2015).
- Owens, D. D. G., et al. Dynamic Runx1 chromatin boundaries affect gene expression in hematopoietic development. Nature Communications. 13 (1), 773(2022).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved