Method Article
Capture Hi-C를 통한 고분해능 3D 염색질 조직 해독
요약
이 프로토콜은 위상 연관 도메인(TAD)의 경계 및 조절 및 기타 DNA 서열 요소 간의 장거리 염색질 상호 작용을 포함하여 고해상도에서 메가 기반 크기의 표적 게놈 영역의 3D 조직을 특성화하는 데 사용되는 Capture Hi-C 방법을 설명합니다.
초록
게놈의 공간적 구성은 전사, 복제, 재조합 및 복구를 포함한 많은 맥락에서 게놈의 기능과 조절에 기여합니다. 따라서 게놈 토폴로지와 기능 사이의 정확한 인과 관계를 이해하는 것이 중요하며 점점 더 집중적인 연구의 주제가 되고 있습니다. 염색체 형태 캡처 기술(3C)을 사용하면 게놈의 모든 영역 간의 상호 작용 빈도를 측정하여 염색질의 3D 구조를 추론할 수 있습니다. 여기에서는 메가 기반 크기의 게놈 표적의 대립 유전자 특이적 3D 조직을 고해상도로 특성화하는 3C 기반 표적 농축 방법인 Capture Hi-C를 수행하는 빠르고 간단한 프로토콜에 대해 설명합니다. Capture Hi-C에서 타겟 영역은 다운스트림 고처리량 시퀀싱 전에 비오티닐화된 프로브 어레이에 의해 캡처됩니다. 따라서 더 높은 분해능과 대립유전자 특이성을 달성하는 동시에 기술의 시간 효율성과 경제성을 향상시킵니다. 그 강점을 입증하기 위해 Capture Hi-C 프로토콜은 X-염색체 불활성화(XCI)의 마스터 조절 유전자좌인 마우스 X-불활성화 센터 (Xic)에 적용되었습니다.
서문
선형 게놈은 유기체가 배아 발달을 겪고 성인이 될 때까지 생존하는 데 필요한 모든 정보를 보유하고 있습니다. 그러나 유전적으로 동일한 세포가 서로 다른 기능을 수행하도록 지시하는 것은 서로 다른 조직 및/또는 발달 단계를 포함하여 특정 상황에서 사용되는 정보를 정확하게 제어하는 데 기본입니다. 게놈의 3차원 조직은 선형 게놈에서 수백 킬로베이스로 분리될 수 있는 조절 요소 간의 물리적 상호작용을 촉진하거나 방지함으로써 유전자 활성의 정확한 시공간 조절에 참여하는 것으로 생각됩니다(리뷰 1,2,3;). 지난 20년 동안 게놈 접힘과 활동 사이의 상호 작용에 대한 우리의 이해는 주로 염색체 형태 캡처 기술(3C)의 개발로 인해 급격히 증가했습니다(검토 4,5,6,7). 이 방법은 게놈의 모든 영역 간의 상호 작용 빈도를 측정하고 핵 내에서 3D로 근접한 DNA 서열의 결찰에 의존합니다. 가장 일반적인 3C 프로토콜은 포름알데히드와 같은 가교제로 세포 집단을 고정하는 것으로 시작합니다. 가교 결합 된 염색질은 제한 효소로 소화되지만 MNase 소화도사용되었습니다 8,9. 소화 후, 공간적으로 가까운 유리 DNA 말단이 다시 결찰되고 가교가 역전됩니다. 이 단계는 3C '라이브러리' 또는 '템플릿'을 생성하는데, 이는 핵에 3D 근접한 서열이 동일한 DNA 단편에서 결찰될 가능성이 더 높은 하이브리드 단편의 혼합 풀입니다. 이러한 하이브리드 단편의 다운스트림 정량화를 통해 선형 게놈에서 수천 개의 염기쌍 떨어져 있지만 3D 공간에서 상호 작용할 수 있는 게놈 영역의 3D 형태를 추론할 수 있습니다.
3C 라이브러리를 특성화하기 위해 다양한 접근 방식이 개발되었으며, 결찰 단편의 하위 집합이 분석되고 다운스트림 정량화에 사용되는 기술이 서로 다릅니다. 원래의 3C 프로토콜은 두 관심 영역의 선택과 PCR10,11에 의한 '일대일' 상호 작용 빈도의 정량화에 의존했습니다. 4C 접근법(원형 염색체 형태 캡처)은 단일 관심 유전자좌(즉, '관점')와 나머지 게놈('하나 대 모두') 간의 상호 작용을 측정합니다12,13,14. 도 4C에서, 3C 라이브러리는 제2 분해 및 재결찰을 거쳐 관점 특이적 프라이머(15)에 의해 PCR 증폭되는 작은 원형 DNA 분자를 생성한다. 5C(염색체 형태 캡처 카본 카피)는 더 큰 관심 영역에서 3D 상호 작용의 특성화를 가능하게 하여 해당 영역 내에서 고차 염색질 접힘에 대한 통찰력을 제공합니다('다수 대 다수')16. 도 5C에서, 3C 라이브러리는 유니버셜 프라이머(15)를 갖는 멀티플렉스 PCR에 의해 후속적으로 증폭될 수 있는 제한 부위를 중첩하는 올리고뉴클레오티드의 풀에 혼성화된다. 4C 및 5C 모두에서, 유익한 DNA 단편은 초기에 마이크로어레이에 의해 정량화되었고, 나중에는 차세대 시퀀싱(NGS)에 의해 정량화되었다17,18,19. 이러한 전략은 표적 관심 영역을 특성화하지만 게놈 전체의 상호 작용을 매핑하는 데는 적용할 수 없습니다. 이 후자의 목표는 3C 템플릿의 대규모 병렬 시퀀싱을 통해 게놈 전체 수준('모두 대 모두')에서 염색질 접힘의 편향 없는 특성화를 허용하는 3C 기반 고처리량 전략인 Hi-C를 통해 달성됩니다('모두 대 모두')20. Hi-C 프로토콜은 소화된 절편의 말단에 비오티닐화된 잔기를 혼입시킨 다음, 결찰된 절편의 회수율을 증가시키기 위해 스트렙타비딘 비드로 결찰 절편을 풀다운하는 것을 포함한다20.
Hi-C는 포유류 게놈이 3D 핵에서 여러 규모로 구조적으로 구성되어 있음을 밝혔습니다. 메가 베이스 규모에서 게놈은 활성 및 비활성 염색질 영역, A 및 B 구획으로 각각20,21로 나뉩니다. 상이한 염색질 및 활동 상태로 대표되는 추가 서브 구획의 존재도 이후에 나타났다22. 더 높은 분해능에서, 게놈은 위상 연관 도메인 (TADs)이라고 불리는 서브 메가 베이스 자기 상호 작용 도메인으로 더 분할되며, 인간 및 마우스 게놈23,24의 Hi-C 및 5C 분석에 의해 처음 밝혀졌습니다. 조직별 방식으로 변하는 구획과 달리 TAD는 일정한 경향이 있습니다(많은 예외가 있음에도 불구하고). 중요한 것은 TAD 경계가 종25에 걸쳐 보존된다는 것입니다. 포유류 세포에서 TAD는 종종 동일한 조절 환경을 공유하는 유전자를 포함하며 인접한 조절 영역과의 상호 작용을 제한하면서 유전자 공동 조절을 용이하게 하는 구조적 프레임워크를 나타내는 것으로 나타났습니다(검토 3,26,27,28). 또한, TAD 내에서, 응집력-압출 루프의 기저부에 있는 CTCF 부위로 인한 상호작용은 프로모터-인핸서 또는 인핸서-인핸서 상호작용의 확률을 증가시킬 수 있다(검토29).
Hi-C에서는 1Mb에서 40kb 해상도로 구획 및 TAD를 감지할 수 있지만 5-10kb 규모에서 원위 요소 간의 루프 상호 작용과 같은 더 작은 규모의 접점을 특성화하기 위해 더 높은 분해능을 달성할 수 있습니다. 그러나, HiC에 의해 이러한 루프를 효율적으로 검출할 수 있도록 분해능을 증가시키려면, 시퀀싱 깊이의 상당한 증가를 필요로 하고, 따라서 시퀀싱 비용도 증가시킨다. 분석이 대립 유전자 특이적이어야 하는 경우 이는 악화됩니다. 실제로, 분해능의 X-배 증가는 염기서열 분석 깊이의X2 증가를 필요로 하며, 이는 고분해능 및 대립유전자 특이적 게놈 전체 접근법이 엄청나게 비쌀 수 있음을 의미합니다30.
고분해능을 유지하면서 비용 효율성과 경제성을 개선하기 위해 다운스트림 염기서열 분석 전에 상보적인 비오틴 표지 올리고뉴클레오티드 프로브와 하이브리드화한 후 게놈 전체의 3C 또는 Hi-C 라이브러리에서 표적 관심 영역을 물리적으로 추출할 수 있습니다. 이러한 표적 농축 전략은 Capture-C 방법이라고 하며 게놈 전체에 흩어져 있는 수백 개의 표적 유전자좌(즉, Promoter Capture(PC) Hi-C; 차세대(NG) Capture-C; 낮은 입력(LI) 캡처-C; 핵 적정(NuTi) Capture-C; Tri-C)31,32,33,34,35,36,37,38,39,40 또는 최대 여러 메가베이스에 걸쳐 있는 지역(예: Capture HiC; HYbrid 캡처 Hi-C (Hi-C2); 타일-C)41,42,43. 2가지 측면은 포획 기반 방법에서 다양할 수 있다: (1) 비오티닐화 올리고뉴클레오티드의 성질 및 설계 (즉, RNA 또는 DNA, 분산된 게놈 표적을 포획하는 단일 올리고 또는 관심 영역을 타일링하는 다중 올리고); 및 (2) 3C 또는 Hi-C 라이브러리가 될 수 있는 타겟을 풀다운하는 데 사용되는 템플릿이며, 후자는 3C 라이브러리에서 풀다운된 비오티닐화된 제한 단편으로 구성됩니다.
여기서는 3C 라이브러리에서 타겟 접점의 강화를 기반으로 하는 Capture Hi-C 프로토콜에 대해 설명합니다. 이 프로토콜은 비오티닐화 RNA 프로브의 맞춤형 타일링 어레이 설계에 의존하며 3C 라이브러리 준비에서 NGS 시퀀싱까지 1주일 이내에 수행할 수 있습니다. 이 프로토콜은 빠르고 간단하며 5kb 해상도에서 메가베이스 크기의 관심 영역의 고차 3D 구성을 특성화하는 동시에 다른 3C 방법에 비해 시간 효율성과 경제성을 향상시킬 수 있습니다. Capture Hi-C 프로토콜은 X-염색체 불활성화(XCI)의 마스터 조절 유전자좌인 Xist 비암호화 RNA를 호스트하는 X-불활성화 센터(Xic)에 적용되었습니다. Xic은 이전에 광범위한 구조 및 기능 분석의 대상이었습니다 (검토44,45). 포유류에서 XCI는 암컷(XX)과 수컷(XY) 사이의 X-연관 유전자의 투여량을 보상하고 암컷 세포에서 두 개의 X 염색체 중 하나의 거의 전부의 전사 침묵을 포함합니다. Xic은 3D 게놈 토폴로지 및 유전자 조절44와의 상호 작용에 대한 연구를 위한 강력한 황금 표준 궤적을 나타냅니다. 마우스 배아 줄기 세포(mESC)에서 Xic에 대한 5C 분석은 TAD의 발견 및 명명으로 이어졌으며, 토폴로지 분할 및 유전자 공동 조절의 기능적 관련성에 대한 첫 번째 통찰력을 제공했습니다24. Xic의 토폴로지 조직은 이후 Xist 상향 조절 및 XCI 46의 적절한 발달 시기에 결정적으로 관여하는 것으로 나타났으며, TAD 내부 및 TAD 사이의 유전자 활성에 영향을 미칠 수 있는 의심되지 않는 시스 조절 요소도 최근 Xic47,48,49 내에서 발견되었습니다. Xic에 걸쳐 있는 마우스 X 염색체의 3Mb에 Capture Hi-C를 적용하면 고분해능에서 대규모 염색질 접힘을 해부하는 이 접근 방식의 힘을 알 수 있습니다. 관심 영역 내의 모든 DpnII 제한 부위에 걸친 비오티닐화 프로브 어레이의 설계부터 게놈 차원의 3C 라이브러리 생성, 표적 접촉의 혼성화 및 캡처, 다운스트림 데이터 분석에 이르기까지 상세하고 따르기 쉬운 프로토콜이 제공됩니다. 적절한 품질 관리 및 예상 결과에 대한 개요도 포함되어 있으며 접근 방식의 강점과 한계는 유사한 기존 방법에 비추어 논의됩니다.
프로토콜
이 연구에 사용된 마우스 배아 줄기 세포(mESC)는 Institut Curie(파리)51의 동물 관리 지침에 따라 Mus musculus castaneus 수컷과 TX/TX R26 rtTA/rtTA 암컷50의 교배에서 파생되었습니다.
1. 프로브 설계
- 관심 대상 영역을 커버하는 비오티닐화 프로브(120-mer RNA 올리고뉴클레오티드) 어레이를 설계합니다.
- 관심 영역을 겹치는 올리고뉴클레오타이드로 바둑판식으로 배열하여 표적 내의 각 염기서열이 평균적으로 두 개의 고유한 프로브(2x 커버리지)로 커버되도록 합니다(그림 1).
- 프로브 커버리지에서 반복적인 염기서열을 제외하여 비특이적 상호작용의 강화를 방지합니다.
참고: 정보 결찰 단편의 농축을 최대화하기 위해 표적에 걸쳐 각 DpnII 제한 부위의 300bp 업스트림 및 다운스트림에 걸친 영역을 정의하고(ChrX: 102,475,000-105,475,000), 28,913개의 비오티닐화 프로브를 Sure Design 플랫폼52를 통해 SureSelect DNA 표적 농축 기술에 따라 설계했습니다. 이 전략에 따르면, 최대 40개의 반복 서열 염기가 각 올리고뉴클레오티드에 허용되어 비특이적 상호작용의 농축을 최소화합니다. 프로브 어레이는 애질런트에 의해 합성되었다. 여기서, DpnII는 두 가지 이유로 제한 효소로서 사용된다: (1) 여러 3C-기반 방법(53)에서 일상적으로 사용되는 4-커터이다; (2) 이 연구에 사용된 F1 하이브리드 라인에서 인실리코(in silico )에서 테스트된 다른 제한 효소와 비교하여 절단 부위 근처에서 유익한 단일 뉴클레오티드 다형성(SNP)을 포착할 가능성을 최대화합니다(C57BL/6J x CASTEi/J).
2. 실험 절차
- 세포 준비
- 하나 또는 다수의 세포 배양 플레이트에 적절한 수의 세포를 시딩하여 고정 당일 ≥ 5 x 107 세포의 총 세포 수를 달성합니다.
참고: 이 연구에는 마우스 배아 줄기 세포(mESC)가 사용되었습니다. mESC는 2i + LIF를 함유하는 mESC 배지에서 젤라틴화된(1x PBS 중 0.1% 젤라틴 - 37°C에서 o/n, 5% CO2 인큐베이터) 세포 배양 플레이트 및 배치 테스트 태아 송아지 혈청(DMEM, 15% FBS, 0.1mM β-메르캅토에탄올, 1,000U/mL-1 백혈병 억제 인자(LIF), CHIR99021(3μM) 및 PD0325901(1μM)). 이 세포 유형의 경우 하나의 80% 융합 10cm 플레이트에는 약 2 x 107 개의 세포가 포함되어 있습니다. - 세포 계수를 위해 하나의 추가 세포 배양 플레이트를 준비합니다.
참고: 배지 사용량을 줄이기 위해 더 작은 세포 배양 플레이트를 사용할 수 있습니다. 이 경우 더 작은 플레이트에 시딩할 세포의 수를 그에 따라 조정해야 합니다(예: 15cm 플레이트에 비해 10cm 플레이트에서 3배 더 적은 셀).
- 하나 또는 다수의 세포 배양 플레이트에 적절한 수의 세포를 시딩하여 고정 당일 ≥ 5 x 107 세포의 총 세포 수를 달성합니다.
- 포름알데히드 고정
- 가교될 총 셀 수를 추정합니다.
- 가교 반응을 시작하기 전에 제조업체의 지침에 따라 자동 세포 계수기를 사용하여 세포 계수를 위해 특별히 준비된 대조군 플레이트에서 트립신 처리 및 세포 계수합니다.
- 생존 세포의 백분율을 결정하기 위해 생존율 염색 (예를 들어, 트리판 블루)을 포함한다54. 이 세포 수에서 가교를 위해 준비된 플레이트의 총 세포 수를 추정합니다.
- 가교를 위해 준비된 플레이트에서 배양액을 제거하고 적절한 양의 고정 용액(세포 배양액 내 2% 포름알데히드)으로 교체한다. 10cm 플레이트에 10mL를 사용합니다(예: 20cm 플레이트의 경우 ~15mL).
알림: 정확한 양의 고정 용액을 추가합니다. 부착 세포를 고정할 수 없는 경우 이 단계를 트립신 처리된 세포에 적용하고 50mL 원추형 원심분리기 튜브의 고정 용액 30mL에서 수행할 수 있습니다. 포름알데히드는 1년을 넘지 않아야 합니다. 일회용 바이알을 사용하는 것이 바람직합니다. 고정 용액은 사용하기 전에 실온(RT)에 가져와야 합니다.
주의 : 포름알데히드는 위험하므로 적절한 건강 및 안전 규정에 따라 취급해야 합니다. - 셰이커에서 부드럽게 혼합하면서 RT에서 10분 동안 고정합니다.
- 2.5 M 글리신-1x PBS를 최종 농도 0.125 M에 첨가하여 고정 반응을 소멸시키고, 10 cm 플레이트 상에 10 mL에 530 μL의 2.5 M 글리신-1x PBS를 첨가한다 (예를 들어, 15 cm 플레이트 상에 1060 μL 내지 20 mL).
참고: 세포가 용액에 고정된 경우 1590μL의 2.5M 글리신-1x PBS로 고정 반응을 소멸시킵니다. - RT에서 5분 동안 배양하고 셰이커에서 부드럽게 혼합합니다.
- 접시를 얼음으로 옮기고 셰이커에서 부드럽게 혼합하면서 얼음에서 추가로 15분 동안 배양합니다.
참고: 이제부터는 세포를 얼음 위에 보관해야 하며 완충액은 더 이상의 가교를 피하기 위해 미리 냉각되어야 합니다. 많은 플레이트를 처리해야 하는 경우 냉장실로 이동하십시오. - 빠른 취급을 위해 고정 용액을 비커에 부어 세포에서 제거합니다.
알림: 포름알데히드 함유 액체 폐기물은 적절한 건강 및 안전 규정에 따라 폐기하십시오. - 10cm 플레이트를 5mL의 차가운 0.125M 글리신-1x PBS(15cm 플레이트의 경우 8mL)로 빠르게 두 번 헹구어 파편과 죽은 세포를 씻어냅니다. 빠른 취급을 위해 비커에 액체를 부어 접시에서 액체를 제거합니다.
- 5 mL의 차가운 0.125 M 글리신-1x PBS를 10 cm 플레이트 (15 cm 플레이트의 경우 10 mL)에 넣고 플라스틱 셀 스크레이퍼를 사용하여 플레이트에서 세포를 빠르게 긁어냅니다.
- 혈청학적 피펫을 사용하여 세포 현탁액을 예냉된 50mL 원추형 원심분리 튜브로 옮깁니다.
- 차가운 0.125M 글리신-1x PBS 5mL로 플레이트를 두 번 헹구고 세포 현탁액을 원추형 원심분리기 튜브에 추가합니다.
- 4 °C에서 10분 동안 480 x g 로 회전합니다.
참고: 세포가 용액에 고정된 경우 세포를 미리 냉각된 원추형 원심분리기 튜브로 옮기고 4°C에서 10분 동안 480 x g 에서 회전합니다. 고정 용액을 비커에 붓고 차가운 0.125M 글리신-1x PBS 10mL로 3회 세척하여 제거합니다. 각 세척 단계에서 세포를 다시 일시 중단해야 합니다. - 탁상형 흡인 시스템으로 흡인하여 상층액을 제거합니다. P1000 피펫으로 조심스럽게 위아래로 피펫팅하여 1 x 107 세포당 1x PBS 500μL에 세포를 재현탁합니다. 정확한 볼륨의 셀을 다시 일시 중단하려면 2.2.1에서 얻은 총 셀 수 추정치를 참조하십시오.
- 세포 현탁액 500μL를 계산된 수의 1.5mL 미세원심분리기 튜브(1 x 107 세포/튜브)에 분취합니다.
- 4 °C에서 10분 동안 480 x g 로 회전합니다.
- 탁상형 흡인 시스템으로 상층액을 제거하고 액체 질소에서 세포 펠릿을 급속 동결합니다. 건전지 펠릿을 -80°C에서 보관한다.
참고: 샘플은 최소 1년 동안 보관할 수 있습니다.
- 가교될 총 셀 수를 추정합니다.
- 세포 용해
- 얼린 펠릿을 얼음 위에서 해동합니다.
- 시료 당 H 2 0에서 1.5 mL의 용해 완충액을 준비하십시오 : 10 mM Tris-HCl, pH 8.0, 10 mM NaCl 및0.2% NP40을 첨가하십시오.
- 600 μL의 저온 용해 완충액을 첨가하고 얼음 위에 잘 재현탁합니다.
- 세포가 부풀어 오르도록 얼음에서 15분 동안 배양합니다.
- 4°C에서 5분 동안 2655 x g 로 스핀다운하고 탁상형 흡인 시스템을 사용하여 상층액을 제거합니다.
- 이물질을 제거하려면 펠릿을 저온 용해 완충액 1mL에 재현탁하고 4°C에서 5분 동안 2655 x g 에서 회전시킨 다음 상층액을 제거합니다.
- 2655 x g 및 4 °C에서 다시 짧게 회전하고 P200 팁이 장착된 탁상용 흡인 시스템을 사용하여 남은 상층액을 최대한 제거합니다.
- 0.5%(vol/vol) SDS 100μL에서 재현탁합니다.
- 62°C의 열혼합기에서 1400rpm으로 10분 동안 소용돌이치면서 배양합니다.
- 290 μL의 H2O+ 50 μL의 10 % TritonX-100을 첨가하고 기포를 피하면서 잘 섞는다.
- 37°C의 열혼합기에서 1400rpm으로 15분 동안 소용돌이치면서 배양합니다.
- 50μL의 10x Dpnll 버퍼를 추가하고 튜브를 뒤집어 혼합합니다.
- 품질 관리를 위해 소화되지 않은 DNA 50μL를 별도의 튜브에 넣습니다. 소화되지 않은 대조군 샘플을 섭취하는 것을 잊지 마십시오.
- DpnII 소화
- 10 μL의 Dpnll 고농도(총 500 U)를 첨가하고 반전하여 혼합합니다.
- 샘플과 소화되지 않은 대조군을 37°C의 열혼합기에서 인큐베이션하고, 1400rpm에서 >4시간 동안 소용돌이칩니다.
- 하루가 끝날 때 10 μL의 Dpnll 고농도 (총 500 U)를 추가하십시오.
- 샘플과 소화되지 않은 대조군을 37°C에서 배양하고, 밤새 1400 rpm에서 소용돌이치게 한다.
- 다음날 초에 10 μL의 Dpnll 고농도 (총 500 U)를 샘플에 첨가합니다.
- 샘플과 소화되지 않은 대조군을 37°C의 열혼합기에서 인큐베이션하고, 1400rpm에서 4시간 동안 소용돌이치게 한다.
- 결찰 및 가교 반전
- 튜브를 65°C에서 1400rpm으로 20분 동안 배양합니다.
참고: 이 시점에서 SDS를 추가하지 마십시오. 이 아이디어는 핵 무결성을 보존하는 것이므로 결찰은 핵 내부에서 수행되어 극단적 인 희석의 필요성을 피합니다. - 최대 5-10분 동안 얼음에서 샘플을 식히십시오. SDS 침전을 피하려면 샘플을 이보다 더 오래 얼음 위에 두지 마십시오.
- 품질 관리를 위해 50μL의 결찰되지 않은 소화된 DNA를 별도의 튜브에 담습니다. 소화되지 않고 결찰되지 않은 대조군을 -20°C에서 보관하십시오.
참고: 결찰되지 않은 대조군 샘플을 채취하는 것을 잊지 마십시오. - 800 μL의 결찰 칵테일을 추가합니다: 122 μL의 10x 리가아제 완충액, 8 μL의 T4 리가아제(30 U/μL) 및 670 μL의 H20.
- 16°C에서 배양하고 밤새 1000rpm으로 소용돌이칩니다.
- 7.5 μL의 Proteinase K(20 mg/mL)를 샘플에 추가하고 2 μL를 대조군에 추가합니다.
- 65°C에서 1000rpm으로 4시간 동안 배양합니다.
- 튜브를 65°C에서 1400rpm으로 20분 동안 배양합니다.
- DNA 정제
- 얼음 위의 샘플을 사전 냉각된 15mL 원추형 원심분리기 튜브로 옮기고 물 2mL, 얼음처럼 차가운 EtOH 10.5mL, 3M NaAC 583μL를 추가합니다.
알림: 추가 물은 DTT가 펠릿으로 이월되는 것을 방지하는 것을 목표로 합니다. - 얼음처럼 차가운 EtOH 200 μL, NaAC 10.8 μL, 공침제 1 μL를 소화되지 않고 결찰되지 않은 품질 관리에 추가합니다.
- -80°C에서 최소 4시간 동안 하룻밤 동안 배양합니다.
- 15mL 튜브를 2200 x g 에서 4°C에서 45분 동안 회전시킵니다.
- 1.5mL 대조군 튜브를 4°C에서 20,500 x g 로 30분 동안 회전시킵니다.
- 얼음처럼 차가운 70% EtOH 3mL(샘플) 및 1mL(대조군)로 한 번 세척합니다.
- 4°C에서 2200 x g(샘플) 또는 20,500 x g (대조군)에서 10분 동안 회전합니다.
- 조심스럽게 EtOH를 제거하고 RT에서 10-15분 동안 자연 건조합니다. 과도하게 사용하지 마십시오.
- 샘플과 대조군을 각각 100μL 및 20μL의 H20에 재현탁합니다.
- 1μL의 RNAseA를 추가하고 37°C에서 1400rpm으로 30분 동안 소용돌이치면서 배양합니다.
- 얼음 위의 샘플을 사전 냉각된 15mL 원추형 원심분리기 튜브로 옮기고 물 2mL, 얼음처럼 차가운 EtOH 10.5mL, 3M NaAC 583μL를 추가합니다.
- 3C 템플릿 준비의 품질 관리
- 고감도 DNA 농도 측정을 위해 형광계 키트를 사용하여 각 시료 및 대조군을 정량화합니다.
- 각 샘플 100-200 ng과 각 대조군을 1% 아가로스/1x TBE 젤에 로드합니다.
- 그림 2A와 같이 대조군과 3C 템플릿의 DNA 단편 크기 차이를 비교하여 겔 이미지가 예상 결과를 나타내는지 확인합니다.
- 샘플과 대조군을 -20°C에서 보관하십시오.
- 멀티플렉스 염기서열 분석을 위한 하이브리드화, 캡처 및 시료 처리
- 비오티닐화된 RNA 프로브 어레이를 3C 주형에 혼성화하고, 표적 결찰 단편을 포획하고, 쌍말단 다중화 시퀀싱을 위해 본 연구에서 사용된 표적 농축 시스템에 따라 다중화 시퀀싱을 위한 샘플을 준비합니다( 재료 표). 제조업체의 지침에 따라 프로토콜을 따르고 다음과 같은 사소한 수정 사항을 도입하십시오.
- 제조업체 프로토콜의 섹션 2: 샘플 준비
- 3μg의 gDNA 입력에서 시작하는 표적 농축에 대한 지침을 따르십시오.
- 다음 사양을 사용하여 초음파 처리기에서 DNA를 전단하십시오: 10% 듀티 사이클, 4 강도, 200 cyc/버스트 및 130초. 각 포획 반응에 대해 130μL의 물에 4μg의 3C 주형을 재현탁하여 3μg의 전단된 DNA로 샘플 준비를 계속하기에 충분한 물질을 확보합니다.
- 전단된 DNA의 품질을 평가합니다. 고감도 프로토콜에 따라 DNA 바이오분석기에서 전단된 DNA 1μL를 실행합니다. 150-700bp 사이의 단편 크기 분포를 예상합니다(그림 2).
- SPRI(Solid Phase Reversible Immobilization) 비드를 사용하여 시료를 정제합니다. 124 μL의 SPRI 비드를 124 μL의 DNA 샘플에 첨가하여 제조업체의 지침에 따라 1:1 왼쪽 크기 선택을 수행하고 25 μL의 뉴클레아제가 없는 물에서 용리합니다. 이 정제 단계는 더 짧은 단편을 제거하여 약 300bp의 단편을 농축합니다(그림 2).
참고: 이 단계에서 사용되는 샘플 및 SPRI 비드의 양은 샘플을 새 튜브로 옮기고 Bioanalyzer에서 품질 관리를 실행하는 동안 발생한 부피 손실을 고려합니다. 모든 후속 크기 선택 단계는 제조업체의 프로토콜에서 권장하는 비율에 따라 수행됩니다. SPRI 비드로부터의 DNA 용출은 프로토콜 전반에 걸쳐 RT에서 수행된다. - 크기에 따라 선택된 전단 DNA의 품질을 평가합니다. 고감도(HS) 프로토콜에 따라 DNA 바이오분석기에서 전단된 DNA 1μL를 실행합니다. 300bp에서 가장 높은 농축을 가진 단편 크기의 분포를 예상합니다(그림 2). 전단이 성공한 경우 전단된 DNA의 정량화를 진행하십시오.
- HS DNA 농도 측정을 위해 형광계 키트로 전단된 DNA를 정량합니다.
참고: DNA 전단 결과 <3μg의 DNA 수율이 나오면 다른 4μg의 DNA로 두 번째 DNA 전단을 수행하고 첫 번째 SPRI 비드 정제 단계 후 전단된 DNA 샘플을 결합하여 총 3μg의 전단된 DNA를 얻습니다. - 뉴클레아제가 없는 물을 크기가 선택된 세척된 DNA 샘플(총 3μg)에 최종 부피 48μL로 추가하고 제조업체의 프로토콜에 따라 최종 복구 반응을 진행합니다.
- paired-end adaptors를 결찰한 후, 제조사의 지침에 따라 5주기의 pre-capture PCR을 수행하여 라이브러리를 증폭합니다(PCR 및 프라이머에 대한 조건은 키트에 제공됨).
- 제조업체 프로토콜의 섹션 4: 하이브리드화 및 캡처
- 준비된 DNA 샘플을 표적 특이적 RNA 프로브에 혼성화하기 위해 750ng의 DNA 샘플을 최종 부피 3.4μL로 희석하여 초기 농도가 221ng/μL가 되도록 합니다. 더 큰 부피로 희석된 DNA 샘플의 경우 속도 진공 농축기를 사용하여 최종 부피로 줄입니다. 15-20분 동안의 속도-진공 농도(250 x g; ≤45°C)는 일반적으로 10μL로 재현탁된 샘플에 충분합니다. 속도-진공 농축기를 시작하기 전에 각 샘플에 대해 동일한 입력 부피가 있는지 확인하십시오.
- 하이브리드화 혼합물을 제조자의 지시에 따라 105°C에서 가열된 뚜껑과 함께 65°C에서 16-18시간 동안 인큐베이션한다.
- 제조업체 프로토콜의 섹션 5: 다중화 시퀀싱을 위한 인덱싱 및 샘플 처리
- 인덱싱 프라이머로 캡처된 라이브러리를 증폭하려면 제조업체의 지침에 따라 12주기의 포획 후 PCR을 수행합니다(PCR 및 프라이머에 대한 조건은 키트에 제공됨).
- 제조업체 프로토콜의 섹션 2: 샘플 준비
- 비오티닐화된 RNA 프로브 어레이를 3C 주형에 혼성화하고, 표적 결찰 단편을 포획하고, 쌍말단 다중화 시퀀싱을 위해 본 연구에서 사용된 표적 농축 시스템에 따라 다중화 시퀀싱을 위한 샘플을 준비합니다( 재료 표). 제조업체의 지침에 따라 프로토콜을 따르고 다음과 같은 사소한 수정 사항을 도입하십시오.
- 차세대 염기서열 분석
- 동일한 플로우 셀에서 여러 캡처 Hi-C 라이브러리를 실행하려면 캡처 라이브러리와 라이브러리당 100-120M 읽기를 시퀀스로 혼합하여 준비합니다.
- 대립유전자 특이적 분석이 필요한 경우, 충분한 SNP 커버리지를 보장하기 위해 150bp paired-end를 시퀀싱한다.
3. 데이터 분석
- HiC-Pro 파이프라인을 적용하여 Capture Hi-C 데이터 분석(55)을 수행합니다. HiC-Pro는 다음을 포함하여 처리의 각 단계에서 품질 관리를 제공합니다(그림 3).
(나는) 결찰 부위에 걸친 판독 비율과 쌍 및 싱글톤의 수를 지정하는 참조 게놈의 정렬률.
(ii) 유효한 결찰 산물과 비정보 판독 쌍(dangling-end, self-ligation 등)의 비율.
(iii) 단/장거리 및 염색체 내/간 접촉의 비율.
(iv) Capture Hi-C에 대한 타겟 접촉의 비율입니다.
(v) 지정된 경우 대립 유전자 관련 읽기의 비율입니다.
참고: HiC-Pro는 현장 Hi-C 및 Capture Hi-C를 포함한 광범위한 프로토콜을 지원합니다. 후자의 경우 사용자는 구성 파일에서 대상 지역(BED 형식)을 지정하기만 하면 됩니다. 일단 데이터가 처리되면, HiC-Pro 출력은 다운스트림 분석(56)을 위해 더 차가운 물체로 쉽게 변환될 수 있다. 이 단계에서, 다양한 해상도에서의 접촉 맵은 이전에 Imakaev 및 동료57에 의해 기술된 ICE 방법을 사용하여 정규화된다. 그런 다음 염색체 구획, TAD 또는 염색질 루프를 호출하기 위해 여러 분석을 실행할 수 있습니다(검토58용). 프로토콜의 워크플로는 그림 4에 나와 있습니다. 여기서 'cooltools' 제품군은 그림 5 및 그림 659와 같이 절연 점수와 TAD 경계를 계산하는 데 적용됩니다.
결과
설명된 Capture Hi-C 프로토콜은 4염기 절단기(DpnII)를 사용하여 게놈 전체 3C 템플릿의 준비를 기반으로 합니다. 관심 게놈 영역에 걸친 결찰 단편의 후속 농축은 이 연구에서 사용된 표적 농축 시스템에 따라 타일링 RNA 프로브 어레이와 스트렙타비딘 기반 포획의 혼성화에 의해 얻어집니다(그림 1). 비오티닐화된 RNA 프로브는 DNA 프로브52,60에 비해 표적에 대해 더 긴밀한 결합 친화도를 나타내기 때문에 선택되었습니다. 그런 다음 캡처된 라이브러리는 멀티플렉싱된 고처리량 시퀀싱을 위해 인덱싱되고 풀링됩니다. 캡처 Hi-C 데이터는 고해상도 Hi-C 상호 작용 맵으로 시각화할 수 있을 뿐만 아니라 4C와 유사한 단일 시점 접촉 맵으로 시각화하여 캡처된 전체 영역 내에서 프로모터 또는 인핸서와 같은 더 작은 시퀀스의 상호 작용을 구체적으로 시각화할 수 있습니다. 프로토콜의 워크플로는 그림 4에 나와 있습니다. 사전 시퀀싱 품질 관리는 그림 2에 나와 있으며 3C 템플릿의 적절한 분해 및 재결찰 평가와 프로토콜의 여러 단계에 걸친 효율적인 전단 및 정제를 포함합니다. 전단된 3C 주형 DNA는 150 내지 700 bp 사이에서 실행될 것으로 예상되며, 단편의 농축 >2 kb는 검출되지 않아야 한다. 다음 단계 동안 여러 비드 기반 DNA 클린업 및 크기 선택 단계가 먼저 전단 후, 그 다음 사전 포획 및 사후 포획 PCR 후에 수행됩니다. 세척된 라이브러리는 고감도 DNA 바이오분석기에서 시각화된 뚜렷한 단편 농축 프로파일을 보여줍니다(그림 2). 평균 단편 크기는 어댑터의 결찰, 시퀀싱 및 인덱싱 프라이머로 인해 라이브러리 준비 과정에서 증가합니다. 시퀀싱 후 품질 관리는 Hi-C Pro를 통해 얻을 수 있으며 그림 3에 나와 있습니다. 3C와 같은 데이터 처리 및 분석을 위해 다양한 생물 정보학 소프트웨어 응용 프로그램이 제안되었습니다. 그 중에서도, HiC-Pro 파이프라인은 가장 인기 있는 솔루션 중 하나이며, 다양한 해상도(55)에서 최종 접촉 맵에 대한 원시 시퀀싱 데이터를 처리할 수 있다. HiC-Pro는 2단계 매핑 전략을 사용하여 참조 게놈에서 염기서열 분석 판독을 정렬합니다. 그런 다음 3C 제품을 재구성하고 필터링하여 정보가 없는 연락처 쌍을 제거하고 연락처 맵을 생성합니다. 또한 알려진 다형성 목록을 사용하여 대립 유전자 특이 분석을 수행하고 별개의 접촉 맵에서 두 부모 대립 유전자에서 오는 접촉을 분리 할 수 있습니다. 보다 최근에, HiC-Pro가 포함되고 nf-코어 프레임워크(nf-core-hic)로 확장되어, 확장성이 뛰어나고 재현 가능한 커뮤니티 기반 파이프라인(61,62)을 제공한다.
마우스 Xic을 포획하기 위해 X 염색체의 3Mb를 타일링하는 28,913개의 RNA 프로브 어레이를 설계했습니다. 이 영역에는 XCI의 핵심 요소인 긴 비암호화 유전자 Xist 및 알려진 ~800kb 조절 환경이 포함됩니다(그림 5). 이 ~800 kb 영역은 2개의 TAD로 분할된다: 하나는 Xist 프로모터 및 그의 공지된 양성 조절인자 (즉, 비암호화 전사체 Ftx, Jpx 및 Xert 및 단백질-코딩 유전자 Rnf12)를 포함하고, Xist의 음성 시스-조절인자(즉, 그의 안티센스 전사체 Tsix, 인핸서 요소 Xite 및 비암호화 전사체 Linx)를 포함하는 인접 TAD (검토44를 위해, 45).
설명된 Capture Hi-C 프로토콜을 Xic에 적용함으로써 이 유전자좌의 토폴로지 구성을 전례 없는 해상도로 얻을 수 있었습니다(그림 6 및 그림 7). 이는 Capture Hi-C 프로파일을 이전에 공개된 5C47과 비교할 때 특히 명확합니다(그림 6 및 그림 7; 보충표 1) 및 Hi-C61(그림 6 및 그림 7; 보충표 1) 프로필. 예를 들어, 하위 TAD 구조가 더 분명한데, Xist 프로모터(Xist-TAD)를 포함하는 TAD는 명확하게 두 개의 작은 도메인으로 세분화됩니다(그림 6A, 파란색 화살촉). 이전에는 5C 프로파일(그림 6B)에서만 시각적으로 "추측"할 수 있었지만 절연 점수 알고리즘을 사용하여 이 영역의 경계를 감지했습니다. 마찬가지로, Capture Hi-C 프로파일의 분해능은 Tsix 유전자좌(Tsix-TAD)의 프로모터를 포함하는 이웃 TAD(그림 6A, B)에서 두 개의 더 작은 도메인을 식별할 수 있게 합니다. 이것은 이전에 5C에서 달성되지 않았습니다(그림 6B). 참고로, Capture Hi-C 및 5C 데이터의 절연 점수에 의해 결정된 토폴로지 경계는 일반적으로 약간 다른 위치와 다른 상대 강도로 감지됩니다.
또한, 접촉 루프와 같은 다른 서브-TAD 구조는 이전에 Capture-C63으로 식별된 Xist와 Ftx 사이의 루프(그림 7A) 및 최근에 Capture Hi-C48에 대해 유사한 프로토콜을 사용하여 식별된 Xist와 Xert 사이의 루프(그림 7B)와 같은 Capture Hi-C 데이터에서 명확하게 볼 수 있습니다. Linx, Chic1 및 Xite 유전자좌 사이의 Tsix-TAD 내에서 알려진 접촉 핫스팟을 형성하는 것과 같은 Capture Hi-C 프로파일의 증가된 분해능으로 인해 다른 접점도 더 정확하게 매핑할 수 있습니다(그림 7A).
그림 7에 표시된 Hi-C 데이터와 비교하여 Capture Hi-C는 분해능을 4배 증가시킬 수 있었지만 시퀀싱 깊이의 1/4(즉, 126M 판독 대 571M)만 필요했습니다(보충 표 1). 이러한 분해능 증가를 통해 그림 6 및 그림 7에 표시된 시퀀싱 깊이에서 Hi-C로 감지할 수 없는 subTAD 및 루핑 상호 작용을 감지할 수 있습니다. 따라서 Capture Hi-C에 대해 설명된 프로토콜은 이전 접근 방식과 비교할 때 대규모 게놈 관심 영역에 대한 훨씬 더 상세한 고해상도 특성화를 가능하게 합니다.
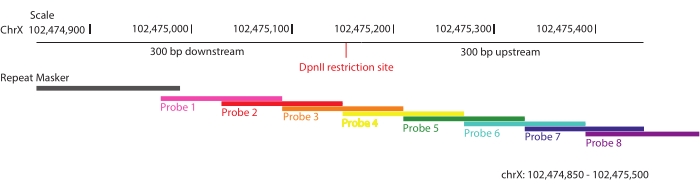
그림 1: 프로브 설계. 프로브 설계에 사용되는 전략의 개략도. 3Mb 표적 영역에 걸쳐 각 DpnII 제한 부위의 300bp 상류 및 하류 영역을 선택하고 중첩된 비오티닐화 RNA 프로브로 타일링했습니다. 이러한 선택된 영역 중 하나가 chrX: 102,474,805-102,475,500으로 표시됩니다. 각 프로브에는 40개 이하의 반복 염기가 허용됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
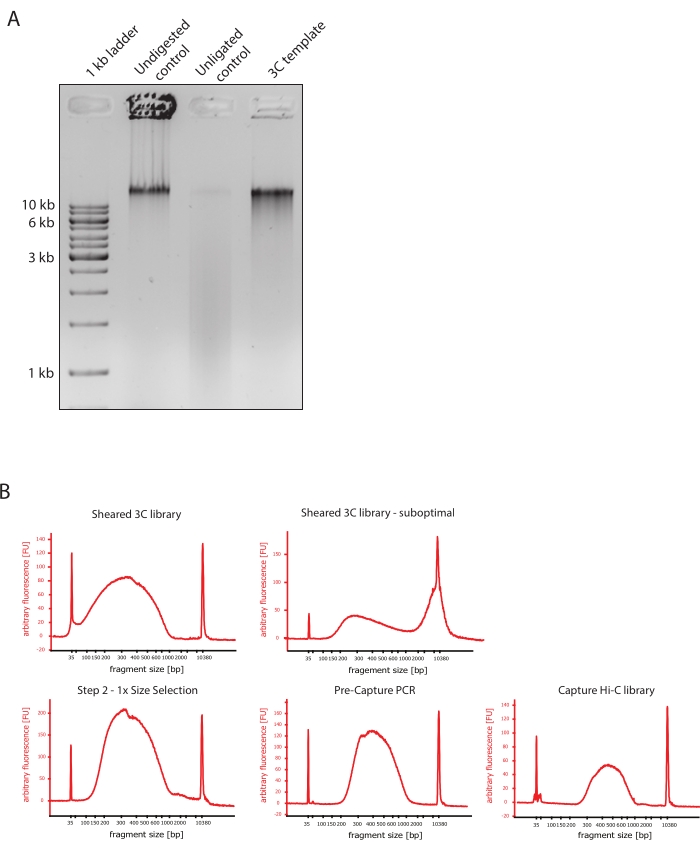
그림 2: Hi-C 사전 시퀀싱 품질 제어를 캡처합니다 . (A) 3C 템플릿 품질 관리의 대표적인 예. 200 ng의 DNA를 1% 아가로스 겔에 로딩하였다. 레인 1 : 1kb 사다리. 레인 2: 소화되지 않고 가교된 온전한 염색질은 >10kb에서 날카로운 띠로 실행됩니다. 레인 3: DpnII 소화 가교 염색질은 크기가 1kb에서 3kb 사이인 도말로 실행됩니다. 레인 4 : 최종 3C 라이브러리 또는 템플릿; 소화된 가교 결합된 DNA 단편의 자유 말단은 재결찰됩니다. 더 낮은 분자 크기의 DNA 도말은 거의 검출할 수 없으며 결찰 생성물은 >10kb의 밴드로 검출됩니다. (B) 고감도 바이오분석기 DNA 프로파일의 대표적인 예. 왼쪽 상단: 150bp에서 700bp 사이의 단편 크기 분포를 보여주는 성공적으로 전단된 3C 라이브러리. 오른쪽 상단: 불만족스러운 전단 3C 라이브러리. 전단되지 않은 DNA는 >2kb)의 광범위한 단편 농축으로 검출됩니다. (C) 왼쪽 하단: SPRI 비드를 사용하여 1:1 왼쪽 크기 선택 후 전단된 DNA 샘플. ~ 300 bp의 단편이 풍부합니다. 하단 중앙: 제조업체의 프로토콜에 따라 paired-end 어댑터를 결찰한 후 사전 캡처 PCR 프로필. 오른쪽 하단: 어댑터, 시퀀싱 및 멀티플렉스 시퀀싱을 위한 인덱싱 프라이머를 포함한 최종 Capture Hi-C 라이브러리. 약어: bp = 염기쌍, FU = 임의의 형광 단위. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
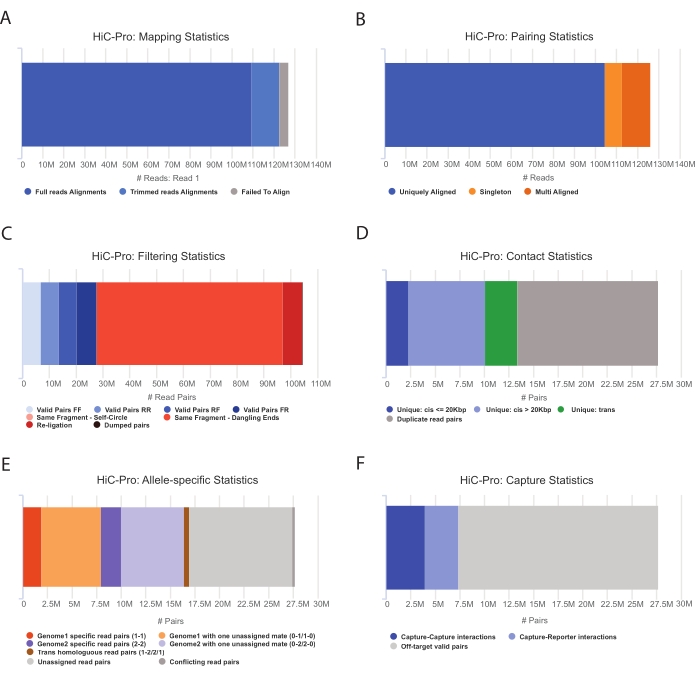
그림 3: HiC-Pro로 Hi-C 시퀀싱 후 품질 제어 캡처 . (A) 시퀀싱 쌍의 첫 번째 메이트에 대한 참조 게놈에 대한 매핑 속도의 예. 연한 파란색 분수는 HiC-Pro에 의해 정렬되고 결찰 접합부에 걸쳐 있는 판독값을 나타냅니다. 따라서 이 메트릭을 사용하여 실험적 결찰 단계를 검증할 수 있습니다. (B) 시퀀싱 메이트가 게놈에 정렬되면 분석을 위해 고유하게 정렬된 읽기 쌍만 유지됩니다. (C) dangling-end, self-circle 또는 re-ligation과 같은 유효하지 않은 쌍(빨간색)은 분석에서 제외됩니다. 유효한 쌍의 비율은 결찰 및 풀다운 효율의 좋은 지표입니다. (D) 유효한 쌍은 염색체 내/간 및 단/장거리 접촉으로 더 나눌 수 있습니다. PCR 아티팩트를 나타낼 가능성이 있는 중복된 읽기 쌍은 분석에서 삭제됩니다. (E) 대립유전자 특이적 분석을 위해 HiC-Pro는 각 부모 게놈(즉, C57BL/6J x CASTEi/J)에 대해 하나 또는 두 개의 메이트가 지원하는 대립유전자 판독 수를 보고합니다. 모계 및 부계 대립 유전자에 할당 된 동일한 비율의 판독이 예상됩니다. (F) 마지막으로, 캡처 영역과 겹치는 유효한 쌍만 선택되어 접촉 지도를 작성합니다. 캡처-캡처 쌍은 대상 영역 내의 접촉을 나타내는 반면, 캡처-리포터 쌍은 대상 영역과 대상을 벗어난 영역 간의 상호 작용을 포함합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
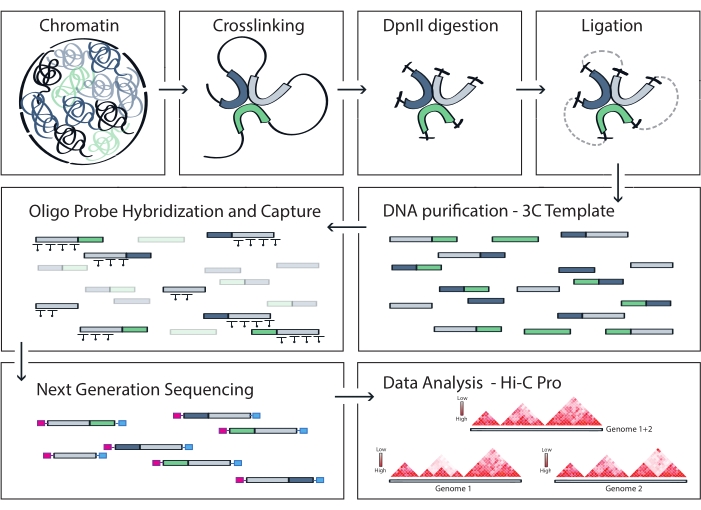
그림 4: Capture Hi-C 프로토콜의 워크플로. 다양한 프로토콜 단계의 개략도. 게놈 차원의 3C 주형을 생성하기 위해 염색질은 먼저 포름알데히드와 가교된 다음 DpnII 제한 효소로 분해됩니다. 그런 다음 유리 DNA 말단이 다시 결찰되고 가교가 역전되고 DNA가 정제됩니다. 표적 영역을 포함하는 단편을 풍부하게 하기 위해, 비오티닐화된 RNA 프로브의 어레이를 3C 주형에 혼성화하고 스트렙타비딘 매개 풀다운에 의해 포획합니다. 캡처 라이브러리는 멀티플렉스 시퀀싱을 위해 처리되며, 유효한 결찰 단편은 고분해능 상호 작용 맵으로 시각화되는 표적 전체의 염색질 접촉 빈도를 추론하기 위해 정량화됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
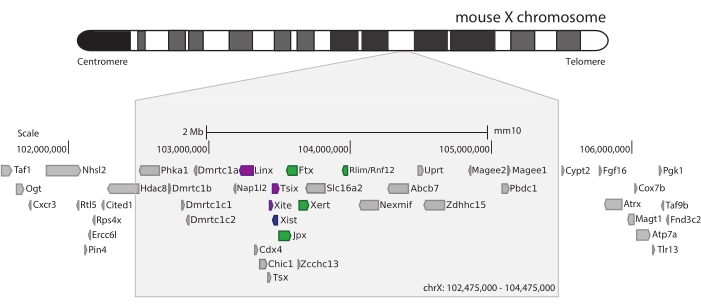
그림 5: 마우스 X 염색체에서 Xic 를 둘러싸고 있는 영역의 개요. 마우스 X 염색체의 개략도 표현 및 3Mb 캡처 영역의 확대(ChrX: 102,475,000-105,475,000). 표적 영역에는 XCI의 마스터 조절 유전자좌인 Xic에 해당하는 ~800kb의 DNA가 포함됩니다. Xic 에는 긴 비암호화 유전자인 Xist, XCI의 핵심 플레이어 및 규제 환경이 포함됩니다. Xist의 포지티브 레귤레이터는 녹색으로, 네거티브 레귤레이터는 보라색으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
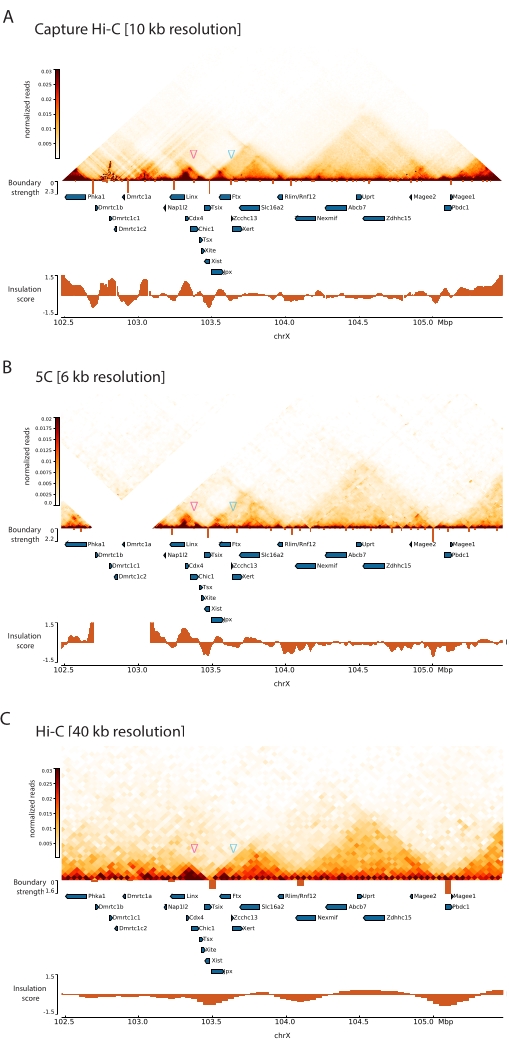
그림 6: 3Mb 캡처 영역에서 Hi-C, 5C 및 Hi-C 상호 작용 맵을 캡처합니다. (A) 마우스 Xic를 포함하는 3Mb 대상의 Hi-C 상호 작용 맵을 10kb 해상도로 캡처합니다(이 연구). (B) 6kb 해상도에서 A에서와 동일한 대상 영역의 5C 상호작용 맵(47에서 재처리된 데이터). 분석에 포함되지 않은 반복 영역은 흰색으로 마스킹됩니다. 5C 데이터는 자체 생물정보학 처리를 필요로 합니다(47 참조). 세척 및 정렬 후 프라이머 분해능의 5C 맵은 실행 중앙값(창 = 30kb, 단계 = 5)을 사용하여 비닝되어 6kb의 최종 분해능에 도달합니다. (C) 40kb 해상도에서 A 및 B와 동일한 게놈 영역의 Hi-C 상호작용 맵(64에서 재처리된 데이터). 모든 상호 작용 맵은 마우스 ESC에서 생성되었습니다. 절연 점수는 cooltools를 사용하여 계산되었으며 TAD 경계에서 절연 최소값이 있는 히스토그램으로 표시됩니다. TAD 경계는 맵 아래에 수직선으로 표시됩니다. 각 선의 높이는 경계 강도를 나타냅니다. 유전자는 전사 방향을 가리키는 화살표로 표시됩니다. Capture Hi-C 맵에서 독점적으로 또는 더 정확하게 검출되는 서브 TAD 경계는 Tsix 및 Xist TAD의 서브 TAD에 대해 각각 마젠타 및 파란색 화살촉으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
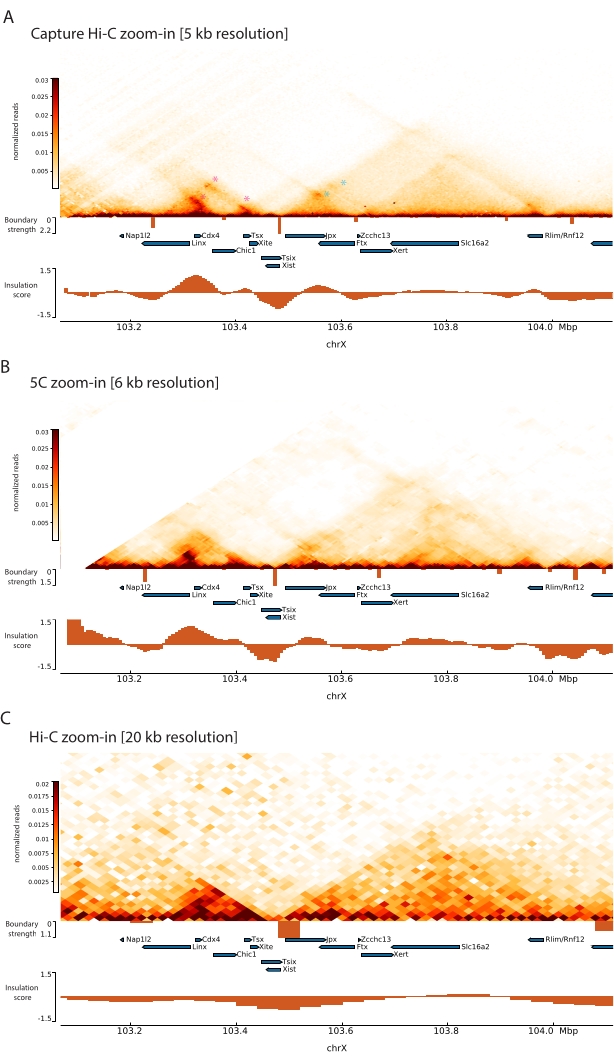
그림 7: 캡처된 영역 내에서 1Mb에 걸쳐 Hi-C, 5C 및 Hi-C 상호 작용 맵을 캡처합니다. (A) 마우스 Xic를 포함하는 1Mb 게놈 영역의 Hi-C 상호작용 맵을 5kb 해상도로 캡처합니다(이 연구). (B) A에서와 동일한 게놈 영역의 5C 상호작용 맵. 6KB 해상도(47에서 데이터 재처리). 분석에 포함되지 않은 반복 영역은 흰색으로 마스킹됩니다. 참고로, 5C 데이터는 자체 생물정보학 처리를 필요로 합니다(47 참조). 세척 및 정렬 후 프라이머 분해능의 5C 맵은 실행 중앙값(창 = 30kb, 단계 = 5)을 사용하여 비닝되어 6kb의 최종 분해능에 도달합니다. (C) 20kb 해상도에서 Hi-C의 A 및 B 와 동일한 게놈 영역의 Hi-C 상호작용 맵(64에서 재처리된 데이터). 모든 상호 작용 맵은 mESC에서 생성되었습니다. 절연 점수는 cooltools를 사용하여 계산되었으며 TAD 경계에서 절연 최소값이 있는 히스토그램으로 표시됩니다. TAD 경계는 맵 아래에 수직선으로 표시됩니다. 각 선의 높이는 경계 강도를 나타냅니다. 유전자는 전사 방향을 가리키는 화살표로 표시됩니다. Capture Hi-C에서 독점적으로 또는 더 정확하게 감지되는 접촉 루프는 Tsix 및 Xist TAD의 루프에 대해 각각 마젠타 및 파란색 별표로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 표 1: 이 원고에 사용된 데이터 세트에 대한 사후 시퀀싱 통계: Capture Hi-C(본 연구), Hi-C64 및 5C47. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
여기에서는 5-10kb 해상도에서 메가베이스 크기 게놈 영역의 고차 조직을 특성화하기 위한 비교적 빠르고 쉬운 Capture Hi-C 프로토콜을 설명합니다. Capture Hi-C는 게놈 전체 3C 또는 Hi-C 템플릿에서 표적 염색질 상호작용을 강화하도록 설계된 Capture-C 기술 제품군에 속합니다. 현재까지 대부분의 Capture-C 애플리케이션은 전체 게놈에 흩어져 있는 비교적 작은 조절 요소의 염색질 접촉을 매핑하는 데 활용되었습니다. 첫 번째 Capture-C 프로토콜에서, 다수의 중첩 RNA 비오티닐화 프로브를 사용하여 적혈구 세포로부터 제조된 3C 라이브러리에서 >400개의 미리 선택된 프로모터를 포획하였다31. 이후 NG(Next Generation) 및 NuTi(Nuclear Titrated) Capture-C에서도 동일한 전략이 개선되어 단일 제한 부위에 걸친 단일 120bp DNA 미끼와 2회의 순차적 포획을 사용하여 >8,000 프로모터의 고분해능 상호작용 프로파일을 달성하여 유익한 결찰 단편의 농축을 극대화했습니다32,40. 이러한 전략은 마우스 배아 발달, 세포 분화, X-염색체 불활성화 및 병리학적 상태에서의 유전자 오조절을 포함하여 다양한 맥락에서 시스 작용 요소의 기능적 해부로 이어졌습니다46,63,65,66,67,68,69,70,71.
프로모터 포획 Hi-C (PCHi-C)에서, 제한 단편을 함유하는 >22,000 주석 달린 프로모터는 제한 단편34,72의 한쪽 또는 양쪽 말단에서 단일 RNA 120-mer 비오티닐화 프로브의 혼성화에 의해 Hi-C 라이브러리로부터 풀다운되었다. 이 방법은 마우스 배아 줄기 세포, 태아 간 세포 및 지방 세포 34,35,72,73뿐만 아니라 인간 림프구 계통, 조혈 전구 세포, 표피 각질 세포 및 만능 세포 37,74,75,76,77.
이러한 표적 농축 기술과 비교하여 Capture Hi-C는 최대 메가베이스 규모까지 인접한 게놈 영역을 표적으로 하여 하나 이상의 TAD에 걸쳐 유전자의 조절 환경을 포괄합니다. 전체 관심 영역은 표적 내의 각 DpnII 제한 부위를 포함하는 비오티닐화 프로브 어레이로 바둑판식으로 배열되어야 합니다. 3C 템플릿에 대한 비오티닐화 어레이의 하이브리드화, 후속 스트렙타비딘 기반 포획 및 다중화 시퀀싱을 위한 처리는 Illumina Paired-End 다중화 시퀀싱을 위한 표적 농축 시스템을 사용하여 수행됩니다. 전체 프로토콜은 3C 라이브러리 준비에서 NGS 시퀀싱에 이르기까지 1주일 만에 수행할 수 있으며 약간의 조정 및/또는 맞춤형 문제 해결만 필요하기 때문에 빠릅니다.
이 프로토콜은 또한 다른 3C 기반 방법과 비교하여 이점을 제공합니다. 5-10kb의 해상도에서 상호 작용 맵을 얻기 위해 100-120M paired-end 읽기를 시퀀싱했습니다. 비교를 위해 여기서는 20kb 해상도64 (GSM2053973)에 도달하기 위해 571M 읽기의 Hi-C 데이터 세트를 사용했으며 염색체 전체 Hi-C22로 5kb 해상도에 도달하려면 최소 10억 개의 읽기가 필요합니다.
본 연구에서 사용된 포획 Hi-C는 6-bp 절단기 제한효소47 에 기초하여 이전에 발표된 5C보다 훨씬 더 높은 분해능에 도달한다(보충표 1). 중요하게도, 5C에서 표적 상호 작용을 풍부하게하고 증폭하도록 설계된 전략은 염색질 상호 작용의 대립 유전자 특이 적 분석을 허용하지 않습니다. 반대로, Capture Hi-C 데이터는 대립유전자-특이적으로 맵핑될 수 있어서, 예를 들어 인간 세포 또는 유전적으로 상이한 마우스 균주를 교배함으로써 유도된 F1 하이브리드 세포주에서 상동성 염색체 쌍의 3D 구조적 풍경을 해부할 수 있다78. 5kb 해상도에서 대립 유전자 특이적 Capture Hi-C 상호 작용 맵을 생성하기 위해 SNP 적용 범위를 늘리기 위해 150bp paired-end read를 시퀀싱했습니다. 유사한 대립유전자-특이적 접근법이 인간 세포주에 적용될 수 있으며, 이에 대해 SNP의 주석이 이용 가능하다22.
중요한 것은 Capture Hi-C가 일반적으로 염기서열 분석 비용의 경제성을 향상시키면서 고분해능을 보장하지만, 맞춤형 비오티닐화 올리고뉴클레오티드의 생산이 이 방법의 전체 비용에 영향을 미친다는 것입니다. 따라서 가장 적합한 3C 분석법의 선택은 응용 분야마다 다르며, 다루고 있는 생물학적 문제와 필요한 분해능, 관심 영역의 크기에 따라 달라집니다. 개발된 다른 Capture Hi-C 프로토콜은 여기에 설명된 프로토콜과 주요 기능을 공유합니다. 예를 들어, 유방암 및 결장직장암 위험과 관련된 비암호화 변이체에 걸쳐 ~50kb에서 1Mb 게놈 영역을 특성화하기 위해 Capture Hi-C 전략이 적용되었습니다. 이 프로토콜에서, 표적 영역은 3배 커버리지33,38,79에서 표적 영역을 타일링하는 120-mer RNA 미끼를 혼성화함으로써 Hi-C 라이브러리로부터 풀다운되었습니다. 유사하게, HYbrid 캡처 Hi-C (Hi-C 2)는 최대2 Mb80의 관심 영역 내에서 상호 작용을 타겟팅하는 데 사용되었습니다. 두 프로토콜 모두에서 비오틴 풀다운 결찰 단편에 대해 강화된 Hi-C 템플릿을 사용하면 프로토콜에 비해 총 정보 판독 비율이 증가했습니다. 예를 들어, 비교64(GSM2053973)를 위해 여기에서 사용한 Hi-C 데이터 세트에서 중복 제거 후 유효한 쌍의 백분율은 그림 3 및 보충 표 1에 설명된 대로 Capture Hi-C에서 얻은 유효한 쌍보다 4.8배 더 높습니다. 그러나 비오티닐화 결찰 단편 및 하이브리드화된 프로브의 연속적인 풀다운은 프로토콜을 훨씬 더 복잡하고 시간 소모적으로 만드는 동시에 포획된 영역의 복잡성을 감소시킬 수 있습니다.
타일링 프로브로 3C 템플릿을 풍부하게 하는 또 다른 방법은 Tiled-C로, 마우스 적혈구 분화 동안 높은 공간 및 시간 분해능에서 염색질 구조를 연구하는 데 적용되었습니다43. Tiled-C에서, 70 bp 비오티닐화 프로브의 패널은 표적 상호작용43,81의 매우 높은 해상도 맵을 생성하기 위해 2개의 연속 포획 라운드에서 대규모 영역 내의 접촉을 풍부하게 하는 데 사용된다. 또한 이중 캡처 강화는 Capture Hi-C와 비교할 때 프로토콜을 더 길고 복잡하게 만듭니다. 그러나, 단일 제한 부위를 표적으로 하는 Capture-C 전략과는 달리, Tiled-C에서 2차 포획은 포획 효율을 유의하게 증가시키지 않는 것으로 보이므로, 아마도 생략될 수 있다(43). 마지막으로, 이 연구에서 사용된 것과 동일한 표적 농축 전략을 기반으로 하는 유사한 타일링 접근 방식이 선천성 기형 환자에서 설명되고 형질전환 마우스에서 재설계된 구조적 변이를 포함하는 규제 환경의 해부에 적용되었습니다41,42. 이 경우, 프로브의 타일링 어레이는 DpnII 제한 부위(41)의 근접보다는 전체 표적에 걸쳐 설계되었다. 그럼에도 불구하고, 이 연구는 다양한 맥락에서 큰 게놈 영역의 고해상도 특성화를 달성하기 위한 이 전략의 민감도와 힘을 강조하는 데 중요했습니다(41,42,48).
결론적으로, 여기에 설명된 프로토콜은 관심 있는 모든 게놈 영역의 고해상도 3D 특성화를 위한 쉽고 강력하며 강력한 전략을 나타냅니다. 다양한 모델 시스템, 세포 유형, 발달 조절 염색질 풍경 및 건강하고 병리학적인 조건에서 유전자 조절에 이 접근 방식을 적용하면 후성 유전학 분야의 근본적인 미해결 질문 중 하나인 게놈 토폴로지와 유전자 조절 사이의 상호 작용 및 인과 관계에 대한 이해를 촉진할 수 있습니다. 또한, Capture Hi-C를 적용하여 GWAS 연구에 의해 확인된 위험 변이체의 장거리 상호 작용 및 고차 염색질 접힘을 매핑하면 다양한 맥락에서 인간 질병과 관련된 비암호화 게놈 유전자좌의 기능적 관련성을 밝힐 수 있는 잠재력이 있으며, 이를 통해 잠재적으로 발병의 기저에 있는 과정에 대한 새로운 통찰력을 제공할 수 있습니다.
공개
Kai Hauschulz는 Agilent Technologies - Diagnostic and Genomics Group의 현장 응용 과학자입니다. 다른 모든 저자는 경쟁 이익을 선언하지 않습니다.
감사의 말
허드 실험실에서의 작업은 유럽 연구위원회 (European Research Council) 고급 조사자 상 (XPRESS-AdG671027)의 지원을 받았습니다. AL은 유럽 연합 Marie Skłodowska-Curie Actions Individual Fellowship(IF-838408)의 지원을 받습니다. AH는 Marie Skłodowska-Curie Grant 계약 813327에 따라 ITN 혁신 및 학제 간 네트워크 ChromDesign의 지원을 받습니다. 저자는 유용한 기술 자문을 제공한 Daniel Ibrahim(MPI for Molecular Genetics, Berlin), Institut Curie(파리)의 NGS 플랫폼, 지원과 지원을 제공한 EMBL(Heidelberg)의 Vladimir Benes 및 Genomics Core Facility에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 10x PBS pH 7.4 | Gibco | 10010-023 | |
| 37% (vol/vol) paraformaldehyde solution | Electron Microscopy Sciences | 15686 | single use glass-vials; do not reuse |
| 50 mL PP conical tube | Falcon | 352070 | |
| Agarose | Sigma | A9539-500g | |
| Bioanalyzer | Agilent | G2939BA | |
| Cell Scrapers - 25 cm Handle and 3.0 cm Blade | Falcon | 353089 | |
| CHIR99021 | Axon Medchem BV | Axon 1386 | |
| cOmplete Mini, Protease inhibitor cocktail (EDTA-free) | Merck | 11836170001 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL | Invitrogen | ZGEXSCCOUNTESS2FL | Automated cell counter |
| Covaris S2 | Covaris | 500217 | Sonicator |
| DNA LoBind tube, 1.5 mL | Eppendorf | 30108051 | |
| DpnII (50000 units/mL) | New England Biolabs | R0543M | |
| Dulbecco's Modified Eagle Medium (DMEM) | Merck | D6429 | |
| Ethanol (100%) | Merck | 1.00983.2500 | |
| Fetal Bovine Serum (FBS) | Thermo Scientific | 10270106 | |
| gelatine from porcine skin | Sigma | G1890 | |
| GeneRuler 1 kb Plus DNA Ladder | Thermo Scientific | SM0313 | |
| GlycoBlue | Thermo Scientific | AM9516 | Coprecipitant |
| High-Sensitivity Bioanlayzer chips | Agilent | 5067-4626 | |
| Large Cooling Centrifuge 5920 R | Eppendorf | 5948000018 | |
| leukaemia inhibitory factor (LIF) | Merck | ESG1107 | |
| Liquiport | KNF | NF300 | Benchtop aspiration system |
| Low-binding filter tips | Biozym | VT0260U, VT0240, VT0220, VT0200U | |
| Molecular biology grade water | Merck | W3500-6x500ML | |
| Next Seq 500 | Illumina | SY-415-1001 | |
| Next Seq 500 High Output v2 Kit (300 cycles) | Illumina | FC-404-2004 | |
| Nonidet P40 Substitute (NP40) | Merck | 11332473001 | |
| PD0325901 | Axon Medchem BV | Axon 1408 | |
| Protease inhibitor cocktail (EDTA-free) | Merck | 11873580001 | |
| Proteinase K - recombinant, PCR-grade (20 mg/mL) | Thermo Scientific | EO0491 | |
| Qubit 2.0 | Thermo Scientific | Q32871 | |
| Qubit assay tubes | Thermo Scientific | Q32856 | |
| Qubit dsDNA High Sensitivity kit | Thermo Scientific | Q32851 | |
| RNase A (10 mg/mL) | Thermo Scientific | EN0531 | |
| Sodium acetate pH 5.2 (3M) | Merck | S7899 | |
| speed vacuum concentrator | Eppendorf | EP5305000100-1EA | |
| Agencourt AMPureXP | Beckman Coulter | A63881 | SPRI beads |
| SureSelect Target Enrichment Box 1 | Agilent | 5190-8645 | |
| SureSelect Target Enrichment Kit ILM Indexing Hyb Module Box 2 | Agilent | 5190-4455 | |
| SureSelect XT Library Prep Kit ILM | Agilent | 5500-0132 | |
| T4 ligase (30 units/µL) | Thermo Scientific | EL0013 | |
| table-top Centrifuge 5427 R | Eppendorf | 5409000012 | |
| Triton-X-100 (500 mL) | Merck | X100-500ML | |
| Trypan Blue | Invitrogen | T10282 | |
| Trypsine | Thermo Scientific | 25300054 | |
| UltraPure Glycine | Thermo Scientific | 15527013 | |
| β-mercaptoethanol | Thermo Scientific | 31350010 |
참고문헌
- Ibrahim, D. M., Mundlos, S. The role of 3D chromatin domains in gene regulation: a multi-facetted view on genome organization. Current Opinion in Genetics & Development. 61, 1-8 (2020).
- Bolt, C. C., Duboule, D. The regulatory landscapes of developmental genes. Development. 147 (3), (2020).
- Glaser, J., Mundlos, S. 3D or not 3D: Shaping the genome during development. Cold Spring Harbor Perspectives in Biology. 14 (5), 040188 (2021).
- Denker, A., De Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Kempfer, R., Pombo, A. Methods for mapping 3D chromosome architecture. Nature Reviews Genetics. 21 (4), 207-226 (2020).
- McCord, R. P., Kaplan, N., Giorgetti, L. Chromosome conformation capture and beyond: Toward an integrative view of chromosome structure and function. Molecular Cell. 77 (4), 688-708 (2020).
- Jerkovic, I., Cavalli, G. Understanding 3D genome organization by multidisciplinary methods. Nature ReviewsMolecular Cell Biology. 22 (8), 511-528 (2021).
- Hsieh, T. -. H. S., et al. Mapping nucleosome resolution chromosome folding in yeast by Micro-C. Cell. 162 (1), 108-119 (2015).
- Krietenstein, N., et al. Ultrastructural details of mammalian chromosome architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Naumova, N., Smith, E. M., Zhan, Y., Dekker, J. Analysis of long-range chromatin interactions using Chromosome Conformation Capture. Methods. 58 (3), 192-203 (2012).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Zhao, Z., et al. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra-and interchromosomal interactions. Nature Genetics. 38 (11), 1341-1347 (2006).
- Würtele, H., Chartrand, P. Genome-wide scanning of HoxB1-associated loci in mouse ES cells using an open-ended Chromosome Conformation Capture methodology. Chromosome Research. 14 (5), 477-495 (2006).
- De Wit, E., De Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes & Development. 26 (1), 11-24 (2012).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Splinter, E., et al. The inactive X chromosome adopts a unique three-dimensional conformation that is dependent on Xist RNA. Genes & Development. 25 (13), 1371-1383 (2011).
- Ferraiuolo, M. A., Sanyal, A., Naumova, N., Dekker, J., Dostie, J. From cells to chromatin: capturing snapshots of genome organization with 5C technology. Methods. 58 (3), 255-267 (2012).
- Kim, J. H., et al. 5C-ID: Increased resolution Chromosome-Conformation-Capture-Carbon-Copy with in situ 3C and double alternating primer design. Methods. 142, 39-46 (2018).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Zhang, Y., et al. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations. Cell. 148 (5), 908-921 (2012).
- Rao, S. S. P., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Dixon, J. R., et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485 (7398), 376-380 (2012).
- Nora, E. P., et al. Spatial partitioning of the regulatory landscape of the X-inactivation centre. Nature. 485 (7398), 381-385 (2012).
- Krefting, J., Andrade-Navarro, M. A., Ibn-Salem, J. Evolutionary stability of topologically associating domains is associated with conserved gene regulation. BMC Biology. 16 (1), 87 (2018).
- Galupa, R., Heard, E. Topologically associating domains in chromosome architecture and gene regulatory landscapes during development, disease, and evolution. Cold Spring Harbor Symposia on Quantitative Biology. 82, 267-278 (2017).
- Tena, J. J., Santos-Pereira, J. M. Topologically associating domains and regulatory landscapes in development, evolution and disease. Frontiers in Cell and Developmental Biology. 9, 702787 (2021).
- Lupiáñez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How alterations of chromatin domains result in disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Davidson, I. F., Peters, J. -. M. Genome folding through loop extrusion by SMC complexes. Nature Reviews Molecular Cell Biology. 22 (7), 445-464 (2021).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews Molecular Cell Biology. 17 (12), 743-755 (2016).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Davies, J. O. J., et al. Multiplexed analysis of chromosome conformation at vastly improved sensitivity. Nature Methods. 13 (1), 74-80 (2016).
- Jäger, R., et al. Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci. Nature Communications. 6, 6178 (2015).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Sahlén, P., et al. Genome-wide mapping of promoter-anchored interactions with close to single-enhancer resolution. Genome Biology. 16, 156 (2015).
- Joshi, O., et al. Dynamic reorganization of extremely long-range promoter-promoter interactions between two states of pluripotency. Cell Stem Cell. 17 (6), 748-757 (2015).
- Mifsud, B., et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nature Genetics. 47 (6), 598-606 (2015).
- Dryden, N. H., et al. Unbiased analysis of potential targets of breast cancer susceptibility loci by Capture Hi-C. Genome Research. 24 (11), 1854-1868 (2014).
- Oudelaar, A. M., Davies, J. O. J., Downes, D. J., Higgs, D. R., Hughes, J. R. Robust detection of chromosomal interactions from small numbers of cells using low-input Capture-C. Nucleic Acids Research. 45 (22), 184 (2017).
- Oudelaar, A. M., et al. Single-allele chromatin interactions identify regulatory hubs in dynamic compartmentalized domains. Nature Genetics. 50 (12), 1744-1751 (2018).
- Franke, M., et al. Formation of new chromatin domains determines pathogenicity of genomic duplications. Nature. 538 (7624), 265-269 (2016).
- Despang, A., et al. Functional dissection of the Sox9-Kcnj2 locus identifies nonessential and instructive roles of TAD architecture. Nature Genetics. 51 (8), 1263-1271 (2019).
- Oudelaar, A. M., et al. Dynamics of the 4D genome during in vivo lineage specification and differentiation. Nature Communications. 11 (1), 1-12 (2020).
- Galupa, R., Heard, E. X-chromosome inactivation: A crossroads between chromosome architecture and gene regulation. Annual Review of Genetics. 52, 535-566 (2018).
- Loda, A., Collombet, S., Heard, E. Gene regulation in time and space during X-chromosome inactivation. Nature Reviews. Molecular Cell Biology. 23 (4), 231-249 (2022).
- van Bemmel, J. G., et al. The bipartite TAD organization of the X-inactivation center ensures opposing developmental regulation of Tsix and Xist. Nature Genetics. 51 (6), 1024-1034 (2019).
- Galupa, R., et al. A conserved noncoding locus regulates random monoallelic Xist expression across a topological boundary. Molecular Cell. 77 (2), 352-367 (2020).
- Gjaltema, R. A. F., et al. Distal and proximal cis-regulatory elements sense X chromosome dosage and developmental state at the Xist locus. Molecular Cell. 82 (1), 190-208 (2022).
- Galupa, R., et al. Inversion of a topological domain leads to restricted changes in its gene expression and affects inter-domain communication. Development. 149 (9), (2022).
- Savarese, F., Flahndorfer, K., Jaenisch, R., Busslinger, M., Wutz, A. Hematopoietic precursor cells transiently reestablish permissiveness for X inactivation. Molecular and Cellular Biology. 26 (19), 7167-7177 (2006).
- Schulz, E. G., et al. The two active X chromosomes in female ESCs block exit from the pluripotent state by modulating the ESC signaling network. Cell Stem Cell. 14 (2), 203-216 (2014).
- Gnirke, A., et al. Solution hybrid selection with ultra-long oligonucleotides for massively parallel targeted sequencing. Nature Biotechnology. 27 (2), 182-189 (2009).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Piccinini, F., Tesei, A., Arienti, C., Bevilacqua, A. Cell counting and viability assessment of 2D and 3D Cell cultures: Expected reliability of the trypan blue assay. Biological Procedures Online. 19 (1), 8 (2017).
- Servant, N., et al. HiC-Pro: an optimized and flexible pipeline for Hi-C data processing. Genome Biology. 16, 259 (2015).
- Abdennur, N., Mirny, L. A. Cooler: scalable storage for Hi-C data and other genomically labeled arrays. Bioinformatics. 36 (1), 311-316 (2020).
- Imakaev, M., et al. Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nature Methods. 9 (10), 999-1003 (2012).
- Forcato, M., et al. Comparison of computational methods for Hi-C data analysis. Nature Methods. 14 (7), 679-685 (2017).
- Venev, S., et al. . open2c/cooltools: v0.4.1. , (2021).
- Wages, J. M. NUCLEIC ACIDS | Immunoassays. Encyclopedia of Analytical Science. , 408-417 (2005).
- Ewels, P. A., et al. The nf-core framework for community-curated bioinformatics pipelines. Nature Biotechnology. 38 (3), 276-278 (2020).
- Servant, N., Peltzer, A. nf-core/hic: Initial release of nf-core/hic. Zenodo. , (2019).
- Furlan, G., et al. The Ftx noncoding locus controls X chromosome inactivation independently of its RNA products. Molecular Cell. 70 (3), 462-472 (2018).
- Giorgetti, L., et al. Structural organization of the inactive X chromosome in the mouse. Nature. 535 (7613), 575-579 (2016).
- Simon, C. S., et al. Functional characterisation of cis-regulatory elements governing dynamic Eomes expression in the early mouse embryo. Development. 144 (7), 1249-1260 (2017).
- Williams, R. M., et al. Reconstruction of the global neural crest gene regulatory network in vivo. Developmental Cell. 51 (2), 255-276 (2019).
- Godfrey, L., et al. DOT1L inhibition reveals a distinct subset of enhancers dependent on H3K79 methylation. Nature Communications. 10 (1), 2803 (2019).
- Hanssen, L. L. P., et al. Tissue-specific CTCF-cohesin-mediated chromatin architecture delimits enhancer interactions and function in vivo. Nature Cell Biology. 19 (8), 952-961 (2017).
- Larke, M. S. C., et al. Enhancers predominantly regulate gene expression during differentiation via transcription initiation. Molecular Cell. 81 (5), 983-997 (2021).
- Oudelaar, A. M., et al. A revised model for promoter competition based on multi-way chromatin interactions at the α-globin locus. Nature Communications. 10 (1), 5412 (2019).
- Long, H. K., et al. Loss of extreme long-range enhancers in human neural crest drives a craniofacial disorder. Cell Stem Cell. 27 (5), 765-783 (2020).
- Schoenfelder, S., Javierre, B. -. M., Furlan-Magaril, M., Wingett, S. W., Fraser, P. Promoter Capture Hi-C: High-resolution, genome-wide profiling of promoter interactions. Journal of Visualized Experiments. (136), e57320 (2018).
- Siersbæk, R., et al. Dynamic rewiring of promoter-anchored chromatin loops during adipocyte differentiation. Molecular Cell. 66 (3), 420-435 (2017).
- Rubin, A. J., et al. Lineage-specific dynamic and pre-established enhancer-promoter contacts cooperate in terminal differentiation. Nature Genetics. 49 (10), 1522-1528 (2017).
- Freire-Pritchett, P., et al. Global reorganisation of cis-regulatory units upon lineage commitment of human embryonic stem cells. eLife. 6, 21926 (2017).
- Javierre, B. M., et al. Lineage-specific genome architecture links enhancers and non-coding disease variants to target gene promoters. Cell. 167 (5), 1369-1384 (2016).
- Miguel-Escalada, I., et al. Human pancreatic islet three-dimensional chromatin architecture provides insights into the genetics of type 2 diabetes. Nature Genetics. 51 (7), 1137-1148 (2019).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Baxter, J. S., et al. Capture Hi-C identifies putative target genes at 33 breast cancer risk loci. Nature Communications. 9 (1), 1028 (2018).
- Sanborn, A. L., et al. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes. Proceedings of the National Academy of Sciences. 112 (47), 6456-6465 (2015).
- Owens, D. D. G., et al. Dynamic Runx1 chromatin boundaries affect gene expression in hematopoietic development. Nature Communications. 13 (1), 773 (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유