Method Article
Yüksek Çözünürlüklü 3D Kromatin Organizasyonunun Capture Hi-C ile Deşifre Edilmesi
Bu Makalede
Özet
Bu protokol, topolojik olarak ilişkili alanların (TAD'ler) sınırları ve düzenleyici ve diğer DNA dizisi elemanları arasındaki uzun menzilli kromatin etkileşimleri de dahil olmak üzere, mega tabanlı boyutlu hedeflenen genomik bölgelerin 3D organizasyonunu yüksek çözünürlükte karakterize etmek için kullanılan Capture Hi-C yöntemini açıklar.
Özet
Genomun mekansal organizasyonu, transkripsiyon, replikasyon, rekombinasyon ve onarım dahil olmak üzere birçok bağlamda işlevine ve düzenlemesine katkıda bulunur. Bu nedenle, genom topolojisi ve fonksiyon arasındaki kesin nedenselliği anlamak çok önemlidir ve giderek daha yoğun araştırmaların konusudur. Kromozom konformasyon yakalama teknolojileri (3C), genomun herhangi bir bölgesi arasındaki etkileşimlerin sıklığını ölçerek kromatinin 3D yapısını çıkarmaya izin verir. Burada, mega tabanlı genomik hedeflerin alele özgü 3D organizasyonunu yüksek çözünürlükte karakterize eden 3C tabanlı bir hedef zenginleştirme yöntemi olan Capture Hi-C'yi gerçekleştirmek için hızlı ve basit bir protokol açıklıyoruz. Capture Hi-C'de hedef bölgeler, aşağı akış yüksek verimli dizilemeden önce bir dizi biyotinile prob tarafından yakalanır. Böylece, teknolojinin zaman etkinliğini ve satın alınabilirliğini geliştirirken daha yüksek çözünürlük ve alele özgüllük elde edilir. Güçlü yönlerini göstermek için, Capture Hi-C protokolü, X-kromozom inaktivasyonunun (XCI) ana düzenleyici lokusu olan fare X-inaktivasyon merkezine (Xic) uygulandı.
Giriş
Doğrusal genom, bir organizmanın embriyonik gelişimden geçmesi ve yetişkinlik boyunca hayatta kalması için gerekli tüm bilgileri tutar. Bununla birlikte, genetik olarak özdeş hücrelere farklı işlevleri yerine getirmeleri talimatını vermek, farklı dokular ve / veya gelişim aşamaları da dahil olmak üzere belirli bağlamlarda hangi bilgilerin kullanıldığını doğru bir şekilde kontrol etmek için esastır. Genomun üç boyutlu organizasyonunun, doğrusal genomda birkaç yüz kilobaz ile ayrılabilen düzenleyici elemanlar arasındaki fiziksel etkileşimi kolaylaştırarak veya önleyerek gen aktivitesinin bu doğru mekansal-zamansal düzenlemesine katıldığı düşünülmektedir (incelemeler için 1,2,3). Son 20 yılda, genom katlanması ve aktivitesi arasındaki etkileşimi anlamamız, büyük ölçüde kromozom konformasyon yakalama teknolojilerinin (3C) gelişmesi nedeniyle hızla artmıştır (inceleme için 4,5,6,7). Bu yöntemler, genomun herhangi bir bölgesi arasındaki etkileşimlerin sıklığını ölçer ve çekirdek içinde yakın 3B yakınlıkta bulunan DNA dizilerinin bağlanmasına dayanır. En yaygın 3C protokolleri, hücre popülasyonlarının formaldehit gibi çapraz bağlayıcı bir ajanla sabitlenmesiyle başlar. Çapraz bağlı kromatin daha sonra bir kısıtlama enzimi ile sindirilir, ancak MNaz sindirimi de 8,9 kullanılmıştır. Sindirimden sonra, yakın uzamsal yakınlıkta serbest DNA uçları yeniden bağlanır ve çapraz bağlanma tersine çevrilir. Bu adım, çekirdeğe 3B yakınlığında olan dizilerin aynı DNA parçasına bağlanma şansının daha yüksek olduğu karışık bir melez parça havuzu olan 3C 'kütüphanesi' veya 'şablonu'na yol açar. Bu hibrid fragmanların aşağı akış nicelleştirilmesi, doğrusal genomda binlerce baz çifti ayrı ayrı bulunan ancak 3B uzayda etkileşime girebilecek genomik bölgelerin 3B konformasyonunu çıkarmayı sağlar.
3C kütüphanesini karakterize etmek için, hem ligasyon parçalarının hangi alt kümelerinin analiz edildiği hem de aşağı akış nicelleştirmesi için hangi teknolojinin kullanıldığı açısından farklılık gösteren birçok farklı yaklaşım geliştirilmiştir. Orijinal 3C protokolü, iki ilgi alanının seçimine ve PCR10,11 ile 'bire bir' etkileşim frekanslarının ölçülmesine dayanıyordu. 4C yaklaşımı (dairesel kromozom konformasyonu yakalama), tek bir ilgi odağı (yani, 'bakış açısı') ile genomun geri kalanı ('bir tanesine karşı hepsi') arasındaki etkileşimleri ölçer12,13,14. 4C'de, 3C kütüphanesi, bakış açısına özgü primerler15 tarafından güçlendirilmiş PCR olan küçük dairesel DNA molekülleri üretmek için ikinci bir sindirim ve yeniden bağlanma turuna uğrar. 5C (kromozom konformasyonu yakalama karbon kopyası), daha büyük ilgi alanlarındaki 3D etkileşimlerin karakterizasyonunu sağlar ve bu bölgedeki daha yüksek dereceli kromatin katlanmasına ilişkin içgörüler sağlar ("çoğuna karşı çok")16. 5C'de, 3C kütüphanesi, daha sonra evrensel primerler15 ile multipleks PCR ile güçlendirilebilen kısıtlama bölgeleriyle örtüşen oligonükleotidlerden oluşan bir havuza hibritleştirilir. Hem 4C hem de 5C'de, bilgilendirici DNA fragmanları başlangıçta mikrodiziler ve daha sonra yeni nesil dizileme (NGS) 17,18,19 ile ölçülmüştür. Bu stratejiler hedeflenen ilgi alanlarını karakterize eder, ancak genom çapındaki etkileşimleri haritalamak için uygulanamaz. Bu ikinci hedefe, 3C şablonunun büyük ölçüde paralel diziliminin, genom çapında ("hepsine karşı") kromatin katlanmasının tarafsız karakterizasyonuna izin verdiği 3C tabanlı yüksek verimli bir strateji olan Hi-C ile ulaşılır20. Hi-C protokolü, sindirilmiş parçaların uçlarına biyotinile edilmiş bir kalıntının dahil edilmesini içerir, bunu takiben, bağlı parçaların geri kazanımını arttırmak için streptavidin boncukları ile ligasyon parçalarının aşağı çekilmesi takip edilir20.
Hi-C, memeli genomlarının 3D çekirdekte yapısal olarak çoklu ölçeklerde organize olduğunu ortaya koydu. Megabaz ölçeğinde, genom aktif ve inaktif kromatin bölgelerine, sırasıyla A ve B bölmelerine,sırasıyla 20,21'e ayrılır. Farklı kromatin ve aktivite durumları ile temsil edilen diğer alt bölmelerin varlığı da daha sonra gösterilmiştir22. Daha yüksek çözünürlükte, genom ayrıca topolojik olarak ilişkilendiren alanlar (TAD'lar) adı verilen alt megabaz kendi kendine etkileşime giren alanlara bölünür ve ilk olarak insan ve fare genomlarının Hi-C ve 5C analizi ile ortaya çıkar23,24. Dokuya özgü bir şekilde değişen bölmelerin aksine, TAD'lar sabit olma eğilimindedir (birçok istisna olmasına rağmen). Daha da önemlisi, TAD sınırlarıtür 25 arasında korunmaktadır. Memeli hücrelerinde, TAD'lar sıklıkla aynı düzenleyici manzarayı paylaşan genleri kapsar ve komşu düzenleyici alanlarla etkileşimleri sınırlarken gen birlikte düzenlemesini kolaylaştıran yapısal bir çerçeveyi temsil ettiği gösterilmiştir (gözden geçirme için 3,26,27,28). Ayrıca, TAD'lar içinde, kohesin-ekstrüde döngülerin tabanındaki CTCF bölgelerine bağlı etkileşimler, promotör-arttırıcı veya arttırıcı-arttırıcı etkileşimlerinin olasılığını artırabilir (gözden geçirme29 için).
Hi-C'de, bölmeler ve TAD'lar 1 Mb ila 40 kb çözünürlükte algılanabilir, ancak 5-10 kb ölçeğinde distal elemanlar arasındaki döngü etkileşimleri gibi daha küçük ölçekli temasları karakterize etmek için daha yüksek çözünürlük elde edilebilir. Bununla birlikte, bu tür döngüleri HiC ile verimli bir şekilde tespit edebilmek için çözünürlüğün arttırılması, sıralama derinliğinde ve dolayısıyla sıralama maliyetlerinde önemli bir artış gerektirir. Analizin alele özgü olması gerekiyorsa bu durum daha da kötüleşir. Gerçekten de, çözünürlükteki bir X-kat artışı, dizileme derinliğinde bir X2 artışı gerektirir, bu da yüksek çözünürlüklü ve alele özgü genom çapında yaklaşımların engelleyici derecede pahalı olabileceği anlamına gelir30.
Yüksek çözünürlüğü korurken maliyet etkinliğini ve satın alınabilirliği artırmak için, ilgilenilen hedef bölgeler, aşağı akış diziliminden önce tamamlayıcı biyotin etiketli oligonükleotid probları ile hibridizasyonlarını takiben genom çapında 3C veya Hi-C kütüphanelerinden fiziksel olarak aşağı çekilebilir. Bu hedef zenginleştirme stratejileri Capture-C yöntemleri olarak adlandırılır ve genom boyunca dağılmış yüzlerce hedef lokusun etkileşimlerinin sorgulanmasına izin verir (yani, Promoter Capture (PC) Hi-C; Yeni Nesil (NG) Yakalama-C; Düşük Giriş (LI) Yakalama-C; Nükleer Titrated (NuTi) Yakalama-C; Tri-C)31,32,33,34,35,36,37,38,39,40 veya birkaç megabaza kadar uzanan bölgeler arasında (yani, HiC'yi Yakalamak; HYbrid Yakalama Hi-C (Hi-C2); Kiremitli-C)41,42,43. Yakalama tabanlı yöntemlerde iki yön değişebilir: (1) biyotinile oligonükleotidlerin doğası ve tasarımı (yani, RNA veya DNA, dağınık genomik hedefleri yakalayan tek oligolar veya ilgi çekici bir bölgeyi döşeyen çoklu oligolar); ve (2) 3C veya Hi-C kütüphanesi olabilen hedefleri aşağı çekmek için kullanılan şablon, ikincisi 3C kütüphanesinden aşağı çekilen biyotinile kısıtlama parçalarından oluşur.
Burada, 3C kütüphanesinden hedef kişilerin zenginleştirilmesine dayanan bir Capture Hi-C protokolü açıklanmaktadır. Protokol, biyotinile RNA problarının özel olarak uyarlanmış bir döşeme dizisinin tasarımına dayanır ve 3C kütüphanesi hazırlığından NGS dizilemesine kadar 1 hafta içinde gerçekleştirilebilir. Protokol hızlı, basittir ve diğer 3C yöntemlerine kıyasla zaman etkinliğini ve satın alınabilirliği artırırken, 5 kb çözünürlükte megabaz boyutundaki ilgi alanlarının daha yüksek dereceli 3D organizasyonunu karakterize etmeyi sağlar. Capture Hi-C protokolü, Xist kodlamayan RNA'yı barındıran X-kromozom inaktivasyonunun (XCI) ana düzenleyici lokusuna, X-inaktivasyon merkezine (Xic) uygulandı. Xic daha önce kapsamlı yapısal ve fonksiyonel analizlere konu olmuştur (inceleme için44,45). Memelilerde, XCI, dişiler (XX) ve erkekler (XY) arasındaki X'e bağlı genlerin dozajını telafi eder ve dişi hücrelerdeki iki X kromozomundan birinin neredeyse tamamının transkripsiyonel susturulmasını içerir. Xic, 3D genom topolojisindeki çalışmalar ve gen regülasyonu44 ile etkileşim için güçlü, altın standart bir lokusu temsil etmiştir. Fare embriyonik kök hücrelerinde (mESC'ler) Xic'in 5C analizi, TAD'ların keşfedilmesine ve adlandırılmasına yol açarak, topolojik bölümleme ve gen ortak düzenlemesinin işlevsel alaka düzeyine dair ilk bilgileri sağladı24. Xic'in topolojik organizasyonunun daha sonra Xist upregülasyonunun ve XCI 46'nın uygun gelişimsel zamanlamasına kritik olarak dahil olduğu gösterildi ve TAD'lar içindeki ve arasındaki gen aktivitesini etkileyebilecek beklenmedik cis-düzenleyici unsurlar da yakın zamanda Xic47,48,49 içinde keşfedildi. Xic'i kapsayan fare X kromozomunun 3 Mb'ına Capture Hi-C uygulamak, bu yaklaşımın büyük ölçekli kromatin katlanmasını yüksek çözünürlükte parçalamadaki gücünü gösterir. İlgilenilen bölgedeki her DpnII kısıtlama bölgesinde biyotinile prob dizisinin tasarımından başlayarak, genom çapında 3C kütüphanesinin oluşturulmasına, hedef temasların hibridizasyonuna ve yakalanmasına ve aşağı akış veri analizine kadar ayrıntılı ve takip edilmesi kolay bir protokol sağlanmaktadır. Uygun kalite kontrollerine ve beklenen sonuçlara genel bir bakış da dahil edilmekte ve yaklaşımın hem güçlü hem de sınırlı yönleri benzer mevcut yöntemler ışığında tartışılmaktadır.
Protokol
Bu çalışmada kullanılan fare embriyonik kök hücreleri (mESC'ler), Institut Curie (Paris)51'in hayvan bakım kılavuzlarına göre bir TX/TX R26rtTA/rtTA dişi50 ile Mus musculus castaneus erkeğinin bir haçından türetilmiştir.
1. Prob tasarımı
- İlgilenilen hedef bölgeyi kapsayan bir dizi biyotinile prob (120-mer RNA oligonükleotid) tasarlayın.
- İlgilenilen bölgeyi üst üste binen oligonükleotidlerle döşeyin, böylece hedef içindeki her sekans ortalama olarak iki benzersiz prob (2x kapsama) tarafından kaplanır (Şekil 1).
- Spesifik olmayan etkileşimlerin zenginleşmesini önlemek için tekrarlayan dizileri prob kapsamının dışında tutun.
NOT: Bilgilendirici ligasyon parçalarının zenginleştirilmesini en üst düzeye çıkarmak için, hedef boyunca her bir DpnII kısıtlama bölgesinin yukarı ve aşağı yönünde 300 bp yukarı ve aşağı akış yayılan bölgeler tanımlanmıştır (ChrX: 102.475.000-105.475.000) ve Sure Design platformu52 aracılığıyla SureSelect DNA hedef zenginleştirme teknolojisine göre 28.913 biyotinile prob tasarlanmıştır. Bu stratejiye göre, spesifik olmayan etkileşimlerin zenginleşmesini en aza indirmek için her oligonükleotidde maksimum 40 baza kadar tekrarlayan dizilere izin verilir. Prob dizisi Agilent tarafından sentezlendi. Burada, DpnII iki nedenden dolayı bir kısıtlama enzimi olarak kullanılır: (1) birkaç 3C tabanlı yöntemde rutin olarak kullanılan dört kesicidir53; ve (2) bu çalışmada kullanılan F1 hibrid hatlarında (C57BL / 6J x CASTEi / J) siliko olarak test edilen diğer kısıtlama enzimlerine kıyasla, kesme bölgelerinin yakınında bilgilendirici tek nükleotid polimorfizmlerini (SNP'ler) yakalama şansını en üst düzeye çıkarır.
2. Deneysel prosedür
- Hücre hazırlama
- Fiksasyon gününde toplam hücre sayısı ≥ 5 x 107 hücre elde etmek için bir veya daha fazla hücre kültürü plakasına uygun sayıda hücre tohumlayın.
NOT: Bu çalışmada fare embriyonik kök hücreleri (mESC'ler) kullanılmıştır. mESC'ler, 2i + LIF ve toplu olarak test edilmiş fetal buzağı serumu (DMEM, %15 FBS, 0.1 mM β-merkaptoetanol, 1.000 U/mL−1 lösemi inhibitör faktörü (LIF), CHIR99021 (3 μM) ve PD0325901 (1 μM)) içeren mESC'ler ortamında jelatinize (%0,1 PBS - 37 °C'de o/n, %5 CO2 inkübatör) hücre kültürü plakaları üzerine kaplanır. Bu hücre tipi için,% 80 birleşik 10 cm'lik bir plaka yaklaşık 2 x 107 hücre içerir. - Hücre sayımı için ek bir hücre kültürü plakası hazırlayın.
NOT: Medya kullanımını azaltmak için daha küçük bir hücre kültürü plakası kullanılabilir. Bu durumda, daha küçük plakaya tohumlanacak hücre sayısının buna göre ayarlanması gerekir (örneğin, 10 cm'lik bir plakada 15 cm'lik bir plakaya kıyasla 3 kat daha az hücre).
- Fiksasyon gününde toplam hücre sayısı ≥ 5 x 107 hücre elde etmek için bir veya daha fazla hücre kültürü plakasına uygun sayıda hücre tohumlayın.
- Formaldehit fiksasyonu
- Çapraz bağlanacak toplam hücre sayısını tahmin edin.
- Çapraz bağlama reaksiyonuna başlamadan önce, üreticinin talimatlarına göre otomatik bir hücre sayacı kullanarak hücre sayımı için özel olarak hazırlanan kontrol plakasındaki hücreleri deneyin ve sayın.
- Canlı hücrelerin yüzdesini belirlemek için canlı bir boyama (örneğin, Tripan Mavisi) ekleyin54. Bu hücre sayısından, çapraz bağlanma için hazırlanan plaka (lar) daki toplam hücre sayısını tahmin edin.
- Kültür ortamını çapraz bağlanma için hazırlanan plakalardan çıkarın ve uygun miktarda fiksasyon çözeltisi (hücre kültürü ortamında% 2 formaldehit) ile değiştirin. 10 cm'lik bir plaka üzerinde 10 mL kullanın (örneğin, 15 cm'lik bir plaka için ~20 mL).
NOT: Tam bir sabitleme çözeltisi hacmi ekleyin. Yapışkan hücrelerin sabitlenmesi mümkün değilse, bu adım tripnize hücrelere uyarlanabilir ve 50 mL konik santrifüj tüplerinde 30 mL fiksasyon çözeltisinde gerçekleştirilebilir. Formaldehit 1 yaşından büyük olmamalıdır. Tek kullanımlık şişelerin kullanılması tercih edilir. Fiksasyon çözeltisi kullanımdan önce oda sıcaklığına (RT) getirilmelidir.
DİKKAT: Formaldehit tehlikelidir ve uygun sağlık ve güvenlik yönetmeliklerine göre ele alınması gerekir. - RT'de bir çalkalayıcı üzerinde nazik karıştırma altında 10 dakika boyunca sabitleyin.
- Fiksasyon reaksiyonunu, 0,125 M'lik bir nihai konsantrasyona 2,5 M glisin-1x PBS ekleyerek söndürün. 10 cm'lik bir plaka üzerinde 10 mL'ye 530 μL 2,5 M glisin-1x PBS ekleyin (örneğin, 15 cm'lik bir plaka üzerinde 1060 μL ila 20 mL).
NOT: Hücreler çözelti içinde sabitlenmişse, fiksasyon reaksiyonunu 1590 μL 2.5 M glisin-1x PBS ile söndürün. - RT'de 5 dakika boyunca inkübe edin, bir çalkalayıcı üzerinde hafifçe karıştırın.
- Plakaları buza aktarın ve bir çalkalayıcıda hafifçe karıştırırken buz üzerinde 15 dakika daha inkübe edin.
NOT: Şu andan itibaren, hücreler buz üzerinde tutulmalı ve daha fazla çapraz bağlanmayı önlemek için tamponlar önceden soğutulmalıdır. Çok sayıda plakanın işlenmesi gerekiyorsa soğuk bir odaya geçin. - Hızlı kullanım sağlamak için fiksasyon çözeltisini bir beher içine dökerek hücrelerden çıkarın.
NOT: Formaldehit içeren sıvı atıkları uygun sağlık ve güvenlik yönetmeliklerine uygun şekilde attığınızdan emin olun. - Kalıntıları ve ölü hücreleri temizlemek için 10 cm'lik plakayı 5 mL soğuk 0,125 M glisin-1x PBS (15 cm'lik bir plaka için 8 mL) ile iki kez hızlı bir şekilde durulayın. Hızlı kullanım sağlamak için sıvıyı bir beher içine dökerek plakadan çıkarın.
- 10 cm'lik plakaya 5 mL soğuk 0.125 M glisin-1x PBS ekleyin (15 cm'lik bir plaka için 10 mL) ve plastik bir hücre kazıyıcı kullanarak hücreleri plakadan hızla kazıyın.
- Hücre süspansiyonunu serolojik bir pipet kullanarak önceden soğutulmuş 50 mL konik santrifüj tüpüne aktarın.
- Plakayı 5 mL soğuk 0.125 M glisin-1x PBS ile iki kez durulayın ve hücre süspansiyonunu konik santrifüj tüpüne ekleyin.
- 4 °C'de 10 dakika boyunca 480 x g'de aşağı doğru döndürün.
NOT: Hücreler çözelti içinde sabitlenmişse, hücreyi önceden soğutulmuş bir konik santrifüj tüpüne aktarın ve 4 ° C'de 10 dakika boyunca 480 x g'de aşağı doğru döndürün. Sabitleme çözeltisini bir beher içine dökerek çıkarın ve 10 mL soğuk 0.125 M glisin-1x PBS'de üç kez yıkayın. Her yıkama adımında hücreleri yeniden askıya aldığınızdan emin olun. - Bir tezgah üstü aspirasyon sistemi ile aspire ederek süpernatantı çıkarın. P1000 pipetle dikkatlice yukarı ve aşağı pipetleme yaparak hücreleri 1 x 107 hücre başına 500 μL 1x PBS'de yeniden askıya alın. Hücreleri doğru hacimde yeniden askıya almak için, 2.2.1'de elde edilen toplam hücre sayısı tahminine bakın.
- Hücre süspansiyonunun 500 μL'sini hesaplanan 1.5 mL mikrosantrifüj tüpüne (1 x 107 hücre / tüp) alikot edin.
- 4 °C'de 10 dakika boyunca 480 x g'de aşağı doğru döndürün.
- Süpernatantı bir tezgah üstü aspirasyon sistemi ile çıkarın ve hücre peletlerini sıvı azot içinde dondurun. Kuru hücreli peletleri -80 °C'de saklayın.
NOT: Numuneler en az 1 yıl saklanabilir.
- Çapraz bağlanacak toplam hücre sayısını tahmin edin.
- Hücre lizisi
- Dondurulmuş peletleri buz üzerinde çözün.
- Numune başınaH20'da 1,5 mL lizis tamponu hazırlayın: 10 mM Tris-HCl, pH 8,0, 10 mM NaCl ve% 0,2 NP40 ekleyin.
- Soğuk lizis tamponundan 600 μL ekleyin ve buz üzerinde iyice askıya alın.
- Hücrelerin şişmesine izin vermek için 15 dakika boyunca buz üzerinde inkübe edin.
- 4 °C'de 5 dakika boyunca 2655 x g'de aşağı doğru döndürün ve bir tezgah üstü aspirasyon sistemi kullanarak süpernatantı çıkarın.
- Kalıntıları gidermek için, peleti soğuk lizis tamponunun 1 mL'sinde yeniden askıya alın, 4 ° C'de 5 dakika boyunca 2655 x g'de döndürün ve süpernatanı çıkarın.
- Kısa bir süre sonra 2655 x g ve 4 ° C'de tekrar döndürün ve P200 ucu ile donatılmış bir tezgah üstü aspirasyon sistemi kullanarak kalan süpernatantın mümkün olduğunca çoğunu çıkarın.
- %0,5 (hacim/hacim) SDS'nin 100 μL'sinde yeniden askıya alın.
- 62 ° C'de bir termomikserde inkübe edin, 10 dakika boyunca 1400 rpm'de dönün.
- 290 μLH2O + 50 μL% 10 TritonX-100 ekleyin ve hava kabarcıklarından kaçınarak iyice karıştırın.
- 37 ° C'de bir termomikserde inkübe edin, 15 dakika boyunca 1400 rpm'de dönün.
- 50 μL 10x Dpnll tampon ekleyin ve karıştırmak için tüpü ters çevirin.
- Kalite kontrol için 50 μL sindirilmemiş DNA'yı ayrı bir tüpe alın. Sindirilmemiş kontrol örneğini almayı unutmayın.
- DpnII sindirim
- 10 μL Dpnll yüksek konsantrasyon (toplam 500 U) ekleyin ve ters çevirerek karıştırın.
- Numuneleri ve sindirilmemiş kontrolü 37 °C'de bir termomikserde inkübe edin, 1400 rpm'de >4 saat boyunca döndürün.
- Günün sonunda 10 μL Dpnll yüksek konsantrasyon (toplam 500 U) ekleyin.
- Numuneleri ve sindirilmemiş kontrolü 37 ° C'de inkübe edin, gece boyunca 1400 rpm'de döndürün.
- Numunelere ertesi günün başında 10 μL Dpnll yüksek konsantrasyon (toplam 500 U) ekleyin.
- Numuneleri ve sindirilmemiş kontrolü 37 °C'de bir termomikserde inkübe edin, 4 saat boyunca 1400 rpm'de döndürün.
- Çapraz bağlamanın bağlanması ve tersine çevrilmesi
- Tüpleri 65 ° C'de 1400 rpm'de 20 dakika boyunca inkübe edin.
NOT: Bu noktada SDS eklemeyin. Buradaki fikir, nükleer bütünlüğü korumaktır, bu nedenle ligasyon, aşırı seyreltme ihtiyacını ortadan kaldırarak çekirdeklerin içinde gerçekleştirilir. - Numuneleri buz üzerinde maksimum 5-10 dakika soğutun. SDS yağışını önlemek için, numuneleri bundan daha uzun süre buz üzerinde bırakmayın.
- Ayrı bir tüpte kalite kontrolü için bağlanmamış sindirilmiş DNA'nın 50 μL'sini alın. Sindirilmemiş ve bağlanmamış kontrolleri -20 °C'de saklayın.
NOT: Bağlanmamış kontrol numunesini almayı unutmayınız. - 800 μL ligasyon kokteyli ekleyin: 122 μL 10x ligaz tamponu, 8 μL T4 ligaz (30 U / μL) ve 670 μL H20.
- 16 ° C'de inkübe edin, gece boyunca 1000 rpm'de dönün.
- Numunelere 7.5 μL Proteinaz K (20 mg / mL) ve kontrollere 2 μL ekleyin.
- 65 ° C'de 1000 rpm'de 4 saat boyunca inkübe edin.
- Tüpleri 65 ° C'de 1400 rpm'de 20 dakika boyunca inkübe edin.
- DNA saflaştırma
- Buz üzerindeki numuneleri önceden soğutulmuş 15 mL konik santrifüj tüplerine aktarın ve 2 mL su, 10,5 mL buz gibi soğuk EtOH ve 583 μL 3 M NaAC ekleyin.
NOT: Ek su, DTT'nin pelet içine taşınmasını önlemeyi amaçlar. - Sindirilmemiş ve bağlanmamış kalite kontrollerine 200 μL buz gibi soğuk EtOH, 10,8 μL NaAC ve 1 μL koprecipitant ekleyin.
- Gece boyunca en az 4 saat -80 ° C'de inkübe edin.
- 15 mL tüpleri 4 °C'de 2200 x g'de 45 dakika boyunca döndürün.
- 1,5 mL kontrol tüplerini 20.500 x g'de 4 °C'de 30 dakika boyunca döndürün.
- Bir kez 3 mL (numuneler) ve 1 mL (kontroller) buz gibi soğuk% 70 EtOH ile yıkayın.
- 10 dakika boyunca 4 °C'de 2200 x g (numuneler) veya 20.500 x g (kontroller) hızla döndürün.
- EtOH'yi dikkatlice çıkarın ve RT'de 10-15 dakika boyunca hava kurutun; Fazla kurumayın.
- Numuneleri ve kontrolleri sırasıyla 100 μL ve 20 μL H20'de yeniden askıya alın.
- 1 μL RNAseA ekleyin ve 37 ° C'de inkübe edin, 30 dakika boyunca 1400 rpm'de dönün.
- Buz üzerindeki numuneleri önceden soğutulmuş 15 mL konik santrifüj tüplerine aktarın ve 2 mL su, 10,5 mL buz gibi soğuk EtOH ve 583 μL 3 M NaAC ekleyin.
- 3C şablon hazırlama kalite kontrolü
- Yüksek hassasiyetli DNA konsantrasyonu ölçümleri için bir florometre kiti kullanarak her numuneyi ölçün ve kontrol edin.
- Her numunenin 100-200 ng'sini ve her kontrolü% 1 agaroz / 1x TBE jeli üzerine yükleyin.
- Kontrollerin DNA fragman boyutlarındaki farklılıkları ve Şekil 2A'da gösterildiği gibi 3C şablonunu karşılaştırarak jel görüntüsünün beklenen sonucu gösterdiğini doğrulayın.
- Numuneleri ve kontrolleri -20 °C'de saklayın.
- Çok katlı sıralama için hibridizasyon, yakalama ve numune işleme
- Biyotinile RNA probları dizisini 3C şablonuna hibritize etmek, hedeflenen ligasyon parçalarını yakalamak ve bu çalışmada çift uçlu çoklanmış dizileme için kullanılan hedef zenginleştirme sistemine göre örnekleri çoklanmış dizileme için hazırlamak (bkz. Malzeme Tablosu). Aşağıdaki küçük değişiklikleri yaparken protokolü üreticinin talimatlarına göre izleyin:
- Üretici protokolünün 2. Bölümü: Numune hazırlama
- 3 μg gDNA girişinden başlayarak hedef zenginleştirme talimatlarını izleyin.
- Aşağıdaki özellikleri kullanarak DNA'yı bir sonikatörde kesin:% 10 görev döngüsü, 4 yoğunluk, 200 döngü / patlama ve 130 s. Numune hazırlamaya 3 μg kesilmiş DNA ile devam etmek için yeterli malzemeden emin olmak için her yakalama reaksiyonu için 130 μL su içinde yeniden askıya alınmış 4 μg 3C şablonu ile başlayın.
- Kesilmiş DNA'nın kalitesini değerlendirin. Kesilen DNA'nın 1 μL'sini yüksek hassasiyet protokolüne göre bir DNA biyoanalizöründe çalıştırın. Parça boyutunun 150-700 bp arasında bir dağılım olmasını bekleyin (Şekil 2).
- Katı faz geri dönüşümlü immobilizasyon (SPRI) boncukları kullanarak numuneyi saflaştırın. Üreticinin talimatlarına göre 1:1 sol taraf boyutu seçimi yapmak için DNA örneğinin 124 μL'sine 124 μL SPRI boncuk ekleyin ve 25 μL nükleaz içermeyen sudan yararlanın. Bu saflaştırma adımı, yaklaşık 300 bp'lik parçaları zenginleştirmek için daha kısa parçaları kaldıracaktır (Şekil 2).
NOT: Bu adımda kullanılan numunelerin ve SPRI boncuklarının miktarı, numunelerin yeni tüplere aktarılması ve Biyoanalizörde kalite kontrollerinin yapılması sırasında meydana gelen hacim kaybını dikkate alır. Sonraki tüm boyut seçim adımları, üreticinin protokolü tarafından önerilen oranlara göre gerçekleştirilir. SPRI boncuklarından DNA elüsyonu, protokol boyunca RT'de gerçekleştirilir. - Boyut olarak seçilmiş kesilmiş DNA'nın kalitesini değerlendirin. Yüksek hassasiyet (HS) protokolüne göre DNA biyoanalizöründe kesilen DNA'nın 1 μL'sini çalıştırın. 300 bp'de en yüksek zenginleştirmeye sahip parça boyutlarının dağılımını bekleyin (Şekil 2). Kesme başarılı olduysa, kesilen DNA'nın nicelleştirilmesine devam edin.
- HS DNA konsantrasyon ölçümleri için kesilen DNA'yı bir florometre kiti ile sayısallaştırın.
NOT: DNA kesme işlemi <3 μg'lık bir DNA verimi ile sonuçlanırsa, ikinci bir DNA kesme turunu başka bir 4 μg DNA ile gerçekleştirin ve toplam 3 μg kesilmiş DNA elde etmek için ilk SPRI boncuk saflaştırma adımından sonra kesilen DNA örneklerini birleştirin. - Boyut olarak seçilen temizlenmiş DNA örneğine (toplam 3 μg) nükleaz içermeyen suyu 48 μL'lik son bir hacme kadar ekleyin ve üreticinin protokolüne göre son onarım reaksiyonuna devam edin.
- Eşleştirilmiş uçlu adaptörlerin bağlanmasından sonra, üreticinin talimatlarına göre beş çekim öncesi PCR döngüsü gerçekleştirerek kütüphaneyi güçlendirin (PCR ve primerler için koşullar kitte sağlanmıştır).
- Üreticinin protokolünün 4. Bölümü: Hibridizasyon ve yakalama
- Hazırlanan DNA örneklerini hedefe özgü RNA problarına hibritleştirmek için, 750 ng DNA örneğini 3.4 μL'lik son bir hacimde seyreltin, bu da 221 ng / μL'lik bir başlangıç konsantrasyonu ile sonuçlanır. Daha büyük hacimlerde seyreltilmiş DNA örnekleri için, son hacme indirgemek için bir hız-vakum yoğunlaştırıcı kullanın. 15-20 dakika boyunca bir hız-vakum konsantrasyonu (250 x g; ≤45 ° C) normalde 10 μL'de yeniden askıya alınan numuneler için yeterlidir.
- Hibridizasyon karışımını, üreticinin talimatlarına göre 105 ° C'de ısıtılmış bir kapakla 65 ° C'de 16-18 saat boyunca inkübe edin.
- Üretici protokolünün 5. Bölümü: Çok katlı sıralama için indeksleme ve numune işleme
- Yakalanan kütüphaneleri indeksleme primerleri ile güçlendirmek için, üreticinin talimatlarına göre 12 çekim sonrası PCR döngüsü gerçekleştirin (PCR ve primerler için koşullar kit içinde sağlanmıştır).
- Üretici protokolünün 2. Bölümü: Numune hazırlama
- Biyotinile RNA probları dizisini 3C şablonuna hibritize etmek, hedeflenen ligasyon parçalarını yakalamak ve bu çalışmada çift uçlu çoklanmış dizileme için kullanılan hedef zenginleştirme sistemine göre örnekleri çoklanmış dizileme için hazırlamak (bkz. Malzeme Tablosu). Aşağıdaki küçük değişiklikleri yaparken protokolü üreticinin talimatlarına göre izleyin:
- Yeni nesil sıralama
- Aynı akış hücresinde birden fazla yakalama Hi-C kitaplığı çalıştırmak için, yakalama kitaplıklarının eşit molar bir karışımını hazırlayın ve kitaplık başına 100-120 M okuma sıralayın.
- Alele özgü analize ihtiyaç duyulursa, yeterli SNP kapsamı sağlamak için 150 bp çift uçlu sıralayın.
3. Veri analizi
- Capture Hi-C veri analizi55 gerçekleştirmek için HiC-Pro işlem hattını uygulayın. HiC-Pro, aşağıdakiler dahil olmak üzere işlemenin her adımında kalite kontrolleri sağlar (Şekil 3):
(i) Bir ligasyon bölgesini kapsayan okumaların fraksiyonunu ve ayrıca çiftlerin ve singletonların sayısını belirten referans genom üzerindeki hizalama oranı.
(ii) Geçerli ligasyon ürünlerinin ve bilgilendirici olmayan okuma çiftlerinin (sarkan uç, kendinden ligasyon, vb.) fraksiyonu.
(iii) Kısa / uzun menzilli ve intra / kromozomal temasların fraksiyonu.
(iv) Capture Hi-C için hedefteki kontakların oranı.
(v) Alele özgü fraksiyon belirtilmişse okur.
NOT: HiC-Pro, in situ Hi-C ve Capture Hi-C dahil olmak üzere çok çeşitli protokolleri destekler. İkinci durumda, kullanıcının yapılandırma dosyasında hedeflenen bölgeyi (BED biçimi) belirtmesi yeterlidir. Veriler işlendikten sonra, HiC-Pro çıkışları aşağı akış analizi için kolayca daha soğuk bir nesneye dönüştürülebilir56. Bu adımda, çeşitli çözünürlüklerdeki temas haritaları, daha önce Imakaev ve meslektaşları57 tarafından açıklanan ICE yöntemi kullanılarak normalleştirilir. Daha sonra kromozom bölmelerini, TAD'ları veya kromatin döngülerini çağırmak için çeşitli analizler yapılabilir (inceleme58 için). Protokolün iş akışı Şekil 4'te gösterilmiştir. Burada, Şekil 5 ve Şekil 659'da gösterildiği gibi, yalıtım puanını ve TAD sınırlarını hesaplamak için 'cooltools' paketi uygulanır.
Sonuçlar
Açıklanan Capture Hi-C protokolü, dört tabanlı bir kesici (DpnII) kullanılarak genom çapında 3C şablonunun hazırlanmasına dayanmaktadır. İlgilenilen genomik bölge boyunca ligasyon fragmanlarının daha sonraki zenginleştirilmesi, bir dizi döşeme RNA probunun hibridizasyonu ve bu çalışmada kullanılan hedef zenginleştirme sistemine göre streptavidin bazlı yakalanması ile elde edilir (Şekil 1). Biyotinile RNA probları, DNA problarına kıyasla hedeflerine daha sıkı bağlanma afinitesi gösterdikleri için seçildi52,60. Yakalanan kitaplıklar daha sonra dizine alınır ve çoğullanmış yüksek verimli sıralama için havuza alınır. Hi-C verilerini yakalamak, yüksek çözünürlüklü Hi-C etkileşim haritaları olarak görselleştirilebilir, ancak aynı zamanda yakalanan bölgenin tamamındaki destekleyiciler veya arttırıcılar gibi daha küçük dizilerin etkileşimlerini özel olarak görselleştirmek için 4C benzeri tek görüş noktası temas haritaları olarak da görselleştirilebilir. Protokolün iş akışı Şekil 4'te gösterilmiştir. Ön sıralama kalite kontrolleri Şekil 2'de gösterilmiştir ve 3C şablonunun uygun sindiriminin ve yeniden ligasyonunun değerlendirilmesini ve protokolün farklı adımlarında etkili kesilmesini ve saflaştırılmasını içerir. Kesilen 3C şablon DNA'nın 150 ila 700 bp arasında çalışması bekleniyor ve parçaların >2 kb zenginleştirilmesi tespit edilmemelidir. Aşağıdaki adımlarda, önce kesmeden sonra, daha sonra yakalama öncesi ve yakalama sonrası PCR'lerden sonra birkaç boncuk bazlı DNA temizleme ve boyut seçimi adımı gerçekleştirilir. Temizlenen kütüphaneler, yüksek hassasiyetli bir DNA biyoanalizöründe görselleştirildiği gibi farklı bir parça zenginleştirme profili gösterir (Şekil 2). Ortalama parça boyutu, bağdaştırıcıların bağlanması, dizileme ve indeksleme primerleri nedeniyle kütüphane hazırlığı boyunca artar. Sıralama sonrası kalite kontrolleri Hi-C Pro ile elde edilir ve Şekil 3'te gösterilmiştir. 3C benzeri veri işleme ve analizi için birçok farklı biyoinformatik yazılım uygulaması önerilmiştir. Bunlar arasında, HiC-Pro boru hattı, ham sıralama verilerinin çeşitli çözünürlüklerde son temas haritalarına işlenmesine izin veren en popüler çözümlerden biridir55. HiC-Pro, referans genom üzerindeki dizileme okumalarını hizalamak için iki adımlı bir haritalama stratejisi kullanır. 3C ürünleri daha sonra bilgilendirici olmayan temas çiftlerini kaldırmak ve temas haritalarını oluşturmak için yeniden yapılandırılır ve filtrelenir. Ek olarak, alele özgü analiz yapmak ve iki ebeveyn alelinden gelen temasları farklı temas haritalarında ayırmak için bilinen polimorfizmlerin bir listesini kullanabilir. Daha yakın zamanlarda, HiC-Pro, nf-core çerçevesine (nf-core-hic) dahil edildi ve genişletildi, bu da yüksek oranda ölçeklenebilir ve tekrarlanabilir topluluk odaklı bir boru hattı61,62 sağladı.
Xic faresini yakalamak için, X kromozomunun 3 Mb'sini döşeyen 28.913 RNA probu dizisi tasarlandı. Bu bölge, XCI'daki kilit oyuncuyu, uzun kodlamayan gen Xist'i ve bilinen ~ 800 kb düzenleyici manzarasını içerir (Şekil 5). Bu ~ 800 kb bölgesi iki TAD'a bölünmüştür: biri Xist promotörü ve bilinen pozitif düzenleyicileri (yani, kodlamayan transkriptler Ftx, Jpx ve Xert ve protein kodlayan gen Rnf12) ve Xist'in negatif cis-düzenleyicilerini kapsayan komşu TAD (yani, antisens transkript Tsix, geliştirici element Xite ve kodlamayan transkript Linx) (inceleme için44, 45).
Tanımlanan Capture Hi-C protokolünü Xic'e uygulayarak, bu lokusun topolojik organizasyonu benzeri görülmemiş bir çözünürlükte elde edildi (Şekil 6 ve Şekil 7). Bu, özellikle Capture Hi-C profili ile daha önce yayınlanmış 5C47 (Şekil 6 ve Şekil 7; Ek Tablo 1) ve Hi-C61 (Şekil 6 ve Şekil 7; Ek Tablo 1) Profil. Örneğin, alt-TAD yapıları daha belirgindir – Xist promotörünü (Xist-TAD) içeren TAD açıkça iki küçük alana bölünmüştür (Şekil 6A, mavi ok ucu). Önceden, bu, yalıtım puanı algoritması kullanılarak bu bölgede bir sınırın tespit edilmesine rağmen, yalnızca 5C profilinden (Şekil 6B) görsel olarak "tahmin edilebilir". Benzer şekilde, Capture Hi-C profilinin çözünürlüğü, Tsix lokusunun (Tsix-TAD) destekleyicisini içeren komşu TAD'de (Şekil 6A, B) iki küçük etki alanının tanımlanmasına izin verir; bu daha önce 5C ile elde edilememişti (Şekil 6B). Not olarak, Capture Hi-C ve 5C verilerinden yalıtım skoru ile belirlenen topolojik sınırlar genellikle biraz farklı konumlarda ve farklı göreceli güçlerde tespit edilir.
Ayrıca, temas döngüleri gibi diğer alt-TAD yapıları, daha önce Capture-C63 ile tanımlanan Xist ve Ftx arasındaki döngü (Şekil 7A) ve yakın zamanda Capture Hi-C48 için benzer bir protokol kullanılarak tanımlanan Xist ve Xert arasındaki döngü (Şekil 7B) gibi Capture Hi-C verilerinden açıkça görülebilir. Linx, Chic1 ve Xite lokusları arasındaki Tsix-TAD içinde bilinen temas noktalarını oluşturanlar gibi Capture Hi-C profillerinin artan çözünürlüğü nedeniyle diğer kontaklar da daha hassas bir şekilde eşlenebilir (Şekil 7A).
Şekil 7'de gösterilen Hi-C verileriyle karşılaştırıldığında, Capture Hi-C çözünürlükte dört kat artışa izin verdi, ancak sıralama derinliğinin yalnızca dörtte birini gerektiriyordu (yani, 126 M okumalara karşı 571 M) (Ek Tablo 1). Çözünürlükteki bu artış, Şekil 6 ve Şekil 7'de gösterilen sıralama derinliğinde Hi-C tarafından tespit edilemeyen alt TAD'lerin ve döngü etkileşimlerinin algılanmasını sağlar. Capture Hi-C için açıklanan protokol, önceki yaklaşımlara kıyasla, geniş bir genomik bölgenin çok daha ayrıntılı, yüksek çözünürlüklü bir karakterizasyonuna izin verir.
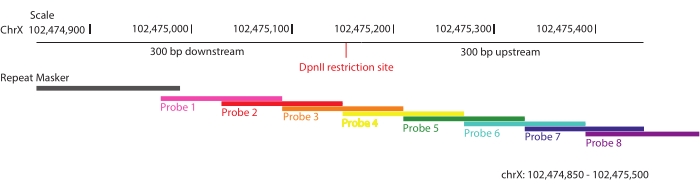
Resim 1: Prob tasarımı. Prob tasarımı için kullanılan stratejinin şematik gösterimi. 3 Mb hedef bölge boyunca her bir DpnII kısıtlama bölgesinin 300 bp yukarı ve aşağı akış bölgeleri seçildi ve üst üste binen biyotinile RNA probları ile döşendi. Bu seçilen bölgelerden biri gösterilir, chrX: 102,474,805-102,475,500. Her probda 40'tan fazla baz tekrarlayan diziye izin verilmez. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
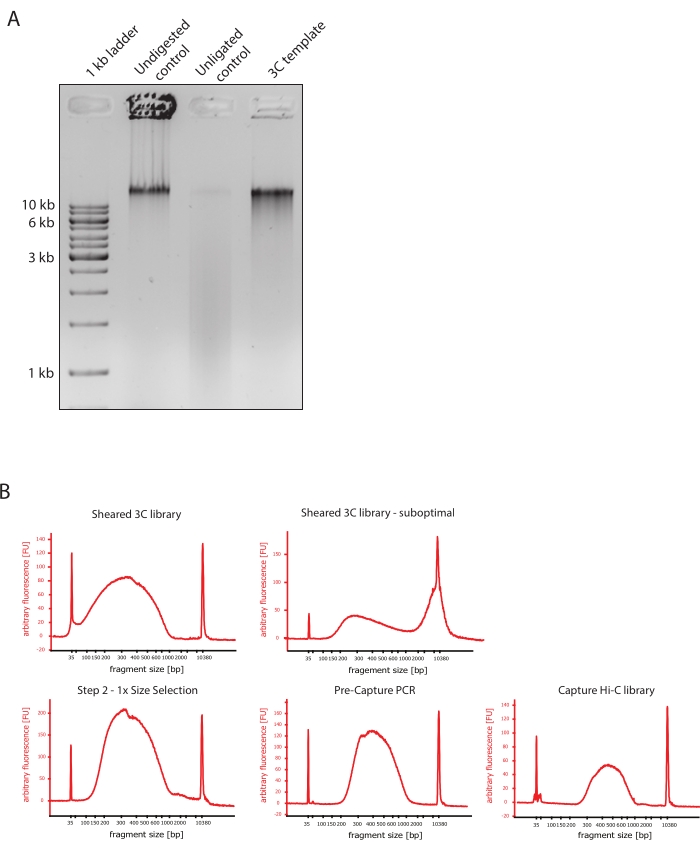
Şekil 2: Hi-C ön sıralama kalite kontrollerini yakalayın . (A) 3C şablon kalite kontrollerinin temsili örneği. 200 ng DNA,% 1'lik bir agaroz jeline yüklendi. Şerit 1: 1 kb merdiven. Şerit 2: Sindirilmemiş, çapraz bağlı ve bozulmamış kromatin, >10 kb'de keskin bir bant olarak çalışır. Şerit 3: DpnII ile sindirilmiş çapraz bağlı kromatin, 1 kb ila 3 kb arasında bir yayma olarak çalışır. Şerit 4: Son 3C kütüphanesi veya şablonu; Sindirilmiş çapraz bağlı DNA fragmanlarının serbest uçları yeniden bağlanır. Düşük moleküler boyuttaki DNA yayması neredeyse tespit edilemez ve ligasyon ürünü >10 kb'lik bir bant olarak tespit edilir. (B) Yüksek hassasiyetli biyoanalizör DNA profillerinin temsili örnekleri. Sol üst: 150 bp ile 700 bp arasında parça boyutunun dağılımını gösteren başarıyla kesilmiş 3C kütüphanesi. Sağ üst: yetersiz kesilmiş 3C kütüphanesi. Kesilmemiş DNA, parçaların geniş zenginleşmesi olarak tespit edilir >2 kb. (C) Sol altta: SPRI boncukları kullanılarak 1:1 sol taraf boyutunda bir seçimi takiben kesilmiş DNA örneği. ~300 bp'lik parçalar zenginleştirilmiştir. Alt orta: Üreticinin protokolüne göre çift uçlu adaptörlerin bağlanmasından sonra PCR profilini önceden yakalayın. Sağ altta: çoğullanmış sıralama için adaptörler, sıralama ve dizin oluşturma primerleri dahil olmak üzere son Capture Hi-C kitaplığı. Kısaltmalar: bp = baz çiftleri, FU = keyfi floresan birimi. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
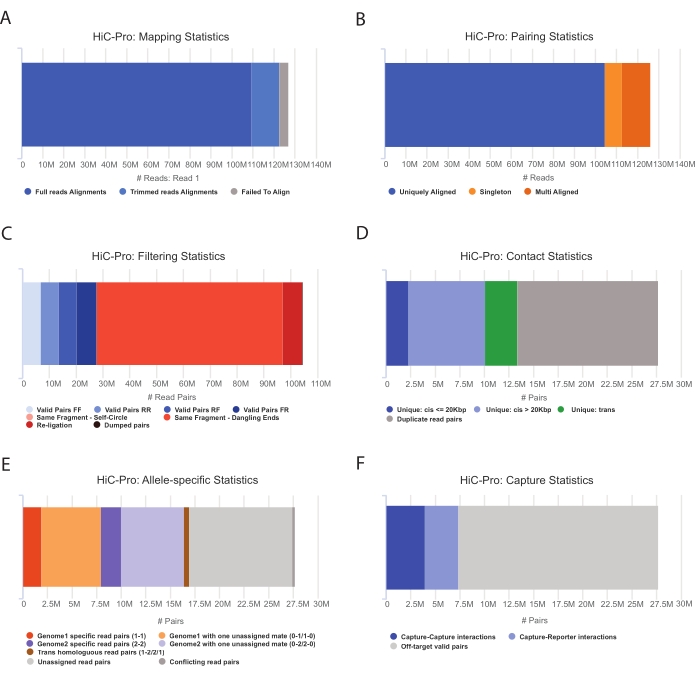
Şekil 3: HiC-Pro ile Hi-C dizileme sonrası kalite kontrollerini yakalayın . (A) Dizileme çiftlerinin ilk eşi için referans genom üzerindeki haritalama hızı örneği. Açık mavi fraksiyon, HiC-Pro tarafından hizalanan ve bir ligasyon kavşağını kapsayan okumaları temsil eder. Bu metrik böylece deneysel ligasyon adımını doğrulamak için kullanılabilir. (B) Dizileme eşleri genom üzerinde hizalandıktan sonra, analiz için yalnızca benzersiz şekilde hizalanmış okuma çiftleri tutulur. (C) Sarkan uç, kendi kendine daire veya yeniden ligasyon gibi geçerli olmayan çiftler (kırmızı renkte) analizden atılır. Geçerli çiftlerin oranı, ligasyonun ve aşağı çekme verimliliğinin iyi bir göstergesidir. (D) Geçerli çiftler ayrıca intra/interkromozomal ve kısa/uzun menzilli temaslara ayrılabilir. PCR yapıtlarını temsil etmesi muhtemel yinelenen okuma çiftleri analizden atılır. (E) Alele özgü analiz için HiC-Pro, her ebeveyn genomu için bir veya iki eş tarafından desteklenen allelik okuma sayısını bildirir (yani, C57BL / 6J x CASTEi / J). Anne ve baba aleline atanan okumaların aynı kısmı beklenir. (F) Son olarak, temas haritalarını oluşturmak için yalnızca yakalama bölgesiyle çakışan geçerli çiftler seçilir. Yakalama-yakalama çiftleri hedeflenen bölge içindeki kişileri temsil ederken, yakalama-raporlayıcı çiftleri hedeflenen bölge ile hedef dışı bölge arasındaki etkileşimi içerir. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
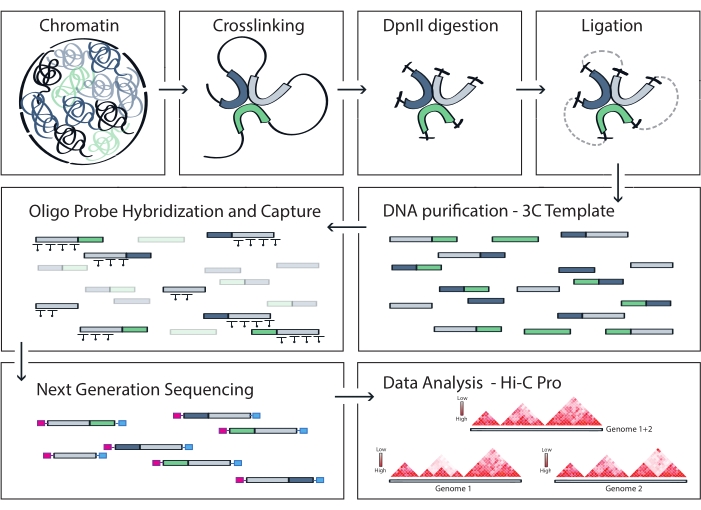
Şekil 4: Capture Hi-C protokolünün iş akışı. Farklı protokol adımlarının şematik gösterimi. Genom çapında 3C şablonunu oluşturmak için, kromatin önce formaldehit ile çapraz bağlanır ve daha sonra DpnII kısıtlama enzimi ile sindirilir. Serbest DNA uçları daha sonra yeniden bağlanır, çapraz bağlanma tersine çevrilir ve DNA saflaştırılır. Hedef bölgeyi kapsayan parçaları zenginleştirmek için, bir dizi biyotinillenmiş RNA probu 3C şablonuna hibridize edilir ve streptavidin aracılı aşağı çekme ile yakalanır. Yakalama kitaplıkları çoğullanmış sıralama için işlenir ve yüksek çözünürlüklü etkileşim haritaları olarak görselleştirilen hedef boyunca kromatin temaslarının sıklığını çıkarmak için geçerli ligasyon parçaları ölçülür. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
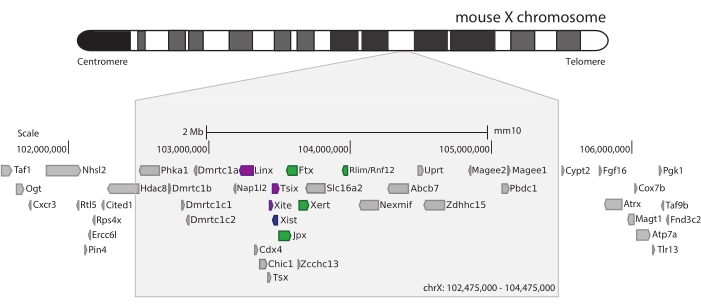
Şekil 5: Fare X kromozomu üzerindeki Xic'i kapsayan bölgeye genel bakış. Fare X kromozomunun şematik gösterimi ve yakalanan 3 Mb bölgesinin yakınlaştırılması (ChrX: 102.475.000-105.475.000). Hedeflenen bölge, XCI'nın ana düzenleyici lokusu olan Xic'e karşılık gelen ~ 800 kb DNA içerir. Xic , uzun kodlamayan genleri, XCI'nın önemli bir oyuncusu olan Xist'i ve düzenleyici manzarasını içerir. Xist'in pozitif düzenleyicileri yeşil, negatif regülatörleri mor renkte gösterilmiştir. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
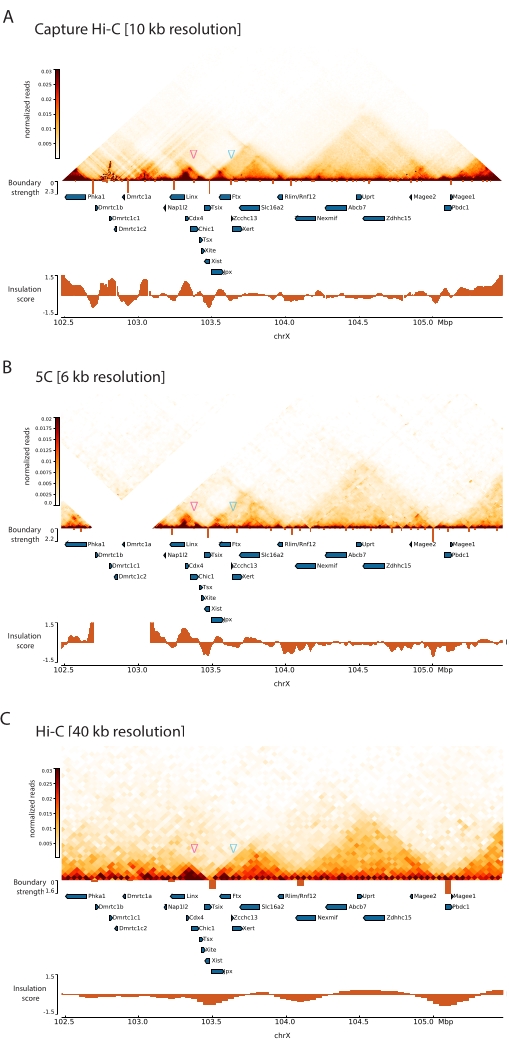
Şekil 6: Yakalanan 3 Mb bölge boyunca Hi-C, 5C ve Hi-C etkileşim haritalarını yakalayın. (A) Fare Xic'ini kapsayan 3 Mb hedefin Hi-C etkileşim haritasını 10 kb çözünürlükte yakalayın (bu çalışma). (B) 6 kb çözünürlükte A'dakiyle aynı hedef bölgenin 5C etkileşim haritası (47'den yeniden işlenen veriler). Analizlerde yer almayan tekrarlayan bölgeler beyaz renkte maskelenir. 5C verileri kendi biyoinformatik işlemlerini gerektirir (bkz.47). Temizleme ve hizalamadan sonra, astar çözünürlüğündeki 5C haritaları, 6 kb'lik bir nihai çözünürlüğe ulaşmak için çalışan bir medyan (pencere = 30 kb, adım = 5) kullanılarak bağlanır. (C) 40 kb çözünürlükte A ve B'deki ile aynı genomik bölgenin Hi-C etkileşim haritası (64'ten yeniden işlenen veriler). Tüm etkileşim haritaları fare ESC'lerinden oluşturulmuştur. İzolasyon skoru cooltools kullanılarak hesaplanmıştır ve TAD sınırlarında yalıtım minimleri ile histogramlar olarak temsil edilir. TAD sınırları haritanın altında dikey çizgiler olarak gösterilir. Her çizginin yüksekliği sınır kuvvetini gösterir. Genler, transkripsiyon yönünü gösteren oklar olarak gösterilir. Yalnızca veya daha kesin olarak Capture Hi-C haritalarında algılanan alt TAD sınırları, sırasıyla Tsix ve Xist TAD'lardaki alt-TAD'lar için macenta ve mavi ok uçlarıyla gösterilir. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
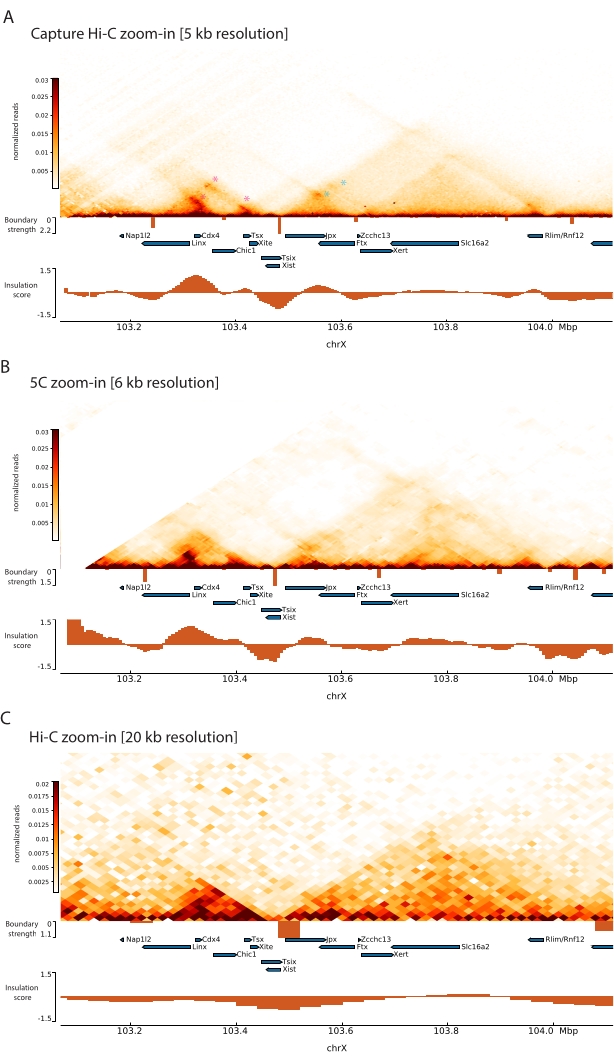
Şekil 7: Yakalanan bölge içinde 1 Mb boyunca Hi-C, 5C ve Hi-C etkileşim haritalarını yakalayın. (A) Fare Xic'ini kapsayan 1 Mb genomik bölgenin Hi-C etkileşim haritasını 5 kb çözünürlükte yakalayın (bu çalışma). (B) A'dakiyle aynı genomik bölgenin 5C etkileşim haritası. 6 KB çözünürlükte (47'den yeniden işlenen veriler). Analizlerde yer almayan tekrarlayan bölgeler beyaz renkte maskelenir. Not olarak, 5C verileri kendi biyoinformatik işlemlerini gerektirir (bkz.47). Temizleme ve hizalamadan sonra, astar çözünürlüğündeki 5C haritaları, 6 kb'lik bir nihai çözünürlüğe ulaşmak için çalışan bir medyan (pencere = 30 kb, adım = 5) kullanılarak bağlanır. (C) Hi-C'nin A ve B'sindeki ile aynı genomik bölgenin Hi-C etkileşim haritası 20 kb çözünürlükte (veriler 64'ten yeniden işlenir). Tüm etkileşim haritaları mESC'lerden oluşturulmuştur. İzolasyon skoru cooltools kullanılarak hesaplanmıştır ve TAD sınırlarında yalıtım minimleri ile histogramlar olarak temsil edilir. TAD sınırları haritanın altında dikey çizgiler olarak gösterilir. Her çizginin yüksekliği sınır kuvvetini gösterir. Genler, transkripsiyon yönüne işaret eden oklar olarak gösterilir. Capture Hi-C'de özel olarak veya daha hassas bir şekilde algılanan temas döngüleri, sırasıyla Tsix ve Xist TAD'lardaki döngüler için macenta ve mavi yıldız işaretleriyle gösterilir. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
Ek Tablo 1: Bu makalede kullanılan veri kümeleri için sıralama sonrası istatistikler: Capture Hi-C (bu çalışma), Hi-C64 ve 5C47. Bu Dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Burada, megabaz boyutlu genomik bölgelerin 5-10 kb çözünürlükte daha üst düzey organizasyonunu karakterize etmek için nispeten hızlı ve kolay bir Capture Hi-C protokolünü açıklıyoruz. Capture Hi-C, genom çapında 3C veya Hi-C şablonlarından hedeflenen kromatin etkileşimlerini zenginleştirmek için tasarlanmış Capture-C teknolojileri ailesine aittir. Bugüne kadar, Capture-C uygulamalarının büyük çoğunluğu, tüm genoma dağılmış nispeten küçük düzenleyici elementlerin kromatin temaslarını haritalamak için kullanılmıştır. İlk Capture-C protokolünde, eritroid hücrelerden hazırlanan 3C kütüphanelerinde önceden seçilmiş >400 promotör yakalamak için çoklu örtüşen RNA biyotinile probları kullanıldı31. Aynı strateji daha sonra Yeni Nesil (NG) ve Nükleer Titrated (NuTi) Yakalama-C'de, bilgilendirici ligasyon parçalarının zenginleştirilmesini en üst düzeye çıkarmak için tek kısıtlama bölgelerini kapsayan tek 120 bp DNA yemleri ve iki sıralı Yakalama turu kullanarak 8.000 > destekçinin yüksek çözünürlüklü etkileşim profillerini elde etmek için geliştirildi32,40. Bu stratejiler, fare embriyonik gelişimi, hücre farklılaşması, X-kromozomu inaktivasyonu ve patolojik durumlarda gen yanlış regülasyonu dahil olmak üzere birçok farklı bağlamda cis etkili elementlerin fonksiyonel diseksiyonuna yol açmıştır 46,63,65,66,67,68,69,70,71.
Promoter Capture Hi-C'de (PCHi-C), kısıtlama fragmanları içeren >22.000 açıklamalı promotör, kısıtlama fragmanı 34,72'nin herhangi birinde veya her iki ucunda tek RNA 120-mer biyotinile probların hibridizasyonu ile Hi-C kütüphanelerinden aşağı çekildi. Bu yöntem, fare embriyonik kök hücreleri, fetal karaciğer hücreleri ve adipositler 34,35,72,73, aynı zamanda insan lenfoblastoid çizgileri, hematopoetik progenitörler, epidermal keratinositler ve pluripotent hücreler 37,74,75,76,77 dahil olmak üzere hızla artan sayıda hücre tipinde binlerce promotörün interaktomunun diseksiyonuna izin verdi.
Bu hedef zenginleştirme teknolojileriyle karşılaştırıldığında, Capture Hi-C, megabaz ölçeğine kadar bitişik genomik bölgeleri hedefler, böylece bir veya daha fazla TAD'yi kapsar ve genlerin düzenleyici manzaralarını kapsar. İlgilenilen bölgenin tamamı, hedef içindeki her bir DpnII kısıtlama bölgesini kapsayan bir dizi biyotinile prob ile döşenmelidir. Biyotinillenmiş dizinin 3C şablonuna hibridizasyonu, ardından streptavidin tabanlı yakalama ve çoğullanmış dizileme için işleme, Illumina Paired-End çoklanmış dizileme için bir hedef zenginleştirme sistemi kullanılarak gerçekleştirilir. Tüm protokol hızlıdır, çünkü 3C kütüphane hazırlığından NGS sıralamasına kadar 1 hafta içinde gerçekleştirilebilir ve yalnızca küçük uyarlamalar ve / veya özel özel sorun giderme gerektirir.
Protokol ayrıca diğer 3C tabanlı yöntemlere kıyasla avantajlar sağlar. 5-10 kb çözünürlükte etkileşim haritaları elde etmek için 100-120 M çift uçlu okumaları sıraladık. Bir karşılaştırma olarak, burada 20 kb çözünürlük64'e (GSM2053973) ulaşmak için 571 M okumalık bir Hi-C veri kümesi kullandık ve kromozom çapındaHi-C 22 ile 5 kb çözünürlüğe ulaşmak için en az 1 milyar okuma gerekecekti.
Bu çalışmada kullanılan Hi-C'yi yakalamak, 6-bp kesici kısıtlama enzimi47'ye dayanan daha önce yayınlanmış 5C'den çok daha yüksek bir çözünürlüğe ulaşmaktadır (Ek Tablo 1). Önemli olarak, 5C'de hedeflenen etkileşimleri zenginleştirmek ve güçlendirmek için tasarlanan strateji, kromatin etkileşimlerinin alele özgü analizine izin vermemektedir. Aksine, Capture Hi-C verileri spesifik olarak alel ile eşlenebilir, bu da homolog kromozom çiftlerinin 3D yapısal manzaralarının, örneğin insan hücrelerinde veya genetik olarak farklı fare suşlarının78'i geçerek türetilen F1 hibrid hücre çizgilerinde diseksiyonuna izin verir. 5 kb çözünürlükte alele özgü Capture Hi-C etkileşim haritaları oluşturmak için, SNP kapsamını artırmak amacıyla 150 bp çift uçlu okumaları sıraladık. Benzer alele özgü yaklaşımlar, SNP'lerin ek açıklamasının mevcut olduğu insan hücre hatlarına uygulanabilir22.
Daha da önemlisi, Capture Hi-C, sıralama maliyetlerinin karşılanabilirliğini artırırken genellikle yüksek çözünürlük sağlasa da, özel olarak uyarlanmış biyotinile oligonükleotidlerin üretimi, bu yöntemin genel maliyeti üzerinde bir etkiye sahiptir. Bu nedenle, en uygun 3C yönteminin seçimi farklı uygulamalar için farklılık gösterecek ve ele alınan biyolojik soruya ve gerekli çözüme ve ayrıca ilgili bölgenin büyüklüğüne bağlı olacaktır. Geliştirilen diğer Capture Hi-C protokolleri, temel özellikleri burada açıklanan protokolle paylaşır. Örneğin, meme ve kolorektal kanser riski ile ilişkili kodlamayan varyantları kapsayan ~ 50 kb ila 1 Mb genomik bölgeleri karakterize etmek için bir Capture Hi-C stratejisi uygulanmıştır; Bu protokolde, hedef bölgeler Hi-C kütüphanelerinden aşağı çekilerek, hedef bölgeleri 3x kapsama alanı33,38,79'da döşeyen 120-mer RNA yemleri hibridize edildi. Benzer şekilde, HYbrid Capture Hi-C (Hi-C 2),2 Mb80'e kadar ilgi duyulan bölgelerdeki etkileşimleri hedeflemek için kullanıldı. Her iki protokolde de, biyotin aşağı çekilmiş ligasyon fragmanları için zenginleştirilmiş bir Hi-C şablonunun kullanılması, protokolümüze kıyasla toplam bilgilendirici okumaların yüzdesini arttırmıştır. Örneğin, karşılaştırma64 (GSM2053973) için burada kullandığımız Hi-C veri kümesinde, kopyaların kaldırılmasından sonraki geçerli çiftlerin yüzdesi, Şekil 3 ve Ek Tablo 1'de açıklandığı gibi Capture Hi-C'de elde edilen geçerli çiftlerden 4,8 kat daha yüksektir. Bununla birlikte, biyotinile bağlanmış parçaların ve hibridize probların art arda aşağı çekilmesi, protokolü önemli ölçüde daha karmaşık ve zaman alıcı hale getirirken, muhtemelen yakalanan bölgenin karmaşıklığını azaltır.
3C şablonlarını döşeme problarıyla zenginleştirmek için mevcut bir başka yöntem, fare eritroid farklılaşması43 sırasında kromatin mimarisini yüksek uzamsal ve zamansal çözünürlükte incelemek için uygulanan Tiled-C'dir. Tiled-C'de, hedeflenen etkileşimlerin çok yüksek çözünürlüklü haritalarını oluşturmak için iki ardışık yakalama turunda büyük ölçekli bölgelerdeki temasları zenginleştirmek için70 bp biyotinile problardan oluşan bir panel kullanılır 43,81. Çift yakalama zenginleştirme ayrıca protokolü Capture Hi-C'ye kıyasla daha uzun ve daha karmaşık hale getirir. Bununla birlikte, tek kısıtlama alanlarını hedefleyen Capture-C stratejilerinden farklı olarak, Tiled-C'de ikinci yakalama turu yakalama verimliliğini önemli ölçüde artırmıyor gibi görünmektedir ve bu nedenle muhtemelen43 atlanabilir. Son olarak, bu çalışmada kullanılan aynı hedef zenginleştirme stratejisine dayanan benzer bir döşeme yaklaşımı, konjenital malformasyonlu hastalarda tanımlanan ve transgenik farelerde yeniden tasarlanan yapısal varyantları kapsayan düzenleyici manzaraların diseksiyonuna uygulanmıştır41,42. Bu durumda, probların döşeme dizisi, DpnII kısıtlama bölgeleri41'in yakınından ziyade tüm hedef boyunca tasarlanmıştır. Bununla birlikte, bu çalışma, farklı bağlamlarda büyük genomik bölgelerin yüksek çözünürlüklü karakterizasyonunu elde etmek için bu stratejinin duyarlılığını ve gücünü vurgulamada ufuk açıcıydı41,42,48.
Sonuç olarak, burada açıklanan protokol, ilgilenilen herhangi bir genomik bölgenin yüksek çözünürlüklü 3D karakterizasyonu için kolay, sağlam ve güçlü bir stratejiyi temsil etmektedir. Bu yaklaşımın farklı model sistemlere, hücre tiplerine, gelişimsel olarak düzenlenmiş kromatin manzaralarına ve sağlıklı ve patolojik koşullarda gen düzenlemesine uygulanmasının, epigenetik alanındaki temel açık sorulardan biri olan genom topolojisi ile gen regülasyonu arasındaki etkileşim ve nedensellik hakkındaki anlayışımızı kolaylaştırması muhtemeldir. Ayrıca, GWAS çalışmaları tarafından tanımlanan risk varyantlarının uzun menzilli etkileşimlerini ve daha yüksek dereceli kromatin katlanmasını haritalamak için Capture Hi-C'nin uygulanması, farklı bağlamlarda insan hastalıklarıyla ilişkili kodlamayan genomik lokusların fonksiyonel alaka düzeyini ortaya çıkarma potansiyeline sahiptir ve böylece potansiyel olarak altta yatan patogenezin süreçlerine yeni bakış açıları sağlar.
Açıklamalar
Kai Hauschulz, Agilent Technologies - Teşhis ve Genomik Grubu'nda Saha Uygulama Bilimcisidir. Diğer tüm yazarlar birbiriyle çelişen çıkarlar beyan etmemektedir.
Teşekkürler
Heard laboratuvarındaki çalışmalar Avrupa Araştırma Konseyi İleri Araştırmacı ödülü (XPRESS - AdG671027) ile desteklenmiştir. A.L., Avrupa Birliği Marie Skłodowska-Curie Eylemleri Bireysel Bursu (IF-838408) tarafından desteklenmektedir. A.H., ITN Yenilikçi ve Disiplinlerarası Ağ ChromDesign tarafından Marie Skłodowska-Curie Hibe anlaşması 813327 kapsamında desteklenmektedir. Yazarlar, Daniel Ibrahim'e (MPI for Molecular Genetics, Berlin) yararlı teknik tavsiyeler için, Institut Curie'deki (Paris) NGS platformuna ve Vladimir Benes'e ve EMBL'deki (Heidelberg) Genomik Çekirdek Tesisi'ne destek ve yardım için teşekkür eder.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 10x PBS pH 7.4 | Gibco | 10010-023 | |
| 37% (vol/vol) paraformaldehyde solution | Electron Microscopy Sciences | 15686 | single use glass-vials; do not reuse |
| 50 mL PP conical tube | Falcon | 352070 | |
| Agarose | Sigma | A9539-500g | |
| Bioanalyzer | Agilent | G2939BA | |
| Cell Scrapers - 25 cm Handle and 3.0 cm Blade | Falcon | 353089 | |
| CHIR99021 | Axon Medchem BV | Axon 1386 | |
| cOmplete Mini, Protease inhibitor cocktail (EDTA-free) | Merck | 11836170001 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL | Invitrogen | ZGEXSCCOUNTESS2FL | Automated cell counter |
| Covaris S2 | Covaris | 500217 | Sonicator |
| DNA LoBind tube, 1.5 mL | Eppendorf | 30108051 | |
| DpnII (50000 units/mL) | New England Biolabs | R0543M | |
| Dulbecco's Modified Eagle Medium (DMEM) | Merck | D6429 | |
| Ethanol (100%) | Merck | 1.00983.2500 | |
| Fetal Bovine Serum (FBS) | Thermo Scientific | 10270106 | |
| gelatine from porcine skin | Sigma | G1890 | |
| GeneRuler 1 kb Plus DNA Ladder | Thermo Scientific | SM0313 | |
| GlycoBlue | Thermo Scientific | AM9516 | Coprecipitant |
| High-Sensitivity Bioanlayzer chips | Agilent | 5067-4626 | |
| Large Cooling Centrifuge 5920 R | Eppendorf | 5948000018 | |
| leukaemia inhibitory factor (LIF) | Merck | ESG1107 | |
| Liquiport | KNF | NF300 | Benchtop aspiration system |
| Low-binding filter tips | Biozym | VT0260U, VT0240, VT0220, VT0200U | |
| Molecular biology grade water | Merck | W3500-6x500ML | |
| Next Seq 500 | Illumina | SY-415-1001 | |
| Next Seq 500 High Output v2 Kit (300 cycles) | Illumina | FC-404-2004 | |
| Nonidet P40 Substitute (NP40) | Merck | 11332473001 | |
| PD0325901 | Axon Medchem BV | Axon 1408 | |
| Protease inhibitor cocktail (EDTA-free) | Merck | 11873580001 | |
| Proteinase K - recombinant, PCR-grade (20 mg/mL) | Thermo Scientific | EO0491 | |
| Qubit 2.0 | Thermo Scientific | Q32871 | |
| Qubit assay tubes | Thermo Scientific | Q32856 | |
| Qubit dsDNA High Sensitivity kit | Thermo Scientific | Q32851 | |
| RNase A (10 mg/mL) | Thermo Scientific | EN0531 | |
| Sodium acetate pH 5.2 (3M) | Merck | S7899 | |
| speed vacuum concentrator | Eppendorf | EP5305000100-1EA | |
| Agencourt AMPureXP | Beckman Coulter | A63881 | SPRI beads |
| SureSelect Target Enrichment Box 1 | Agilent | 5190-8645 | |
| SureSelect Target Enrichment Kit ILM Indexing Hyb Module Box 2 | Agilent | 5190-4455 | |
| SureSelect XT Library Prep Kit ILM | Agilent | 5500-0132 | |
| T4 ligase (30 units/µL) | Thermo Scientific | EL0013 | |
| table-top Centrifuge 5427 R | Eppendorf | 5409000012 | |
| Triton-X-100 (500 mL) | Merck | X100-500ML | |
| Trypan Blue | Invitrogen | T10282 | |
| Trypsine | Thermo Scientific | 25300054 | |
| UltraPure Glycine | Thermo Scientific | 15527013 | |
| β-mercaptoethanol | Thermo Scientific | 31350010 |
Referanslar
- Ibrahim, D. M., Mundlos, S. The role of 3D chromatin domains in gene regulation: a multi-facetted view on genome organization. Current Opinion in Genetics & Development. 61, 1-8 (2020).
- Bolt, C. C., Duboule, D. The regulatory landscapes of developmental genes. Development. 147 (3), (2020).
- Glaser, J., Mundlos, S. 3D or not 3D: Shaping the genome during development. Cold Spring Harbor Perspectives in Biology. 14 (5), 040188(2021).
- Denker, A., De Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Kempfer, R., Pombo, A. Methods for mapping 3D chromosome architecture. Nature Reviews Genetics. 21 (4), 207-226 (2020).
- McCord, R. P., Kaplan, N., Giorgetti, L. Chromosome conformation capture and beyond: Toward an integrative view of chromosome structure and function. Molecular Cell. 77 (4), 688-708 (2020).
- Jerkovic, I., Cavalli, G. Understanding 3D genome organization by multidisciplinary methods. Nature ReviewsMolecular Cell Biology. 22 (8), 511-528 (2021).
- Hsieh, T. -H. S., et al. Mapping nucleosome resolution chromosome folding in yeast by Micro-C. Cell. 162 (1), 108-119 (2015).
- Krietenstein, N., et al. Ultrastructural details of mammalian chromosome architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Naumova, N., Smith, E. M., Zhan, Y., Dekker, J. Analysis of long-range chromatin interactions using Chromosome Conformation Capture. Methods. 58 (3), 192-203 (2012).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Zhao, Z., et al. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra-and interchromosomal interactions. Nature Genetics. 38 (11), 1341-1347 (2006).
- Würtele, H., Chartrand, P. Genome-wide scanning of HoxB1-associated loci in mouse ES cells using an open-ended Chromosome Conformation Capture methodology. Chromosome Research. 14 (5), 477-495 (2006).
- De Wit, E., De Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes & Development. 26 (1), 11-24 (2012).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Splinter, E., et al. The inactive X chromosome adopts a unique three-dimensional conformation that is dependent on Xist RNA. Genes & Development. 25 (13), 1371-1383 (2011).
- Ferraiuolo, M. A., Sanyal, A., Naumova, N., Dekker, J., Dostie, J. From cells to chromatin: capturing snapshots of genome organization with 5C technology. Methods. 58 (3), 255-267 (2012).
- Kim, J. H., et al. 5C-ID: Increased resolution Chromosome-Conformation-Capture-Carbon-Copy with in situ 3C and double alternating primer design. Methods. 142, 39-46 (2018).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Zhang, Y., et al. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations. Cell. 148 (5), 908-921 (2012).
- Rao, S. S. P., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Dixon, J. R., et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485 (7398), 376-380 (2012).
- Nora, E. P., et al. Spatial partitioning of the regulatory landscape of the X-inactivation centre. Nature. 485 (7398), 381-385 (2012).
- Krefting, J., Andrade-Navarro, M. A., Ibn-Salem, J. Evolutionary stability of topologically associating domains is associated with conserved gene regulation. BMC Biology. 16 (1), 87(2018).
- Galupa, R., Heard, E. Topologically associating domains in chromosome architecture and gene regulatory landscapes during development, disease, and evolution. Cold Spring Harbor Symposia on Quantitative Biology. 82, 267-278 (2017).
- Tena, J. J., Santos-Pereira, J. M. Topologically associating domains and regulatory landscapes in development, evolution and disease. Frontiers in Cell and Developmental Biology. 9, 702787(2021).
- Lupiáñez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How alterations of chromatin domains result in disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Davidson, I. F., Peters, J. -M. Genome folding through loop extrusion by SMC complexes. Nature Reviews Molecular Cell Biology. 22 (7), 445-464 (2021).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews Molecular Cell Biology. 17 (12), 743-755 (2016).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Davies, J. O. J., et al. Multiplexed analysis of chromosome conformation at vastly improved sensitivity. Nature Methods. 13 (1), 74-80 (2016).
- Jäger, R., et al. Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci. Nature Communications. 6, 6178(2015).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Sahlén, P., et al. Genome-wide mapping of promoter-anchored interactions with close to single-enhancer resolution. Genome Biology. 16, 156(2015).
- Joshi, O., et al. Dynamic reorganization of extremely long-range promoter-promoter interactions between two states of pluripotency. Cell Stem Cell. 17 (6), 748-757 (2015).
- Mifsud, B., et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nature Genetics. 47 (6), 598-606 (2015).
- Dryden, N. H., et al. Unbiased analysis of potential targets of breast cancer susceptibility loci by Capture Hi-C. Genome Research. 24 (11), 1854-1868 (2014).
- Oudelaar, A. M., Davies, J. O. J., Downes, D. J., Higgs, D. R., Hughes, J. R. Robust detection of chromosomal interactions from small numbers of cells using low-input Capture-C. Nucleic Acids Research. 45 (22), 184(2017).
- Oudelaar, A. M., et al. Single-allele chromatin interactions identify regulatory hubs in dynamic compartmentalized domains. Nature Genetics. 50 (12), 1744-1751 (2018).
- Franke, M., et al. Formation of new chromatin domains determines pathogenicity of genomic duplications. Nature. 538 (7624), 265-269 (2016).
- Despang, A., et al. Functional dissection of the Sox9-Kcnj2 locus identifies nonessential and instructive roles of TAD architecture. Nature Genetics. 51 (8), 1263-1271 (2019).
- Oudelaar, A. M., et al. Dynamics of the 4D genome during in vivo lineage specification and differentiation. Nature Communications. 11 (1), 1-12 (2020).
- Galupa, R., Heard, E. X-chromosome inactivation: A crossroads between chromosome architecture and gene regulation. Annual Review of Genetics. 52, 535-566 (2018).
- Loda, A., Collombet, S., Heard, E. Gene regulation in time and space during X-chromosome inactivation. Nature Reviews. Molecular Cell Biology. 23 (4), 231-249 (2022).
- van Bemmel, J. G., et al. The bipartite TAD organization of the X-inactivation center ensures opposing developmental regulation of Tsix and Xist. Nature Genetics. 51 (6), 1024-1034 (2019).
- Galupa, R., et al. A conserved noncoding locus regulates random monoallelic Xist expression across a topological boundary. Molecular Cell. 77 (2), 352-367 (2020).
- Gjaltema, R. A. F., et al. Distal and proximal cis-regulatory elements sense X chromosome dosage and developmental state at the Xist locus. Molecular Cell. 82 (1), 190-208 (2022).
- Galupa, R., et al. Inversion of a topological domain leads to restricted changes in its gene expression and affects inter-domain communication. Development. 149 (9), (2022).
- Savarese, F., Flahndorfer, K., Jaenisch, R., Busslinger, M., Wutz, A. Hematopoietic precursor cells transiently reestablish permissiveness for X inactivation. Molecular and Cellular Biology. 26 (19), 7167-7177 (2006).
- Schulz, E. G., et al. The two active X chromosomes in female ESCs block exit from the pluripotent state by modulating the ESC signaling network. Cell Stem Cell. 14 (2), 203-216 (2014).
- Gnirke, A., et al. Solution hybrid selection with ultra-long oligonucleotides for massively parallel targeted sequencing. Nature Biotechnology. 27 (2), 182-189 (2009).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Piccinini, F., Tesei, A., Arienti, C., Bevilacqua, A. Cell counting and viability assessment of 2D and 3D Cell cultures: Expected reliability of the trypan blue assay. Biological Procedures Online. 19 (1), 8(2017).
- Servant, N., et al. HiC-Pro: an optimized and flexible pipeline for Hi-C data processing. Genome Biology. 16, 259(2015).
- Abdennur, N., Mirny, L. A. Cooler: scalable storage for Hi-C data and other genomically labeled arrays. Bioinformatics. 36 (1), 311-316 (2020).
- Imakaev, M., et al. Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nature Methods. 9 (10), 999-1003 (2012).
- Forcato, M., et al. Comparison of computational methods for Hi-C data analysis. Nature Methods. 14 (7), 679-685 (2017).
- Venev, S., et al. open2c/cooltools: v0.4.1. , (2021).
- Wages, J. M. NUCLEIC ACIDS | Immunoassays. Encyclopedia of Analytical Science. , Elsevier. 408-417 (2005).
- Ewels, P. A., et al. The nf-core framework for community-curated bioinformatics pipelines. Nature Biotechnology. 38 (3), 276-278 (2020).
- Servant, N., Peltzer, A. nf-core/hic: Initial release of nf-core/hic. Zenodo. , (2019).
- Furlan, G., et al. The Ftx noncoding locus controls X chromosome inactivation independently of its RNA products. Molecular Cell. 70 (3), 462-472 (2018).
- Giorgetti, L., et al. Structural organization of the inactive X chromosome in the mouse. Nature. 535 (7613), 575-579 (2016).
- Simon, C. S., et al. Functional characterisation of cis-regulatory elements governing dynamic Eomes expression in the early mouse embryo. Development. 144 (7), 1249-1260 (2017).
- Williams, R. M., et al. Reconstruction of the global neural crest gene regulatory network in vivo. Developmental Cell. 51 (2), 255-276 (2019).
- Godfrey, L., et al. DOT1L inhibition reveals a distinct subset of enhancers dependent on H3K79 methylation. Nature Communications. 10 (1), 2803(2019).
- Hanssen, L. L. P., et al. Tissue-specific CTCF-cohesin-mediated chromatin architecture delimits enhancer interactions and function in vivo. Nature Cell Biology. 19 (8), 952-961 (2017).
- Larke, M. S. C., et al. Enhancers predominantly regulate gene expression during differentiation via transcription initiation. Molecular Cell. 81 (5), 983-997 (2021).
- Oudelaar, A. M., et al. A revised model for promoter competition based on multi-way chromatin interactions at the α-globin locus. Nature Communications. 10 (1), 5412(2019).
- Long, H. K., et al. Loss of extreme long-range enhancers in human neural crest drives a craniofacial disorder. Cell Stem Cell. 27 (5), 765-783 (2020).
- Schoenfelder, S., Javierre, B. -M., Furlan-Magaril, M., Wingett, S. W., Fraser, P. Promoter Capture Hi-C: High-resolution, genome-wide profiling of promoter interactions. Journal of Visualized Experiments. (136), e57320(2018).
- Siersbæk, R., et al. Dynamic rewiring of promoter-anchored chromatin loops during adipocyte differentiation. Molecular Cell. 66 (3), 420-435 (2017).
- Rubin, A. J., et al. Lineage-specific dynamic and pre-established enhancer-promoter contacts cooperate in terminal differentiation. Nature Genetics. 49 (10), 1522-1528 (2017).
- Freire-Pritchett, P., et al. Global reorganisation of cis-regulatory units upon lineage commitment of human embryonic stem cells. eLife. 6, 21926(2017).
- Javierre, B. M., et al. Lineage-specific genome architecture links enhancers and non-coding disease variants to target gene promoters. Cell. 167 (5), 1369-1384 (2016).
- Miguel-Escalada, I., et al. Human pancreatic islet three-dimensional chromatin architecture provides insights into the genetics of type 2 diabetes. Nature Genetics. 51 (7), 1137-1148 (2019).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Baxter, J. S., et al. Capture Hi-C identifies putative target genes at 33 breast cancer risk loci. Nature Communications. 9 (1), 1028(2018).
- Sanborn, A. L., et al. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes. Proceedings of the National Academy of Sciences. 112 (47), 6456-6465 (2015).
- Owens, D. D. G., et al. Dynamic Runx1 chromatin boundaries affect gene expression in hematopoietic development. Nature Communications. 13 (1), 773(2022).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır