A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
المعالجة التلقائية للصور لتحديد هيكل حجم المجتمع لللافقاريات النهرية الكبيرة
In This Article
Summary
تستند المقالة إلى إنشاء بروتوكول مكيف لمسح واكتشاف وفرز وتحديد الأشياء الرقمية المقابلة لللافقاريات الكبيرة في الأنهار القاعية باستخدام إجراء تصوير شبه آلي. يسمح هذا الإجراء بالحصول على توزيعات الحجم الفردية ومقاييس الحجم لمجتمع اللافقاريات الكبيرة في حوالي 1 ساعة.
Abstract
حجم الجسم هو سمة وظيفية مهمة يمكن استخدامها كمؤشر حيوي لتقييم آثار الاضطرابات في المجتمعات الطبيعية. يستجيب هيكل حجم المجتمع للتدرجات الأحيائية وغير الحيوية ، بما في ذلك الاضطرابات البشرية المنشأ عبر الأصناف والنظم الإيكولوجية. ومع ذلك، فإن القياس اليدوي للكائنات الصغيرة الجسم مثل اللافقاريات القاعية الكبيرة (على سبيل المثال، >500 ميكرومتر إلى بضعة سنتيمترات) يستغرق وقتا طويلا. لتسريع تقدير هيكل حجم المجتمع ، قمنا هنا بتطوير بروتوكول لقياس حجم الجسم الفردي لللافقاريات النهرية المحفوظة بشكل شبه تلقائي ، والتي تعد واحدة من المؤشرات الحيوية الأكثر استخداما لتقييم الحالة البيئية للنظم الإيكولوجية للمياه العذبة. تم تكييف هذا البروتوكول من منهجية موجودة تم تطويرها لمسح العوالق البحرية المتوسطة باستخدام نظام مسح مصمم لعينات المياه. ويتألف البروتوكول من ثلاث خطوات رئيسية هي: (1) مسح العينات الفرعية (كسور حجم العينة الدقيقة والخشنة) من اللافقاريات النهرية الكبيرة ومعالجة الصور الرقمية لإضفاء الطابع الفردي على كل كائن مكتشف في كل صورة؛ و (2) مسح العينات الفرعية (كسور حجم العينات الدقيقة والخشنة) من اللافقاريات النهرية الكبيرة ومعالجة الصور الرقمية لإضفاء الطابع الفردي على كل كائن مكتشف في كل صورة؛ و (2) مسح العينات الفرعية (كسور حجم العينات الدقيقة والخشنة) من اللافقاريات النهرية الكبيرة ومعالجة الصور الرقمية لإضفاء الطابع الفردي على كل كائن مكتشف في كل صورة؛ و (2) مسح (2) إنشاء وتقييم والتحقق من صحة مجموعة التعلم من خلال الذكاء الاصطناعي لفصل الصور الفردية لللافقاريات الكبيرة بشكل شبه تلقائي عن المخلفات والتحف في العينات الممسوحة ضوئيا ؛ و (3) يصور هيكل حجم مجتمعات اللافقاريات الكبيرة. بالإضافة إلى البروتوكول ، يتضمن هذا العمل نتائج المعايرة ويعدد العديد من التحديات والتوصيات لتكييف الإجراء مع عينات اللافقاريات الكبيرة والنظر في مزيد من التحسينات. وبشكل عام، تدعم النتائج استخدام نظام المسح المقدم للقياس التلقائي لحجم جسم اللافقاريات النهرية الكبيرة وتشير إلى أن تصوير طيف حجمها هو أداة قيمة للتقييم البيولوجي السريع للنظم الإيكولوجية للمياه العذبة.
Introduction
تستخدم اللافقاريات القاعية الكبيرة على نطاق واسع كمؤشرات حيوية لتحديد الحالة البيئية للمسطحات المائية1. تركز معظم المؤشرات لوصف مجتمعات اللافقاريات الكبيرة على المقاييس التصنيفية. ومع ذلك ، يتم تشجيع أدوات التقييم البيولوجي الجديدة التي تدمج حجم الجسم لتوفير منظور بديل أو تكميلي لنهج التصنيف 2,3.
يعتبر حجم الجسم مشط شعر مرتبط بسمات حيوية أخرى مثل التمثيل الغذائي والنمو والتنفس والحركة4. علاوة على ذلك ، يمكن أن يحدد حجم الجسم الموقف الغذائي والتفاعلات5. تعرف العلاقة بين حجم الجسم الفردي والكتلة الحيوية الطبيعية (أو الوفرة) حسب فئة الحجم في المجتمع على أنها طيف الحجم6 وتتبع النمط العام للانخفاض الخطي في الكتلة الحيوية الطبيعية مع زيادة الحجم الفردي على مقياس لوغاريتمي7. تمت دراسة منحدر هذه العلاقة الخطية على نطاق واسع نظريا ، واستخدمتها الدراسات التجريبية عبر النظم الإيكولوجية كمؤشر بيئي لهيكل حجم المجتمع4. ومن المؤشرات التركيبية الأخرى لهيكل حجم المجتمع الذي تم استخدامه بنجاح في دراسات أداء النظم الإيكولوجية للتنوع البيولوجي تنوع حجم المجتمع ، والذي يتم تمثيله كمؤشر شانون لفئات الحجم في طيف الحجم أو نظيره ، والذي يتم حسابه بناء على توزيعات الحجم الفردية8.
وفي النظم الإيكولوجية للمياه العذبة، يستخدم هيكل حجم المجموعات الحيوانية المختلفة كمؤشر رننحي لتقييم استجابة المجتمعات الأحيائية للتدرجات البيئية9،10،11 والاضطرابات البشرية المنشأ12،13،14،15،16. اللافقاريات الكبيرة ليست استثناء ، ويستجيب هيكل حجمها أيضا للتغيرات البيئية17,18 والاضطرابات البشرية المنشأ ، مثل التعدين 19 ، أو استخدام الأراضي 20 ، أو تخصيب النيتروجين (N) والفوسفور (P)20,21,22. ومع ذلك ، فإن قياس مئات الأفراد لوصف هيكل حجم المجتمع هو مهمة شاقة وتستغرق وقتا طويلا وغالبا ما يتم تجنبها كقياس روتيني في المختبرات بسبب ضيق الوقت. وبالتالي ، تم تطوير العديد من طرق التصوير شبه الأوتوماتيكية أو الأوتوماتيكية لتصنيف وقياس العينات23،24،25،26. ومع ذلك ، فإن معظم هذه الطرق تركز على التصنيف التصنيفي أكثر من التركيز على الحجم الفردي للكائنات الحية وليست جاهزة للاستخدام لجميع أنواع اللافقاريات الكبيرة. في بيئة العوالق البحرية ، تم استخدام نظام تحليل صور المسح على نطاق واسع لتحديد الحجم والتكوين التصنيفي لمجتمعات العوالق الحيوانية27،28،29،30،31. يمكن العثور على هذه الأداة في العديد من المعاهد البحرية في جميع أنحاء العالم ، ويتم استخدامها لمسح عينات العوالق الحيوانية المحفوظة للحصول على صور رقمية عالية الدقة للعينة بأكملها. يكيف البروتوكول الحالي استخدام هذه الأداة لتقدير طيف مجتمع اللافقاريات الكبيرة في الأنهار بطريقة تلقائية سريعة دون الاستثمار في إنشاء جهاز جديد.
يتكون البروتوكول من مسح عينة ومعالجة الصورة بأكملها للحصول تلقائيا على صور مفردة (أي المقالات القصيرة) للكائنات الموجودة في العينة. تميز العديد من مقاييس الشكل والحجم وميزات المستوى الرمادي كل كائن وتسمح بالتصنيف التلقائي للكائنات إلى فئات ، والتي يتم التحقق من صحتها بعد ذلك بواسطة خبير. يتم حساب الحجم الفردي لكل كائن حي باستخدام الحجم الحيوي الإهليلجي (مم3) ، والذي يتم اشتقاقه من مساحة الكائن الحي المقاسة بالبكسل. هذا يسمح بالحصول على طيف حجم العينة بطريقة سريعة. على حد علمنا ، تم استخدام نظام التصوير الضوئي هذا فقط لمعالجة عينات العوالق الحيوانية المتوسطة ، ولكن من المحتمل أن يسمح الجهاز بالعمل مع اللافقاريات القاعية في المياه العذبة.
وبالتالي ، فإن الهدف العام من هذه الدراسة هو تقديم طريقة للحصول بسرعة على الحجم الفردي لللافقاريات النهرية المحفوظة من خلال تكييف بروتوكول موجود كان يستخدم سابقا مع العوالق البحرية المتوسطة27،32،33. يتكون الإجراء من استخدام نهج شبه تلقائي يعمل بجهاز مسح ضوئي لمسح عينات المياه وثلاثة برامج مفتوحة لمعالجة الصور الممسوحة ضوئيا. يتم هنا تقديم بروتوكول معدل لمسح واكتشاف وتحديد اللافقاريات النهرية الرقمية للحصول تلقائيا على هيكل حجم المجتمع ومقاييس الحجم ذات الصلة. كما يتم تقديم تقييم الإجراء والمبادئ التوجيهية لتعزيز الكفاءة بناء على 42 صورة ممسوحة ضوئيا لعينات اللافقاريات النهرية الكبيرة التي تم جمعها من ثلاثة أحواض في شبه الجزيرة الأيبيرية الشمالية الشرقية (NE) (Ter و Segre-Ebre و Besòs).
تم جمع العينات على امتدادات النهر 100 متر وفقا لبروتوكول أخذ العينات الميدانية والتحليل المختبري لللافقاريات الكبيرة في الأنهار القاعية في الأنهار القابلة للعبور من الحكومة الإسبانية34. تم جمع العينات باستخدام جهاز أخذ العينات (الإطار: 0.3 م × 0.3 م ، شبكة: 250 ميكرومتر) بعد مسح متعدد الموائل. في المختبر ، تم تنظيف العينات وغربلتها من خلال شبكة 5 مم و 500 ميكرومتر للحصول على عينتين فرعيتين: عينة فرعية خشنة (شبكة 5 مم) وعينة فرعية دقيقة (شبكة 500 ميكرومتر) ، والتي تم تخزينها في قوارير منفصلة وحفظها في 70٪ من الإيثانول. يسمح فصل العينة إلى كسرين بحجم تقدير أفضل لهيكل حجم المجتمع ، نظرا لأن الكائنات الكبيرة أكثر ندرة وأقل من الكائنات الصغيرة. خلاف ذلك ، فإن العينة الممسوحة ضوئيا لها تمثيل متحيز للجزء كبير الحجم.
Protocol
ملاحظة: يعتمد البروتوكول الموصوف هنا على النظام الذي طوره Gorsky et al.27 للعوالق البحرية المتوسطة. يمكن العثور على وصف محدد للماسح الضوئي (ZooSCAN) ، وبرنامج المسح الضوئي (VueScan 9x64 [9.5.09]) ، وبرنامج معالجة الصور (Zooprocess ، ImageJ) ، وبرنامج التعرف التلقائي (معرف العوالق) في المراجع السابقة32،33. لضبط أحجام اللافقاريات القاعية بشكل أفضل فيما يتعلق بالعوالق الحيوانية المتوسطة ، بمجرد إنشاء المشروع وفقا للبروتوكولالأصلي 32,33 ، قم بتغيير معلمة الحجم الأدنى (minsizeesd_mm) إلى 0.3 مم والمعلمة من الحجم الأقصى (maxsizeesd_mm) إلى 100 مم في ملف التكوين. للمساعدة في اتباع البروتوكول ، يتم تلخيص ذلك في مخطط العمل (الشكل 1). يتم تخزين المشروع الذي تم إنشاؤه في المجلد C الخاص بالكمبيوتر ويتم تنظيمه في المجلدات التالية: PID_process و Zooscan_back و Zooscan_check و Zooscan_config و Zooscan_meta و Zooscan_results و Zooscan_scan. يتكون كل مجلد من عدة مجلدات فرعية تستخدمها تطبيقات البرامج المختلفة في الخطوات التالية من البروتوكول.
1. اقتناء الصور الرقمية لعينات اللافقاريات الكبيرة
- مسح ومعالجة الفراغ
ملاحظة: قم بإنشاء صورتين فارغتين يوميا قبل المسح الضوئي لاستخراج عمليات المسح الضوئي في الخلفية أثناء معالجة الصور الممسوحة ضوئيا في نفس اليوم.- قم بتشغيل الماسحة الضوئية وقم بتشغيل الضوء في الوضع المزدوج لعرض الضوء الأبيض من الأعلى ومن الأسفل.
ملاحظة: عند مسح عينات العوالق الحيوانية المتوسطة ، يتم استخدام اتجاه الضوء الصاعد ، ولكن نظرا لأن اللافقاريات الكبيرة أكثر غموضا ، فمن المستحسن تبديل الضوء إلى وضع مزدوج. - نظف وشطف درج المسح بماء الصنبور.
- صب 110 مل من ماء الصنبور المخزن في درجة حرارة الغرفة (RT) في درج المسح الضوئي حتى يتم تغطية الزجاج. ضع الإطار الكبير (24.5 سم × 15.8 سم) على درج المسح الضوئي في الموضع الصحيح (مع وجود الزاوية في الجزء العلوي الأيسر من درج المسح الضوئي)، واملأه بماء الصنبور حتى يتم تغطية خطوة الإطار لتجنب تأثير الغضروف المفصلي، والذي من شأنه أن يغير الصورة الممسوحة ضوئيا. أغلق غطاء الماسحة الضوئية.
ملاحظة: استخدم الماء في RT لتجنب التكثيف وتكوين الفقاعات. نظف الإطار بدون علامات أو قطرات لتجنب انعكاس الضوء. - انتقل إلى برنامج معالجة الصور ، وحدد مشروع العمل ، وانقر فوق مسح (تحويل) صورة الخلفية.
- انتقل إلى برنامج المسح وانقر على معاينة. تأكد من معاينة الصورة الممسوحة ضوئيا ، وتحقق من عدم وجود خطوط أو بقع ، وانتظر لمدة 30 ثانية على الأقل قبل بدء مسح آخر. انقر فوق Scan (مسح ضوئي) واضغط على OK (موافق) في نافذة الإرشادات قبل الفحص الثاني لإرسال البيانات من برنامج المسح الضوئي إلى برنامج معالجة الصور.
ملاحظة: قم بإجراء المسح الضوئي مرتين للحصول على عمليتي المسح الضوئي في الخلفية اللتين ستكونان من الفراغ. تتم هذه الخطوة مرة واحدة كل يوم قبل بدء معالجة العينة ، ويتم تخزين الصور في مجلد Zooscan_back. - أغلق برنامج المسح الضوئي بعد الانتهاء من الفحص.
- قم بتشغيل الماسحة الضوئية وقم بتشغيل الضوء في الوضع المزدوج لعرض الضوء الأبيض من الأعلى ومن الأسفل.
- تحضير العينات ومسحها ضوئيا
تنبيه: الإيثانول سائل قابل للاشتعال ويمكن أن يسبب تلفا / تهيجا خطيرا للعين.- املأ عينة البيانات الوصفية. انتقل إلى برنامج معالجة الصور وحدد تعبئة عينة البيانات الوصفية. أدخل نموذج الهوية، وانقر فوق موافق، واملأ البيانات الوصفية.
ملاحظة: تم إنشاء ملف التعريف خصيصا لعينات العوالق الحيوانية المتوسطة ، لذلك لا يتناسب مع منهجية أخذ عينات اللافقاريات القاعية الكبيرة ، ومع ذلك يجب ملء جميع حقول الملف قبل الفحص ، أو ستظهر علامة خطأ. - صب 110 مل من الإيثانول بنسبة 70٪ في درج المسح حتى يتم تغطية الزجاج ووضع الإطار الكبير (24.5 سم × 15.8 سم) مع الزاوية في الجزء العلوي الأيسر من درج المسح.
ملاحظة: العمل مع الإيثانول بدلا من الماء ، حيث يتم حفظ اللافقاريات الكبيرة في الإيثانول. في الماء ، تطفو وتنجرف في درج المسح الضوئي ، مما يمنع الصورة الحادة ، وبالتالي قياسات الحجم الموثوقة. يجب حفظ الإيثانول في RT لتجنب التكثيف وتكوين الفقاعات. - صب عينة اللافقاريات الكبيرة في درج المسح الذي يحده الإطار ، وقم بتغطية خطوة الإطار بمزيد من الإيثانول إذا لزم الأمر.
ملاحظة: الامتناع عن إضافة الكثير من الإيثانول لتجنب الكائنات الحية العائمة والانجراف. - قم بتجانس العينة في جميع أنحاء منطقة الإطار ، ووضع أكبر الأفراد في وسط الدرج لمعالجة الصور بشكل صحيح ، وإغراق الكائنات الحية العائمة باستخدام إبرة خشبية.
ملاحظة: إذا كانت العينة الفرعية تحتوي عدديا على أكثر من 1000 فرد، فقم بتقسيم العينة الفرعية إلى كسرين أو أكثر لتقليل لمس الكائنات الحية في الصورة الممسوحة ضوئيا، وامسح الكسور ضوئيا بشكل منفصل. - افصل الكائنات الحية التي تلامس والكائنات الحية التي تلامس حواف الإطار باستخدام الإبرة الخشبية.
ملاحظة: تتطلب هذه الخطوة 5-20 دقيقة. تعتبر الكائنات الحية التي تلمس كائنا واحدا بواسطة البرنامج. وبالتالي ، في هذه الحالات ، لا تتوافق الأحجام الفردية المحسوبة مع الكائنات الفردية الفعلية ويمكن أن تؤدي إلى تحيز تقدير هيكل حجم المجتمع. هناك إمكانية لتحرير الصورة باستخدام برنامج معالجة الصور لفصلها ، ولكن هذه الخطوة الإضافية تتضمن 1.5 ساعة على الأقل من إعادة المعالجة ؛ وبالتالي ، يوصى بشدة بالفصل اليدوي. - لمسح العينة ضوئيا ، أغلق غطاء الماسحة الضوئية ، وانتقل إلى برنامج معالجة الصور ، وحدد مشروع العمل ، وانقر فوق SCAN Sample with Zooscan (للأرشفة ، بدون عملية).
- حدد العينة واتبع التعليمات.
- انتقل إلى برنامج المسح وانقر على معاينة. تأكد من معاينة الصورة الممسوحة ضوئيا ، وتحقق من عدم وجود خطوط أو بقع ، وانتظر 30 ثانية على الأقل قبل بدء مسح آخر.
- بعد 30 ثانية على الأقل ، انقر فوق الزر Scan في برنامج المسح.
ملاحظة: اضغط على OK في برنامج معالجة الصور بعد الضغط على Scan (مسح ضوئي ) في برنامج المسح الضوئي. لا تضغط على أي مفتاح على لوحة مفاتيح الكمبيوتر ، وتجنب اهتزازات الفحص أثناء المسح. يتم إنشاء ثلاثة ملفات في مجلد Zooscan_scan > _raw : (i) تنسيق ملف صورة موسومة (.tif) (16 بت) ؛ (ii) مستند نصي قياسي يسمى LOG (.txt) يسجل معلومات عن معلمات المسح الضوئي ؛ و (iii) وثيقة نصية قياسية تسمى META (.txt) مع معلومات عن طرق أخذ العينات. - تحقق من صحة الفحص الخام.
ملاحظة: إذا كان الفحص يحتوي على خطوط ضوئية أو مشكلات مرئية أخرى، ففكر في تكرار الفحص لتجنب حدوث مشكلات في الخطوات التالية.
- املأ عينة البيانات الوصفية. انتقل إلى برنامج معالجة الصور وحدد تعبئة عينة البيانات الوصفية. أدخل نموذج الهوية، وانقر فوق موافق، واملأ البيانات الوصفية.
- استعادة العينة
- قم بإزالة الإطار وشطفه فوق درج المسح باستخدام زجاجة ضغط مملوءة بنسبة 70٪ من الإيثانول لاستعادة أي لافقاريات كبيرة متصلة.
- ارفع الجزء العلوي من الماسح الضوئي لاسترداد جميع الكائنات الحية والإيثانول من الدرج عبر قمع استرجاع المسح الضوئي إلى دورق. مع استمرار رفع الجزء العلوي من الماسح الضوئي ، اشطف الدرج بزجاجة الضغط لمسح أي كائنات متبقية.
- مرر العينات والإيثانول من الدورق عبر شبكة 500 ميكرومتر للاحتفاظ باللافقاريات في الشبكة ، وتخزينها مرة أخرى في قنينة تحتوي على 70٪ من الإيثانول.
- بمجرد استرداد جميع العينات في القارورة ، قم بتنظيف الدرج بماء الصنبور.
ملاحظة: اغسل الدرج بماء الصنبور بين العينات لتقليل ترسيب الإيثانول ، مما يغير معالجة الصورة. اشطف الإطار بماء الصنبور لتجنب الأضرار المحتملة المتعلقة باستخدام الإيثانول. في نهاية اليوم ، قم بتنظيف الدرج باستخدام ماء الصنبور وجففه برفق بالورق لتجنب الخدوش.
- معالجة الصور
- انتقل إلى برنامج معالجة الصور وحدد تحويل ومعالجة الصور والكائنات الحية في وضع الدفعات ثم تحويل ومعالجة الصورة والجسيمات (الصورة في مجلد RAW). احتفظ بالإعدادات الافتراضية وانقر فوق موافق. ستظهر النهاية العادية في نهاية العملية.
ملاحظة: سيتم إنشاء ملف PID والمقالات القصيرة المقابلة لجميع الكائنات المكتشفة في الصورة الممسوحة ضوئيا (في ملف مجموعة الصور المشتركة [.jpg]) في مجلد Zooscan_scan > _work. ملف PID هو ملف واحد يخزن جميع البيانات الوصفية (ملف التعريف) ، والبيانات الفنية المرتبطة بملف السجل ، وجدول يحتوي على 36 متغيرا مقاسا لجميع الكائنات المكتشفة في الصورة. تتوافق المتغيرات المقاسة مع تقديرات مختلفة للمستوى الرمادي والبعد الكسوري والشكل والحجم. المتغيرات التي يمكن استخدامها لتقدير الحجم هي المساحة والمحاور الرئيسية والثانوية للقطع الناقص بمساحة مساوية للكائن (انظر القسم 3 من البروتوكول). يعتمد وقت المعالجة على كثافة الصورة وخصائص الكمبيوتر ، ويمكن إطلاقها بين العينات أثناء استعادة وإعداد العينة التالية. خلاف ذلك ، يوصى ببدء معالجة العينات الممسوحة ضوئيا كل يوم في وضع الدفعات أثناء الليل والتحقق من معالجة الصور المناسبة في صباح اليوم التالي. - تحقق مما إذا كانت الخلفية في الصورة المعالجة قد تم طرحها بشكل مناسب من صورة العينة باستخدام برنامج معالجة الصور أو عن طريق التحقق من صور القناع (المنتهية في MSK1.gif) الموجودة في Zooscan_scan > _work. إذا كانت الخلفية تحتوي على مناطق مشبعة أو العديد من النقاط ، ففكر في تكرار الفحص لضمان صور عالية الجودة.
ملاحظة: لتجنب المناطق المشبعة في الخلفية ، يجب شطف درج المسح بماء الصنبور بعد كل مسح بالإيثانول. من المهم أيضا (1) تقليل عدد الأفراد الممسوحين ضوئيا (عن طريق تجزئة العينة والمسح الضوئي في طيات مختلفة) ؛ (2) تأكد من وضع الكائنات الحية الكبيرة في وسط درج المسح ؛ (3) استخدام الإيثانول النظيف والمصفى ؛ (4) تقليل الأوساخ على العينات ؛ (5) التأكد من أن حجم الإيثانول للمسح كاف ؛ و (6) تأكد من أن التأخير بين معاينة العينة والمسح الضوئي لا يقل عن 30 ثانية.
- انتقل إلى برنامج معالجة الصور وحدد تحويل ومعالجة الصور والكائنات الحية في وضع الدفعات ثم تحويل ومعالجة الصورة والجسيمات (الصورة في مجلد RAW). احتفظ بالإعدادات الافتراضية وانقر فوق موافق. ستظهر النهاية العادية في نهاية العملية.
- فصل الكائنات الحية التي تلامس
ملاحظة: عندما يكون هناك العديد من المقالات القصيرة مع الكائنات الحية اللامسة ، من الضروري فصل صور الكائنات الملامسة عن الكائنات الأخرى و / أو عن الألياف / الحطام لضمان تقدير مناسب لهيكل حجم المجتمع.- انتقل إلى برنامج معالجة الصور لاكتشاف المقالات القصيرة ذات الكائنات المتعددة. حدد الفصل باستخدام المقالات القصيرة واضغط على موافق. في نافذة تحديد التكوين ، احتفظ بالإعدادات الافتراضية وانقر فوق موافق.
- في النافذة الفصل عن المقالات القصيرة ، احتفظ بالإعدادات الافتراضية، بالإضافة إلى تحديد إضافة مخططات تفصيلية على المقالات الكاملة، ثم حدد العينة المراد تحريرها.
- افصل الكائنات الحية المؤثرة في كل مقالة قصيرة تنبثق عن طريق رسم خط بالماوس (اضغط على زر اللفة للرسم). بمجرد اكتمال الفصل في المقالة القصيرة ، انقر فوق الزر X في الزاوية العلوية اليمنى من النافذة ، واضغط على نعم لمعالجة النافذة التالية. اضغط على NO لإنهاء التغييرات وحفظها. في نهاية العملية ، ستظهر NORMAL END إذا كان كل شيء صحيحا.
- بعد الفصل ، أعد معالجة الصورة للحصول على بيانات الكائن المحدثة. انتقل إلى برنامج معالجة الصور ، وانقر فوق معالجة الصورة (المحولة) (العملية الأولى) ، وحدد معالجة الجسيمات مرة أخرى من الصور المعالجة في مجلدات العمل الفرعية. حدد العينة ، وفي نافذة معالجة الصورة الواحدة ، احتفظ بالإعدادات الافتراضية ، وحدد العمل مع قناع الفصل (CREATE-EDIT-INCLUDE) ، ثم انقر فوق موافق. في نهاية العملية ، ستظهر NORMAL END إذا كان كل شيء صحيحا.
- في نافذة التحكم في الفصل ، اضغط على موافق لحفظ الصورة مع الخطوط قبل المعالجة ؛ في حالة وجود صورة سابقة ، سيتم استبدالها.
- في نافذة قناع التحكم في الفصل ، إذا لزم الأمر، حدد تحرير لإضافة خطوط فصل إلى القناع باستخدام الماوس لفصل الكائنات الحية التي لم تظهر من قبل في خطوة الفصل باستخدام المقالات القصيرة. عند الانتهاء، قم بإنهاء العملية، وفي نافذة التحكم في قناع الفصل ، حدد نعم لقبول القناع. في نهاية العملية ، ستظهر NORMAL END إذا كان كل شيء صحيحا.
ملاحظة: تستغرق إعادة معالجة عينة باستخدام قناع فصل وقتا طويلا (قد يستغرق ذلك أكثر من 1.5 ساعة لكل عينة). يفضل تخصيص الوقت المطلوب في الخطوة 1.2.5 لتجنب هذه الخطوة الإضافية.
2. التعرف التلقائي على الكائنات
ملاحظة: قم بإنشاء مجموعة تعلم للتنبؤ تلقائيا بهوية الكائنات المكتشفة ، وبالتالي فصل الكائنات الحية عن الحطام في العينة.
- إنشاء مجموعة التعلم
- انسخ الصور وملفات .pid المقترنة بالصور التي سيتم استخدامها لإنشاء فئات مجموعة التعليم من Zooscan_scan > _work إلى PID_process > Unsorted_vignettes_pid.
ملاحظة: اختر مجموعة فرعية من العينات ذات التنوع التصنيفي العالي ومواقع أخذ العينات المختلفة و/أو مواسم أخذ العينات لضمان أقصى قدر من التمثيل للكائنات الحية في العينات. - في مجلد مجموعة التعلم PID_process >، قم بإنشاء مجلد فرعي باسم مجموعة التعلم الجديدة (أي yyyymmdd_raw_LS)، وبداخله، قم بإنشاء المجلدات الفرعية التي تتوافق مع كل فئة من فئات مجموعة التعلم (أي اللافقاريات الكبيرة والحطام واللافقاريات الأخرى).
ملاحظة: للحصول بكفاءة على هيكل حجم المجتمع لعينات اللافقاريات الكبيرة النهرية ، يوصى باستخدام مجموعة تعليمية تستند إلى ثلاث فئات فقط: اللافقاريات الكبيرة ، واللافقاريات الأخرى ، والحطام. تفصل مجموعة التعلم هذه بشكل أساسي المقالات القصيرة للأشياء المقابلة للكائنات الحية عن تلك المقابلة للحطام (مثل الألياف أو الجسيمات أو الطحالب الخيطية). - انتقل إلى برنامج معالجة الصور (الوضع المتقدم فقط) واختر EXTRACT Vignettes ل PLANKTON IDENTIFIER (المقالات القصيرة غير المصنفة للتدريب). احتفظ بالخيارات الافتراضية وحدد المربع إضافة مخططات تفصيلية .
- انتقل إلى برنامج التعريف التلقائي ، وانقر فوق التعلم ، وحدد من PID_process > Learning_set المجلد الفرعي الذي تم إنشاؤه لمجموعة التعلم الجديدة (الخطوة 2.1.2) ، واضغط على موافق.
- في القسم الأيسر (إبهام غير مفروز) من النافذة المفتوحة ، حدد المجلد غير المصنف vignettes_pid. حدد المقالات القصيرة واسحبها بالماوس من الإبهام غير المصنف إلى مجلد الفئة المقابلة لها في القسم الأيمن ، الإبهام الذي تم فرزه ، لتصنيف كل كائن إلى فئات محددة. سيتم تمييز المقالات القصيرة المنقولة بعلامة X حمراء.
ملاحظة: حدد الفئات يدويا عن طريق إنشاء مجلدات فرعية في مجلد الإبهام الذي تم فرزه أو قم بإنشائها بالنقر فوق رمز المجلدات في البرنامج. لا تحرك أكثر من 50 مقالة قصيرة في نفس الوقت. - بمجرد اكتمال جميع الفئات بالكائنات المحددة (حوالي 300 كائن لكل فئة) ، انقر فوق إنشاء ملف تعليمي واحفظه بالاسم المطلوب.
ملاحظة: سيتم حفظ مجموعة التعليم كملف .pid في مجلد مجموعة التعلم PID_process > الخاص بالمشروع. يوصى بإنشاء واختبار عدة مجموعات تعليمية بمستويات مختلفة من الفئات (من الأشكال الخشنة إلى الأشكال الدقيقة) وبتوازن مختلف لعدد الكائنات داخل كل فئة. ابدأ بمجموعة تعلم خشنة تحتوي على عدد قليل من الفئات و 50 عنصرا على الأقل لكل فئة ، ثم قم بزيادة عدد الكائنات في كل فئة و / أو قم بإنشاء مجموعات تعلم أدق. يجب أن تكون الفئة ممثلة لتباينها في مجموعة العينات.
- انسخ الصور وملفات .pid المقترنة بالصور التي سيتم استخدامها لإنشاء فئات مجموعة التعليم من Zooscan_scan > _work إلى PID_process > Unsorted_vignettes_pid.
- تقييم مجموعة التعلم
ملاحظة: قم بإجراء التحقق المتقاطع مع ثنيتين وخمس تجارب باستخدام طريقة Random Forest مع برنامج التعرف التلقائي للحصول على مصفوفة ارتباك للتصنيف الناتج للكائنات.- انتقل إلى برنامج التصنيف التلقائي وانقر على تحليل البيانات.
- في تحديد ملف التعلم، حدد ملف مجموعة التعليم الذي تم إنشاؤه من PID_process > مجموعة التعلم.
- في تحديد طريقة، اختر أسلوب الغابة العشوائية للتحقق التبادلي . في المتغيرات الأصلية، قم بإلغاء تحديد متغيرات الموضع (X وY وXM وYM وBX وBY والارتفاع). في المتغيرات المخصصة ، حدد ESD فقط.
ملاحظة: تستخدم هذه الطريقة جزءا عشوائيا واحدا من مجموعة التعلم للتعرف على الجزء الآخر (ضعفان) ، ويتكرر ذلك خمس مرات للتأكد من أنه قوي إحصائيا. - انقر فوق بدء التحليل ، واحفظ النتائج كما Analysis_name.txt في مجلد التنبؤ PID_process >. عند اكتمال التحليل بنجاح، قم بإنهاء تحليل البيانات.
- انتقل إلى مجلد PID_process > Prediction وانقر على ملف التحقق المتبادل. ستظهر نافذة مع مصفوفة الارتباك للتصنيف الحقيقي (الصفوف) مقابل التصنيف التلقائي (الأعمدة).
ملاحظة: الاستدعاء هو النسبة المئوية للكائنات الحية التي تنتمي إلى مجموعة تم التعرف عليها تلقائيا بشكل جيد ، في حين أن 1-precision هي النسبة المئوية للكائنات الحية المصنفة بواسطة الخوارزمية كمجموعة لم يتم التعرف عليها (التلوث في مجموعة). يجب أن يكون الاستدعاء أعلى من 70٪ ، ويجب أن يكون التلوث (1-الدقة) أقل من 20٪. - كرر الخطوات 2.1-2.5 إذا تم إنشاء عدة مجموعات تعليمية ويجب الحصول على استدعاء ودقة واحدة لكل مجموعة.
ملاحظة: إذا تم إنشاء عدة مجموعات تعلم، فاختر المجموعة التي تتمتع بأكبر قدر من التذكر (التعرف الجيد) والدقة (التلوث المنخفض) للمجموعة ذات الاهتمام (أي اللافقاريات الكبيرة) لاختبار التنبؤ التلقائي لمجموعة من العينات في الخطوة التالية.
- التنبؤ بتحديد اللافقاريات الكبيرة
ملاحظة: استخدم مجموعة التعلم المحددة للتنبؤ بهوية جميع الكائنات في مجموعة فرعية من العينات باستخدام برنامج التعرف التلقائي مع خوارزمية غابة عشوائية.- انتقل إلى برنامج التعريف التلقائي وانقر على تحليل البيانات.
- في تحديد ملف التعلم، حدد ملف مجموعة التعليم من مجموعة التعلم PID_process > التي يجب استخدامها للتنبؤ.
- في تحديد نموذج (ملفات) ، حدد من مجلد PID_results العينات (ملفات PID) التي سيتم التنبؤ بها.
ملاحظة: معالجة 20 ملف .pid كحد أقصى في نفس الوقت لتجنب الأخطاء المتعلقة بمشاكل الذاكرة. إذا تمت معالجة عدد كبير جدا من ملفات .pid في نفس الوقت ، فستظهر العملية نهاية صحيحة ولكن قد لا تتم معالجتها بشكل جيد ، وقد يحدث خطأ في الخطوات التالية عند المعالجة باستخدام برنامج معالجة الصور. - في تحديد طريقة، اختر أسلوب الغابة العشوائية . ضع علامة على حفظ النتائج التفصيلية لكل عينة. في المتغيرات الأصلية، قم بإلغاء تحديد متغيرات الموضع (X وY وXM وYM وBX وBY والارتفاع). في المتغيرات المخصصة ، حدد ESD فقط.
- انقر فوق بدء التحليل ، واحفظ النتائج كما Analysis_name.txt في مجلد التنبؤ PID_process >.
- التحقق اليدوي
ملاحظة: يقوم أحد الخبراء يدويا بالتحقق من صحة التنبؤ من الخطوة السابقة لإعادة تصنيف الكائنات المصنفة بشكل خاطئ إلى الفئة الصحيحة.- انسخ ملفات Analysis_sample_dat1.txt المراد التحقق من صحتها من مجلد توقع PID_process > إلى مجلد PID_process > Pid_results.
- انتقل إلى برنامج معالجة الصور وحدد استخراج المقالات القصيرة في المجلدات وفقا للتنبؤ أو التحقق من الصحة. ثم حدد استخدام الملفات المتوقعة من المجلد "pid_results". احتفظ بالإعدادات الافتراضية واضغط على موافق.
- يقوم البرنامج بإنشاء مجلد يسمى sample_yyyymmdd_hhmm_to_validate مع الكائنات المتوقعة في مجلد المقالات القصيرة PID_process > التي تم فرزها.
- انتقل إلى مجلد PID_process > المقالات القصيرة التي تم فرزها، وانسخ المجلد sample_yyyymmdd_ hhmm_to_validate. استبدل اسم المجلد _to التحقق من صحته ب _validated.
- للتحقق يدويا من صحة التصنيف التلقائي ، افتح sample_yyyymmdd_ hhmm_validated المجلد ، وراجع جميع المقالات القصيرة من كل مجلد فرعي (فئة) لتحديد ما إذا كانت هناك كائنات مصنفة بشكل خاطئ. عندما يتم تصنيف كائن واحد بشكل خاطئ ، اسحب المقالة القصيرة باستخدام الماوس إلى المجلد الصحيح (الفئة).
- انتقل إلى برنامج معالجة الصور وحدد LOAD Identifications من المقالات القصيرة التي تم فرزها. احتفظ بالإعدادات الافتراضية وحدد yyyymmdd_hhmm_name_validated المراد معالجتها.
- انتقل إلى PID_process > Pid_results > Dat1_validated، حيث تم إنشاء ملف باسم Id_from_sorted_vignettes_yyyymmdd_hhmm.txt وملف .txt واحد لكل نموذج من العينات التي تم التحقق من صحتها (sample_tot_1_dat1.txt).
ملاحظة: تحتوي هذه الملفات .txt على عمود جديد يعرض التنبؤ ، يسمى pred_valid_Id_yyyymmdd_hhmm ، والذي يحدد تصنيف الخبراء لكل كائن (أي التصنيف الذي تم التحقق من صحته). ويمكن إنشاء فئات جديدة (مثل فئات تصنيفية أدق) في هذه المرحلة، أثناء عملية التحقق. ومع ذلك ، احتفظ باسم الفئة الأصلية بالاسم الجديد (على سبيل المثال ، macroinvertebrate_chironomidae). وهذا يسمح بتتبع الفئة الأصلية عند حساب الاستدعاء والدقة وتجميع جميع اللافقاريات الكبيرة بسهولة لحساب معلمات بنية حجم المجتمع (أي طيف الحجم وتنوع الحجم). يوفر الملف النصي البيانات المرتبطة بكل كائن ، بما في ذلك المحاور الثانوية والرئيسية المستخدمة للحصول على الحجم الإهليلجي لكل كائن حي كمقياس لحجم الجسم الفردي. علاوة على ذلك ، يحتوي العمودين الأخيرين من الجدول على الفئات المتوقعة والتحقق من صحتها لكل كائن (صف) ، والتي تسمح بحساب ، حسب الفئة ، استدعاء ودقة مجموعة التعلم على مجموعة فرعية من العينات.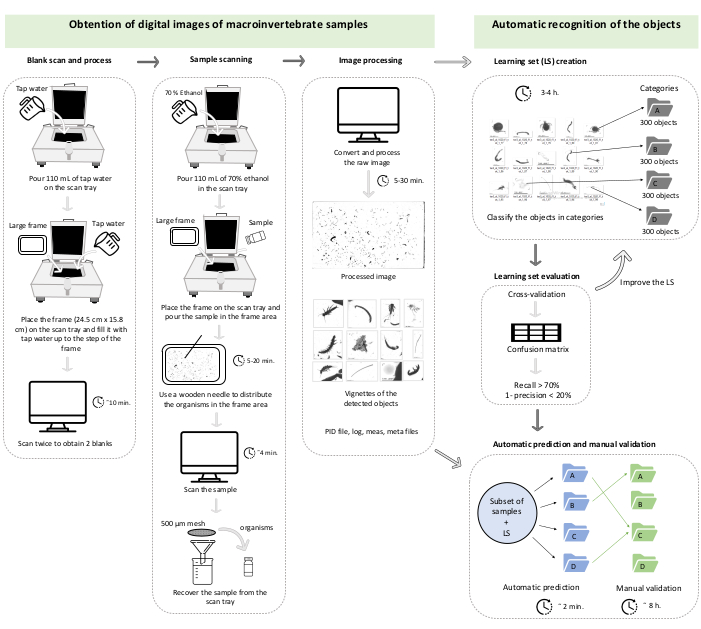
الشكل 1: مخطط عمل يمثل القسم 1 والقسم 2 من البروتوكول. الأوقات توضيحية ويمكن أن تتغير اعتمادا على الكمبيوتر ، ووفرة المقالات القصيرة للمعالجة ، وعدد فئات مجموعة التعلم. تتوافق هذه الحالة مع التحقق من صحة مجموعة تعليمية من ثلاث فئات على مجموعة من 42 عينة فرعية (في المجموع ، 47473 مقالة قصيرة). الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
3. حساب توزيع الحجم الفردي وأطياف الحجم ومقاييس الحجم
ملاحظة: تم إجراء الحسابات المذكورة في هذا القسم باستخدام Matlab (انظر البرنامج النصي كملف تكميلي 1).
- توزيع الحجم الفردي
- يحتوي العمود الأخير من ملف Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt على التصنيف الذي تم التحقق من صحته للكائنات. حدد فقط العناصر المصنفة على أنها لافقاريات كبيرة لتصوير توزيع حجمها الفردي في العينة.
ملاحظة: حجم الجسم الفردي يتوافق مع الحجم الإهليلجي للكائنات اللافقارية الكبيرة. يوفر النظام القياسات بالبكسل. - قم بربط المتجهات بقياسات الحجم من كلا المسحين ، لأن كل كسر له أس أخذ عينات فرعية مختلف. قبل التسلسل ، صحح التجزئة عن طريق تكرار متجهات الحجم عدة مرات كما تم تجزئة العينة الفرعية المقابلة.
ملاحظة: هذه الخطوة مطلوبة إذا كان الفحص يتوافق مع جزء من العينة (أي خشن أو ناعم). - احسب الحجم الإهليلجي من المحاور الرئيسية (M) والصغرى (M) للإهليلجية البرولات بنفس مناطق البكسل مثل الكائنات الحية. قبل حساب الحجم الإهليلجي ، قم بتحويل المحاور الرئيسية (M) والثانوية (m) من وحدات البكسل إلى المليمترات (mm) باستخدام عامل التحويل التالي (cf):
1 بكسل = 2,400 نقطة لكل بوصة
1 بوصة = 25.4 مم
cf = 25.4/2400
الحجم الإهليلجي (بيضاوي الشكل مع وحدات في مم3) يتوافق مع: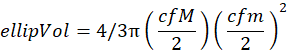
- تصوير دالة كثافة الاحتمال لتوزيع الحجم الفردي على مقياس log2 .
- يحتوي العمود الأخير من ملف Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt على التصنيف الذي تم التحقق من صحته للكائنات. حدد فقط العناصر المصنفة على أنها لافقاريات كبيرة لتصوير توزيع حجمها الفردي في العينة.
- تنوع الحجم
- احسب تنوع الحجم (Sd) بعد Quintana et al. (2008) 8 ، كما في García-Comas et al. (2016) 35:
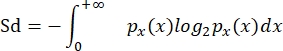
حيث p x (x) هي دالة كثافة الاحتمال للحجم x ، و x تمثل log2 (ellipVol). وبالتالي ، فإن هذا المقياس هو مؤشر شانون للتنوع الذي تم تكييفه مع مقياس مستمر ، مثل توزيع الحجم الفردي في المجتمع.
- احسب تنوع الحجم (Sd) بعد Quintana et al. (2008) 8 ، كما في García-Comas et al. (2016) 35:
- طيف حجم الحجم الحيوي الطبيعي (NBSS)
- تحديد فئات الحجم في NBSS ، وتحديد الحد الأدنى من الطيف على أنه 0.01 كمي لتوزيع حجم اللافقاريات الكبيرة في العينات وإنشاء فئات الحجم بمقياس هندسي للقاعدة 2 حتى يتم تضمين أكبر كائن حي في العينات.
ملاحظة: يزداد عرض فئة الحجم مع الحجم لحساب التباين الأكبر المرتبط بأحجام أكبر. كان لدى NBSS لمجتمعات اللافقاريات الكبيرة التي تم تحليلها هنا 14 فئة حجم (الجدول 1). - احصل على الحجم الحيوي الطبيعي بقسمة الحجم الحيوي الكلي في كل فئة حجم على عرض فئة الحجم.
- تحديد فئات الحجم في NBSS ، وتحديد الحد الأدنى من الطيف على أنه 0.01 كمي لتوزيع حجم اللافقاريات الكبيرة في العينات وإنشاء فئات الحجم بمقياس هندسي للقاعدة 2 حتى يتم تضمين أكبر كائن حي في العينات.
- حجم منحدر الطيف
- احسب المنحدر الخطي لل NBSS.
ملاحظة: يتم حساب المنحدر (μ) بناء على العلاقة بين log 2 (نقطة منتصف فئة الحجم) و log2 (الكتلة الحيوية الطبيعية) في فئات الحجم الأكبر من الوضع ، مع تجاهل أي فئات فارغة (في هذه الدراسة ، فئات الحجم من 3 إلى 14).
- احسب المنحدر الخطي لل NBSS.
| حدود فئة الحجم (مم3) | فئة الحجم نقطة المنتصف (مم3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
الجدول 1: فئات الحجم من طيف حجم الكتلة الحيوية الطبيعية (NBSS). يوضح الجدول أيضا نهايات فئة الحجم ال 15 ونقاط منتصف فئة الحجم للكائنات الحية.
النتائج
اقتناء الصور الرقمية لعينات اللافقاريات الكبيرة
الفروق الدقيقة في المسح الضوئي: ترسيب الإيثانول في درج المسح الضوئي
أثناء اختبار النظام بحثا عن اللافقاريات الكبيرة ، كانت العديد من عمليات المسح ذات جودة رديئة. منعت منطقة مشبعة داكنة في الخلفية المعالجة الطبيعية لل?...
Discussion
يسمح تكييف المنهجية التي وصفها Gorsky et al. 2010 لللافقاريات النهرية الكبيرة بدقة تصنيف عالية في تقدير هيكل حجم المجتمع في اللافقاريات الكبيرة للمياه العذبة. تشير النتائج إلى أن البروتوكول يمكن أن يقلل من الوقت اللازم لتقدير بنية الحجم الفردية في العينة إلى حوالي 1 ساعة. وهكذا، فإن البروتوكول ا?...
Disclosures
يعلن المؤلفون عدم وجود مصالح متنافسة محتملة.
Acknowledgements
تم دعم هذا العمل من قبل وزارة العلوم والابتكار والجامعات الإسبانية (رقم المنحة RTI2018-095363-B-I00). نشكر أعضاء CERM-UVic-UCC إيليا بريتكسا ، وآنا كوستاروسا ، ولايا خيمينيز ، وماريا إيزابيل غونزاليس ، ومارتا جوتجلار ، وفرانسيسك لاخ ، ونوريا سيلاريس على عملهم في أخذ العينات الميدانية لللافقاريات الكبيرة والفرز المختبري وديفيد ألبيسا على التعاون في مسح العينات. وأخيرا نشكر جوزيب ماريا جيلي ومعهد علوم مار (ICM-CSIC) على استخدام مرافق المختبر وجهاز الماسح الضوئي.
Materials
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
References
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved