Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Автоматическая обработка изображений для определения структуры размера сообщества речных макробеспозвоночных
В этой статье
Резюме
Статья основана на создании адаптированного протокола сканирования, обнаружения, сортировки и идентификации оцифрованных объектов, соответствующих бентическим речным макробеспозвоночным с использованием полуавтоматической процедуры визуализации. Эта процедура позволяет получить индивидуальные распределения размеров и показатели размера сообщества макробеспозвоночных примерно за 1 ч.
Аннотация
Размер тела является важной функциональной чертой, которая может быть использована в качестве биоиндикатора для оценки воздействия возмущений в природных сообществах. Структура размера сообщества реагирует на биотические и абиотические градиенты, включая антропогенные возмущения в таксонах и экосистемах. Однако ручное измерение мелкотелых организмов, таких как бентические макробеспозвоночные (например, >500 мкм до нескольких сантиметров в длину), занимает много времени. Чтобы ускорить оценку структуры размера сообщества, здесь мы разработали протокол полуавтоматического измерения индивидуального размера тела сохранившихся речных макробеспозвоночных, которые являются одним из наиболее часто используемых биоиндикаторов для оценки экологического состояния пресноводных экосистем. Этот протокол адаптирован из существующей методологии, разработанной для сканирования морского мезозоопланктона с помощью системы сканирования, предназначенной для проб воды. Протокол состоит из трех основных этапов: (1) сканирование подвыборок (мелких и грубых фракций размера выборки) речных макробеспозвоночных и обработка оцифрованных изображений для индивидуализации каждого обнаруженного объекта на каждом изображении; (2) создание, оценка и проверка обучающего набора с помощью искусственного интеллекта для полуавтоматического отделения отдельных изображений макробеспозвоночных от обломков и артефактов в отсканированных образцах; и 3) изображение размерной структуры сообществ макробеспозвоночных. В дополнение к протоколу эта работа включает результаты калибровки и перечисляет несколько проблем и рекомендаций по адаптации процедуры к образцам макробеспозвоночных и рассмотрению дальнейших улучшений. В целом, результаты подтверждают использование представленной сканирующей системы для автоматического измерения размеров тела речных макробеспозвоночных и свидетельствуют о том, что изображение их спектра размеров является ценным инструментом для быстрой биооценки пресноводных экосистем.
Введение
Бентические макробеспозвоночные широко используются в качестве биоиндикаторов для определения экологического состояния водных объектов1. Большинство индексов для описания сообществ макробеспозвоночных сосредоточены на таксономических показателях. Тем не менее, новые инструменты биооценки, которые интегрируют размер тела, поощряются для обеспечения альтернативной или дополнительной перспективы таксономических подходов 2,3.
Размер тела считается метатравмой, которая связана с другими жизненно важными чертами, такими как метаболизм, рост, дыхание и движение4. Кроме того, размер тела может определять трофическое положение и взаимодействия5. Связь между индивидуальным размером тела и нормализованной биомассой (или численностью) по классу размеров в сообществе определяется как спектр размеров6 и следует общей схеме линейного уменьшения нормализованной биомассы по мере увеличения индивидуального размера по логарифмической шкале7. Наклон этой линейной зависимости был тщательно изучен теоретически, и эмпирические исследования экосистем использовали его в качестве экологического индикатора структуры размера сообщества4. Другим синтетическим показателем структуры размеров сообществ, который успешно используется в исследованиях функционирования биоразнообразия и экосистем, является разнообразие размеров сообществ, которое представлено в виде индекса Шеннона классов размеров спектра размеров или его аналога, который рассчитывается на основе индивидуальных распределений размеров8.
В пресноводных экосистемах размерная структура различных групп фауны используется в качестве атаксического показателя для оценки реакции биотических сообществ на градиенты окружающей среды 9,10,11 и на антропогенные возмущения 12,13,14,15,16. Макробеспозвоночные не являются исключением, и их размерная структура также реагирует на изменения окружающей среды 17,18 и антропогенные возмущения, такие как добыча19, землепользование20 или обогащение азотом (N) и фосфором (P) 20,21,22. Тем не менее, измерение сотен людей для описания структуры размера сообщества является утомительной и трудоемкой задачей, которую часто избегают в качестве рутинного измерения в лабораториях из-за нехватки времени. Так, было разработано несколько полуавтоматических или автоматических методов визуализации для классификации и измерения образцов 23,24,25,26. Однако большинство этих методов ориентированы на таксономическую классификацию больше, чем на индивидуальные размеры организмов и не готовы к использованию для всех видов макробеспозвоночных. В экологии морского планктона система анализа сканирующих изображений широко используется для определения размера и таксономического состава сообществ зоопланктона 27,28,29,30,31. Этот инструмент можно найти в нескольких морских институтах по всему миру, и он используется для сканирования сохранившихся образцов зоопланктона для получения цифровых изображений с высоким разрешением всего образца. Настоящий протокол адаптирует использование этого инструмента для быстрой автоматической оценки спектра размеров сообщества макробеспозвоночных в реках без затрат на создание нового устройства.
Протокол состоит из сканирования образца и обработки всего изображения для автоматического получения отдельных изображений (т.е. виньеток) объектов в образце. Несколько измерений формы, размера и особенностей уровня серого характеризуют каждый объект и позволяют автоматически классифицировать объекты по категориям, которые затем проверяются экспертом. Индивидуальный размер каждого организма рассчитывается с использованием эллипсоидального биообъема (мм3), который выводится из области организма, измеренной в пикселях. Это позволяет быстро получить спектр размеров образца. Насколько нам известно, эта сканирующая система визуализации использовалась только для обработки образцов мезозупланктона, но устройство потенциально может позволить работать с пресноводными бентическими макробеспозвоночными.
Таким образом, общая цель этого исследования состоит в том, чтобы внедрить метод быстрого получения индивидуального размера сохраненных речных макробеспозвоночных путем адаптации существующего протокола, ранее использовавшегося с морским мезозоопланктоном 27,32,33. Процедура состоит из использования полуавтоматического подхода, который работает со сканирующим устройством для сканирования образцов воды и тремя открытыми программными средствами для обработки отсканированных изображений. Здесь представлен адаптированный протокол для сканирования, обнаружения и идентификации оцифрованных речных макробеспозвоночных для автоматического получения структуры размера сообщества и связанных с ним показателей размера. Оценка процедуры и руководящие принципы повышения эффективности также представлены на основе 42 отсканированных изображений образцов речных макробеспозвоночных, собранных из трех бассейнов северо-восточного (NE) Пиренейского полуострова (Ter, Segre-Ebre и Besòs).
Пробы были взяты на 100-метровых речных участках в соответствии с протоколом отбора проб в полевых условиях и лабораторного анализа бентических речных макробеспозвоночных в переходных реках от правительстваИспании 34. Образцы были собраны с помощью пробоотборника (рамка: 0,3 м х 0,3 м, сетка: 250 мкм) после обследования нескольких мест обитания. В лаборатории образцы очищали и просеивали через сетку 5 мм и 500 мкм для получения двух подвыборок: грубого подвыборки (сетка 5 мм) и тонкой подвыборки (сетка 500 мкм), которые хранились в отдельных флаконах и сохранялись в 70% этаноле. Разделение выборки на две размерные фракции позволяет лучше оценить структуру размера сообщества, поскольку крупные организмы встречаются реже и меньше, чем мелкие организмы. В противном случае отсканированный образец имеет предвзятое представление фракции большого размера.
протокол
ПРИМЕЧАНИЕ: Протокол, описанный здесь, основан на системе, разработанной Gorsky et al.27 для морского мезозоопланктона. Конкретное описание сканера (ZooSCAN), программного обеспечения для сканирования (VueScan 9x64 [9.5.09]), программного обеспечения для обработки изображений (Zooprocess, ImageJ) и программного обеспечения автоматической идентификации (Plankton Identifier) можно найти в предыдущих ссылках32,33. Чтобы наилучшим образом отрегулировать размеры бентических макробеспозвоночных по отношению к мезозоопланктону, после создания проекта по оригинальному протоколу32,33 измените параметр минимального размера (minsizeesd_mm) на 0,3 мм и параметр максимального размера (maxsizeesd_mm) на 100 мм в конфигурационном файле. Чтобы помочь следовать протоколу, это обобщено в рабочей диаграмме (рисунок 1). Созданный проект хранится в папке C компьютера и организован в следующие папки: PID_process, Zooscan_back, Zooscan_check, Zooscan_config, Zooscan_meta, Zooscan_results и Zooscan_scan. Каждая папка состоит из нескольких вложенных папок, которые различные программные приложения используют на следующих этапах протокола.
1. Получение цифровых изображений для образцов макробеспозвоночных
- Сканирование и обработка заготовки
ПРИМЕЧАНИЕ: Создавайте два пустых изображения ежедневно перед сканированием, чтобы извлечь фоновые сканы при обработке отсканированных изображений в тот же день.- Включите сканер и включите свет в двойном положении, чтобы проецировать белый свет сверху и снизу.
ПРИМЕЧАНИЕ: При сканировании образцов мезозоопланктона используется направление света вверх, но поскольку макробеспозвоночные более непрозрачны, рекомендуется переключать свет в двойное положение. - Очистите и промойте лоток для сканирования водопроводной водой.
- Налейте 110 мл водопроводной воды, хранящейся при комнатной температуре (RT), в сканирующий лоток до тех пор, пока стекло не будет закрыто. Поместите большую рамку (24,5 см х 15,8 см) на лоток для сканирования в правильном положении (с углом в верхней левой части лотка для сканирования) и заполните ее водопроводной водой до тех пор, пока ступень рамки не будет покрыта, чтобы избежать эффекта мениска, который изменил бы отсканированное изображение. Закройте крышку сканера.
ПРИМЕЧАНИЕ: Используйте воду в RT, чтобы избежать конденсации и образования пузырьков. Очистите раму без следов или капель, чтобы избежать отражения света. - Перейдите в программное обеспечение для обработки изображений, выберите рабочий проект и нажмите «Сканировать (конвертировать) фоновое изображение».
- Перейдите в программное обеспечение для сканирования и нажмите «Просмотр». Убедитесь, что вы просматриваете отсканированное изображение, убедитесь, что нет линий или пятен, и подождите не менее 30 секунд, прежде чем начинать другое сканирование. Нажмите « Сканировать» и нажмите OK в окне инструкций перед вторым сканированием, чтобы отправить данные из программного обеспечения сканирования в программное обеспечение для обработки изображений.
ПРИМЕЧАНИЕ: Выполните сканирование дважды, чтобы получить два фоновых сканирования, которые будут содержать пустоту. Этот шаг выполняется один раз в день перед началом обработки образца, и изображения сохраняются в папке Zooscan_back. - Закройте программное обеспечение сканирования после завершения сканирования.
- Включите сканер и включите свет в двойном положении, чтобы проецировать белый свет сверху и снизу.
- Пробоподготовка и сканирование
ВНИМАНИЕ: Этанол является легковоспламеняющейся жидкостью и может вызвать серьезное повреждение / раздражение глаз.- Заполните образец метаданных. Перейдите в программное обеспечение для обработки изображений и выберите Заполнить образец метаданных. Введите образец удостоверения, нажмите кнопку ОК и заполните метаданные.
ПРИМЕЧАНИЕ: Метафайл специально создан для образцов мезозооопланктона, поэтому он не соответствует методологии выборки бентических макробеспозвоночных, но все поля файла должны быть заполнены перед сканированием, иначе появится флаг ошибки. - Налейте 110 мл 70% этанола в сканирующий лоток до тех пор, пока стекло не будет закрыто, и поместите большую рамку (24,5 см х 15,8 см) с углом в верхней левой части сканирующего лотка.
ПРИМЕЧАНИЕ: Работайте с этанолом вместо воды, так как макробеспозвоночные сохраняются в этаноле. В воде они плавают и дрейфуют в скан-лотке, препятствуя получению четкого изображения и, таким образом, надежным измерениям размеров. Этанол следует хранить на РТ, чтобы избежать конденсации и образования пузырьков. - Вылейте образец макробеспозвоночных в лоток для сканирования, окаймленный рамкой, и при необходимости накройте ступень рамки большим количеством этанола.
ПРИМЕЧАНИЕ: Воздержитесь от добавления слишком большого количества этанола, чтобы избежать плавания и дрейфа организмов. - Гомогенизируйте образец по всей области кадра, разместив самых крупных особей в центре лотка для правильной обработки изображения, и потопите плавающие организмы с помощью деревянной иглы.
ПРИМЕЧАНИЕ: Если подвыборка численно содержит более 1000 особей, разделите подвыборку на две или более фракций, чтобы свести к минимуму прикосновение организмов на сканируемом изображении, и отсканируйте фракции отдельно. - Разделите трогающие организмы и организмы, касающиеся краев каркаса с помощью деревянной иглы.
ПРИМЕЧАНИЕ: Этот шаг занимает 5-20 минут. Соприкасающиеся организмы рассматриваются программным обеспечением как единый объект; таким образом, в этих случаях рассчитанные индивидуальные размеры не соответствуют фактическим единичным организмам и могут искажать оценку структуры размера сообщества. Существует возможность редактирования изображения с помощью программного обеспечения для обработки изображений для их разделения, но этот дополнительный этап включает в себя не менее 1,5 ч повторной обработки; таким образом, настоятельно рекомендуется ручное разделение. - Чтобы отсканировать образец, закройте крышку сканера, перейдите в программное обеспечение для обработки изображений, выберите рабочий проект и нажмите «Сканировать образец с помощью Zooscan (Для архива, без процесса)».
- Выберите образец и следуйте инструкциям.
- Перейдите в программное обеспечение для сканирования и нажмите «Просмотр». Убедитесь, что отсканированное изображение предварительно просмотрено, нет ли линий или пятен, и подождите не менее 30 секунд, прежде чем начинать еще одно сканирование.
- По прошествии не менее 30 секунд нажмите кнопку «Сканировать » в программном обеспечении для сканирования.
ПРИМЕЧАНИЕ: Нажмите OK в программном обеспечении для обработки изображений после нажатия кнопки Сканировать в сканирующем программном обеспечении. Не нажимайте никаких клавиш на клавиатуре компьютера и избегайте вибраций сканирования во время сканирования. В папке Zooscan_scan > _raw генерируются три файла: (i) формат файла изображения с тегами (.tif) (16 бит); (ii) стандартный текстовый документ под названием LOG (.txt), в котором записывается информация о параметрах сканирования; и iii) стандартный текстовый документ под названием МЕТА (.txt) с информацией о методах отбора проб. - Проверьте правильность необработанного сканирования.
ПРИМЕЧАНИЕ: Если сканирование имеет светлые полосы или другие видимые проблемы, рассмотрите возможность повторения сканирования, чтобы избежать проблем в следующих шагах.
- Заполните образец метаданных. Перейдите в программное обеспечение для обработки изображений и выберите Заполнить образец метаданных. Введите образец удостоверения, нажмите кнопку ОК и заполните метаданные.
- Восстановление образцов
- Снимите рамку и промойте ее над сканирующим лотком, используя бутылку для сжатия, наполненную 70% этанолом, чтобы восстановить любых прикрепленных макробеспозвоночных.
- Поднимите верхнюю часть сканера, чтобы извлечь все организмы и этанол из лотка через воронку извлечения сканирования в стакан. Когда верхняя часть сканера все еще поднята, промойте лоток с бутылкой для сжатия, чтобы промести все оставшиеся организмы.
- Пропустите образцы и этанол из стакана через сетку 500 мкм, чтобы удержать беспозвоночных в сетке, и храните их обратно во флаконе с 70% этанолом.
- После того, как все образцы будут извлечены во флаконе, очистите лоток водопроводной водой.
ПРИМЕЧАНИЕ: Промывайте лоток водопроводной водой между образцами, чтобы свести к минимуму осаждение этанола, которое изменяет обработку изображения. Промойте раму водопроводной водой, чтобы избежать потенциальных повреждений, связанных с использованием этанола. В конце дня очистите лоток водопроводной водой и аккуратно высушите его бумагой, чтобы избежать царапин.
- Обработка изображений
- Перейдите в программное обеспечение для обработки изображений и выберите КОНВЕРТИРОВАТЬ и ОБРАБАТЫВАТЬ изображения и организмы в пакетном режиме, а затем Конвертировать и обрабатывать изображения и частицы (изображение в папке RAW). Сохраните настройки по умолчанию и нажмите OK. НОРМАЛЬНЫЙ КОНЕЦ появится в конце процесса.
ПРИМЕЧАНИЕ: PID-файл и виньетки, соответствующие всем обнаруженным объектам на отсканированном изображении (в файле Joint Photographic Group [.jpg]), будут созданы в папке Zooscan_scan > _work. PID-файл — это один файл, в котором хранятся все метаданные (метафайл), технические данные, связанные с файлом журнала, и таблица с 36 измеренными переменными всех объектов, обнаруженных в изображении. Измеренные переменные соответствуют различным оценкам уровня серого, фрактального размера, формы и размера. Переменными, которые могут быть использованы для оценки размера, являются площадь и большая и второстепенная оси эллипса с равной площадью объекта (см. раздел 3 протокола). Время обработки зависит от плотности изображения и характеристик компьютера и может быть запущено между образцами во время восстановления и подготовки следующего образца. В противном случае рекомендуется запускать обработку сканируемых образцов каждый день в пакетном режиме в течение ночи и проверять правильность обработки изображений на следующее утро. - Проверьте, правильно ли вычитается фон в обработанном изображении из образца изображения с помощью программного обеспечения для обработки изображений или путем проверки изображений-масок (заканчивающихся в msk1.gif), расположенных в Zooscan_scan > _work. Если фон содержит насыщенные области или много точек, рассмотрите возможность повторения сканирования, чтобы обеспечить высокое качество изображения.
ПРИМЕЧАНИЕ: Чтобы избежать насыщенных областей на заднем плане, сканирующий лоток следует промывать водопроводной водой после каждого сканирования этанолом. Также важно (1) уменьшить количество сканируемых лиц (путем фракционирования образца и сканирования в разные складки); (2) обеспечить, чтобы крупные организмы были помещены в центр сканирующего лотка; (3) использовать чистый, отфильтрованный этанол; (4) уменьшить загрязнение образцов; (5) обеспечить достаточный объем этанола для сканирования; и (6) обеспечить, чтобы задержка между предварительным просмотром образца и сканированием составляла не менее 30 с.
- Перейдите в программное обеспечение для обработки изображений и выберите КОНВЕРТИРОВАТЬ и ОБРАБАТЫВАТЬ изображения и организмы в пакетном режиме, а затем Конвертировать и обрабатывать изображения и частицы (изображение в папке RAW). Сохраните настройки по умолчанию и нажмите OK. НОРМАЛЬНЫЙ КОНЕЦ появится в конце процесса.
- Разделение соприкасающихся организмов
ПРИМЕЧАНИЕ: При наличии нескольких виньеток с соприкасающимися организмами необходимо отделить изображения соприкасающихся организмов от других организмов и/или от волокон/мусора, чтобы обеспечить надлежащую оценку структуры размера сообщества.- Перейдите в программное обеспечение для обработки изображений, чтобы обнаружить виньетки с несколькими объектами. Выберите РАЗДЕЛЕНИЕ с помощью виньеток и нажмите OK. В окне выбора конфигурации сохраните настройки по умолчанию и нажмите кнопку ОК.
- В окне ОТДЕЛЕНИЕ от ВИНЬЕТОК сохраните параметры по умолчанию, дополнительно выберите ДОБАВИТЬ контуры для виньеток, а затем выберите образец для редактирования.
- Разделите трогательные организмы в каждой виньетке, которая всплывает, нарисовав линию мышью (нажмите кнопку рулона, чтобы нарисовать). После завершения разделения в виньетке нажмите кнопку X в правом верхнем углу окна и нажмите YES , чтобы обработать следующую. Нажмите NO , чтобы закончить и сохранить изменения. В конце процесса появится NORMAL END , если все правильно.
- После разделения повторно обработайте изображение, чтобы получить обновленные данные объекта. Перейдите в программное обеспечение для обработки изображений, нажмите «ОБРАБОТАТЬ (преобразованное) изображение (Process One)» и выберите « Обработать снова частицы из обработанных изображений» в подпапках WORK. Выберите образец и в окне Обработка одного изображения сохраните параметры по умолчанию, установите флажок Работа с маской разделения (CREATE-MODIFY-INCLUDE) и нажмите кнопку ОК. В конце процесса появится NORMAL END , если все правильно.
- В окне Управление разделением нажмите OK , чтобы сохранить изображение с контурами перед обработкой; если предыдущее изображение существует, оно будет заменено.
- При необходимости в окне Маска элемента управления разделением выберите ИЗМЕНИТЬ , чтобы добавить разделительные линии в маску с помощью мыши для разделения затрагивающих организмов, которые ранее не появлялись на этапе разделения с помощью виньеток. По завершении завершите процесс и в окне Элемент управления маской разделения выберите ДА , чтобы принять маску. В конце процесса появится NORMAL END , если все правильно.
ПРИМЕЧАНИЕ: Повторная обработка образца с помощью маски разделения занимает много времени (это может занять более 1,5 ч на образец). Желательно посвятить требуемое время на этапе 1.2.5, чтобы избежать этого дополнительного этапа.
2. Автоматическое распознавание объектов
ПРИМЕЧАНИЕ: Создайте обучающий набор для автоматического прогнозирования идентичности обнаруженных объектов, тем самым отделяя организмы от обломков в образце.
- Создание обучающего набора
- Скопируйте изображения и PID-файлы, связанные с изображениями, которые будут использоваться для создания категорий обучающего набора из Zooscan_scan > _work в PID_process > Unsorted_vignettes_pid.
ПРИМЕЧАНИЕ: Выберите подмножество образцов с высоким разнообразием таксонов и различными участками отбора проб и/или сезонами отбора проб для обеспечения максимальной репрезентативности организмов в образцах. - В папке PID_process > Learning set создайте подпапку с именем нового обучающего набора (т.е. yyyymmdd_raw_LS), а внутри нее создайте подпапки, которые будут соответствовать каждой категории обучающего набора (т.е. макробеспозвоночные, мусор, другие беспозвоночные).
ПРИМЕЧАНИЕ: Для эффективного получения структуры размера сообщества речных макробеспозвоночных рекомендуется использовать обучающий набор, основанный всего на трех категориях: макробеспозвоночные, другие беспозвоночные и мусор. Этот обучающий набор в основном отделяет виньетки объектов, соответствующих организмам, от тех, которые соответствуют мусору (например, волокна, частицы или нитевидные водоросли). - Перейдите в программное обеспечение для обработки изображений (только расширенный режим) и выберите ИЗВЛЕЧЬ виньетки для PLANKTON IDENTIFIER (несортированные виньетки для обучения). Оставьте параметры по умолчанию и установите флажок Добавить контуры .
- Перейдите в программное обеспечение автоматической идентификации, нажмите « Обучение», выберите из PID_process > Learning_set созданную вложенную папку для нового набора обучения (шаг 2.1.2) и нажмите OK.
- В левом разделе (Несортированные большие пальцы) открывшегося окна выберите папку Несортированные vignettes_pid. Выделите виньетки и перетащите их мышью из несортированных больших пальцев в папку соответствующей категории в правом разделе «Отсортированные большие пальцы», чтобы классифицировать каждый объект по определенным категориям. Перемещенные виньетки будут помечены красным крестиком.
ПРИМЕЧАНИЕ: Определите категории вручную, создав вложенные папки в папке отсортированных больших пальцев или создайте их, щелкнув значок папок в программном обеспечении. Не перемещайте более 50 виньеток одновременно. - После того, как все категории будут заполнены выбранными объектами (около 300 объектов на категорию), нажмите «Создать обучающий файл» и сохраните его с нужным именем.
ПРИМЕЧАНИЕ: Обучающий набор будет сохранен в виде файла .pid в папке PID_process > Learning set проекта. Рекомендуется создать и протестировать несколько обучающих наборов с разными уровнями категорий (от грубых до тонких форм) и с разным балансом количества объектов внутри каждой категории. Начните с грубого набора обучения с небольшим количеством категорий и не менее 50 объектами в каждой категории, а затем увеличьте количество объектов в каждой категории и / или создайте более тонкие наборы обучения. Категория должна быть репрезентативной с точки зрения ее изменчивости в наборе выборок.
- Скопируйте изображения и PID-файлы, связанные с изображениями, которые будут использоваться для создания категорий обучающего набора из Zooscan_scan > _work в PID_process > Unsorted_vignettes_pid.
- Оценка учебного набора
ПРИМЕЧАНИЕ: Выполните перекрестную проверку с двумя и пятью испытаниями с использованием метода Random Forest с программным обеспечением автоматической идентификации для получения матрицы путаницы результирующей классификации объектов.- Перейдите в программное обеспечение автоматической классификации и нажмите «Анализ данных».
- В разделе Выбор файла обучения выберите созданный файл набора обучения из PID_process > Набор обучения.
- В разделе Выбор метода выберите метод Случайного леса перекрестной проверки . В разделе Исходные переменные снимите флажки с переменных положения (X, Y, XM, YM, BX, BY и Height). В разделе Настраиваемые переменные установите флажок Только ESD.
ПРИМЕЧАНИЕ: Этот метод использует одну случайную часть обучающего набора для распознавания другой части (два раза), и это повторяется пять раз, чтобы обеспечить его статистическую надежность. - Нажмите кнопку Начать анализ и сохраните результаты как Analysis_name.txt в папке прогнозирования PID_process >. После успешного завершения анализа прекратите анализ данных.
- Перейдите в папку PID_process > Prediction и щелкните файл перекрестной проверки. Появится окно с матрицей путаницы истинной классификации (строк) по сравнению с автоматической классификацией (столбцы).
ПРИМЕЧАНИЕ: Отзыв - это процент организмов, принадлежащих к группе, которая была автоматически хорошо распознана, тогда как 1-точность - это процент организмов, классифицированных алгоритмом как группа, которая не распознается (загрязнение в группе). Отзыв должен быть выше 70%, а загрязнение (1-точность) должно быть ниже 20%. - Повторите шаги 2.1-2.5, если было создано несколько обучающих наборов и необходимо получить отзыв и 1-точность каждого из них.
ПРИМЕЧАНИЕ: Если было создано несколько обучающих наборов, выберите тот, который имеет наибольшую запоминаемость (хорошее распознавание) и точность (низкое загрязнение) интересующей группы (т.е. макробеспозвоночных), чтобы проверить автоматическое прогнозирование набора образцов на следующем этапе.
- Прогнозирование идентификации макробеспозвоночных
ПРИМЕЧАНИЕ: Используйте выбранный обучающий набор для прогнозирования идентичности всех объектов в подмножестве образцов с помощью программного обеспечения автоматической идентификации с алгоритмом случайного леса.- Перейдите в программное обеспечение автоматической идентификации и нажмите «Анализ данных».
- В разделе Выбор файла обучения выберите файл набора обучения из PID_process > Набор обучения , который должен использоваться для прогнозирования.
- В разделе Выбор образцов файлов выберите в папке PID_results образцы (PID-файлы), которые будут предсказаны.
ПРИМЕЧАНИЕ: Одновременно обрабатывайте максимум 20 файлов .pid, чтобы избежать ошибок, связанных с проблемами в памяти. Если одновременно обрабатывается слишком много файлов .pid, процесс покажет правильный конец, но может быть обработан неправильно, и на следующих шагах может возникнуть ошибка при обработке с помощью программного обеспечения для обработки изображений. - В разделе Выбор метода выберите метод Случайный лес . Установите флажок Сохранять подробные результаты для каждого образца. В разделе Исходные переменные снимите флажки с переменных положения (X, Y, XM, YM, BX, BY и Height). В разделе Настраиваемые переменные установите флажок Только ESD.
- Нажмите кнопку Начать анализ и сохраните результаты как Analysis_name.txt в папке прогнозирования PID_process >.
- Ручная проверка
ПРИМЕЧАНИЕ: Эксперт вручную проверяет прогноз с предыдущего шага, чтобы переклассифицировать неправильно классифицированные объекты в правильную категорию.- Скопируйте Analysis_sample_dat1.txt файлы для проверки из папки прогнозирования PID_process > в папку PID_process > Pid_results.
- Перейдите в программное обеспечение для обработки изображений и выберите ИЗВЛЕЧЬ виньетки в папках в соответствии с ПРОГНОЗИРОВАНИЕМ или ПРОВЕРКОЙ. Затем выберите «Использовать прогнозируемые файлы из папки «pid_results». Сохраните настройки по умолчанию и нажмите OK.
- Программа создает папку с именем sample_yyyymmdd_hhmm_to_validate с предсказанными объектами в папке PID_process > Sorted vignettes.
- Перейдите в папку PID_process > Sorted vignettes и скопируйте папку sample_yyyymmdd_ hhmm_to_validate. Замените имя папки , _to проверить, на _validated.
- Чтобы вручную проверить автоматическую классификацию, откройте папку sample_yyyymmdd_ hhmm_validated и просмотрите все виньетки из каждой вложенной папки (категории), чтобы определить, есть ли неправильно классифицированные объекты. Если один объект неправильно классифицирован, перетащите виньетку мышью в нужную папку (категорию).
- Перейдите в программное обеспечение для обработки изображений и выберите LOAD Identifications из Sorted Vignettes. Сохраните настройки по умолчанию и выберите yyyymmdd_hhmm_name_validated для обработки.
- Перейдите в PID_process > Pid_results > Dat1_validated, где были созданы файл с именем Id_from_sorted_vignettes_yyyymmdd_hhmm.txt и один .txt файл для каждого из проверенных образцов (sample_tot_1_dat1.txt).
ПРИМЕЧАНИЕ: Эти файлы .txt содержат новый столбец, представляющий прогноз, называемый pred_valid_Id_yyyymmdd_hhmm, который определяет экспертную классификацию каждого объекта (т.е. проверенную классификацию). Новые категории (например, более тонкие таксономические категории) могут быть созданы на этом этапе, во время проверки. Однако сохраните название исходной категории в новом названии (например, macroinvertebrate_chironomidae). Это позволяет проследить исходную категорию при расчете отзыва и точности и легко сгруппировать всех макробеспозвоночных для расчета параметров структуры размера сообщества (т. Е. Спектра размеров и разнообразия размеров). Текстовый файл предоставляет данные, связанные с каждым объектом, включая второстепенную и большую оси, которые используются для получения эллипсоидального объема каждого организма в качестве меры индивидуального размера тела. Кроме того, последние два столбца таблицы содержат прогнозируемые и проверенные категории каждого объекта (строки), которые позволяют рассчитать по категориям отзыв и точность обучающего набора на подмножестве образцов.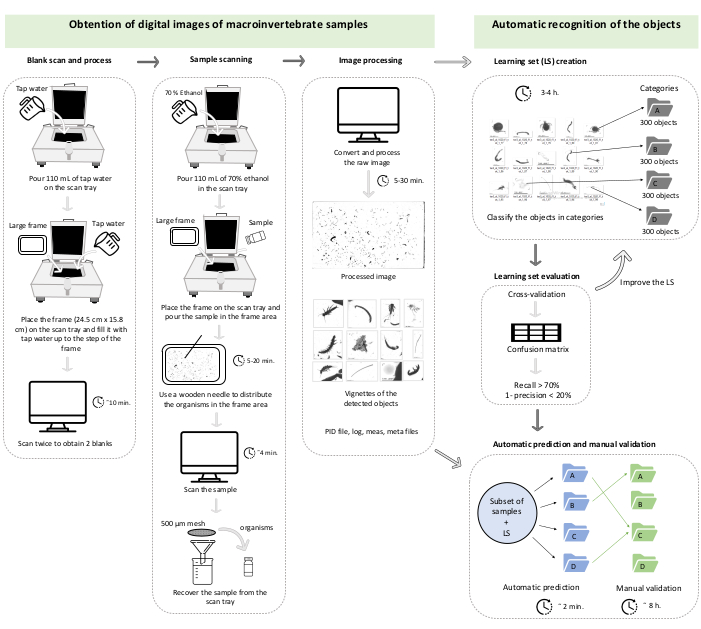
Рисунок 1: Рабочая диаграмма, представляющая раздел 1 и раздел 2 протокола. Время показательно и может меняться в зависимости от компьютера, обилия виньеток для обработки и количества категорий учебного набора. Этот случай соответствует валидации обучающего набора из трех категорий на наборе из 42 подвыборок (всего 47 473 виньетки). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
3. Вычисление индивидуального распределения размеров, спектров размеров и метрик размеров
ПРИМЕЧАНИЕ: Расчеты, упомянутые в этом разделе, были выполнены с использованием Matlab (см. скрипт как Дополнительный файл 1).
- Индивидуальное распределение размеров
- Последний столбец файла Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt содержит проверенную классификацию объектов. Выберите только объекты, классифицированные как макробеспозвоночные, чтобы отобразить их индивидуальное распределение размеров в выборке.
ПРИМЕЧАНИЕ: Индивидуальный размер тела соответствует эллипсоидному объему организмов макробеспозвоночных. Система обеспечивает измерения в пикселях. - Объедините векторы с измерениями размера из обоих сканирований, так как каждая дробь имеет различную экспоненту поддискретизации. Перед конкатенизацией исправьте фракционирование, реплицируя векторы размера столько раз, сколько была фракционирована соответствующая подвыборка.
ПРИМЕЧАНИЕ: Этот шаг необходим, если сканирование соответствует части образца (т.е. грубому или мелкому). - Вычислите эллипсоидальный объем из большой (M) и минорной (m) осей выступающих эллипсоидов с теми же областями пикселей, что и организмы. Перед вычислением эллипсоидального объема преобразуйте большую (M) и минорную (m) оси из пикселей в миллиметры (мм) со следующим коэффициентом пересчета (cf):
1 пиксель = 2 400 dpi
1 дюйм = 25,4 мм
cf = 25.4/2400
Эллипсоидальный объем (эллипВол с единицами вмм3) соответствует: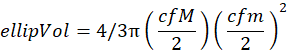
- Отображение функции плотности вероятности индивидуального распределения размеров на шкалеlog 2 .
- Последний столбец файла Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt содержит проверенную классификацию объектов. Выберите только объекты, классифицированные как макробеспозвоночные, чтобы отобразить их индивидуальное распределение размеров в выборке.
- Разнообразие размеров
- Рассчитайте разнообразие размеров (Sd) после Quintana et al. (2008)8, как в García-Comas et al. (2016)35:
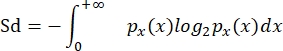
где px(x) — функция плотности вероятности размера x, а x — log2 (ellipVol). Таким образом, эта мера представляет собой индекс разнообразия Шеннона, адаптированный к непрерывной мере, такой как индивидуальное распределение размеров в сообществе.
- Рассчитайте разнообразие размеров (Sd) после Quintana et al. (2008)8, как в García-Comas et al. (2016)35:
- Нормализованный спектр размеров биообъема (NBSS)
- Определите классы размеров NBSS, установив нижнюю границу спектра как квантиль 0,01 распределения размеров макробеспозвоночных в образцах и создав классы размеров по геометрической шкале основания 2 до тех пор, пока не будет охвачен самый большой организм в образцах.
ПРИМЕЧАНИЕ: Ширина класса размеров увеличивается с размером, чтобы учесть большую изменчивость, связанную с большими размерами. NBSS сообществ макробеспозвоночных, проанализированных здесь, имели 14 классов размеров (таблица 1). - Получение нормализованного биообъема путем деления общего биообъема в каждом классе размеров на ширину класса размеров.
- Определите классы размеров NBSS, установив нижнюю границу спектра как квантиль 0,01 распределения размеров макробеспозвоночных в образцах и создав классы размеров по геометрической шкале основания 2 до тех пор, пока не будет охвачен самый большой организм в образцах.
- Наклон размерного спектра
- Рассчитайте линейный наклон NBSS.
ПРИМЕЧАНИЕ: Уклон (μ) рассчитывается на основе соотношения между log2 (средний класс размеров) и log2 (нормализованная биомасса) в классах размеров больше режима, игнорируя любые пустые (в данном исследовании классы размеров от 3 до 14).
- Рассчитайте линейный наклон NBSS.
| Ограничения по классу размеров (мм 3) | Класс размеров средней точки (мм3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
Таблица 1: Классы размеров нормализованного спектра размеров биомассы (NBSS). В таблице также показаны 15 пределов класса размеров и средние точки класса размеров организмов.
Результаты
Получение цифровых изображений образцов макробеспозвоночных
Нюансы сканирования: осаждение этанола в сканном лотке
При тестировании системы на макробеспозвоночных несколько сканирований были низкого качества. Темная насыщенная область на заднем плане препятс...
Обсуждение
Адаптация методологии, описанной Gorsky et al. 2010 для речных макробеспозвоночных, позволяет обеспечить высокую точность классификации при оценке структуры размеров сообщества у пресноводных макробеспозвоночных. Результаты показывают, что протокол может сократить время оценки индивидуал...
Раскрытие информации
Авторы заявляют об отсутствии потенциальных конкурирующих интересов.
Благодарности
Эта работа была поддержана Министерством науки, инноваций и университетов Испании (номер гранта RTI2018-095363-B-I00). Мы благодарим членов CERM-UVic-UCC Элию Бречу, Анну Костарросу, Лайю Хименес, Марию Изабель Гонсалес, Марту Ютглар, Франческа Ллаха и Нурию Селларес за их работу в области отбора проб макробеспозвоночных и лабораторной сортировки и Дэвида Альбеса за сотрудничество в сканировании образцов. Наконец, мы благодарим Хосепа Марию Гили и Институт сертификации Мара (ICM-CSIC) за использование лабораторной базы и сканера.
Материалы
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
Ссылки
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены