Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Riverine Makroomurgasızlarının Topluluk Boyut Yapısını Belirlemek için Otomatik Görüntü İşleme
Bu Makalede
Özet
Makale, yarı otomatik bir görüntüleme prosedürü kullanarak bentik nehir makroomurgasızlarına karşılık gelen sayısallaştırılmış nesneleri taramak, tespit etmek, sıralamak ve tanımlamak için uyarlanmış bir protokolün oluşturulmasına dayanmaktadır. Bu prosedür, makroomurgasız bir topluluğun bireysel boyut dağılımlarının ve boyut metriklerinin yaklaşık 1 saat içinde elde edilmesini sağlar.
Özet
Vücut büyüklüğü, doğal topluluklardaki pertürbasyonların etkilerini değerlendirmek için biyoendikatör olarak kullanılabilecek önemli bir fonksiyonel özelliktir. Topluluk büyüklüğü yapısı, taksonlar ve ekosistemler arasındaki antropojenik pertürbasyonlar da dahil olmak üzere biyotik ve abiyotik gradyanlara yanıt verir. Bununla birlikte, bentik makroomurgasızlar gibi küçük gövdeli organizmaların (örneğin, >500 μm ila birkaç santimetre uzunluğunda) manuel ölçümü zaman alıcıdır. Topluluk büyüklüğü yapısının tahminini hızlandırmak için, burada, tatlı su ekosistemlerinin ekolojik durumunu değerlendirmek için en yaygın kullanılan biyogöstergelerden biri olan korunmuş nehir makroomurgasızlarının bireysel vücut büyüklüğünü yarı otomatik olarak ölçmek için bir protokol geliştirdik. Bu protokol, deniz mezozooplanktonlarını su örnekleri için tasarlanmış bir tarama sistemi ile taramak için geliştirilen mevcut bir metodolojiden uyarlanmıştır. Protokol üç ana adımdan oluşur: (1) nehir makroomurgasızlarının alt örneklerinin (ince ve kaba örneklem boyutu kesirleri) taranması ve her görüntüde tespit edilen her nesneyi kişiselleştirmek için sayısallaştırılmış görüntülerin işlenmesi; (2) taranan örneklerdeki makroomurgasızların bireysel görüntülerini detritus ve eserlerden yarı otomatik olarak ayırmak için yapay zeka aracılığıyla bir öğrenme seti oluşturmak, değerlendirmek ve doğrulamak; ve (3) makroomurgasız topluluklarının boyut yapısını tasvir etmek. Protokole ek olarak, bu çalışma kalibrasyon sonuçlarını içerir ve prosedürü makroomurgasız numunelere uyarlamak ve daha fazla iyileştirme için dikkate almak için çeşitli zorlukları ve önerileri sıralar. Genel olarak, sonuçlar, nehir makroomurgasızlarının otomatik vücut büyüklüğü ölçümü için sunulan tarama sisteminin kullanımını desteklemektedir ve boyut spektrumlarının tasvirinin, tatlı su ekosistemlerinin hızlı biyo-değerlendirmesi için değerli bir araç olduğunu göstermektedir.
Giriş
Bentik makroomurgasızlar, su kütlelerinin ekolojik durumunu belirlemek için biyogöstergeler olarak yaygın olarak kullanılmaktadır1. Makroomurgasız toplulukları tanımlayan endekslerin çoğu taksonomik metriklere odaklanır. Bununla birlikte, vücut büyüklüğünü bütünleştiren yeni biyodeğerlendirme araçları, taksonomik yaklaşımlara alternatif veya tamamlayıcı bir bakış açısı sağlamak için teşvik edilmektedir 2,3.
Vücut büyüklüğü, metabolizma, büyüme, solunum ve hareket4 gibi diğer hayati özelliklerle ilişkili bir metaözellik olarak kabul edilir. Ayrıca, vücut büyüklüğü trofik pozisyonu ve etkileşimleri belirleyebilir5. Bireysel vücut büyüklüğü ile bir topluluktaki boyut sınıfına göre normalleştirilmiş biyokütle (veya bolluk) arasındaki ilişki, boyut spektrumu6 olarak tanımlanır ve logaritmik bir ölçek7'de bireysel boyut arttıkça normalleştirilmiş biyokütlede doğrusal bir azalmanın genel modelini izler. Bu doğrusal ilişkinin eğimi teorik olarak kapsamlı bir şekilde incelenmiştir ve ekosistemler arasındaki ampirik çalışmalar bunu topluluk büyüklüğü yapısının ekolojik bir göstergesi olarak kullanmıştır4. Biyoçeşitlilik-ekosistem işleyişi çalışmalarında başarıyla kullanılan topluluk büyüklüğü yapısının bir diğer sentetik göstergesi, boyut spektrumunun boyut sınıflarının Shannon indeksi veya analogu olarak temsil edilen ve bireysel boyut dağılımlarına göre hesaplanan topluluk büyüklüğü çeşitliliğidir8.
Tatlı su ekosistemlerinde, farklı faunal grupların boyut yapısı, biyotik toplulukların çevresel gradyanlara 9,10,11 ve antropojenik pertürbasyonlara 12,13,14,15,16 tepkisini değerlendirmek için ataksik bir gösterge olarak kullanılır. Makroomurgasızlar bir istisna değildir ve boyut yapıları aynı zamanda çevresel değişikliklere 17,18 ve madencilik 19, arazi kullanımı 20 veya azot (N) ve fosfor (P) zenginleştirme20,21,22 gibi antropojenik bozulmalara da yanıt verir. Bununla birlikte, topluluk büyüklüğü yapısını tanımlamak için yüzlerce kişiyi ölçmek, zaman yetersizliği nedeniyle laboratuvarlarda rutin bir ölçüm olarak kaçınılan sıkıcı ve zaman alıcı bir iştir. Bu nedenle, örnekleri sınıflandırmak ve ölçmek için çeşitli yarı otomatik veya otomatik görüntüleme yöntemleri geliştirilmiştir 23,24,25,26. Bununla birlikte, bu yöntemlerin çoğu, organizmaların bireysel büyüklüğünden daha fazla taksonomik sınıflandırmaya odaklanmıştır ve her türlü makroomurgasız için kullanıma hazır değildir. Deniz plankton ekolojisinde, zooplankton topluluklarının büyüklüğünü ve taksonomik bileşimini belirlemek için bir tarama görüntü analiz sistemi yaygın olarak kullanılmıştır 27,28,29,30,31. Bu cihaz dünya çapında çeşitli deniz enstitülerinde bulunabilir ve tüm numunenin yüksek çözünürlüklü dijital görüntülerini elde etmek için korunmuş zooplankton örneklerini taramak için kullanılır. Mevcut protokol, nehirlerdeki makroomurgasız topluluk büyüklüğü spektrumunu yeni bir cihaz oluşturmaya yatırım yapmadan hızlı bir şekilde otomatik bir şekilde tahmin etmek için bu aracın kullanımını uyarlamaktadır.
Protokol, bir numunenin taranmasından ve numunedeki nesnelerin tek bir görüntüsünü (yani vinyetleri) otomatik olarak elde etmek için tüm görüntünün işlenmesinden oluşur. Şekil, boyut ve gri seviyeli özelliklerin çeşitli ölçümleri her nesneyi karakterize eder ve nesnelerin kategorilere otomatik olarak sınıflandırılmasına izin verir ve bunlar daha sonra bir uzman tarafından doğrulanır. Her organizmanın bireysel boyutu, piksel cinsinden ölçülen organizmanın alanından türetilen elipsoidal biyohacim (mm3) kullanılarak hesaplanır. Bu, numunenin boyut spektrumunun hızlı bir şekilde elde edilmesini sağlar. Bildiğimiz kadarıyla, bu tarama görüntüleme sistemi sadece mezozooplankton örneklerini işlemek için kullanılmıştır, ancak cihaz potansiyel olarak tatlı su bentik makroomurgasızlarla çalışmaya izin verebilir.
Bu nedenle bu çalışmanın genel amacı, daha önce deniz mezozooplankton 27,32,33 ile kullanılan mevcut bir protokolü uyarlayarak korunmuş nehir makroomurgasızlarının bireysel boyutlarını hızlı bir şekilde elde etmek için bir yöntem sunmaktır. Prosedür, su örneklerini taramak için bir tarama cihazı ve taranan görüntüleri işlemek için üç açık yazılım ile çalışan yarı otomatik bir yaklaşım kullanmaktan oluşur. Topluluk büyüklüğü yapısını ve ilgili boyut metriklerini otomatik olarak elde etmek için sayısallaştırılmış nehir makroomurgasızlarını taramak, tespit etmek ve tanımlamak için uyarlanmış bir protokol burada sunulmuştur. Prosedürün değerlendirilmesi ve verimliliği artırmak için kılavuzlar, Kuzeydoğu (NE) İber Yarımadası'ndaki (Ter, Segre-Ebre ve Besòs) üç havzadan toplanan nehir makroomurgasız örneklerinin taranmış 42 görüntüsüne dayanarak da sunulmaktadır.
Örnekler, İspanyol Hükümeti34'ten sürülebilir nehirlerdeki bentik nehir makroomurgasızlarının saha örneklemesi ve laboratuvar analizi protokolünü takiben 100 m nehir uzanımlarında toplandı. Örnekler, çok habitatlı bir anketi takiben bir sörber örnekleyici (çerçeve: 0.3 m x 0.3 m, ağ: 250 μm) ile toplandı. Laboratuvarda, numuneler iki alt numune elde etmek için 5 mm'lik ve 500 μm'lik bir ağ üzerinden temizlendi ve elendi: kaba bir alt numune (5 mm ağ) ve ayrı şişelerde saklanan ve% 70 etanolde saklanan ince bir alt numune (500 μm ağ). Numuneyi iki boyut fraksiyonuna ayırmak, topluluk büyüklüğü yapısının daha iyi tahmin edilmesini sağlar, çünkü büyük organizmalar küçük organizmalardan daha nadir ve daha azdır. Aksi takdirde, taranan numune büyük boyutlu fraksiyonun önyargılı bir temsiline sahiptir.
Protokol
NOT: Burada açıklanan protokol, Gorsky ve ark.27 tarafından deniz mezozooplanktonları için geliştirilen sisteme dayanmaktadır. Tarayıcı (ZooSCAN), tarama yazılımı (VueScan 9x64 [9.5.09]), görüntü işleme yazılımı (Zooprocess, ImageJ) ve otomatik tanımlama yazılımı (Plankton Identifier) adımlarının belirli bir açıklaması önceki referanslarda bulunabilir32,33. Bentik makroomurgasızların boyutlarını mezozooplanktona göre en iyi şekilde ayarlamak için, proje orijinal protokol32,33'ü izleyerek oluşturulduktan sonra, yapılandırma dosyasında minimum boyut parametresini (minsizeesd_mm) 0,3 mm ve maksimum boyut parametresini (maxsizeesd_mm) 100 mm olarak değiştirin. Protokolün izlenmesine yardımcı olmak için, bu bir çalışma çizelgesinde özetlenmiştir (Şekil 1). Oluşturulan proje bilgisayarın C klasöründe depolanır ve aşağıdaki klasörlerde düzenlenir: PID_process, Zooscan_back, Zooscan_check, Zooscan_config, Zooscan_meta, Zooscan_results ve Zooscan_scan. Her klasör, farklı yazılım uygulamalarının protokolün aşağıdaki adımlarında kullandığı birkaç alt klasörden oluşur.
1. Makroomurgasız örnekleri için dijital görüntülerin elde edilmesi
- Boşluğun taranması ve işlenmesi
NOT: Taranan görüntüleri aynı gün içinde işlerken arka plan taramalarını ayıklamak için taramadan önce günde iki boş görüntü oluşturun.- Tarayıcıyı açın ve üstten ve alttan beyaz ışık yansıtmak için ışığı çift konumda açın.
NOT: Mezozooplankton örneklerini tararken, yukarı doğru ışık yönü kullanılır, ancak makroomurgasızlar daha opak olduğundan, ışığın çift konuma getirilmesi önerilir. - Tarama tepsisini temizleyin ve musluk suyuyla durulayın.
- Oda sıcaklığında (RT) depolanan 110 mL musluk suyunu, cam kaplanana kadar tarama tepsisine dökün. Büyük çerçeveyi (24,5 cm x 15,8 cm) tarama tepsisine doğru konuma (köşesi tarama tepsisinin sol üst kısmında olacak şekilde) yerleştirin ve taranan görüntüyü değiştirecek bir menisküs etkisinden kaçınmak için çerçevenin basamağı kaplanana kadar musluk suyuyla doldurun. Tarayıcı kapağını kapatın.
NOT: Yoğuşma ve kabarcık oluşumunu önlemek için RT'de su kullanın. Işık yansımasını önlemek için çerçeveyi iz veya damlacık olmadan temizleyin. - Görüntü işleme yazılımına gidin, çalışan projeyi seçin ve Arka Plan Görüntüsünü Tara (Dönüştür) seçeneğine tıklayın.
- Tarama yazılımına gidin ve Önizleme'ye tıklayın. Taranan görüntüyü önizlediğinizden emin olun, çizgi veya nokta olmadığından emin olun ve başka bir taramaya başlamadan önce en az 30 saniye bekleyin. Verileri tarama yazılımından görüntü işleme yazılımına göndermek için ikinci taramadan önce Tara'ya tıklayın ve talimatlar penceresinde Tamam'a basın.
NOT: Boşluğu oluşturacak iki arka plan taramasını elde etmek için iki kez tarayın. Bu adım, örnek işlemeye başlamadan önce günde bir kez yapılır ve görüntüler Zooscan_back klasöründe saklanır. - Taramayı bitirdikten sonra tarama yazılımını kapatın.
- Tarayıcıyı açın ve üstten ve alttan beyaz ışık yansıtmak için ışığı çift konumda açın.
- Numune hazırlama ve tarama
DİKKAT: Etanol yanıcı bir sıvıdır ve ciddi göz hasarına / tahrişine neden olabilir.- Örnek meta verileri doldurun. Görüntü işleme yazılımına gidin ve Örnek Meta Verileri Doldur'u seçin. Örnek kimliği girin, Tamam'a tıklayın ve meta verileri doldurun.
NOT: Meta dosya özellikle mezozooplankton örnekleri için oluşturulmuştur, bu nedenle bentik makroomurgasız örnekleme metodolojisine uymaz, ancak taramadan önce dosyanın tüm alanlarının doldurulması gerekir, aksi takdirde bir hata bayrağı açılır. - Cam kaplanana kadar tarama tepsisine 110 mL% 70 etanol dökün ve köşesi tarama tepsisinin sol üst kısmına gelecek şekilde büyük çerçeveyi (24,5 cm x 15,8 cm) yerleştirin.
NOT: Makroomurgasızlar etanolde korunduğundan, su yerine etanol ile çalışın. Suda, tarama tepsisinde yüzer ve sürüklenirler, böylece keskin bir görüntü ve dolayısıyla güvenilir boyut ölçümleri önlerler. Yoğuşma ve kabarcık oluşumunu önlemek için RT'de etanol korunmalıdır. - Makroomurgasızlar örneğini çerçevenin kenarındaki tarama tepsisine dökün ve gerekirse çerçeve adımını daha fazla etanol ile örtün.
NOT: Organizmaların yüzmesini ve sürüklenmesini önlemek için çok fazla etanol eklemekten kaçının. - Numuneyi çerçeve alanı boyunca homojenize edin, en büyük bireyleri uygun görüntü işleme için tepsinin ortasına yerleştirin ve yüzen organizmaları ahşap bir iğne kullanarak batırın.
NOT: Bir alt örnek sayısal olarak 1.000'den fazla birey içeriyorsa, taranan görüntüdeki dokunan organizmaları en aza indirmek için alt numuneyi iki veya daha fazla fraksiyona bölün ve fraksiyonları ayrı ayrı tarayın. - Dokunan organizmaları ve çerçeve kenarlarına dokunan organizmaları ahşap iğneyi kullanarak ayırın.
NOT: Bu adım 5-20 dakika sürer. Dokunan organizmalar yazılım tarafından tek bir nesne olarak kabul edilir; Bu nedenle, bu durumlarda, hesaplanan bireysel boyutlar gerçek tek organizmalara karşılık gelmez ve topluluk büyüklüğü yapısının tahminini önyargılı hale getirebilir. Görüntüyü ayırmak için görüntü işleme yazılımıyla düzenleme olasılığı vardır, ancak bu ek adım en az 1,5 saat yeniden işlemeyi içerir; Bu nedenle, manuel ayırma şiddetle tavsiye edilir. - Örneği taramak için tarayıcı kapağını kapatın, görüntü işleme yazılımına gidin, çalışan projeyi seçin ve Zooscan ile SCAN Sample'a tıklayın (Arşiv İçin, İşlem Yok).
- Örneği seçin ve yönergeleri izleyin.
- Tarama yazılımına gidin ve Önizleme'ye tıklayın. Taranan görüntüyü önizlediğinizden emin olun, çizgi veya nokta olmadığından emin olun ve başka bir taramaya başlamadan önce en az 30 saniye bekleyin.
- En az 30 saniye sonra, tarama yazılımındaki Tara düğmesine tıklayın.
NOT: Tarama yazılımında Tara düğmesine bastıktan sonra görüntü işleme yazılımında OK düğmesine basın. Bilgisayar klavyesinde herhangi bir tuşa basmayın ve tarama sırasında taramanın titreşimlerinden kaçının. Zooscan_scan > _raw klasöründe üç dosya oluşturulur: (i) etiketli bir görüntü dosyası formatı (.tif) (16 bit); (ii) tarama parametreleri hakkında bilgi kaydeden LOG (.txt) adlı standart bir metin belgesi; ve (iii) örnekleme yöntemleri hakkında bilgi içeren META (.txt) adlı standart bir metin belgesi. - Ham taramanın doğru olduğundan emin olun.
NOT: Taramada açık çizgiler veya başka görünür sorunlar varsa, aşağıdaki adımlarda sorun yaşamamak için taramayı tekrarlamayı düşünün.
- Örnek meta verileri doldurun. Görüntü işleme yazılımına gidin ve Örnek Meta Verileri Doldur'u seçin. Örnek kimliği girin, Tamam'a tıklayın ve meta verileri doldurun.
- Örnek kurtarma
- Çerçeveyi çıkarın ve bağlı makroomurgasızları kurtarmak için% 70 etanol ile doldurulmuş bir sıkma şişesi kullanarak tarama tepsisinin üzerinde durulayın.
- Tüm organizmaları ve etanolleri tepsiden tarama alma hunisinden bir beherin içine almak için tarayıcının üst kısmını kaldırın. Tarayıcının üst kısmı hala kaldırılıyken, kalan organizmaları süpürmek için tepsiyi sıkma şişesiyle durulayın.
- Omurgasızları ağda tutmak için örnekleri ve etanolleri beherden 500 μm'lik bir ağdan geçirin ve% 70 etanol içeren bir şişede saklayın.
- Tüm örnekler şişede geri kazanıldıktan sonra, tepsiyi musluk suyuyla temizleyin.
NOT: Görüntü işlemeyi değiştiren etanol çökelmesini en aza indirmek için tepsiyi numuneler arasında musluk suyuyla yıkayın. Etanol kullanımıyla ilgili olası hasarları önlemek için çerçeveyi musluk suyuyla durulayın. Günün sonunda, musluk suyu kullanarak tepsiyi temizleyin ve çizilmeleri önlemek için kağıtla hafifçe kurulayın.
- Görüntü işleme
- Görüntü işleme yazılımına gidin ve Toplu Modda Görüntüleri ve Organizmaları DÖNÜŞTÜR ve İŞLEME ET'i seçin ve ardından Görüntü VE Parçacıkları Dönüştür ve İşle (RAW Klasöründeki Görüntü) öğesini seçin. Varsayılan ayarları koruyun ve Tamam'a tıklayın. NORMAL END işlemin sonunda görünecektir.
NOT: Bir PID dosyası ve taranan görüntüdeki algılanan tüm nesnelere karşılık gelen vinyetler (bir Birleşik Fotoğraf Grubu dosyasında [.jpg]) Zooscan_scan > _work klasöründe oluşturulur. PID dosyası, tüm meta verileri (meta dosya), günlük dosyasıyla ilişkili teknik verileri ve görüntüde algılanan tüm nesnelerin 36 ölçülen değişkenini içeren bir tabloyu depolayan tek bir dosyadır. Ölçülen değişkenler gri seviye, fraktal boyut, şekil ve boyut gibi farklı tahminlere karşılık gelir. Boyut tahmini için kullanılabilecek değişkenler, nesneye eşit alana sahip bir elipsin alanı ve majör ve minör eksenleridir (protokolün 3. bölümüne bakınız). İşlem süresi, görüntü yoğunluğuna ve bilgisayar özelliklerine bağlıdır ve bir sonraki numuneyi kurtarırken ve hazırlarken numuneler arasında başlatılabilir. Aksi takdirde, her gün taranan numunelerin gece boyunca toplu modda işlenmesinin başlatılması ve ertesi sabah uygun görüntü işlemenin kontrol edilmesi önerilir. - Görüntü işleme yazılımını kullanarak veya .gif bulunan maske görüntülerini (msk1 Zooscan_scan > _work sonlandırılmış) denetleyerek işlenen görüntüdeki arka planın örnek görüntüden uygun şekilde çıkarılıp çıkarılmadığını kontrol edin. Arka planda doygun alanlar veya çok sayıda nokta varsa, yüksek kaliteli görüntüler elde etmek için taramayı tekrarlamayı düşünün.
NOT: Arka planda doymuş alanları önlemek için, tarama tepsisi etanol ile yapılan her taramadan sonra musluk suyuyla durulanmalıdır. (1) taranan bireylerin sayısını azaltmak (numuneyi parçalayarak ve farklı kıvrımlarda tarayarak); (2) büyük organizmaların tarama tepsisinin ortasına yerleştirildiğinden emin olmak; (3) temiz, filtrelenmiş etanol kullanın; (4) numunelerdeki kirliliği azaltmak; (5) tarama için etanol hacminin yeterli olduğundan emin olmak; ve (6) numunenin önizlemesi ile tarama arasındaki gecikmenin en az 30 s olmasını sağlamak.
- Görüntü işleme yazılımına gidin ve Toplu Modda Görüntüleri ve Organizmaları DÖNÜŞTÜR ve İŞLEME ET'i seçin ve ardından Görüntü VE Parçacıkları Dönüştür ve İşle (RAW Klasöründeki Görüntü) öğesini seçin. Varsayılan ayarları koruyun ve Tamam'a tıklayın. NORMAL END işlemin sonunda görünecektir.
- Dokunan organizmaların ayrılması
NOT: Dokunan organizmalara sahip birkaç vinyet olduğunda, topluluk büyüklüğü yapısının doğru bir şekilde tahmin edilmesini sağlamak için dokunan organizmaların görüntülerini diğer organizmalardan ve / veya liflerden / döküntülerden ayırmak gerekir.- Birden fazla nesneye sahip vinyetleri algılamak için görüntü işleme yazılımına gidin. Vinyet Kullanarak Ayırma (SEPARATION Using Vinnettes ) seçeneğini belirleyin ve OK (Tamam) düğmesine basın. Yapılandırma seçimi penceresinde, varsayılan ayarları koruyun ve Tamam'a tıklayın.
- VİNYETLERDEN AYIRMA penceresinde, varsayılan ayarları koruyun, ayrıca Vinyetlere Anahatlar EKLE'yi seçin ve ardından düzenlenecek örneği seçin.
- Açılan her vinyetteki dokunan organizmaları fare ile bir çizgi çizerek ayırın (çizmek için rulo düğmesine basın). Vinyetteki ayırma işlemi tamamlandıktan sonra, pencerenin sağ üst köşesindeki X düğmesine tıklayın ve bir sonrakini işlemek için EVET düğmesine basın. Değişiklikleri sonlandırmak ve kaydetmek için HAYIR'a basın. İşlemin sonunda, her şey doğruysa NORMAL END görünecektir.
- Ayırmadan sonra, güncelleştirilmiş nesne verilerini elde etmek için görüntüyü yeniden işleyin. Görüntü işleme yazılımına gidin, PROCESS (Converted) Image (Process One) üzerine tıklayın ve WORK Alt Klasörlerindeki İşlenmiş Görüntülerden Parçacıkları Yeniden İşle'yi seçin. Örneği seçin ve Tek Görüntü İşlemi penceresinde varsayılan ayarları koruyun, Ayırma Maskesiyle Çalış (CREATE-MODIFY-INCLUDE) seçeneğini işaretleyin ve ardından Tamam'a tıklayın. İşlemin sonunda, her şey doğruysa NORMAL END görünecektir.
- Ayırma Denetimi penceresinde, işlemeden önce görüntüyü konturlarla kaydetmek için Tamam'a basın; önceki bir görüntü varsa, değiştirilir.
- Ayırma Denetim Maskesi penceresinde, gerekirse, vinyet kullanarak ayırma adımında daha önce görünmeyen dokunaklı organizmaları ayırmak üzere fareyi kullanarak maskeye ayırma çizgileri eklemek için DÜZENLE'yi seçin. İşiniz bittiğinde işlemi sonlandırın ve Ayırma Maskesi Denetimi penceresinde maskeyi kabul etmek için Evet'i seçin. İşlemin sonunda, her şey doğruysa NORMAL END görünecektir.
NOT: Bir numunenin ayırma maskesiyle yeniden işlenmesi zaman alıcıdır (bu, numune başına 1,5 saatten fazla sürebilir). Bu ek adımdan kaçınmak için adım 1.2.5'te gerekli zamanın ayrılması tercih edilir.
2. Nesnelerin otomatik olarak tanınması
NOT: Algılanan nesnelerin kimliğini otomatik olarak tahmin etmek için bir öğrenme kümesi oluşturun, böylece organizmaları numunedeki enkazdan ayırın.
- Öğrenme seti oluşturma
- Öğrenme kümesinin kategorilerini oluşturmak için kullanılacak görüntüleri ve görüntülerle ilişkili .pid dosyalarını Zooscan_scan > _work PID_process > Unsorted_vignettes_pid kopyalayın.
NOT: Numunelerdeki organizmaların maksimum temsilini sağlamak için yüksek takson çeşitliliğine ve farklı numune alma alanlarına ve/veya numune alma mevsimlerine sahip numunelerin bir alt kümesini seçin. - PID_process > Öğrenme kümesi klasöründe, yeni öğrenme kümesinin adını taşıyan bir alt klasör oluşturun (yani, yyyymmdd_raw_LS) ve içinde, öğrenme kümesinin her kategorisine karşılık gelecek alt klasörleri oluşturun (örneğin, makroomurgasızlar, enkaz, diğer omurgasızlar).
NOT: Nehir makroomurgasız örneklerinin topluluk büyüklüğü yapısını verimli bir şekilde elde etmek için, yalnızca üç kategoriye dayanan bir öğrenme seti kullanılması önerilir: makroomurgasızlar, diğer omurgasızlar ve enkaz. Bu öğrenme seti temel olarak organizmalara karşılık gelen nesnelerin vinyetlerini enkazlara karşılık gelenlerden (örneğin, lifler, parçacıklar veya filamentli algler) ayırır. - Görüntü işleme yazılımına gidin (yalnızca Gelişmiş mod) ve PLANKTON IDENTIFIER için EXTRACT Vinyetleri (eğitim için sıralanmamış vinyetler) öğesini seçin. Varsayılan seçenekleri koruyun ve Anahat Ekle kutusunu işaretleyin.
- Otomatik tanımlama yazılımına gidin, Öğrenme'ye tıklayın, yeni öğrenme kümesi için oluşturulan alt klasörü PID_process > Learning_set seçin (adım 2.1.2) ve Tamam'a basın.
- Açık pencerenin sol bölümünde (Sıralanmamış Başparmaklar), vignettes_pid Sıralanmamış klasörünü seçin. Vinyetleri seçin ve her nesneyi tanımlanmış kategorilere ayırmak için fareyle sıralanmamış başparmaklardan sağ bölümdeki ilgili kategorinin klasörüne, Sıralanmış Başparmaklar'a sürükleyin. Taşınan vinyetler kırmızı X ile işaretlenir.
NOT: Sıralanmış başparmak klasöründe alt klasörler oluşturarak kategorileri manuel olarak tanımlayın veya yazılımdaki klasörler simgesine tıklayarak oluşturun. Aynı anda 50'den fazla vinyet taşımayın. - Tüm kategoriler seçilen nesnelerle tamamlandıktan sonra (kategori başına yaklaşık 300 nesne), Öğrenme Dosyası Oluştur'a tıklayın ve istediğiniz adla kaydedin.
NOT: Öğrenme kümesi, projenin PID_process > Öğrenme kümesi klasörüne bir .pid dosyası olarak kaydedilir. Farklı kategori seviyelerine (kabadan ince formlara) ve her kategorideki nesne sayısının farklı bir dengesine sahip birkaç öğrenme seti oluşturulması ve test edilmesi önerilir. Düşük sayıda kategori ve kategori başına en az 50 nesne içeren kaba bir öğrenme seti ile başlayın ve ardından her kategorideki nesne sayısını artırın ve / veya daha ince öğrenme kümeleri oluşturun. Bir kategori, numune kümesindeki değişkenliğini temsil etmelidir.
- Öğrenme kümesinin kategorilerini oluşturmak için kullanılacak görüntüleri ve görüntülerle ilişkili .pid dosyalarını Zooscan_scan > _work PID_process > Unsorted_vignettes_pid kopyalayın.
- Öğrenme setinin değerlendirilmesi
NOT: Nesnelerin sonuçta sınıflandırılmasının bir karışıklık matrisini elde etmek için otomatik tanımlama yazılımıyla Rastgele Orman yöntemini kullanarak iki kat ve beş deneme ile çapraz doğrulama gerçekleştirin.- Otomatik sınıflandırma yazılımına gidin ve Veri Analizi'ne tıklayın.
- Öğrenme dosyası seç'te, PID_process > Öğrenme kümesinden oluşturulan öğrenme kümesi dosyasını seçin.
- Bir yöntem seçin alanında, Çapraz Doğrulama Rasgele Orman yöntemini seçin. Orijinal Değişkenler'de, konum değişkenlerinin (X, Y, XM, YM, BX, BY ve Height) işaretini kaldırın. Özelleştirilmiş Değişkenler'de, yalnızca ESD'yi işaretleyin.
NOT: Bu yöntem, diğer kısmı tanımak için öğrenme kümesinin rastgele bir bölümünü kullanır (iki kat) ve bu, istatistiksel olarak sağlam olduğundan emin olmak için beş kez tekrarlanır. - Analizi Başlat'a tıklayın ve sonuçları Analysis_name.txt Tahmin klasörüne PID_process > olarak kaydedin. Analiz başarıyla tamamlandığında, veri analizinden çıkın.
- PID_process > Tahmin klasörüne gidin ve çapraz doğrulama dosyasına tıklayın. Gerçek sınıflandırmanın (satırlar) karışıklık matrisi ile otomatik sınıflandırmanın (sütunlar) karışıklık matrisini içeren bir pencere açılacaktır.
NOT: Geri çağırma, otomatik olarak iyi tanınan bir gruba ait organizmaların yüzdesidir, oysa 1-hassasiyet, algoritma tarafından tanınmayan bir grup olarak sınıflandırılan organizmaların yüzdesidir (bir grupta kontaminasyon). Geri çağırma %70'in üzerinde olmalı ve kontaminasyon (1 hassasiyetli) %20'den düşük olmalıdır. - Birkaç öğrenme kümesi oluşturulduysa ve her birinin geri çağırma ve 1 duyarlılığının elde edilmesi gerekiyorsa, 2.1-2.5 arasındaki adımları tekrarlayın.
NOT: Birkaç öğrenme seti oluşturulduysa, bir sonraki adımda bir numune kümesinin otomatik tahminini test etmek için ilgili grubun (yani makroomurgasızlar) en yüksek hatırlama (iyi tanıma) ve hassasiyetine (düşük kontaminasyon) sahip olanı seçin.
- Makroomurgasızların tanımlanmasının tahmini
NOT: Rastgele orman algoritmasına sahip otomatik tanımlama yazılımını kullanarak örneklerin bir alt kümesindeki tüm nesnelerin kimliğini tahmin etmek için seçilen öğrenme kümesini kullanın.- Otomatik tanımlama yazılımına gidin ve Veri Analizi'ne tıklayın.
- Öğrenme dosyası seç'te, tahmin için kullanılması gereken PID_process > Öğrenme kümesinden öğrenme kümesi dosyasını seçin.
- Örnek Dosyaları Seç bölümünde, PID_results klasörden tahmin edilecek örnekleri (PID dosyaları) seçin.
NOT: Bellek sorunlarıyla ilgili hataları önlemek için aynı anda en fazla 20 .pid dosyasını işleyin. Aynı anda çok fazla .pid dosyası işlenirse, işlem doğru bir son gösterir, ancak iyi işlenmeyebilir ve görüntü işleme yazılımıyla işlenirken sonraki adımlarda bir hata oluşabilir. - Bir Yöntem Seç'te, Rastgele Orman yöntemini seçin. Her örnek için Ayrıntılı Sonuçları Kaydet'i işaretleyin. Orijinal Değişkenler'de, konum değişkenlerinin (X, Y, XM, YM, BX, BY ve Height) işaretini kaldırın. Özelleştirilmiş Değişkenler'de yalnızca ESD'yi işaretleyin.
- Analizi Başlat'a tıklayın ve sonuçları Analysis_name.txt Tahmin klasörüne PID_process > olarak kaydedin.
- El ile doğrulama
NOT: Bir uzman, yanlış sınıflandırılmış nesneleri doğru kategoriye yeniden sınıflandırmak için önceki adımdaki tahmini el ile doğrular.- Doğrulanacak Analysis_sample_dat1.txt dosyaları PID_process > Tahmini klasöründen PID_process > Pid_results klasörüne kopyalayın.
- Görüntü işleme yazılımına gidin ve PREDICTION veya VALIDATION'a Göre Klasörlerdeki Vinyetleri EXTRACT öğesini seçin. Ardından, " pid_results" Klasöründen PREDICTED Files Kullan'ı seçin. Varsayılan ayarları koruyun ve Tamam düğmesine basın.
- Yazılım , sample_yyyymmdd_hhmm_to_validate Sıralanmış vinyetler klasöründe tahmin edilen nesnelerle PID_process > adlı bir klasör oluşturur.
- PID_process > Sıralanmış vinyetler klasörüne gidin ve klasörü sample_yyyymmdd_ hhmm_to_validate kopyalayın. Doğrulamak _to klasör adını _validated ile değiştirin.
- Otomatik sınıflandırmayı el ile doğrulamak için, sample_yyyymmdd_ hhmm_validated klasörünü açın ve yanlış sınıflandırılmış nesneler olup olmadığını belirlemek için her bir alt klasördeki (kategori) tüm vinyetleri gözden geçirin. Bir nesne yanlış sınıflandırıldığında, fareyi kullanarak vinyeti doğru klasöre (kategori) sürükleyin.
- Görüntü işleme yazılımına gidin ve Sıralanmış Vinyetler'den YÜK Tanımlamaları'nı seçin. Varsayılan ayarları koruyun ve yyyymmdd_hhmm_name_validated seçin.
- PID_process > Pid_results > Dat1_validated adlı bir dosyanın ve doğrulanmış örneklerin (Id_from_sorted_vignettes_yyyymmdd_hhmm.txt) her biri için bir .txt dosyasının oluşturulduğu sample_tot_1_dat1.txt gidin.
NOT: Bu .txt dosyaları, her nesnenin uzman sınıflandırmasını (yani, doğrulanmış sınıflandırmayı) belirten pred_valid_Id_yyyymmdd_hhmm adı verilen tahmini sunan yeni bir sütun içerir. Bu noktada, doğrulama sırasında yeni kategoriler (örneğin, daha ince taksonomik kategoriler) oluşturulabilir. Ancak, orijinal kategorinin adını yeni adda tutun (ör. macroinvertebrate_chironomidae). Bu, geri çağırma ve hassasiyeti hesaplarken orijinal kategorinin yeniden izlenmesine ve topluluk boyutu yapı parametrelerini (yani, boyut spektrumu ve boyut çeşitliliği) hesaplamak için tüm makroomurgasızların kolayca gruplandırılmasına olanak tanır. Metin dosyası, her organizmanın elipsoidal hacmini bireysel vücut boyutunun bir ölçüsü olarak elde etmek için kullanılan küçük ve büyük eksenler de dahil olmak üzere her nesneyle ilişkili verileri sağlar. Ayrıca, tablonun son iki sütunu, her nesnenin (satır) tahmin edilen ve doğrulanan kategorilerini içerir ve bu da örneklerin alt kümesindeki öğrenme kümesinin geri çağrılmasını ve hassasiyetini kategoriye göre hesaplamaya olanak tanır.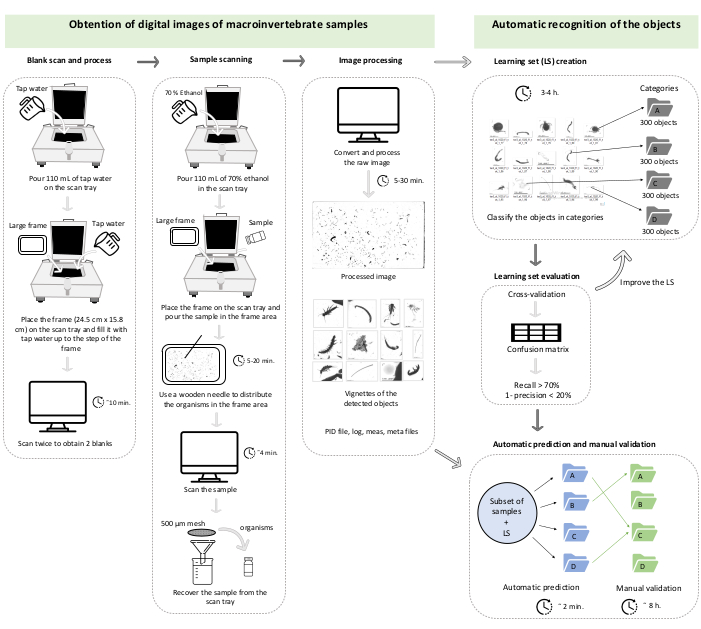
Şekil 1: Protokolün bölüm 1 ve bölüm 2'sini temsil eden çalışma şeması. Zamanlar açıklayıcıdır ve bilgisayara, işlenecek vinyetlerin bolluğuna ve öğrenme kümesinin kategori sayısına bağlı olarak değişebilir. Bu durum, 42 alt örneklemden oluşan bir kümede (toplamda 47.473 vinyet) üç kategoriden oluşan bir öğrenme kümesinin doğrulanmasına karşılık gelir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
3. Tek tek boyut dağılımını, boyut spektrumlarını ve boyut metriklerini hesaplama
NOT: Bu bölümde bahsedilen hesaplamalar Matlab kullanılarak gerçekleştirilmiştir (Ek Dosya 1 olarak komut dosyasına bakın).
- Bireysel boyut dağılımı
- Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt dosyasının son sütunu, nesnelerin doğrulanmış sınıflandırmasını içerir. Örnekteki bireysel boyut dağılımlarını göstermek için yalnızca makroomurgasız olarak sınıflandırılan nesneleri seçin.
NOT: Bireysel vücut büyüklüğü, makroomurgasız organizmaların elipsoidal hacmine karşılık gelir. Sistem ölçümleri piksel cinsinden sağlar. - Vektörleri her iki taramadaki boyut ölçümleriyle birleştirin, çünkü her kesirin farklı bir alt örnekleme üssü vardır. Birleştirmeden önce, boyut vektörlerini karşılık gelen alt örneklemin fraksiyone edildiği kadar çoğaltarak fraksiyonasyon için düzeltin.
NOT: Bu adım, tarama bir numunenin bir kısmına karşılık geliyorsa (yani, kaba veya ince) gereklidir. - Elipsoidal hacmi, organizmalarla aynı piksel alanlarına sahip prolat elipsoidlerin majör (M) ve minör (m) eksenlerinden hesaplayın. Elipsoidal hacmi hesaplamadan önce, aşağıdaki dönüştürme faktörü (cf) ile majör (M) ve minör (m) eksenleri pikselden milimetreye (mm) dönüştürün:
1 piksel = 2.400 dpi
1 inç = 25,4 mm
cf = 25,4/2400
Elipsoidal hacim (mm3 cinsinden birimlere sahip elipVol) aşağıdakilere karşılık gelir: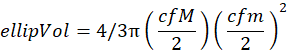
- Günlük2 ölçeğinde tek tek boyut dağılımının olasılık yoğunluğu işlevini gösterin.
- Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt dosyasının son sütunu, nesnelerin doğrulanmış sınıflandırmasını içerir. Örnekteki bireysel boyut dağılımlarını göstermek için yalnızca makroomurgasız olarak sınıflandırılan nesneleri seçin.
- Boyut çeşitliliği
- García-Comas et al. (2016)35'te olduğu gibi, Quintana et al. (2008)8'i takiben boyut çeşitliliğini (Sd) hesaplayın:
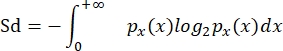
burada p x(x), x boyutunun olasılık yoğunluğu fonksiyonudur ve x, log2'yi (ellipVol) temsil eder. Bu nedenle, bu ölçüm, bir topluluktaki bireysel boyut dağılımı gibi sürekli bir ölçüme uyarlanmış Shannon çeşitlilik endeksidir.
- García-Comas et al. (2016)35'te olduğu gibi, Quintana et al. (2008)8'i takiben boyut çeşitliliğini (Sd) hesaplayın:
- Normalleştirilmiş biyohacim boyutu spektrumu (NBSS)
- NBSS'nin boyut sınıflarını tanımlayın, spektrumun alt sınırını örneklerdeki makroomurgasız boyut dağılımının 0.01 kantilini olarak belirleyin ve örneklerdeki en büyük organizma kuşatılana kadar taban 2'nin geometrik ölçeği ile boyut sınıfları oluşturun.
NOT: Boyut sınıfı genişliği, daha büyük boyutlarla ilişkili daha büyük değişkenliği hesaba katmak için boyutla birlikte artar. Burada analiz edilen makroomurgasız toplulukların NBSS'si 14 boyut sınıfına sahipti (Tablo 1). - Her boyut sınıfındaki toplam biyohacmi boyut sınıfı genişliğine bölerek normalleştirilmiş biyohacmi elde edin.
- NBSS'nin boyut sınıflarını tanımlayın, spektrumun alt sınırını örneklerdeki makroomurgasız boyut dağılımının 0.01 kantilini olarak belirleyin ve örneklerdeki en büyük organizma kuşatılana kadar taban 2'nin geometrik ölçeği ile boyut sınıfları oluşturun.
- Boyut spektrum eğimi
- NBSS'nin doğrusal eğimini hesaplayın.
NOT: Eğim (μ), moddan daha büyük boyut sınıflarındaki log 2 (boyut sınıfı orta noktası) ile log2 (normalleştirilmiş biyokütle) arasındaki ilişkiye dayanarak, boş olanları (bu çalışmada, 3'ten 14'e kadar olan boyut sınıfları) göz ardı ederek hesaplanır.
- NBSS'nin doğrusal eğimini hesaplayın.
| Boyut sınıfı sınırları (mm3) | Boyut sınıfı orta noktası (mm3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
Tablo 1: Normalleştirilmiş biyokütle boyut spektrumunun (NBSS) boyut sınıfları. Tablo ayrıca organizmaların 15 boyut sınıfı sınırını ve boyut sınıfı orta noktalarını göstermektedir.
Sonuçlar
Makroomurgasız örneklerinin dijital görüntülerinin elde edilmesi
Tarama nüansları: Tarama tepsisinde etanol birikimi
Sistemi makroomurgasızlar için test ederken, birkaç tarama kalitesizdi. Arka plandaki koyu doymuş bir alan, görüntünün normal olarak işlenmesini ve makroomurgasızların bireysel boyutlarının ölçülmesini engelledi (Şekil 2). Arka planda doymuş alanların veya yüksek pikselli görüntülerin ortaya çıkması için...
Tartışmalar
Gorsky ve ark. 2010 tarafından nehir makroomurgasızları için açıklanan metodolojinin uyarlanması, tatlı su makroomurgasızlarında topluluk büyüklüğü yapısını tahmin etmede yüksek sınıflandırma doğruluğuna izin verir. Sonuçlar, protokolün bir örneklemdeki bireysel boyut yapısını tahmin etme süresini yaklaşık 1 saate indirebileceğini göstermektedir. Bu nedenle, önerilen protokol, tatlı su ekosistemlerindeki bozulmaların etkisini değerlendirmek için makroomurgasız boyut spektrumların...
Açıklamalar
Yazarlar potansiyel rakip çıkarlar olmadığını beyan ederler.
Teşekkürler
Bu çalışma İspanya Bilim, İnovasyon ve Üniversiteler Bakanlığı tarafından desteklenmiştir (hibe numarası RTI2018-095363-B-I00). CERM-UVic-UCC üyeleri Èlia Bretxa, Anna Costarrosa, Laia Jiménez, María Isabel González, Marta Jutglar, Francesc Llach ve Núria Sellarès'e makroomurgasız alan örneklemesi ve laboratuvar sıralamasındaki çalışmaları için ve David Albesa'ya örnek taramasında işbirliği yaptıkları için teşekkür ederiz. Son olarak Josep Maria Gili'ye ve Institut de Ciències del Mar'a (ICM-CSIC) laboratuvar tesislerinin ve tarayıcı cihazının kullanımı için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
Referanslar
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır