A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
עיבוד תמונה אוטומטי לקביעת מבנה גודל הקהילה של מקרו-חסרי חוליות בנהרות
In This Article
Summary
המאמר מבוסס על יצירת פרוטוקול מותאם לסריקה, זיהוי, מיון וזיהוי אובייקטים דיגיטליים המתאימים למקרו-חסרי חוליות של נהרות בנטיים באמצעות הליך הדמיה חצי אוטומטי. הליך זה מאפשר רכישה של התפלגויות הגודל האינדיבידואליות ומדדי הגודל של קהילת מקרו-חסרי חוליות תוך כשעה.
Abstract
גודל הגוף הוא תכונה פונקציונלית חשובה שיכולה לשמש כביו-אינדיקטור להערכת ההשפעות של הפרעות בקהילות טבעיות. מבנה גודל הקהילה מגיב לשיפועים ביוטיים וא-ביוטיים, כולל הפרעות אנתרופוגניות בין טקסונים ומערכות אקולוגיות. עם זאת, המדידה הידנית של אורגניזמים בעלי גוף קטן כגון מקרו-חסרי חוליות בנטיים (למשל, >500 מיקרומטר עד כמה סנטימטרים אורך) גוזלת זמן רב. כדי לזרז את ההערכה של מבנה גודל הקהילה, כאן, פיתחנו פרוטוקול למדידה אוטומטית למחצה של גודל הגוף האינדיבידואלי של מקרו-חסרי חוליות בנהרות שהשתמרו, שהם אחד הביו-אינדיקטורים הנפוצים ביותר להערכת המצב האקולוגי של מערכות אקולוגיות של מים מתוקים. פרוטוקול זה מותאם ממתודולוגיה קיימת שפותחה לסריקת מזוזואופלנקטון ימי באמצעות מערכת סריקה המיועדת לדגימות מים. הפרוטוקול מורכב משלושה שלבים עיקריים: (1) סריקת תת-דגימות (שברים בגודל דגימה עדין וגס) של מקרו-חוליות בנהר ועיבוד התמונות הדיגיטליות כדי להתאים אישית כל אובייקט שזוהה בכל תמונה; (2) יצירה, הערכה ואימות של מערך למידה באמצעות בינה מלאכותית כדי להפריד באופן אוטומטי למחצה את התמונות הבודדות של מקרו-חוליות מדטריטוס וממצאים בדגימות הסרוקות; ו-(3) תיאור מבנה הגודל של קהילות המקרו-חסרי חוליות. בנוסף לפרוטוקול, עבודה זו כוללת את תוצאות הכיול ומפרטת מספר אתגרים והמלצות להתאמת ההליך לדגימות מקרו-חסרי חוליות ולשקול שיפורים נוספים. בסך הכל, התוצאות תומכות בשימוש במערכת הסריקה המוצגת למדידת גודל הגוף האוטומטית של מקרו-חסרי חוליות בנהרות ומציעות כי תיאור ספקטרום הגודל שלהם הוא כלי רב ערך להערכה ביולוגית מהירה של מערכות אקולוגיות של מים מתוקים.
Introduction
מקרו-חסרי חוליות בנטיים נמצאים בשימוש נרחב כביו-אינדיקטורים כדי לקבוע את המצב האקולוגי של גופי מים1. רוב המדדים לתיאור קהילות מקרו-חסרי חוליות מתמקדים במדדים טקסונומיים. עם זאת, כלי הערכה ביולוגית חדשים המשלבים את גודל הגוף מעודדים לספק נקודת מבט חלופית או משלימה לגישות טקסונומיות 2,3.
גודל הגוף נחשב למטא-טראיט הקשור לתכונות חיוניות אחרות כגון חילוף חומרים, גדילה, נשימה ותנועה4. יתר על כן, גודל הגוף יכול לקבוע מיקום טרופי ואינטראקציות5. הקשר בין גודל הגוף האינדיבידואלי לבין הביומסה המנורמלת (או השפע) לפי גודל בקהילה מוגדר כספקטרום הגודל6 ועוקב אחר התבנית הכללית של ירידה ליניארית בביומסה המנורמלת ככל שגודל הפרט גדל בסולם לוגריתמי7. השיפוע של מערכת יחסים ליניארית זו נחקר בהרחבה באופן תיאורטי, ומחקרים אמפיריים על פני מערכות אקולוגיות השתמשו בו כאינדיקטור אקולוגי למבנה גודל הקהילה4. אינדיקטור סינתטי נוסף למבנה גודל הקהילה ששימש בהצלחה במחקרי תפקוד של מגוון ביולוגי-מערכות אקולוגיות הוא מגוון גודל הקהילה, המיוצג כמדד שאנון של מחלקות הגודל של ספקטרום הגודל או האנלוגי שלו, המחושב על סמך התפלגויות הגודל האינדיבידואליות8.
במערכות אקולוגיות של מים מתוקים, מבנה הגודל של קבוצות חיות שונות משמש כאינדיקטור אטקסי להערכת התגובה של קהילות ביוטיות לשיפועים סביבתיים 9,10,11 ולהפרעות אנתרופוגניות 12,13,14,15,16. מקרו-חסרי חוליות אינם יוצאי דופן, ומבנה גודלם מגיב גם לשינויים סביבתיים17,18 ולהפרעות אנתרופוגניות, כגון כרייה 19, שימוש בקרקע 20, או העשרת חנקן (N) וזרחן (P)20,21,22. עם זאת, מדידת מאות אנשים כדי לתאר את מבנה גודל הקהילה היא משימה מייגעת וגוזלת זמן, שלעתים קרובות נמנעת ממנה כמדידה שגרתית במעבדות בשל חוסר זמן. לפיכך, מספר שיטות הדמיה חצי אוטומטיות או אוטומטיות לסיווג ומדידת דגימות פותחו23,24,25,26. עם זאת, רוב השיטות הללו מתמקדות בסיווג טקסונומי יותר מאשר בגודל הפרט של האורגניזמים ואינן מוכנות לשימוש עבור כל מיני מקרו-חסרי חוליות. באקולוגיה של פלנקטון ימי, נעשה שימוש נרחב במערכת ניתוח תמונות סורקת כדי לקבוע את הגודל וההרכב הטקסונומי של קהילות זואופלנקטון 27,28,29,30,31. ניתן למצוא מכשיר זה במספר מכונים ימיים ברחבי העולם, והוא משמש לסריקת דגימות זואופלנקטון שהשתמרו כדי לקבל תמונות דיגיטליות ברזולוציה גבוהה של הדגימה כולה. הפרוטוקול הנוכחי מתאים את השימוש במכשיר זה כדי להעריך את ספקטרום גודל קהילת המקרו-חסרי חוליות בנהרות באופן אוטומטי מהיר מבלי להשקיע ביצירת מכשיר חדש.
הפרוטוקול מורכב מסריקת דגימה ועיבוד התמונה כולה כדי לקבל באופן אוטומטי תמונות בודדות (כלומר, vignettes) של האובייקטים בדגימה. מספר מדדים של צורה, גודל ותכונות ברמת האפור מאפיינים כל אובייקט ומאפשרים סיווג אוטומטי של האובייקטים לקטגוריות, אשר מאומתות לאחר מכן על ידי מומחה. הגודל האינדיבידואלי של כל אורגניזם מחושב באמצעות הביו-נפח האליפסואידי (מ"מ3), הנגזר משטח האורגניזם הנמדד בפיקסלים. זה מאפשר לקבל את ספקטרום הגודל של המדגם בצורה מהירה. למיטב ידיעתנו, מערכת הדמיית סריקה זו שימשה רק לעיבוד דגימות מזוזואופלנקטון, אך המכשיר עשוי לאפשר עבודה עם מקרו-חסרי חוליות בנטיים במים מתוקים.
המטרה הכוללת של מחקר זה היא, אם כן, להציג שיטה להשגת הגודל האינדיבידואלי של מקרו-חסרי חוליות בנהר שהשתמרו על ידי התאמת פרוטוקול קיים ששימש בעבר עם מזוזואופלנקטון ימי 27,32,33. ההליך מורכב משימוש בגישה חצי אוטומטית הפועלת עם מכשיר סריקה לסריקת דגימות מים ושלוש תוכנות פתוחות לעיבוד התמונות הסרוקות. פרוטוקול מותאם לסריקה, זיהוי וזיהוי של מקרו-חסרי חוליות בנהרות שעברו דיגיטציה כדי לרכוש באופן אוטומטי את מבנה גודל הקהילה ומדדי גודל קשורים מוצג כאן. הערכת הנוהל וההנחיות לשיפור היעילות מוצגות גם על סמך 42 תמונות סרוקות של דגימות מקרו-חוליות בנהרות שנאספו משלושה אגנים בחצי האי האיברי הצפון-מזרחי (NE) (Ter, Segre-Ebre ו-Besòs).
הדגימות נאספו בקטעי נהרות בגובה 100 מטרים בעקבות פרוטוקול לדיגום שדה וניתוח מעבדה של מקרו-חולייתנים של נהרות בנטיים בנהרות יבשתיים מממשלת ספרד34. הדגימות נאספו באמצעות סמפלר סורבר (מסגרת: 0.3 מ' x 0.3 מ', רשת: 250 מיקרומטר) בעקבות סקר רב-בתי גידול. במעבדה, הדגימות נוקו וסוננו באמצעות רשת של 5 מ"מ ו-500 מיקרומטר כדי לקבל שתי תת-דגימות: תת-דגימה גסה (רשת 5 מ"מ) ותת-דגימה עדינה (רשת של 500 מיקרומטר), שאוחסנו בבקבוקונים נפרדים ונשמרו ב-70% אתנול. הפרדת הדגימה לשני שברים בגודל מאפשרת הערכה טובה יותר של מבנה גודל הקהילה, שכן אורגניזמים גדולים הם נדירים יותר ופחות מהאורגניזמים הקטנים. אחרת, למדגם הסרוק יש ייצוג מוטה של שבר הגודל הגדול.
Protocol
הערה: הפרוטוקול המתואר כאן מבוסס על המערכת שפותחה על ידי Gorsky et al.27 עבור מזוזואופלנקטון ימי. תיאור ספציפי של הסורק (ZooSCAN), תוכנת הסריקה (VueScan 9x64 [9.5.09]), תוכנת עיבוד התמונה (Zooprocess, ImageJ) ושלבי תוכנת הזיהוי האוטומטי (מזהה פלנקטון) ניתן למצוא בהפניות קודמות32,33. כדי להתאים בצורה הטובה ביותר את הגדלים של המקרו-חסרי חוליות הבנטיים ביחס למזוזואופלנקטון, לאחר שהפרויקט נוצר בעקבות הפרוטוקול המקורי32,33, שנה את הפרמטר של גודל מינימלי (minsizeesd_mm) ל- 0.3 מ"מ ואת הפרמטר בגודל מרבי (maxsizeesd_mm) ל- 100 מ"מ בקובץ התצורה. כדי לעזור לעקוב אחר הפרוטוקול, זה מסוכם בתרשים עבודה (איור 1). הפרוייקט שנוצר מאוחסן בתיקיית C של המחשב ומאורגן בתיקיות הבאות: PID_process, Zooscan_back, Zooscan_check, Zooscan_config, Zooscan_meta, Zooscan_results ו- Zooscan_scan. כל תיקיה מורכבת ממספר תיקיות משנה שבהן משתמשים יישומי התוכנה השונים בשלבים הבאים של הפרוטוקול.
1. רכישת תמונות דיגיטליות לדגימות מקרו-חסרי חוליות
- סריקה ועיבוד של הריק
הערה: צור שתי תמונות ריקות מדי יום לפני הסריקה כדי לחלץ את סריקות הרקע בעת עיבוד התמונות הסרוקות באותו יום.- הפעל את הסורק והדליק את האור במיקום הכפול כדי להקרין אור לבן מלמעלה ומלמטה.
הערה: בעת סריקת דגימות מזוזואופלנקטון, נעשה שימוש בכיוון האור כלפי מעלה, אך מכיוון שמקרו-חסרי חוליות אטומים יותר, מומלץ להעביר את האור למצב כפול. - נקו ושטפו את מגש הסריקה במי ברז.
- מוזגים 110 מ"ל של מי ברז המאוחסנים בטמפרטורת החדר (RT) למגש הסריקה עד לכיסוי הכוס. הנח את המסגרת הגדולה (24.5 ס"מ x 15.8 ס"מ) על מגש הסריקה במיקום הנכון (עם הפינה בחלק השמאלי העליון של מגש הסריקה), ומלא אותה במי ברז עד לכיסוי שלב המסגרת כדי למנוע אפקט מניסקוס, שישנה את התמונה הסרוקה. סגור את מכסה הסורק.
הערה: יש להשתמש במים ב-RT כדי למנוע עיבוי והיווצרות בועות. נקו את המסגרת ללא סימנים או טיפות כדי למנוע השתקפות אור. - עבור אל תוכנת עיבוד התמונה, בחר את פרויקט העבודה ולחץ על סרוק (המר) תמונת רקע.
- עבור אל תוכנת הסריקה ולחץ על תצוגה מקדימה. הקפד להציג תצוגה מקדימה של התמונה הסרוקה, לבדוק שאין קווים או כתמים ולהמתין לפחות 30 שניות לפני תחילת סריקה נוספת. לחץ על סריקה ולחץ על אישור בחלון ההוראות לפני הסריקה השנייה כדי לשלוח את הנתונים מתוכנת הסריקה לתוכנת עיבוד התמונה.
הערה: סרוק פעמיים כדי לקבל את שתי סריקות הרקע שירכיבו את הריק. שלב זה נעשה פעם ביום לפני תחילת עיבוד הדגימה, והתמונות מאוחסנות בתיקיית Zooscan_back. - סגור את תוכנת הסריקה לאחר סיום הסריקה.
- הפעל את הסורק והדליק את האור במיקום הכפול כדי להקרין אור לבן מלמעלה ומלמטה.
- הכנה וסריקה של דוגמאות
אזהרה: אתנול הוא נוזל דליק שעלול לגרום לנזק/גירוי חמור בעיניים.- מלא את המטה-נתונים לדוגמה. עבור אל תוכנת עיבוד התמונה ובחר מלא מטה-נתונים לדוגמה. הזן את הזהות לדוגמה, לחץ על אישור ומלא את המטא נתונים.
הערה: קובץ המטא-קובץ נוצר במיוחד עבור דגימות מזוזואופלנקטון, כך שהוא אינו מתאים למתודולוגיית דגימת המקרו-חסרי חוליות הבנטית, אך יש למלא את כל שדות הקובץ לפני הסריקה, אחרת יופיע דגל שגיאה. - מוזגים 110 מ"ל של 70% אתנול למגש הסריקה עד לכיסוי הכוס ומניחים את המסגרת הגדולה (24.5 ס"מ על 15.8 ס"מ) עם הפינה בחלק השמאלי העליון של מגש הסריקה.
הערה: עבודה עם אתנול במקום מים, שכן המקרו-חסרי חוליות נשמרים באתנול. במים, הם צפים ונסחפים במגש הסריקה, ומונעים תמונה חדה, ובכך, מדידות גודל אמינות. אתנול צריך להישמר ב- RT כדי למנוע עיבוי והיווצרות בועות. - שפכו את דגימת המקרו-חסרי חוליות למגש הסריקה שבקצהו המסגרת, וכסו את שלב המסגרת באתנול נוסף במידת הצורך.
הערה: הימנעו מהוספת כמות גדולה מדי של אתנול כדי למנוע מהאורגניזמים לצוף ולהיסחף. - הומוגניזציה של הדגימה בכל אזור המסגרת, הצבת הפרטים הגדולים ביותר במרכז המגש לעיבוד תמונה תקין, והטבעת האורגניזמים הצפים באמצעות מחט עץ.
הערה: אם תת-דגימה מכילה באופן מספרי יותר מ-1,000 פרטים, חלק את תת-הדגימה לשני שברים או יותר כדי למזער את המגע באורגניזמים בתמונה הסרוקה, וסרוק את השברים בנפרד. - הפרד את האורגניזמים הנוגעים במגע ואת האורגניזמים הנוגעים בקצוות המסגרת באמצעות מחט העץ.
הערה: שלב זה דורש 5-20 דקות. אורגניזמים נוגעים נחשבים לאובייקט יחיד על ידי התוכנה; לפיכך, במקרים אלה, הגדלים האישיים המחושבים אינם תואמים אורגניזמים בודדים בפועל ויכולים להטות את הערכת מבנה גודל הקהילה. קיימת אפשרות לערוך את התמונה עם תוכנת עיבוד התמונה כדי להפריד ביניהם, אך שלב נוסף זה כרוך לפחות 1.5 שעות של עיבוד מחדש; לכן, הפרדה ידנית מומלצת מאוד. - כדי לסרוק את המדגם, סגור את מכסה הסורק, עבור אל תוכנת עיבוד התמונה, בחר את פרויקט העבודה ולחץ על SCAN Sample with Zooscan (עבור ארכיון, ללא תהליך).
- בחר את הדוגמה ובצע את ההוראות.
- עבור אל תוכנת הסריקה ולחץ על תצוגה מקדימה. הקפד להציג תצוגה מקדימה של התמונה הסרוקה, לבדוק שאין קווים או כתמים ולהמתין לפחות 30 שניות לפני תחילת סריקה נוספת.
- לאחר 30 שניות לפחות, לחץ על כפתור הסריקה בתוכנת הסריקה.
הערה: לחץ על OK בתוכנת עיבוד התמונה לאחר לחיצה על Scan בתוכנת הסריקה. אל תלחץ על מקש כלשהו במקלדת המחשב והימנע מרעידות של הסריקה במהלך הסריקה. שלושה קבצים נוצרים בתיקייה Zooscan_scan > _raw : (i) תבנית קובץ תמונה מתויגת (.tif) (16 סיביות); (ii) מסמך טקסט סטנדרטי בשם LOG (.txt) המתעד מידע בפרמטרים של הסריקה; ו-(iii) מסמך טקסט סטנדרטי בשם META (.txt) עם מידע על שיטות הדגימה. - ודא שהסריקה הגולמית נכונה.
הערה: אם הסריקה כוללת פסים בהירים או בעיות גלויות אחרות, שקול לחזור על הסריקה כדי למנוע בעיות בשלבים הבאים.
- מלא את המטה-נתונים לדוגמה. עבור אל תוכנת עיבוד התמונה ובחר מלא מטה-נתונים לדוגמה. הזן את הזהות לדוגמה, לחץ על אישור ומלא את המטא נתונים.
- שחזור לדוגמה
- הסר את המסגרת ושטוף אותה מעל מגש הסריקה באמצעות בקבוק לחיץ מלא ב-70% אתנול כדי לשחזר את כל המקרו-חסרי חוליות המצורפים.
- הרם את החלק העליון של הסורק כדי לאחזר את כל האורגניזמים והאתנול מהמגש דרך משפך אחזור הסריקה לתוך. כאשר החלק העליון של הסורק עדיין מורם, שטפו את המגש עם בקבוק הלחיצה כדי לטאטא לאורך כל האורגניזמים שנותרו.
- מעבירים את הדגימות והאתנול מהכוס דרך רשת של 500 מיקרומטר כדי לשמור על חסרי החוליות ברשת, ומאחסנים אותם בחזרה בבקבוקון עם 70% אתנול.
- לאחר שכל הדגימות משוחזרות בבקבוקון, נקו את המגש עם מי ברז.
הערה: שטפו את המגש במי ברז בין דגימות כדי למזער את משקעי האתנול, מה שמשנה את עיבוד התמונה. יש לשטוף את המסגרת במי ברז כדי למנוע נזקים אפשריים הקשורים לשימוש באתנול. בסוף היום, נקו את המגש באמצעות מי ברז ויבשו אותו בעדינות עם נייר כדי למנוע שריטות.
- עיבוד תמונה
- עבור אל תוכנת עיבוד התמונה ובחר המר ועבד תמונות ואורגניזמים במצב אצווה ולאחר מכן המר ועבד תמונה וחלקיקים (תמונה בתיקיית RAW). שמור על הגדרות ברירת המחדל ולחץ על אישור. NORMAL END יופיע בסוף התהליך.
הערה: קובץ PID וה-vignettes המתאימים לכל האובייקטים שזוהו בתמונה הסרוקה (בקובץ קבוצת צילום משותפת [.jpg]) ייווצרו בתיקיית Zooscan_scan > _work. קובץ PID הוא קובץ יחיד המאחסן את כל המטא-נתונים (מטא-קובץ), את הנתונים הטכניים המשויכים לקובץ יומן הרישום וטבלה עם 36 משתנים נמדדים של כל העצמים שזוהו בתמונה. המשתנים הנמדדים תואמים לאומדנים שונים של רמת האפור, ממד הפרקטלים, הצורה והגודל. המשתנים שניתן להשתמש בהם להערכת גודל הם השטח והצירים הראשיים והמשניים של אליפסה עם שטח שווה לאובייקט (ראה סעיף 3 של הפרוטוקול). זמן העיבוד תלוי בצפיפות התמונה ובמאפייני המחשב, וניתן לשגר אותו בין דגימות תוך כדי שחזור והכנת הדגימה הבאה. אחרת, מומלץ להפעיל את עיבוד הדגימות שנסרקו מדי יום במצב אצווה במהלך הלילה ולבדוק עיבוד תמונה תקין למחרת בבוקר. - בדוק אם הרקע בתמונה המעובדת מופחת כראוי מהתמונה לדוגמה באמצעות תוכנת עיבוד התמונה או על-ידי בדיקת תמונות המסיכה (שהסתיימו ב- msk1.gif) הממוקמות ב- Zooscan_scan > _work. אם הרקע כולל אזורים רוויים או נקודות רבות, שקול לחזור על הסריקה כדי להבטיח תמונות באיכות גבוהה.
הערה: כדי להימנע מאזורים רוויים ברקע, יש לשטוף את מגש הסריקה במי ברז לאחר כל סריקה עם אתנול. חשוב גם (1) להפחית את מספר האנשים הסרוקים (על ידי פיצול הדגימה וסריקה בקיפולים שונים); (2) לוודא שאורגניזמים גדולים ממוקמים במרכז מגש הסריקה; (3) להשתמש באתנול נקי ומסונן; (4) להפחית את הלכלוך על הדגימות; (5) לוודא שנפח האתנול לסריקה מספיק; וכן (6) לוודא שההשהיה בין התצוגה המקדימה של הדגימה לבין הסריקה היא לפחות 30 שניות.
- עבור אל תוכנת עיבוד התמונה ובחר המר ועבד תמונות ואורגניזמים במצב אצווה ולאחר מכן המר ועבד תמונה וחלקיקים (תמונה בתיקיית RAW). שמור על הגדרות ברירת המחדל ולחץ על אישור. NORMAL END יופיע בסוף התהליך.
- הפרדת אורגניזמים נוגעים
הערה: כאשר ישנם מספר ציורים עם אורגניזמים נוגעים, יש צורך להפריד את התמונות של האורגניזמים הנוגעים ללב מאורגניזמים אחרים ו/או מסיבים /פסולת כדי להבטיח הערכה נכונה של מבנה גודל הקהילה.- עבור אל תוכנת עיבוד התמונה כדי לזהות את vignettes עם אובייקטים מרובים. בחר הפרדת צבע באמצעות Vignettes ולחץ על אישור. בחלון בחירת התצורה, שמור על הגדרות ברירת המחדל ולחץ על אישור.
- בחלון הפרדת צבע מ- VIGNETTES , שמור על הגדרות ברירת המחדל, בחר בנוסף הוסף קווי מתאר ב- Vignettes ולאחר מכן בחר את הדוגמה לעריכה.
- הפרד את האורגניזמים הנוגעים ללב בכל ציור שצץ על-ידי ציור קו עם העכבר (לחץ על לחצן הגליל כדי לצייר). לאחר השלמת ההפרדה ב- vignette, לחץ על לחצן X בפינה השמאלית העליונה של החלון ולחץ על כן כדי לעבד את הלחצן הבא. לחץ על NO כדי לסיים ולשמור את השינויים. בסוף התהליך, NORMAL END יופיע אם הכל נכון.
- לאחר הפרדת הצבע, עבדו מחדש את התמונה כדי לקבל את נתוני האובייקט המעודכנים. עבור אל תוכנת עיבוד התמונה, לחץ על תהליך ( מומר) תמונה (תהליך אחד), ובחר עבד שוב חלקיקים מתמונות מעובדות בתיקיות המשנה של WORK. בחר את הדוגמה, ובחלון תהליך תמונה בודדת , שמור על הגדרות ברירת המחדל, סמן את עבודה עם מסיכת הפרדה (CREATE-CHANGE-INCLUDE) ולאחר מכן לחץ על אישור. בסוף התהליך, NORMAL END יופיע אם הכל נכון.
- בחלון ' בקרת הפרדות צבע ', לחצו על הלחצן 'אשר' לשמירת התמונה עם קווי המתאר שלפני העיבוד; אם קיימת תמונה קודמת, היא תוחלף.
- בחלון ' מסיכת בקרת הפרדות ', במידת הצורך, בחרו 'עריכה ' להוספת קווי הפרדה למסיכה באמצעות העכבר להפרדת אורגניזמים נוגעים שלא הופיעו קודם לכן בשלב ההפרדה באמצעות vignettes. בסיום, סיימו את התהליך, ובחלון ' בקרת מסיכת הפרדה ', בחרו ' כן' כדי לקבל את המסיכה. בסוף התהליך, NORMAL END יופיע אם הכל נכון.
הערה: עיבוד מחדש של דגימה באמצעות מסיכת הפרדה גוזל זמן רב (פעולה זו עשויה להימשך יותר משעה וחצי לכל דגימה). עדיף להקדיש את הזמן הנדרש בשלב 1.2.5 כדי למנוע צעד נוסף זה.
2. זיהוי אוטומטי של האובייקטים
הערה: צור ערכת למידה כדי לחזות באופן אוטומטי את זהות האובייקטים שזוהו, ובכך להפריד את האורגניזמים מהפסולת בדגימה.
- יצירת ערכות למידה
- העתק את התמונות ואת קבצי ה- .pid המשויכים לתמונות שישמשו ליצירת הקטגוריות של ערכת הלמידה מ- Zooscan_scan > _work ל- PID_process > Unsorted_vignettes_pid.
הערה: בחר תת-קבוצה של דגימות עם מגוון טקסונים גבוה ואתרי דגימה שונים ו/או עונות דגימה שונות כדי להבטיח ייצוג מרבי של אורגניזמים בדגימות. - בתיקייה PID_process > Learning set, צור תיקיית משנה עם שם ערכת הלמידה החדשה (כלומר, yyyymmdd_raw_LS), ובתוכה, צור את תיקיות המשנה שיתאימו לכל קטגוריה של ערכת הלמידה (כלומר, מקרו-חסרי חוליות, פסולת, חסרי חוליות אחרים).
הערה: כדי להשיג ביעילות את מבנה גודל הקהילה של דגימות מקרו-חסרי חוליות בנהר, מומלץ להשתמש בערכת למידה המבוססת על שלוש קטגוריות בלבד: מקרו-חסרי חוליות, חסרי חוליות אחרים ופסולת. מערך למידה זה בעצם מפריד בין האובייקטים המתאימים לאורגניזמים לבין אלה המתאימים לפסולת (למשל, סיבים, חלקיקים או אצות נימה). - עבור אל תוכנת עיבוד התמונה (מצב מתקדם בלבד) ובחר EXTRACT Vignettes עבור PLANKTON IDENTIFIER (ציורים לא ממוינים לאימון). השאר את אפשרויות ברירת המחדל וסמן את התיבה הוסף קווי מתאר .
- עבור אל תוכנת הזיהוי האוטומטית, לחץ על למידה, בחר מתוך PID_process > Learning_set תיקיית המשנה שנוצרה עבור ערכת הלמידה החדשה (שלב 2.1.2) ולחץ על אישור.
- בחלק השמאלי (אגודלים לא ממוינים) של החלון הפתוח, בחר את התיקיה לא ממוינת vignettes_pid. בחר את vignettes וגרור אותם עם העכבר מן האגודלים הלא ממוינים לתיקייה של הקטגוריה המתאימה שלהם בחלק הימני, אגודלים ממוינים, כדי לסווג כל אובייקט לקטגוריות המוגדרות. הציורים שהועברו יסומנו ב-X אדום.
הערה: הגדר את הקטגוריות באופן ידני על ידי יצירת תיקיות משנה בתיקיית האגודלים הממוינת או צור אותן על ידי לחיצה על סמל התיקיות בתוכנה. אין להזיז יותר מ-50 ציורים בו-זמנית. - לאחר השלמת כל הקטגוריות עם האובייקטים שנבחרו (כ -300 אובייקטים לקטגוריה), לחץ על צור קובץ למידה ושמור אותו עם השם הרצוי.
הערה: ערכת הלמידה תישמר כקובץ .pid בתיקיה PID_process > ערכת למידה של הפרוייקט. מומלץ ליצור ולבדוק מספר קבוצות למידה עם רמות שונות של קטגוריות (מצורות גסות ועד עדינות) ועם איזון שונה של מספר האובייקטים בתוך כל קטגוריה. התחל עם ערכת למידה גסה עם מספר נמוך של קטגוריות ולפחות 50 אובייקטים לכל קטגוריה, ולאחר מכן הגדל את מספר האובייקטים בכל קטגוריה ו/או צור ערכות למידה עדינות יותר. קטגוריה צריכה לייצג את השתנותה בקבוצת הדגימות.
- העתק את התמונות ואת קבצי ה- .pid המשויכים לתמונות שישמשו ליצירת הקטגוריות של ערכת הלמידה מ- Zooscan_scan > _work ל- PID_process > Unsorted_vignettes_pid.
- הערכת מערך הלמידה
הערה: בצע אימות צולב עם שני קיפולים וחמישה ניסויים באמצעות שיטת Random Forest עם תוכנת הזיהוי האוטומטית כדי להשיג מטריצת בלבול של הסיווג המתקבל של האובייקטים.- עבור אל תוכנת הסיווג האוטומטית ולחץ על ניתוח נתונים.
- תחת בחר קובץ למידה, בחר את קובץ ערכת הלמידה שנוצר מתוך ערכת הלמידה PID_process >.
- בתיבה בחר שיטה, בחר בשיטה 'יער אקראי של אימות צולב' . במשתנים מקוריים, בטל את הסימון של משתני המיקום (X, Y, XM, YM, BX, BY וגובה). במשתנים מותאמים אישית, סמן רק ESD.
הערה: שיטה זו משתמשת בחלק אקראי אחד של ערכת הלמידה כדי לזהות את החלק השני (שני קיפולים), וזה חוזר על עצמו חמש פעמים כדי להבטיח שהוא עמיד סטטיסטית. - לחץ על התחל ניתוח ושמור את התוצאות כ- Analysis_name.txt בתיקיית החיזוי PID_process >. לאחר שהניתוח הושלם בהצלחה, צא מניתוח הנתונים.
- עבור אל תיקיית החיזוי PID_process > ולחץ על קובץ האימות הצולב. חלון יופיע עם מטריצת הבלבול של הסיווג האמיתי (שורות) לעומת הסיווג האוטומטי (עמודות).
הערה: ההיזכרות היא אחוז האורגניזמים השייכים לקבוצה שזוהתה היטב באופן אוטומטי, ואילו 1-דיוק הוא אחוז האורגניזמים המסווגים על ידי האלגוריתם כקבוצה שאינה מוכרת (זיהום בקבוצה). ההחזרה צריכה להיות מעל 70%, והזיהום (דיוק 1) צריך להיות נמוך מ -20%. - חזור על שלבים 2.1-2.5 אם נוצרו מספר ערכות למידה ויש להשיג את ההיזכרות והדיוק 1 של כל אחת מהן.
הערה: אם נוצרו מספר ערכות למידה, בחר את זו עם ההיזכרות הגדולה ביותר (זיהוי טוב) והדיוק (זיהום נמוך) של קבוצת העניין (כלומר, מקרו-חסרי חוליות) כדי לבדוק את החיזוי האוטומטי של קבוצת דגימות בשלב הבא.
- חיזוי זיהוי של מקרו-חסרי חוליות
הערה: השתמש בערכת הלמידה שנבחרה כדי לחזות את הזהות של כל האובייקטים בקבוצת משנה של דגימות באמצעות תוכנת הזיהוי האוטומטית עם אלגוריתם יער אקראי.- עבור אל תוכנת הזיהוי האוטומטית ולחץ על ניתוח נתונים.
- בקובץ 'בחר למידה', בחר את קובץ ערכת הלמידה מתוך ערכת הלמידה PID_process > שיש להשתמש בו לחיזוי.
- תחת בחר קבצים לדוגמה, בחר מהתיקיה PID_results את הדוגמאות (קבצי PID) שיחזו.
הערה: עבד עד 20 קבצי .pid בו-זמנית כדי למנוע שגיאות הקשורות לבעיות זיכרון. אם יותר מדי קבצי .pid מעובדים בו-זמנית, התהליך יציג סוף נכון אך ייתכן שלא יעובד היטב, ושגיאה עלולה להתרחש בשלבים הבאים בעת עיבוד באמצעות תוכנת עיבוד התמונה. - בתיבה בחר שיטה, בחר את שיטת היער האקראי . סמן שמור תוצאות מפורטות עבור כל מדגם. במשתנים מקוריים, בטל את הסימון של משתני המיקום (X, Y, XM, YM, BX, BY וגובה). במשתנים מותאמים אישית, סמן רק ESD.
- לחץ על התחל ניתוח ושמור את התוצאות כ- Analysis_name.txt בתיקיית החיזוי PID_process >.
- אימות ידני
הערה: מומחה מאמת באופן ידני את החיזוי מהשלב הקודם כדי לסווג מחדש אובייקטים שסווגו באופן שגוי לקטגוריה הנכונה.- העתק את קבצי Analysis_sample_dat1.txt לאימות מהתיקיה PID_process > חיזוי לתיקיית PID_process > Pid_results.
- עבור אל תוכנת עיבוד התמונה ובחר חלץ Vignettes בתיקיות על פי חיזוי או אימות. לאחר מכן, בחר השתמש בקבצים חזויים מתיקיית "pid_results". שמור את הגדרות ברירת המחדל ולחץ על OK.
- התוכנה יוצרת תיקייה בשם sample_yyyymmdd_hhmm_to_validate עם האובייקטים החזויים בתיקייה vignettes ממוינים PID_process >.
- עבור אל התיקיה PID_process > vignettes ממוינים והעתק את התיקיה sample_yyyymmdd_ hhmm_to_validate. החלף את שם התיקיה _to לאמת ב - _validated.
- כדי לאמת באופן ידני את הסיווג האוטומטי, פתח את sample_yyyymmdd_ hhmm_validated התיקיה וסקור את כל הציורים מכל תיקיית משנה (קטגוריה) כדי לזהות אם קיימים אובייקטים המסווגים באופן שגוי. כאשר אובייקט אחד מסווג באופן שגוי, גרור את הפריט באמצעות העכבר לתיקיה הנכונה (קטגוריה).
- עבור אל תוכנת עיבוד התמונה ובחר זיהויי עומס מתוך Vignettes ממוינים. שמור את הגדרות ברירת המחדל ובחר yyyymmdd_hhmm_name_validated לעיבוד.
- עבור אל PID_process > Pid_results > Dat1_validated, שבו נוצר קובץ בשם Id_from_sorted_vignettes_yyyymmdd_hhmm.txt וקובץ .txt אחד עבור כל אחת מהדוגמאות המאומתות (sample_tot_1_dat1.txt).
הערה: קובצי .txt אלה מכילים עמודה חדשה המציגה את החיזוי, הנקרא pred_valid_Id_yyyymmdd_hhmm, המציין את סיווג המומחה של כל אובייקט (כלומר, הסיווג המאומת). ניתן ליצור קטגוריות חדשות (למשל, קטגוריות טקסונומיות עדינות יותר) בשלב זה, במהלך האימות. עם זאת, שמור את שם הקטגוריה המקורית בשם החדש (לדוגמה, macroinvertebrate_chironomidae). זה מאפשר להתחקות אחר הקטגוריה המקורית בעת חישוב ההיזכרות והדיוק ולקבץ בקלות את כל המקרו-חסרי חוליות כדי לחשב את הפרמטרים של מבנה גודל הקהילה (כלומר, ספקטרום הגודל ומגוון הגודלים). קובץ הטקסט מספק את הנתונים המשויכים לכל אובייקט, כולל הצירים הקטנים והעיקריים המשמשים לקבלת הנפח האליפסואידי של כל אורגניזם כמדד לגודל גוף אינדיבידואלי. יתר על כן, שתי העמודות האחרונות של הטבלה מכילות את הקטגוריות החזויות והמאומתות של כל אובייקט (שורה), המאפשרות לחשב, לפי קטגוריה, את ההיזכרות והדיוק של קבוצת הלמידה על תת-קבוצה של דגימות.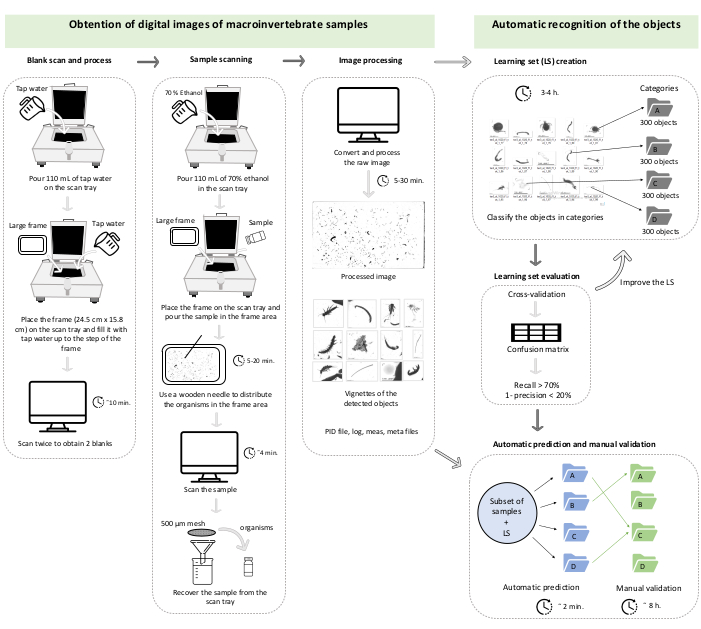
איור 1: תרשים עבודה המייצג את סעיף 1 וסעיף 2 של הפרוטוקול. הזמנים הם להמחשה ויכולים להשתנות בהתאם למחשב, לשפע הווינטות לעיבוד ולמספר הקטגוריות של מערך הלמידה. מקרה זה מתאים לאימות של קבוצת למידה של שלוש קטגוריות על קבוצה של 42 תת-דגימות (בסך הכל, 47,473 vignettes). אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
3. חישוב התפלגות הגודל הבודדת, ספקטרום הגודל ומדדי הגודל
הערה: החישובים המוזכרים בסעיף זה בוצעו באמצעות Matlab (ראה סקריפט כקובץ משלים 1).
- התפלגות גודל אישי
- העמודה האחרונה של קובץ Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt מכילה את הסיווג המאומת של האובייקטים. בחרו רק בעצמים המסווגים כמקרו-חסרי חוליות כדי לתאר את התפלגות הגודל האינדיבידואלי שלהם בדגימה.
הערה: גודל הגוף האינדיבידואלי מתאים לנפח האליפסואידי של האורגניזמים המקרו-חסרי חוליות. המערכת מספקת מדידות בפיקסלים. - שרשררו את הווקטורים עם מדידות הגודל משתי הסריקות, מכיוון שלכל שבר יש מעריך תת-דגימה שונה. לפני שרשור, תקן את הפיצול על ידי שכפול וקטורי הגודל כמה פעמים שתת-הדגימה המתאימה הופצלה.
הערה: שלב זה נחוץ אם סריקה מתאימה לשבריר של דגימה (כלומר, גסה או עדינה). - חשב את הנפח האליפסואידי מהציר הראשי (M) והמינורי (m) של אליפסואידים פרולטים עם אותם אזורי פיקסלים כמו האורגניזמים. לפני חישוב הנפח האליפסואידי, המר את הצירים הראשי (M) והמשני (m) מפיקסלים למילימטרים (מ"מ) באמצעות מקדם ההמרה הבא (cf):
1 פיקסל = 2,400 dpi
1 אינץ' = 25.4 מ"מ
cf = 25.4/2400
הנפח האליפסואידי (אליפוול עם יחידות במ"מ3) מתאים ל: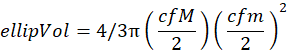
- תאר את פונקציית צפיפות ההסתברות של התפלגות הגודל הבודד בסולם לוג2 .
- העמודה האחרונה של קובץ Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt מכילה את הסיווג המאומת של האובייקטים. בחרו רק בעצמים המסווגים כמקרו-חסרי חוליות כדי לתאר את התפלגות הגודל האינדיבידואלי שלהם בדגימה.
- גיוון בגדלים
- חשב את מגוון הגודל (Sd) בעקבות Quintana et al. (2008)8, כמו ב- García-Comas et al. (2016)35:
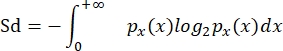
כאשר p x(x) היא פונקציית צפיפות ההסתברות של גודל x, ו-x מייצג את log2 (אליפסה). מדד זה הוא, אם כן, מדד הגיוון של שאנון המותאם למדד רציף, כגון התפלגות הגודל האינדיבידואלי בקהילה.
- חשב את מגוון הגודל (Sd) בעקבות Quintana et al. (2008)8, כמו ב- García-Comas et al. (2016)35:
- ספקטרום גודל ביו-נפח מנורמל (NBSS)
- הגדר את מחלקות הגודל של ה- NBSS, קבע את הגבול התחתון של הספקטרום כקוונטיל 0.01 של התפלגות גודל המקרו-חסרי חוליות בדגימות ויצירת מחלקות גודל על ידי קנה מידה גיאומטרי של בסיס 2 עד להכללת האורגניזם הגדול ביותר בדגימות.
הערה: רוחב מחלקת הגודל גדל עם הגודל כדי לקחת בחשבון את השונות הגדולה יותר המשויכת לגדלים גדולים יותר. ב-NBSS של קהילות המקרו-חסרי חוליות שנותחו כאן היו 14 כיתות גודל (טבלה 1). - השג את ה- biovolume המנורמל על ידי חלוקת ה- biovolume הכולל בכל מחלקת גודל ברוחב מחלקת הגודל.
- הגדר את מחלקות הגודל של ה- NBSS, קבע את הגבול התחתון של הספקטרום כקוונטיל 0.01 של התפלגות גודל המקרו-חסרי חוליות בדגימות ויצירת מחלקות גודל על ידי קנה מידה גיאומטרי של בסיס 2 עד להכללת האורגניזם הגדול ביותר בדגימות.
- שיפוע ספקטרום הגודל
- חישוב השיפוע הליניארי של NBSS.
הערה: השיפוע (μ) מחושב על סמך הקשר בין היומן 2 (נקודת אמצע של מחלקת גודל) לבין היומן2 (ביומסה מנורמלת) במחלקות הגודל הגדולות מהמצב, תוך התעלמות מכל אלה הריקות (במחקר זה, מחלקות הגודל מ-3 עד 14).
- חישוב השיפוע הליניארי של NBSS.
| מגבלות גודל מחלקה (מ"מ3) | גודל מעמד נקודת אמצע (מ"מ3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
טבלה 1: מחלקות גודל של ספקטרום גודל הביומסה המנורמל (NBSS). הטבלה מציגה גם את מגבלות מחלקת הגודל 15 ואת נקודות האמצע של מחלקת הגודל של האורגניזמים.
תוצאות
רכישת תמונות דיגיטליות של דגימות מקרו-חסרי חוליות
ניואנסים בסריקה: תצהיר אתנול במגש הסריקה
בעת בדיקת המערכת עבור מקרו-חסרי חוליות, מספר סריקות היו באיכות ירודה. אזור רווי כהה ברקע מנע עיבוד תקין של התמונה ומדידת הגדלים האינדיבידואליים של המקרו-חסרי חוליות (
Discussion
ההתאמה של המתודולוגיה שתוארה על ידי Gorsky et al. 2010 עבור מקרו-חסרי חוליות של נהרות מאפשרת דיוק סיווג גבוה בהערכת מבנה גודל הקהילה במקרו-חסרי חוליות של מים מתוקים. התוצאות מצביעות על כך שהפרוטוקול יכול לקצר את הזמן להערכת מבנה הגודל הבודד במדגם לכשעה. לפיכך, הפרוטוקול המוצע נועד לקדם את השימוש ה...
Disclosures
המחברים מצהירים שאין אינטרסים מתחרים פוטנציאליים.
Acknowledgements
עבודה זו נתמכה על ידי משרד המדע, החדשנות והאוניברסיטאות הספרדי (מענק מספר RTI2018-095363-B-I00). אנו מודים לחברי CERM-UVic-UCC אליה ברטקסה, אנה קוסטרוסה, לאיה חימנס, מריה איזבל גונזלס, מרתה יוטגלר, פרנצ'סק לאך ונוריה סלארס על עבודתם בדגימת שדה מקרו-חסרי חוליות ומיון מעבדה ולדוד אלבסה על שיתוף הפעולה בסריקת הדגימות. לבסוף אנו מודים לג'וזפ מריה גילי ולמכון Ciències del Mar (ICM-CSIC) על השימוש במתקני המעבדה ובמכשיר הסורק.
Materials
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
References
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved