このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
河川大型無脊椎動物の群集サイズ構造を決定するための自動画像処理
要約
この記事は、半自動イメージング手順を使用して、底生河川大型無脊椎動物に対応するデジタル化されたオブジェクトをスキャン、検出、分類、および識別するための適応プロトコルの作成に基づいています。この手順により、大型無脊椎動物群集の個々のサイズ分布およびサイズメトリックを約1時間で取得できます。
要約
体の大きさは、自然界における摂動の影響を評価するための生物指標として使用できる重要な機能的形質です。群集サイズ構造は、分類群や生態系全体の人為的摂動を含む生物的および非生物的勾配に応答します。しかし、底生大型無脊椎動物などの小体生物(例えば、>500μmから数センチメートルの長さ)の手動測定には時間がかかります。群集サイズの推定を促進するために、ここでは、淡水生態系の生態学的状態を評価するために最も一般的に使用される生物指標の1つである、保存された河川大型無脊椎動物の個々の体のサイズを半自動で測定するプロトコルを開発しました。このプロトコルは、水サンプル用に設計されたスキャンシステムで海洋中動物プランクトンをスキャンするために開発された既存の方法論から適応されています。プロトコルは3つの主要なステップで構成されています:(1)河川大型無脊椎動物のサブサンプル(細かいサンプルサイズと粗いサンプルサイズの割合)をスキャンし、デジタル化された画像を処理して、各画像で検出された各オブジェクトを個別化します。(2)人工知能を通じて学習セットを作成、評価、および検証して、スキャンされたサンプルの残骸やアーティファクトから大型無脊椎動物の個々の画像を半自動的に分離する。(3)大型無脊椎動物群集のサイズ構造を描く。プロトコルに加えて、この作業にはキャリブレーション結果が含まれ、手順を大型無脊椎動物のサンプルに適合させ、さらなる改善を検討するためのいくつかの課題と推奨事項が列挙されています。全体として、結果は、河川大型無脊椎動物の自動体サイズ測定のための提示されたスキャンシステムの使用を支持し、それらのサイズスペクトルの描写が淡水生態系の迅速な生物評価のための貴重なツールであることを示唆しています。
概要
底生大型無脊椎動物は、水域の生態学的状態を決定するための生物指標として広く使用されています1。大型無脊椎動物のコミュニティを説明するためのほとんどの指標は、分類学的指標に焦点を当てています。ただし、体のサイズを統合する新しいバイオアセスメントツールは、分類学的アプローチの代替的または補完的な視点を提供することが奨励されています2,3。
体のサイズは、代謝、成長、呼吸、運動などの他の重要な特性に関連するメタ特性と見なされます4。さらに、体の大きさは栄養の位置と相互作用を決定することができます5。個々の体のサイズとコミュニティのサイズクラスによる正規化バイオマス(または存在量)との関係は、サイズスペクトル6 として定義され、対数スケール7で個々のサイズが増加するにつれて正規化バイオマスの線形減少の一般的なパターンに従います。この線形関係の傾きは理論的に広く研究されており、生態系を横断する実証研究はそれを群集サイズ構造の生態学的指標として使用してきました4。生物多様性生態系機能研究において首尾よく使用されている群集サイズ構造の別の合成指標は、個々のサイズ分布に基づいて計算されるサイズスペクトルまたはその類似体のサイズクラスのシャノン指数として表される群集サイズの多様性である8。
淡水生態系では、さまざまな動物群のサイズ構造が運動失調指標として使用され、環境勾配9,10,11および人為的摂動12,13,14,15,16に対する生物群集の応答を評価します。大型無脊椎動物も例外ではなく、そのサイズ構造は環境変化17,18や、鉱業19、土地利用20、窒素(N)とリン(P)濃縮20,21,22などの人為的摂動にも反応します。ただし、コミュニティのサイズ構造を説明するために数百人の個人を測定することは、退屈で時間のかかる作業であり、時間がないため、ラボでの日常的な測定として避けられることがよくあります。したがって、標本を分類および測定するためのいくつかの半自動または自動イメージング方法が開発されている23、24、25、26。ただし、これらの方法のほとんどは、生物の個々のサイズよりも分類学的分類に焦点を当てており、あらゆる種類の大型無脊椎動物に使用する準備ができているわけではありません。海洋プランクトン生態学では、動物プランクトン群集のサイズと分類学的構成を決定するために、走査画像解析システムが広く使用されています27,28,29,30,31。この装置は、世界中のいくつかの海洋研究所で見つけることができ、保存された動物プランクトンサンプルをスキャンして、サンプル全体の高解像度デジタル画像を取得するために使用されます。本プロトコルは、新しい装置の作成に投資することなく、河川における大型無脊椎動物群集サイズスペクトルを迅速に自動的に推定するために、この装置の使用を適応させる。
このプロトコルは、サンプルをスキャンし、画像全体を処理して、サンプル内のオブジェクトの単一の画像(つまり、ビネット)を自動的に取得することで構成されます。形状、サイズ、グレーレベルの特徴のいくつかの測定値が各オブジェクトを特徴付け、オブジェクトをカテゴリに自動的に分類し、専門家によって検証されます。各生物の個体サイズは、ピクセル単位で測定された生物の面積から導出される楕円体バイオボリューム(mm3)を使用して計算されます。これにより、試料のサイズスペクトルを迅速に得ることができる。私たちの知る限り、このスキャンイメージングシステムは中部動物プランクトンサンプルの処理にのみ使用されていますが、このデバイスは淡水底生大型無脊椎動物での作業を可能にする可能性があります。
したがって、この研究の全体的な目標は、海洋中動物プランクトン27,32,33で以前に使用されていた既存のプロトコルを適応させることにより、保存された河川大型無脊椎動物の個体サイズを迅速に取得する方法を導入することです。この手順は、スキャン装置で動作する半自動アプローチを使用して水サンプルをスキャンし、3つのオープンソフトウェアを使用してスキャンされた画像を処理することで構成されます。群集サイズ構造および関連するサイズメトリクスを自動的に取得するために、デジタル化された河川大型無脊椎動物をスキャン、検出、および識別するための適応されたプロトコルが本明細書に提示される。効率を高めるための手順とガイドラインの評価は、北東(NE)イベリア半島の3つの流域(テル、セグレエブレ、ベソス)から収集された河川大型無脊椎動物サンプルの42のスキャン画像に基づいて提示されます。
サンプルは、スペイン政府からの手頃な価格の河川の底生河川大型無脊椎動物の野外サンプリングと実験室分析のプロトコルに従って、100 mの川域で収集されました34。サンプルは、マルチハビタット調査の後、サーバーサンプラー(フレーム:0.3 m x 0.3 m、メッシュ:250 μm)で収集されました。実験室では、サンプルを洗浄し、5 mmと500 μmのメッシュでふるいにかけ、粗いサブサンプル(5 mmメッシュ)と細かいサブサンプル(500 μmメッシュ)の2つのサブサンプルを取得し、別々のバイアルに保存し、70%エタノールで保存しました。サンプルを2つのサイズ画分に分離すると、大きな生物は小さな生物よりも希少で少ないため、コミュニティのサイズ構造をより適切に推定できます。それ以外の場合、スキャンされたサンプルは、大きなサイズの割合の偏った表現を持ちます。
プロトコル
注:ここで説明するプロトコルは、海洋メソ動物プランクトンのためにGorskyらによって開発されたシステムに基づいています27。スキャナー(ZooSCAN)、スキャンソフトウェア(VueScan 9x64 [9.5.09])、画像処理ソフトウェア(Zooprocess、ImageJ)、および自動識別ソフトウェア(プランクトン識別子)の手順の具体的な説明は、以前の参考文献32,33にあります。中部動物プランクトンに対する底生大型無脊椎動物のサイズを最適に調整するには、元のプロトコル32,33に従ってプロジェクトを作成したら、構成ファイルで最小サイズ(minsizeesd_mm)のパラメーターを0.3 mmに変更し、最大サイズ(maxsizeesd_mm)のパラメーターを100 mmに変更します。プロトコルに従うのを助けるために、これは作業チャートに要約されています(図1)。作成されたプロジェクトは、コンピューターの C フォルダーに格納され、PID_process、Zooscan_back、Zooscan_check、Zooscan_config、Zooscan_meta、Zooscan_results、および Zooscan_scan フォルダーに整理されます。各フォルダーは、プロトコルの次の手順でさまざまなソフトウェアアプリケーションが使用するいくつかのサブフォルダーで構成されています。
1. 大型無脊椎動物試料のデジタル画像取得
- ブランクのスキャンと処理
注:スキャンする前に毎日2つの空白の画像を作成して、同じ日にスキャンした画像を処理しながらバックグラウンドスキャンを抽出します。- スキャナーの電源を入れ、デュアルポジションのライトをオンにして、上と下から白色光を投影します。
注:メソ動物プランクトンサンプルをスキャンするときは、上向きの光の方向が使用されますが、大型無脊椎動物はより不透明であるため、光を二重の位置に切り替えることをお勧めします。 - スキャントレイを清掃し、水道水ですすいでください。
- ガラスが覆われるまで、室温(RT)で保存した水道水110 mLをスキャントレイに注ぎます。大きなフレーム(24.5 cm x 15.8 cm)をスキャントレイの正しい位置(スキャントレイの左上の角)に置き、フレームの段差が覆われるまで水道水を入れて、スキャン画像が変化するメニスカス効果を回避します。スキャナーの蓋を閉じます。
注意: 結露や気泡の形成を避けるために、RTで水を使用してください。光の反射を避けるために、マークや液滴のないフレームを清掃してください。 - 画像処理ソフトウェアに移動し、作業プロジェクトを選択して、[ 背景画像のスキャン(変換)]をクリックします。
- スキャンソフトウェアに移動し、[プレビュー]をクリックします。スキャンした画像をプレビューし、線や斑点がないことを確認し、少なくとも30秒待ってから別のスキャンを開始してください。[スキャン]をクリックし、2回目のスキャンの前に指示ウィンドウで[OK]を押して、スキャンソフトウェアから画像処理ソフトウェアにデータを送信します。
メモ: 2 回スキャンして、空白を構成する 2 つのバックグラウンドスキャンを取得します。このステップは、サンプル処理を開始する前に毎日1回実行され、画像は Zooscan_backフォルダーに保存されます。 - スキャンが終了したら、スキャンソフトウェアを閉じます。
- スキャナーの電源を入れ、デュアルポジションのライトをオンにして、上と下から白色光を投影します。
- サンプル調製とスキャン
注意: エタノールは可燃性の液体であり、深刻な目の損傷/刺激を引き起こす可能性があります。- サンプル メタデータを入力します。画像処理ソフトウェアに移動し、[ サンプルメタデータの入力]を選択します。サンプル ID を入力し、[ OK] をクリックして、メタデータを入力します。
注:メタファイルは中動物プランクトンサンプル用に特別に作成されているため、底生大型無脊椎動物のサンプリング方法に適合しませんが、スキャンの前にファイルのすべてのフィールドに入力する必要があり、そうしないとエラーフラグがポップアップ表示されます。 - ガラスが覆われるまで110 mLの70%エタノールをスキャントレイに注ぎ、大きなフレーム(24.5 cm x 15.8 cm)をスキャントレイの左上部分に置きます。
注:大型無脊椎動物はエタノールに保存されているため、水の代わりにエタノールを使用してください。水中では、スキャントレイに浮遊してドリフトし、鮮明な画像を妨げ、信頼性の高いサイズ測定を妨げます。エタノールは、結露や気泡の形成を避けるためにRTで保存する必要があります。 - 大型無脊椎動物のサンプルをフレームで縁取られたスキャントレイに注ぎ、必要に応じてフレームステップをさらにエタノールで覆います。
注:生物が浮いたり漂ったりするのを防ぐために、エタノールを追加しすぎないようにしてください。 - フレーム領域全体でサンプルを均質化し、適切な画像処理のためにトレイの中央に最大の個体を配置し、木製の針を使用して浮遊生物を沈めます。
注:サブサンプルに数値的に1,000人を超える個人が含まれている場合は、サブサンプルを2つ以上のフラクションに分割して、スキャンされた画像内の生物との接触を最小限に抑え、フラクションを個別にスキャンします。 - 木の針を使って、触れている生物とフレームの端に触れている生物を分離します。
注意: この手順には5〜20分かかります。触れる生物は、ソフトウェアによって単一のオブジェクトと見なされます。したがって、そのような場合、計算された個体サイズは実際の単一生物に対応せず、群集サイズ構造の推定値に偏りが生じる可能性があります。画像処理ソフトウェアで画像を編集して分離する可能性がありますが、この追加のステップには少なくとも1.5時間の再処理が含まれます。したがって、手動で分離することを強くお勧めします。 - サンプルをスキャンするには、スキャナーの蓋を閉じ、画像処理ソフトウェアに移動し、作業プロジェクトを選択して、 Zooscanでサンプルをスキャン(アーカイブ用、プロセスなし)をクリックします。
- サンプルを選択し、指示に従います。
- スキャンソフトウェアに移動し、[ プレビュー]をクリックします。スキャンした画像をプレビューし、線や斑点がないことを確認し、少なくとも30秒待ってから別のスキャンを開始してください。
- 少なくとも30秒後、スキャンソフトウェアの[ スキャン ]ボタンをクリックします。
注意: スキャンソフトウェアでスキャンを押した後、画像処理ソフトウェアでOKを押します。コンピュータのキーボードのキーを押さず、スキャン中のスキャンの振動を避けてください。Zooscan_scan > _rawフォルダには3つのファイルが生成されます:(i)タグ付き画像ファイル形式(.tif)(16ビット)。(ii)スキャンパラメータに関する情報を記録するLOG(.txt)という名前の標準テキスト文書。(iii)サンプリング方法に関する情報を含むMETA(.txt)という名前の標準テキストドキュメント。 - 未加工スキャンが正しいことを確認します。
メモ: スキャンに薄い縞模様やその他の目に見える問題がある場合は、次の手順で問題を回避するためにスキャンを繰り返すことを検討してください。
- サンプル メタデータを入力します。画像処理ソフトウェアに移動し、[ サンプルメタデータの入力]を選択します。サンプル ID を入力し、[ OK] をクリックして、メタデータを入力します。
- サンプル回収
- フレームを取り外し、70%エタノールで満たされたスクイーズボトルを使用してスキャントレイの上ですすぎ、付着した大型無脊椎動物を回収します。
- スキャナーの上部を持ち上げて、トレイからすべての生物とエタノールをスキャン取り出し漏斗を通ってビーカーに入れます。スキャナーの上部を持ち上げたまま、スクイーズボトルでトレイをすすぎ、残っている生物を一掃します。
- 試料とエタノールをビーカーから500μmメッシュに通して無脊椎動物をメッシュに保持し、70%エタノールの入ったバイアルに戻します。
- すべての検体がバイアルに回収されたら、トレイを水道水で洗浄します。
注意: サンプル間の水道水でトレイを洗浄して、画像処理を変更するエタノール沈殿を最小限に抑えます。エタノールの使用に関連する潜在的な損傷を避けるために、フレームを水道水ですすいでください。一日の終わりに、水道水を使用してトレイを掃除し、傷を避けるために紙でやさしく乾かします。
- 画像処理
- 画像処理ソフトウェアに行き、 CONVERT & PROCESS Images and Organisms in Batch Mode を選択してから、Convert AND Process Image AND Particle (Image in RAW Folder) を選択します。デフォルト設定をそのままにして、[ OK]をクリックします。 プロセスの最後に通常の終了 が表示されます。
注:PIDファイルと、スキャンされた画像(共同写真グループファイル[.jpg])で検出されたすべてのオブジェクトに対応するビネットが Zooscan_scan > _workフォルダーに作成されます。PIDファイルは、すべてのメタデータ(メタファイル)、ログファイルに関連付けられた技術データ、および画像で検出されたすべてのオブジェクトの36の測定変数を含むテーブルを格納する単一のファイルです。測定された変数は、グレーレベル、フラクタル次元、形状、およびサイズのさまざまな推定値に対応します。サイズの推定に使用できる変数は、オブジェクトに等しい面積を持つ楕円の面積と長軸と短軸です(プロトコルのセクション3を参照)。処理時間は画像密度とコンピュータ特性に依存し、次のサンプルを回収して準備しながらサンプル間で起動できます。それ以外の場合は、夜間にバッチモードで毎日スキャンしたサンプルの処理を開始し、翌朝に適切な画像処理を確認することをお勧めします。 - 画像処理ソフトを使用するか、 Zooscan_scan > _workにあるマスク画像(msk1.gifで終端)を確認して、処理画像の背景がサンプル画像から適切に差し引かれているか確認してください。背景に飽和領域または多くのドットが含まれている場合は、高品質の画像を確保するためにスキャンを繰り返すことを検討してください。
注意: バックグラウンドの飽和領域を避けるために、エタノールでスキャンするたびにスキャントレイを水道水ですすぐ必要があります。また、(1)スキャンされた個人の数を減らすことも重要です(サンプルを分割し、異なる折り目でスキャンすることにより)。(2)大きな生物がスキャントレイの中央に配置されていることを確認します。(3)清潔でろ過されたエタノールを使用する。(4)サンプルの汚れを減らします。(5)スキャン用のエタノールの量が適切であることを確認してください。(6)サンプルのプレビューとスキャンの間の遅延が少なくとも30秒であることを確認します。
- 画像処理ソフトウェアに行き、 CONVERT & PROCESS Images and Organisms in Batch Mode を選択してから、Convert AND Process Image AND Particle (Image in RAW Folder) を選択します。デフォルト設定をそのままにして、[ OK]をクリックします。 プロセスの最後に通常の終了 が表示されます。
- 触れる生物の分離
注:接触生物が複数のビネットがある場合、群集サイズ構造を適切に推定するために、接触生物の画像を他の生物および/または繊維/破片から分離する必要があります。- 画像処理ソフトウェアに移動して、複数のオブジェクトを持つビネットを検出します。 [ビネットを使用した分離 ]を選択し、[ OK]を押します。構成選択ウィンドウで、デフォルト設定をそのまま使用し、[ OK]をクリックします。
- [ビネットからの分離] ウィンドウで、既定の設定をそのまま使用し、さらに [ビネットにアウトラインを追加] を選択してから、編集するサンプルを選択します。
- マウスで線を引くことで、ポップアップする各ビネットの接触生物を分離します(ロールボタンを押して描画します)。ビネットでの分離が完了したら、ウィンドウの右上隅にある [X ] ボタンをクリックし、[ はい ] を押して次の分離を処理します。 NO を押して終了し、変更を保存します。プロセスの最後に、すべてが正しい場合は NORMAL END が表示されます。
- 色分解後、画像を再処理して、更新されたオブジェクトデータを取得します。画像処理ソフトウェアに移動し、[ 処理(変換済み)画像(プロセス1)]をクリックして、[ WORKサブフォルダー内の処理済み画像からパーティクルを再処理]を選択します。サンプルを選択し、[ 単一イメージ プロセス ] ウィンドウで既定の設定をそのまま使用し、[ 分離マスクの操作 (CREATE-MODIFY-INCLUDE)] をオンにして、[ OK] をクリックします。プロセスの最後に、すべてが正しい場合は NORMAL END が表示されます。
- 「分離制御」ウィンドウで、「OK」を押して、処理前の輪郭を含む画像を保存します。以前のイメージが存在する場合は、置き換えられます。
- 「分離 コントロール マスク」ウィンドウで、必要に応じて 「編集 」を選択し、マウスを使用してマスクに分離線を追加し、「ビネットを使用した分離」ステップで以前に出現しなかった接触生物を分離します。終了したら、プロセスを終了し、[ 分離マスク制御 ]ウィンドウで[ はい ]を選択してマスクを受け入れます。プロセスの最後に、すべてが正しい場合は NORMAL END が表示されます。
注:分離マスクを使用したサンプルの再処理には時間がかかります(サンプルあたり1.5時間以上かかる場合があります)。この追加のステップを回避するために、ステップ1.2.5で必要な時間を捧げることが望ましいです。
2.オブジェクトの自動認識
注意: 検出されたオブジェクトのアイデンティティを自動的に予測する学習セットを作成し、サンプル内の破片から生物を分離します。
- 学習セットの作成
- 学習セットのカテゴリの作成に使用される画像と、画像に関連付けられている .pid ファイルを Zooscan_scan > _work から PID_process > Unsorted_vignettes_pid にコピーします。
注:分類群の多様性が高く、サンプリングサイトやサンプリングシーズンが異なるサンプルのサブセットを選択して、サンプル内の生物の代表性を最大限に高めます。 - PID_process >ラーニングセットフォルダに、新しいラーニングセットの名前(yyyymmdd_raw_LS)のサブフォルダを作成し、その中に、ラーニングセットの各カテゴリ(マクロ無脊椎動物、破片、その他の無脊椎動物)に対応するサブフォルダを作成します。
注:河川大型無脊椎動物サンプルの群集サイズ構造を効率的に取得するには、大型無脊椎動物、その他の無脊椎動物、破片の3つのカテゴリのみに基づく学習セットを使用することをお勧めします。この学習セットは基本的に、生物に対応する物体のビネットを破片(例えば、繊維、粒子、または糸状藻類)に対応するものから分離する。 - 画像処理ソフトウェア(詳細モードのみ)に移動し、 プランクトン識別子(トレーニング用の未ソートビネット)にビネットを抽出を選択します。デフォルトのオプションをそのまま使用し、[ アウトラインの追加 ] チェックボックスをオンにします。
- 自動識別ソフトウェアに移動し、[学習]をクリックし、新しい 学習セット用に作成されたサブフォルダー PID_process > Learning_set から選択して(ステップ2.1.2)、[ OK]を押します。
- 開いているウィンドウの左側のセクション(並べ替えられていない親指)で、フォルダーを選択します 未並べ替え vignettes_pid。ビネットを選択し、ソートされていないサムから右側のセクションの対応するカテゴリのフォルダ(ソートされたサム)にマウスでドラッグして、各オブジェクトを定義されたカテゴリに分類します。移動したビネットは赤いXでマークされます。
注:ソートされたサムフォルダにサブフォルダを作成して手動でカテゴリを定義するか、ソフトウェアのフォルダアイコンをクリックしてサブフォルダを作成します。同時に50を超えるビネットを動かさないでください。 - 選択したオブジェクト(カテゴリごとに約300オブジェクト)ですべてのカテゴリが完成したら、[ 学習ファイルの作成 ]をクリックして、目的の名前で保存します。
注: 学習セットは、プロジェクトの PID_process > 学習セット フォルダーに .pid ファイルとして保存されます。異なるレベルのカテゴリ(粗い形式から細かい形式まで)で、各カテゴリ内のオブジェクト数のバランスが異なる複数の学習セットを作成してテストすることをお勧めします。カテゴリ数が少なく、カテゴリごとに少なくとも50個のオブジェクトを含む粗い学習セットから始めて、各カテゴリのオブジェクト数を増やすか、より細かい学習セットを作成します。カテゴリは、サンプルのセットにおけるその変動性を表す必要があります。
- 学習セットのカテゴリの作成に使用される画像と、画像に関連付けられている .pid ファイルを Zooscan_scan > _work から PID_process > Unsorted_vignettes_pid にコピーします。
- 学習セットの評価
注:自動識別ソフトウェアでランダムフォレスト法を使用して2つのフォールドと5つの試行で交差検証を実行し、結果のオブジェクトの分類の混同行列を取得します。- 自動分類ソフトウェアに移動し、[ データ分析]をクリックします。
- [学習ファイル の選択]で、[学習セット]から作成した 学習セットファイルを選択しますPID_process >。
- [ 方法の選択] で、[ ランダムフォレストの交差検証 ] 方法を選択します。 [元の変数] で、位置変数 (X、Y、XM、YM、BX、BY、および [高さ]) のチェックを外します。 [カスタマイズされた変数] で、[ ESD のみ] にチェックマークを付けます。
注:この方法では、学習セットのランダムな部分の1つを使用して他の部分(2倍)を認識し、統計的に堅牢であることを確認するためにこれを5回繰り返します。 - [分析の開始] をクリックし、結果を [PID_process >予測] フォルダーにAnalysis_name.txtとして保存します。分析が正常に完了したら、データ分析を終了します。
- PID_process >予測フォルダーに移動し、クロス検証ファイルをクリックします。ウィンドウがポップアップし、真の分類(行)と自動分類(列)の混同行列が表示されます。
注:再現率は、自動的に十分に認識されたグループに属する生物の割合であり、1精度は、アルゴリズムによって認識されないグループ(グループ内の汚染)として分類された生物の割合です。再現率は70%を超え、汚染(1精度)は20%未満である必要があります。 - 複数の学習セットが作成され、それぞれの再現率と1精度を取得する必要がある場合は、手順2.1〜2.5を繰り返します。
注:複数の学習セットが作成されている場合は、関心のあるグループ(つまり、大型無脊椎動物)の再現率(良好な認識)と精度(低汚染)を持つものを選択して、次のステップでサンプルのセットの自動予測をテストします。
- 大型無脊椎動物の同定予測
注:選択した学習セットを使用して、ランダムフォレストアルゴリズムを備えた自動識別ソフトウェアを使用して、サンプルのサブセット内のすべてのオブジェクトのIDを予測します。- 自動識別ソフトウェアに移動し、[ データ分析]をクリックします。
- [ 学習ファイルの選択] で、予測に使用する必要がある学習 セットから学習 セット ファイルからPID_process >を選択します。
- [ サンプル ファイルの選択] で、 PID_results フォルダーから予測するサンプル (PID ファイル) を選択します。
注: メモリの問題に関連するエラーを回避するために、最大 20 個の .pid ファイルを同時に処理します。同時に処理される.pidファイルが多すぎると、処理は正しい終了を示しますが、うまく処理されない可能性があり、画像処理ソフトウェアで処理すると次の手順でエラーが発生する場合があります。 - [方法の選択] で、[ランダム フォレスト] 方法を選択します。各サンプルの詳細な結果を保存するにチェックマークを付けます。元の変数で、位置変数(X、Y、XM、YM、BX、BY、および高さ)のチェックを外します。[カスタマイズされた変数] で、[ESD のみ] にチェックマークを付けます。
- [分析の開始] をクリックし、結果を [PID_process >予測] フォルダーにAnalysis_name.txtとして保存します。
- 手動検証
注: エキスパートは、前の手順の予測を手動で検証して、誤って分類されたオブジェクトを正しいカテゴリに再分類します。- 検証する Analysis_sample_dat1.txt ファイルを PID_process > 予測 フォルダーから PID_process > Pid_results フォルダーにコピーします。
- 画像処理ソフトウェアに移動し、 予測または検証に従ってフォルダ内のビネットを抽出を選択します。次に、[ "pid_results" フォルダーの予測ファイルを使用する] を選択します。デフォルト設定のままにして、[ OK]を押します。
- ソフトウェアは、PID_process >ソートされたビネットフォルダに予測されたオブジェクトを含むsample_yyyymmdd_hhmm_to_validateというフォルダを作成します。
- PID_process > [並べ替えられたビネット] フォルダーに移動し、フォルダー sample_yyyymmdd_ hhmm_to_validateコピーします。検証_toフォルダー名を_validatedに置き換えます。
- 自動分類を手動で検証するには、フォルダー sample_yyyymmdd_ hhmm_validatedを開き、各サブフォルダー (カテゴリ) のすべてのビネットを確認して、誤って分類されたオブジェクトがあるかどうかを特定します。1つのオブジェクトが誤って分類されている場合は、マウスを使用してビネットを正しいフォルダー(カテゴリ)にドラッグします。
- 画像処理ソフトウェアに移動し、 ソートされたビネットからLOAD識別を選択します。デフォルト設定をそのまま使用し、処理する yyyymmdd_hhmm_name_validatedを選択します。
- PID_process > Pid_results > Dat1_validated に移動すると、Id_from_sorted_vignettes_yyyymmdd_hhmm.txt という名前のファイルと、検証済みのサンプル (sample_tot_1_dat1.txt) ごとに 1 つの .txt ファイルが作成されます。
注: これらの.txtファイルには、各オブジェクトの専門家分類 (つまり、検証済みの分類) を指定する pred_valid_Id_yyyymmdd_hhmm と呼ばれる予測を表示する新しい列が含まれています。新しいカテゴリ(たとえば、より細かい分類カテゴリ)は、検証中にこの時点で作成できます。ただし、元のカテゴリの名前は新しい名前 (macroinvertebrate_chironomidae など) のままにします。これにより、再現率と精度を計算するときに元のカテゴリをたどり、すべての大型無脊椎動物を簡単にグループ化して、コミュニティサイズ構造パラメータ(つまり、サイズスペクトルとサイズの多様性)を計算することができます。テキストファイルは、個々の体サイズの尺度として各生物の楕円体体積を取得するために使用される短軸と長軸を含む、各オブジェクトに関連付けられたデータを提供します。さらに、表の最後の2つの列には、各オブジェクト(行)の予測および検証されたカテゴリが含まれており、サンプルのサブセットに対する学習セットの再現率と精度をカテゴリ別に計算できます。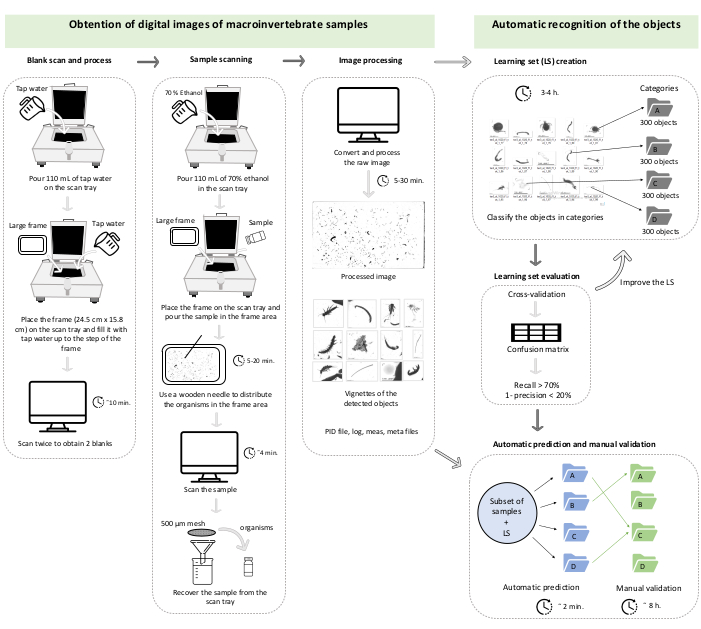
図1:プロトコルのセクション1とセクション2を表す作業チャート。時間は例示的なものであり、コンピューター、処理するビネットの量、および学習セットのカテゴリの数によって変わる可能性があります。このケースは、42のサブサンプルのセット(合計47,473ビネット)の3つのカテゴリの学習セットの検証に対応します。この図の拡大版を表示するには、ここをクリックしてください。
3. 個々のサイズ分布、サイズスペクトル、およびサイズメトリックの計算
注:このセクションに記載されている計算は、Matlabを使用して実行されました(補足ファイル1としてのスクリプトを参照)。
- 個々のサイズ分布
- Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt ファイルの最後の列には、検証済みのオブジェクトの分類が含まれています。大型無脊椎動物として分類されたオブジェクトのみを選択して、サンプル内の個々のサイズ分布を表します。
注:個々の体のサイズは、大型無脊椎動物生物の楕円体の体積に対応します。システムはピクセル単位で測定値を提供します。 - ベクトルを両方のスキャンのサイズ測定値と連結します。これは、各分数が異なるサブサンプリング指数を持つためです。連結する前に、対応するサブサンプルが分画された回数だけサイズベクトルを複製して、分画を修正します。
注:この手順は、スキャンがサンプルの一部(つまり、粗いまたは細かい)に対応する場合に必要です。 - 生物と同じピクセル面積を持つ扁平楕円体の長軸(M)軸と短軸(m)から楕円体体積を計算します。楕円体の体積を計算する前に、次の変換係数(cf)を使用して、長軸(M)軸と短軸(m)をピクセルからミリメートル(mm)に変換します。
1 ピクセル = 2,400 dpi
1 インチ = 25.4 mm
cf = 25.4/2400
楕円体体積(mm3単位の楕円体積)は、次のものに対応します。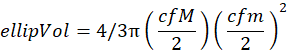
- 個々のサイズ分布の確率密度関数を対数2 スケールで表します。
- Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt ファイルの最後の列には、検証済みのオブジェクトの分類が含まれています。大型無脊椎動物として分類されたオブジェクトのみを選択して、サンプル内の個々のサイズ分布を表します。
- サイズの多様性
- García-Comas et al. (2016)35のように、Quintana et al. (2008)8に従ってサイズの多様性(Sd)を計算します。
ここで、p x(x) はサイズ x の確率密度関数であり、x は対数2 (ellipVol) を表します。したがって、この尺度は、コミュニティ内の個体サイズ分布などの継続的な尺度に適合したシャノン多様性指数です。
- García-Comas et al. (2016)35のように、Quintana et al. (2008)8に従ってサイズの多様性(Sd)を計算します。
- 正規化バイオボリュームサイズスペクトル(NBSS)
- NBSSのサイズクラスを定義し、スペクトルの下限をサンプル内の大型無脊椎動物のサイズ分布の0.01分位数として確立し、サンプル内の最大の生物が含まれるまで、ベース2の幾何学的スケールでサイズクラスを作成します。
注: サイズ クラスの幅は、サイズが大きいほど変動が大きくなるため、サイズとともに大きくなります。ここで分析された大型無脊椎動物群集のNBSSには、14のサイズクラスがありました(表1)。 - 各サイズクラスの総バイオボリュームをサイズクラス幅で割ることにより、正規化されたバイオボリュームを取得します。
- NBSSのサイズクラスを定義し、スペクトルの下限をサンプル内の大型無脊椎動物のサイズ分布の0.01分位数として確立し、サンプル内の最大の生物が含まれるまで、ベース2の幾何学的スケールでサイズクラスを作成します。
- サイズスペクトル勾配
- NBSSの線形勾配を計算します。
注:傾き(μ)は、モードよりも大きいサイズクラスのlog2 (サイズクラスの中間点)とlog2 (正規化バイオマス)の関係に基づいて計算され、空のクラス(この研究では、サイズクラス3から14)は無視されます。
- NBSSの線形勾配を計算します。
| サイズクラス制限(mm3) | サイズクラス中間点(mm3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
表1:正規化バイオマスサイズスペクトル(NBSS)のサイズクラス。 この表には、生物の15のサイズクラス制限とサイズクラスの中間点も示されています。
結果
大型無脊椎動物サンプルのデジタル画像の取得
スキャンニュアンス:スキャントレイへのエタノール堆積
大型無脊椎動物についてシステムをテストしている間、いくつかのスキャンは質が低かった。背景の暗い飽和領域は、画像の通常の処理と大型無脊椎動物の個々のサイズの測定を妨げました(図2)。背景の飽和領域または高度にピクセル...
ディスカッション
Gorsky et al. 2010によって記述された方法論を河川大型無脊椎動物に適応させることで、淡水大型無脊椎動物の群集サイズ構造を推定する際の高い分類精度が可能になります。結果は、プロトコルがサンプル中の個々のサイズ構造を推定する時間を約1時間に短縮できることを示唆しています。したがって、提案されたプロトコルは、淡水生態系における摂動の影響を評価するための高速で統合的...
開示事項
著者らは、潜在的な競合する利益を宣言していません。
謝辞
この研究は、スペイン科学イノベーション大学省(助成金番号RTI2018-095363-B-I00)の支援を受けました。CERM-UVic-UCCのメンバーであるエリア・ブレチャ、アンナ・コスタローザ、ライア・ヒメネス、マリア・イザベル・ゴンザレス、マルタ・ユトグラール、フランチェスク・ラック、ヌリア・セラレスには、大型無脊椎動物のフィールドサンプリングと実験室での選別に協力し、デビッド・アルベサにはサンプルスキャンに協力してもらいました。最後に、ジョセップ・マリア・ギリとマール科学研究所(ICM-CSIC)に、実験施設とスキャナー装置の使用に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
参考文献
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved