Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Traitement automatique d’images pour déterminer la structure granulométrique des macroinvertébrés riverains
Dans cet article
Résumé
L’article est basé sur la création d’un protocole adapté pour scanner, détecter, trier et identifier des objets numérisés correspondant à des macroinvertébrés benthiques de rivière à l’aide d’une procédure d’imagerie semi-automatique. Cette procédure permet l’acquisition des distributions de taille individuelles et des métriques de taille d’une communauté de macroinvertébrés en environ 1 h.
Résumé
La taille corporelle est un trait fonctionnel important qui peut être utilisé comme bioindicateur pour évaluer les impacts des perturbations dans les communautés naturelles. La structure de taille des communautés réagit aux gradients biotiques et abiotiques, y compris les perturbations anthropiques entre les taxons et les écosystèmes. Cependant, la mesure manuelle d’organismes de petite taille tels que les macroinvertébrés benthiques (par exemple, >500 μm à quelques centimètres de long) prend beaucoup de temps. Pour accélérer l’estimation de la structure de la taille des communautés, nous avons développé ici un protocole pour mesurer semi-automatiquement la taille corporelle individuelle des macroinvertébrés de rivière préservés, qui sont l’un des bioindicateurs les plus couramment utilisés pour évaluer l’état écologique des écosystèmes d’eau douce. Ce protocole est adapté d’une méthodologie existante développée pour scanner le mésozooplancton marin avec un système de balayage conçu pour les échantillons d’eau. Le protocole comprend trois étapes principales : (1) balayage de sous-échantillons (fractions fines et grossières) de macroinvertébrés de rivière et traitement des images numérisées pour individualiser chaque objet détecté dans chaque image; (2) créer, évaluer et valider un ensemble d’apprentissage grâce à l’intelligence artificielle pour séparer semi-automatiquement les images individuelles de macroinvertébrés des détritus et des artefacts dans les échantillons numérisés; et (3) la représentation de la structure granulométrique des communautés de macroinvertébrés. En plus du protocole, ce travail comprend les résultats de l’étalonnage et énumère plusieurs défis et recommandations pour adapter la procédure aux échantillons de macroinvertébrés et envisager d’autres améliorations. Dans l’ensemble, les résultats appuient l’utilisation du système de balayage présenté pour la mesure automatique de la taille corporelle des macroinvertébrés de rivière et suggèrent que la représentation de leur spectre de taille est un outil précieux pour la bioévaluation rapide des écosystèmes d’eau douce.
Introduction
Les macroinvertébrés benthiques sont largement utilisés comme bioindicateurs pour déterminer l’état écologique des masses d’eau1. La plupart des indices décrivant les communautés de macroinvertébrés se concentrent sur des mesures taxonomiques. Cependant, de nouveaux outils de bioévaluation qui intègrent la taille corporelle sont encouragés à fournir une perspective alternative ou complémentaire aux approches taxonomiques 2,3.
La taille corporelle est considérée comme un métatrait lié à d’autres traits vitaux tels que le métabolisme, la croissance, la respiration et le mouvement4. De plus, la taille du corps peut déterminer la position trophique et les interactions5. La relation entre la taille corporelle individuelle et la biomasse normalisée (ou l’abondance) par classe de taille dans une communauté est définie comme le spectre de taille6 et suit le schéma général d’une diminution linéaire de la biomasse normalisée à mesure que la taille individuelle augmente sur une échelle logarithmique7. La pente de cette relation linéaire a été largement étudiée théoriquement, et des études empiriques sur les écosystèmes l’ont utilisée comme indicateur écologique de la structure de taille de la communauté4. Un autre indicateur synthétique de la structure de taille des communautés qui a été utilisé avec succès dans les études sur la biodiversité et le fonctionnement des écosystèmes est la diversité de la taille des communautés, qui est représentée par l’indice de Shannon des classes de taille du spectre de taille ou son analogique, qui est calculé sur la base des distributions de tailleindividuelles 8.
Dans les écosystèmes d’eau douce, la structure granulométrique des différents groupes fauniques est utilisée comme indicateur ataxique pour évaluer la réponse des communautés biotiques aux gradients environnementaux 9,10,11 et aux perturbations anthropiques 12,13,14,15,16. Les macroinvertébrés ne font pas exception, et leur structure granulométrique répond également aux changements environnementaux17,18 et aux perturbations anthropiques, telles que l’exploitation minière 19, l’utilisation des terres 20 ou l’enrichissement en azote (N) et en phosphore (P) 20,21,22. Cependant, mesurer des centaines d’individus pour décrire la structure de taille de la communauté est une tâche fastidieuse et chronophage qui est souvent évitée en tant que mesure de routine dans les laboratoires en raison d’un manque de temps. Ainsi, plusieurs méthodes d’imagerie semi-automatiques ou automatiques pour classer et mesurer les échantillons ont été développées23,24,25,26. Cependant, la plupart de ces méthodes sont davantage axées sur la classification taxonomique que sur la taille individuelle des organismes et ne sont pas prêtes à être utilisées pour tous les types de macroinvertébrés. En écologie du plancton marin, un système d’analyse d’images à balayage a été largement utilisé pour déterminer la taille et la composition taxonomique des communautés de zooplancton 27,28,29,30,31. Cet instrument peut être trouvé dans plusieurs instituts marins à travers le monde, et il est utilisé pour scanner des échantillons de zooplancton préservés afin d’obtenir des images numériques haute résolution de l’échantillon entier. Le protocole actuel adapte l’utilisation de cet instrument pour estimer le spectre de taille des communautés de macroinvertébrés dans les rivières de manière automatique rapide sans investir dans la création d’un nouveau dispositif.
Le protocole consiste à scanner un échantillon et à traiter l’image entière pour obtenir automatiquement des images uniques (c.-à-d. des vignettes) des objets de l’échantillon. Plusieurs mesures de forme, de taille et de niveaux de gris caractérisent chaque objet et permettent la classification automatique des objets en catégories, qui sont ensuite validées par un expert. La taille individuelle de chaque organisme est calculée à l’aide du biovolume ellipsoïdal (mm3), qui est dérivé de la surface de l’organisme mesurée en pixels. Cela permet d’obtenir rapidement le spectre de taille de l’échantillon. À notre connaissance, ce système d’imagerie par balayage n’a été utilisé que pour traiter des échantillons de mésozooplancton, mais le dispositif pourrait potentiellement permettre de travailler avec des macroinvertébrés benthiques d’eau douce.
L’objectif global de cette étude est donc d’introduire une méthode permettant d’obtenir rapidement la taille individuelle des macroinvertébrés de rivière préservés en adaptant un protocole existant précédemment utilisé avec le mésozooplanctonmarin 27,32,33. La procédure consiste à utiliser une approche semi-automatique qui fonctionne avec un dispositif de balayage pour numériser des échantillons d’eau et trois logiciels ouverts pour traiter les images numérisées. Un protocole adapté pour scanner, détecter et identifier les macroinvertébrés de rivière numérisés afin d’acquérir automatiquement la structure de taille de la communauté et les mesures de taille connexes est présenté ici. L’évaluation de la procédure et des lignes directrices visant à améliorer l’efficacité est également présentée sur la base de 42 images scannées d’échantillons de macroinvertébrés fluviaux prélevés dans trois bassins du nord-est (NE) de la péninsule ibérique (Ter, Segre-Ebre et Besòs).
Les échantillons ont été prélevés sur des tronçons de rivière de 100 m conformément au protocole d’échantillonnage sur le terrain et d’analyse en laboratoire des macroinvertébrés benthiques dans les rivières guéables du gouvernement espagnol34. Les échantillons ont été prélevés avec un échantillonneur surber, châssis : 0,3 m x 0,3 m, maille : 250 μm) à la suite d’un relevé multi-habitats. En laboratoire, les échantillons ont été nettoyés et tamisés à travers un maillage de 5 mm et un maillage de 500 μm pour obtenir deux sous-échantillons: un sous-échantillon grossier (maille de 5 mm) et un sous-échantillon fin (maille de 500 μm), qui ont été stockés dans des flacons séparés et conservés dans de l’éthanol à 70%. La séparation de l’échantillon en deux fractions granulométriques permet une meilleure estimation de la structure de taille de la communauté, car les grands organismes sont plus rares et moins nombreux que les petits organismes. Sinon, l’échantillon scanné a une représentation biaisée de la fraction de grande taille.
Protocole
REMARQUE : Le protocole décrit ici est basé sur le système développé par Gorsky et al.27 pour le mésozooplancton marin. Une description spécifique des étapes du scanner (ZooSCAN), du logiciel de numérisation (VueScan 9x64 [9.5.09]), du logiciel de traitement d’images (Zooprocess, ImageJ) et du logiciel d’identification automatique (Plankton Identifier) se trouve dans les références précédentes32,33. Pour ajuster au mieux les tailles des macroinvertébrés benthiques par rapport au mésozooplancton, une fois le projet créé selon le protocole original32,33, changer le paramètre de taille minimale (minsizeesd_mm) à 0,3 mm et le paramètre de taille maximale (maxsizeesd_mm) à 100 mm dans le fichier de configuration. Pour aider à suivre le protocole, cela est résumé dans un tableau de travail (Figure 1). Le projet créé est stocké dans le dossier C de l’ordinateur et est organisé dans les dossiers suivants : PID_process, Zooscan_back, Zooscan_check, Zooscan_config, Zooscan_meta, Zooscan_results et Zooscan_scan. Chaque dossier est composé de plusieurs sous-dossiers que les différentes applications logicielles utilisent dans les étapes suivantes du protocole.
1. Acquisition d’images numériques pour des échantillons de macroinvertébrés
- Numérisation et traitement de l’espace vierge
REMARQUE: Créez deux images vierges par jour avant la numérisation pour extraire les numérisations d’arrière-plan tout en traitant les images numérisées le même jour.- Allumez le scanner et allumez la lumière en double position pour projeter la lumière blanche du haut et du bas.
REMARQUE: Lors de la numérisation des échantillons de mésozooplancton, la direction de la lumière vers le haut est utilisée, mais comme les macroinvertébrés sont plus opaques, il est recommandé de basculer la lumière dans une position double. - Nettoyez et rincez le plateau de numérisation avec de l’eau du robinet.
- Versez 110 ml d’eau du robinet stockée à température ambiante (RT) dans le bac de numérisation jusqu’à ce que le verre soit recouvert. Placez le grand cadre (24,5 cm x 15,8 cm) sur le plateau de numérisation dans la bonne position (avec le coin dans la partie supérieure gauche du bac de numérisation) et remplissez-le d’eau du robinet jusqu’à ce que la marche du cadre soit couverte pour éviter un effet ménisque, qui modifierait l’image numérisée. Fermez le couvercle du scanner.
REMARQUE: Utilisez de l’eau à RT pour éviter la condensation et la formation de bulles. Nettoyez le cadre sans marques ni gouttelettes pour éviter la réflexion de la lumière. - Accédez au logiciel de traitement d’image, sélectionnez le projet de travail et cliquez sur Numériser (convertir) l’image d’arrière-plan.
- Accédez au logiciel de numérisation et cliquez sur Aperçu. Assurez-vous de prévisualiser l’image numérisée, vérifiez qu’il n’y a pas de lignes ou de taches et attendez au moins 30 secondes avant de commencer une autre numérisation. Cliquez sur Scan et appuyez sur OK dans la fenêtre d’instructions avant la deuxième numérisation pour envoyer les données du logiciel de numérisation au logiciel de traitement d’image.
REMARQUE: Numérisez deux fois pour obtenir les deux analyses d’arrière-plan qui comprendront l’espace vierge. Cette étape est effectuée une fois par jour avant de commencer le traitement de l’échantillon, et les images sont stockées dans le dossier Zooscan_back. - Fermez le logiciel de numérisation une fois l’analyse terminée.
- Allumez le scanner et allumez la lumière en double position pour projeter la lumière blanche du haut et du bas.
- Préparation et numérisation des échantillons
ATTENTION : L’éthanol est un liquide inflammable qui peut causer de graves lésions ou irritations oculaires.- Renseignez l’exemple de métadonnées. Accédez au logiciel de traitement d’image et sélectionnez Remplir l’exemple de métadonnées. Entrez l’identité de l’exemple, cliquez sur OK et remplissez les métadonnées.
REMARQUE: Le métafichier est spécifiquement créé pour les échantillons de mésozooplancton, il ne correspond donc pas à la méthodologie d’échantillonnage des macroinvertébrés benthiques, mais tous les champs du fichier doivent être remplis avant l’analyse, sinon un indicateur d’erreur apparaîtra. - Versez 110 mL d’éthanol à 70 % dans le bac de numérisation jusqu’à ce que le verre soit recouvert et placez le grand cadre (24,5 cm x 15,8 cm) avec le coin situé dans la partie supérieure gauche du bac à scanner.
NOTE: Travaillez avec de l’éthanol au lieu de l’eau, car les macro-invertébrés sont conservés dans l’éthanol. Dans l’eau, ils flottent et dérivent dans le plateau de balayage, empêchant une image nette et, par conséquent, des mesures de taille fiables. L’éthanol doit être conservé à TA pour éviter la condensation et la formation de bulles. - Versez l’échantillon de macroinvertébrés dans le plateau de balayage bordé par le cadre et couvrez l’étape du cadre avec plus d’éthanol si nécessaire.
REMARQUE: Abstenez-vous d’ajouter trop d’éthanol pour éviter que les organismes ne flottent et ne dérivent. - Homogénéisez l’échantillon dans toute la zone du cadre, en plaçant les plus gros individus au centre du plateau pour un traitement approprié de l’image, et coulez les organismes flottants à l’aide d’une aiguille en bois.
REMARQUE : Si un sous-échantillon contient numériquement plus de 1 000 individus, divisez-le en deux fractions ou plus pour minimiser le contact avec les organismes dans l’image numérisée et numérisez les fractions séparément. - Séparez les organismes qui touchent et les organismes qui touchent les bords du cadre à l’aide de l’aiguille en bois.
REMARQUE: Cette étape nécessite 5-20 min. Le toucher des organismes est considéré comme un objet unique par le logiciel; Ainsi, dans ces cas, les tailles individuelles calculées ne correspondent pas à des organismes individuels réels et peuvent biaiser l’estimation de la structure granulométrique de la Communauté. Il y a la possibilité d’éditer l’image avec le logiciel de traitement d’image pour les séparer, mais cette étape supplémentaire implique au moins 1,5 h de retraitement; Ainsi, la séparation manuelle est fortement recommandée. - Pour numériser l’échantillon, fermez le couvercle du scanner, accédez au logiciel de traitement d’image, sélectionnez le projet de travail et cliquez sur SCAN Sample with Zooscan (For Archive, No Process).
- Sélectionnez l’échantillon et suivez les instructions.
- Accédez au logiciel de numérisation et cliquez sur Aperçu. Assurez-vous de prévisualiser l’image numérisée, vérifiez qu’il n’y a pas de lignes ou de taches et attendez au moins 30 secondes avant de commencer une autre numérisation.
- Après au moins 30 s, cliquez sur le bouton Numériser dans le logiciel de numérisation.
REMARQUE: Appuyez sur OK dans le logiciel de traitement d’image après avoir appuyé sur Numériser dans le logiciel de numérisation. N’appuyez sur aucune touche du clavier de l’ordinateur et évitez les vibrations de la numérisation pendant la numérisation. Trois fichiers sont générés dans le dossier Zooscan_scan > _raw : (i) un format de fichier image balisé (.tif) (16 bits) ; ii) un document texte normalisé appelé LOG (.txt) qui enregistre des informations sur les paramètres de numérisation; et iii) un document texte normalisé intitulé META (.txt) contenant des informations sur les méthodes d’échantillonnage. - Vérifiez que l’analyse brute est correcte.
REMARQUE: Si l’analyse présente des bandes claires ou d’autres problèmes visibles, envisagez de répéter l’analyse pour éviter les problèmes dans les étapes suivantes.
- Renseignez l’exemple de métadonnées. Accédez au logiciel de traitement d’image et sélectionnez Remplir l’exemple de métadonnées. Entrez l’identité de l’exemple, cliquez sur OK et remplissez les métadonnées.
- Récupération d’échantillons
- Retirez le cadre et rincez-le au-dessus du plateau de balayage à l’aide d’un flacon rempli d’éthanol à 70 % pour récupérer les macroinvertébrés attachés.
- Soulevez la partie supérieure du scanner pour récupérer tous les organismes et l’éthanol du plateau à travers l’entonnoir de récupération du scanner dans un bécher. Avec la partie supérieure du scanner encore soulevée, rincez le plateau avec le flacon de pressage pour balayer les organismes restants.
- Passer les échantillons et l’éthanol du bécher à travers un treillis de 500 μm pour retenir les invertébrés dans le maillage et les conserver dans un flacon contenant 70% d’éthanol.
- Une fois que tous les échantillons sont récupérés dans le flacon, nettoyez le plateau avec de l’eau du robinet.
REMARQUE: Lavez le plateau avec de l’eau du robinet entre les échantillons pour minimiser la précipitation de l’éthanol, ce qui modifie le traitement de l’image. Rincez le cadre à l’eau du robinet pour éviter les dommages potentiels liés à l’utilisation d’éthanol. À la fin de la journée, nettoyez le plateau à l’eau du robinet et séchez-le doucement avec du papier pour éviter les rayures.
- Traitement d'images
- Accédez au logiciel de traitement d’image et sélectionnez CONVERTIR et TRAITER les images et les organismes en mode batch, puis Convertir et traiter l’image ET les particules (image dans le dossier RAW). Conservez les paramètres par défaut et cliquez sur OK. NORMAL END apparaîtra à la fin du processus.
REMARQUE : Un fichier PID et les vignettes correspondant à tous les objets détectés dans l’image numérisée (dans un fichier Joint Photographic Group [.jpg]) seront créés dans le dossier Zooscan_scan > _work. Un fichier PID est un fichier unique qui stocke toutes les métadonnées (métafichier), les données techniques associées au fichier journal et un tableau avec 36 variables mesurées de tous les objets détectés dans l’image. Les variables mesurées correspondent à différentes estimations du niveau de gris, de la dimension fractale, de la forme et de la taille. Les variables qui peuvent être utilisées pour l’estimation de la taille sont l’aire et les axes majeur et mineur d’une ellipse avec une aire égale à l’objet (voir section 3 du protocole). Le temps de traitement dépend de la densité de l’image et des caractéristiques de l’ordinateur, et peut être lancé entre les échantillons lors de la récupération et de la préparation de l’échantillon suivant. Sinon, il est recommandé de lancer le traitement des échantillons numérisés chaque jour en mode batch pendant la nuit et de vérifier le bon traitement de l’image le lendemain matin. - Vérifiez si l’arrière-plan de l’image traitée est correctement soustrait de l’exemple d’image à l’aide du logiciel de traitement d’image ou en vérifiant les images de masque (terminées par msk1.gif) situées dans Zooscan_scan > _work. Si l’arrière-plan contient des zones saturées ou de nombreux points, envisagez de répéter l’analyse pour garantir des images de haute qualité.
REMARQUE: Pour éviter les zones saturées en arrière-plan, le plateau de numérisation doit être rincé à l’eau du robinet après chaque balayage avec de l’éthanol. Il est également important (1) de réduire le nombre d’individus scannés (en fractionnant l’échantillon et en scannant dans différents plis); (2) s’assurer que les gros organismes sont placés au centre du bac de balayage; 3° utiliser de l’éthanol propre et filtré; 4° réduire la saleté des échantillons; 5° s’assurer que le volume d’éthanol pour le balayage est adéquat; et (6) s’assurer que le délai entre la prévisualisation de l’échantillon et l’analyse est d’au moins 30 s.
- Accédez au logiciel de traitement d’image et sélectionnez CONVERTIR et TRAITER les images et les organismes en mode batch, puis Convertir et traiter l’image ET les particules (image dans le dossier RAW). Conservez les paramètres par défaut et cliquez sur OK. NORMAL END apparaîtra à la fin du processus.
- Séparation des organismes touchants
REMARQUE : Lorsqu’il y a plusieurs vignettes avec des organismes touchants, il est nécessaire de séparer les images des organismes touchants des autres organismes et/ou des fibres/débris pour assurer une estimation correcte de la structure de la taille de la communauté.- Accédez au logiciel de traitement d’image pour détecter les vignettes avec plusieurs objets. Sélectionnez SEPARATION Using Vignettes et appuyez sur OK. Dans la fenêtre de sélection de la configuration, conservez les paramètres par défaut et cliquez sur OK.
- Dans la fenêtre SÉPARATION des VIGNETTES , conservez les paramètres par défaut, sélectionnez également Ajouter des contours sur les vignettes, puis sélectionnez l’exemple à modifier.
- Séparez les organismes touchants dans chaque vignette qui apparaît en traçant une ligne avec la souris (appuyez sur le bouton de roulement pour dessiner). Une fois la séparation dans une vignette terminée, cliquez sur le bouton X dans le coin supérieur droit de la fenêtre et appuyez sur OUI pour traiter la suivante. Appuyez sur NON pour terminer et enregistrer les modifications. À la fin du processus, NORMAL END apparaîtra si tout est correct.
- Après la séparation, retraitez l’image pour obtenir les données d’objet mises à jour. Accédez au logiciel de traitement d’image, cliquez sur TRAITER (Convertie) Image (Process One) et sélectionnez Traiter à nouveau les particules des images traitées dans les sous-dossiers WORK. Sélectionnez l’exemple et, dans la fenêtre Single Image Process , conservez les paramètres par défaut, cochez Travailler avec le masque de séparation (CREATE-MODIFY-INCLUDE), puis cliquez sur OK. À la fin du processus, NORMAL END apparaîtra si tout est correct.
- Dans la fenêtre Contrôle de séparation , appuyez sur OK pour enregistrer l’image avec les contours avant le traitement ; Si une image précédente existe, elle sera remplacée.
- Dans la fenêtre Masque de contrôle de séparation , si nécessaire, sélectionnez MODIFIER pour ajouter des lignes de séparation au masque à l’aide de la souris afin de séparer les organismes en contact qui n’étaient pas apparus auparavant à l’étape de séparation à l’aide de vignettes. Lorsque vous avez terminé, terminez le processus et, dans la fenêtre Contrôle du masque de séparation , sélectionnez OUI pour accepter le masque. À la fin du processus, NORMAL END apparaîtra si tout est correct.
REMARQUE: Le retraitement d’un échantillon avec un masque de séparation prend beaucoup de temps (cela peut prendre plus de 1,5 heure par échantillon). Il est préférable de consacrer le temps requis à l’étape 1.2.5 pour éviter cette étape supplémentaire.
2. Reconnaissance automatique des objets
REMARQUE : Créez un jeu d’apprentissage pour prédire automatiquement l’identité des objets détectés, séparant ainsi les organismes des débris de l’échantillon.
- Création de jeux d’apprentissage
- Copiez les images et les fichiers .pid associés aux images qui seront utilisés pour créer les catégories de l’ensemble d’apprentissage de Zooscan_scan > _work à PID_process > Unsorted_vignettes_pid.
REMARQUE : Sélectionner un sous-ensemble d’échantillons présentant une grande diversité de taxons et différents sites d’échantillonnage et/ou saisons d’échantillonnage afin d’assurer une représentativité maximale des organismes dans les échantillons. - Dans le dossier PID_process > Jeu d’apprentissage, créez un sous-dossier avec le nom du nouvel ensemble d’apprentissage (c’est-à-dire yyyymmdd_raw_LS) et, à l’intérieur, créez les sous-dossiers qui correspondront à chaque catégorie de l’ensemble d’apprentissage (macroinvertébrés, débris, autres invertébrés).
REMARQUE : Pour obtenir efficacement la structure de taille des communautés d’échantillons de macroinvertébrés de rivière, il est recommandé d’utiliser un ensemble d’apprentissage basé sur seulement trois catégories : macroinvertébrés, autres invertébrés et débris. Cet ensemble d’apprentissage sépare essentiellement les vignettes d’objets correspondant à des organismes de celles correspondant à des débris (par exemple, fibres, particules ou algues filamenteuses). - Accédez au logiciel de traitement d’image (mode avancé uniquement) et choisissez EXTRAIRE les vignettes pour PLANKTON IDENTIFIER (vignettes non triées pour la formation). Conservez les options par défaut et cochez la case Ajouter des contours .
- Accédez au logiciel d’identification automatique, cliquez sur Apprentissage, sélectionnez parmi PID_process > Learning_set le sous-dossier créé pour le nouvel ensemble d’apprentissage (étape 2.1.2) et appuyez sur OK.
- Dans la section de gauche (Pouces non triés) de la fenêtre ouverte, sélectionnez le dossier Non trié vignettes_pid. Sélectionnez les vignettes et faites-les glisser avec la souris des pouces non triés vers le dossier de leur catégorie correspondante dans la section de droite, Pouce trié, pour classer chaque objet dans les catégories définies. Les vignettes déplacées seront marquées d’un X rouge.
REMARQUE: Définissez les catégories manuellement en créant des sous-dossiers dans le dossier des pouces triés ou créez-les en cliquant sur l’icône des dossiers dans le logiciel. Ne déplacez pas plus de 50 vignettes en même temps. - Une fois toutes les catégories complétées avec les objets sélectionnés (environ 300 objets par catégorie), cliquez sur Créer un fichier d’apprentissage et enregistrez-le avec le nom souhaité.
Remarque : l’ensemble d’apprentissage sera enregistré en tant que fichier .pid dans le dossier PID_process > Jeu d’apprentissage du projet. Il est recommandé de créer et de tester plusieurs ensembles d’apprentissage avec différents niveaux de catégories (des formes grossières aux formes fines) et avec un équilibre différent du nombre d’objets dans chaque catégorie. Commencez avec un ensemble d’apprentissage grossier avec un faible nombre de catégories et au moins 50 objets par catégorie, puis augmentez le nombre d’objets dans chaque catégorie et/ou créez des ensembles d’apprentissage plus fins. Une catégorie doit être représentative de sa variabilité dans l’ensemble des échantillons.
- Copiez les images et les fichiers .pid associés aux images qui seront utilisés pour créer les catégories de l’ensemble d’apprentissage de Zooscan_scan > _work à PID_process > Unsorted_vignettes_pid.
- Évaluation de l’ensemble d’apprentissage
REMARQUE : Effectuez une validation croisée avec deux volets et cinq essais à l’aide de la méthode de la forêt aléatoire avec le logiciel d’identification automatique pour obtenir une matrice de confusion de la classification résultante des objets.- Accédez au logiciel de classification automatique et cliquez sur Analyse des données.
- Dans Sélectionner un fichier d’apprentissage, sélectionnez le fichier d’ensemble d’apprentissage créé dans PID_process > jeu d’apprentissage.
- Dans Sélectionner une méthode, choisissez la méthode de forêt aléatoire de validation croisée. Dans Variables d’origine, décochez les variables de position (X, Y, XM, YM, BX, BY et Height). Dans Variables personnalisées, cochez uniquement ESD.
REMARQUE: Cette méthode utilise une partie aléatoire de l’ensemble d’apprentissage pour reconnaître l’autre partie (deux plis), et cela est répété cinq fois pour s’assurer qu’il est statistiquement robuste. - Cliquez sur Démarrer l’analyse et enregistrez les résultats au format Analysis_name.txt dans le dossier PID_process > Prédiction. Une fois l’analyse terminée avec succès, quittez l’analyse des données.
- Accédez au dossier PID_process > Prédiction et cliquez sur le fichier de validation croisée. Une fenêtre apparaîtra avec la matrice de confusion de la classification réelle (lignes) par rapport à la classification automatique (colonnes).
REMARQUE : Le rappel est le pourcentage d’organismes appartenant à un groupe qui a été automatiquement bien reconnu, tandis que la précision 1 est le pourcentage d’organismes classés par l’algorithme comme un groupe qui n’est pas reconnu (contamination dans un groupe). Le rappel doit être supérieur à 70 % et la contamination (précision 1) doit être inférieure à 20 %. - Répétez les étapes 2.1-2.5 si plusieurs ensembles d’apprentissage ont été créés et le rappel et la précision 1 de chacun doivent être obtenus.
REMARQUE : Si plusieurs ensembles d’apprentissage ont été créés, choisissez celui qui présente le plus grand rappel (bonne reconnaissance) et la plus grande précision (faible contamination) du groupe d’intérêt (c.-à-d. les macroinvertébrés) pour tester la prédiction automatique d’un ensemble d’échantillons à l’étape suivante.
- Prédiction de l’identification des macroinvertébrés
Remarque : Utilisez le jeu d’apprentissage sélectionné pour prédire l’identité de tous les objets dans un sous-ensemble d’échantillons à l’aide du logiciel d’identification automatique avec un algorithme de forêt aléatoire.- Accédez au logiciel d’identification automatique et cliquez sur Analyse des données.
- Dans Sélectionner un fichier d’apprentissage, sélectionnez le fichier d’ensemble d’apprentissage dans PID_process > jeu d’apprentissage qui doit être utilisé pour la prédiction.
- Dans Sélectionner le(s) fichier(s) d’exemple, sélectionnez dans le dossier PID_results les exemples (fichiers PID) qui vont être prédits.
Remarque : Traitez un maximum de 20 fichiers .pid en même temps pour éviter les erreurs liées aux problèmes de mémoire. Si trop de fichiers .pid sont traités en même temps, le processus affichera une fin correcte mais peut ne pas être bien traité, et une erreur peut se produire dans les étapes suivantes lors du traitement avec le logiciel de traitement d’image. - Dans Sélectionner une méthode, choisissez la méthode Forêt aléatoire . Cochez Enregistrer les résultats détaillés pour chaque échantillon. Dans Variables d’origine, décochez les variables de position (X, Y, XM, YM, BX, BY et Height). Dans Variables personnalisées, cochez uniquement ESD.
- Cliquez sur Démarrer l’analyse et enregistrez les résultats au format Analysis_name.txt dans le dossier PID_process > Prédiction.
- Validation manuelle
REMARQUE : Un expert valide manuellement la prédiction de l’étape précédente pour reclasser les objets mal classés dans la catégorie appropriée.- Copiez les fichiers Analysis_sample_dat1.txt à valider du dossier PID_process > Prediction vers le dossier PID_process > Pid_results.
- Accédez au logiciel de traitement d’image et sélectionnez EXTRAIRE les vignettes dans les dossiers selon PREDICTION ou VALIDATION. Ensuite, sélectionnez Utiliser les fichiers PREDICTION du dossier « pid_results ». Conservez les paramètres par défaut et appuyez sur OK.
- Le logiciel crée un dossier appelé sample_yyyymmdd_hhmm_to_validate avec les objets prédits dans le dossier PID_process > Vignettes triées.
- Accédez au dossier PID_process > Vignettes triées et copiez le dossier sample_yyyymmdd_ hhmm_to_validate. Remplacez le nom du dossier _to validez par _validated.
- Pour valider manuellement la classification automatique, ouvrez la sample_yyyymmdd_ hhmm_validated du dossier et passez en revue toutes les vignettes de chaque sous-dossier (catégorie) afin d’identifier s’il y a des objets mal classés. Lorsqu’un objet est mal classé, faites glisser la vignette à l’aide de la souris vers le dossier approprié (catégorie).
- Accédez au logiciel de traitement d’image et sélectionnez LOAD Identifications à partir des vignettes triées. Conservez les paramètres par défaut et sélectionnez yyyymmdd_hhmm_name_validated à traiter.
- Accédez à PID_process > Pid_results > Dat1_validated, où un fichier nommé Id_from_sorted_vignettes_yyyymmdd_hhmm.txt et un fichier .txt pour chacun des échantillons validés (sample_tot_1_dat1.txt) ont été créés.
Remarque : Ces fichiers .txt contiennent une nouvelle colonne qui présente la prédiction, appelée pred_valid_Id_yyyymmdd_hhmm, qui spécifie la classification experte de chaque objet (c’est-à-dire la classification validée). De nouvelles catégories (par exemple, des catégories taxonomiques plus fines) pourraient être créées à ce stade, pendant la validation. Cependant, conservez le nom de la catégorie d’origine dans le nouveau nom (par exemple, macroinvertebrate_chironomidae). Cela permet de retracer la catégorie d’origine lors du calcul du rappel et de la précision et de regrouper facilement tous les macroinvertébrés pour calculer les paramètres de structure de taille de la communauté (c.-à-d. le spectre de taille et la diversité de taille). Le fichier texte fournit les données associées à chaque objet, y compris les axes mineurs et majeurs utilisés pour obtenir le volume ellipsoïdal de chaque organisme comme mesure de la taille corporelle individuelle. De plus, les deux dernières colonnes du tableau contiennent les catégories prédites et validées de chaque objet (ligne), ce qui permet de calculer, par catégorie, le rappel et la précision de l’ensemble d’apprentissage sur le sous-ensemble d’échantillons.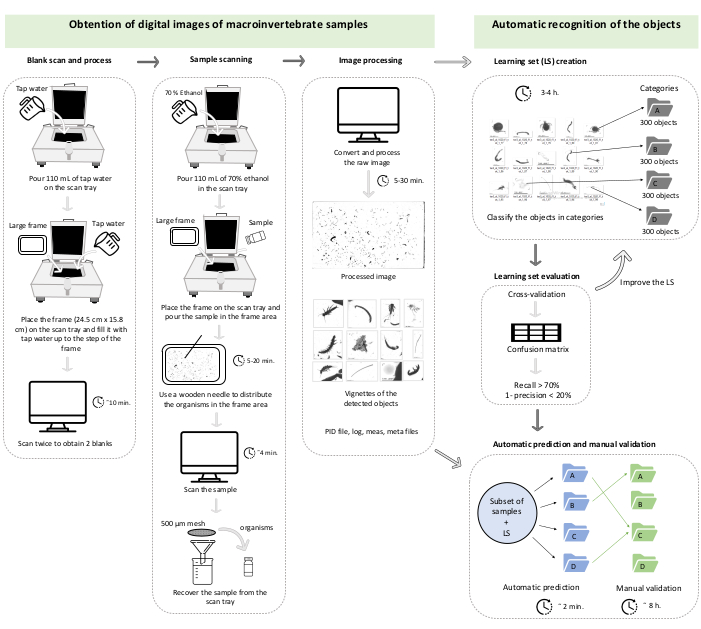
Figure 1 : Diagramme de travail représentant les sections 1 et 2 du protocole. Les horaires sont illustratifs et peuvent changer en fonction de l’ordinateur, de l’abondance de vignettes à traiter et du nombre de catégories de l’ensemble d’apprentissage. Ce cas correspond à la validation d’un ensemble d’apprentissage de trois catégories sur un ensemble de 42 sous-échantillons (au total, 47 473 vignettes). Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Calcul de la distribution de taille individuelle, des spectres de taille et des mesures de taille
REMARQUE: Les calculs mentionnés dans cette section ont été effectués à l’aide de Matlab (voir le script en tant que fichier supplémentaire 1).
- Répartition individuelle par taille
- La dernière colonne du fichier Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt contient la classification validée des objets. Sélectionnez uniquement les objets classés comme macroinvertébrés pour représenter leur distribution de taille individuelle dans l’échantillon.
NOTE: La taille du corps individuel correspond au volume ellipsoïdal des organismes macroinvertébrés. Le système fournit des mesures en pixels. - Concaténer les vecteurs avec les mesures de taille des deux balayages, car chaque fraction a un exposant de sous-échantillonnage différent. Avant la concaténation, corriger le fractionnement en répliquant les vecteurs de taille autant de fois que le sous-échantillon correspondant a été fractionné.
REMARQUE : Cette étape est nécessaire si un balayage correspond à une fraction d’un échantillon (c.-à-d. grossier ou fin). - Calculer le volume ellipsoïdal à partir des axes majeur (M) et mineur (m) des ellipsoïdes prolates ayant les mêmes zones de pixels que les organismes. Avant de calculer le volume ellipsoïdal, convertissez les axes majeur (M) et mineur (m) des pixels en millimètres (mm) avec le facteur de conversion suivant (cf):
1 pixel = 2 400 ppp
1 pouce = 25,4 mm
cf = 25,4/2400
Le volume ellipsoïdal (ellipVol avec unités en mm3) correspond à :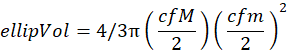
- Représenter la fonction de densité de probabilité de la distribution de taille individuelle sur l’échellelogarithmique 2 .
- La dernière colonne du fichier Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt contient la classification validée des objets. Sélectionnez uniquement les objets classés comme macroinvertébrés pour représenter leur distribution de taille individuelle dans l’échantillon.
- Diversité des tailles
- Calculer la diversité de taille (Sd) d’après Quintana et al. (2008)8, comme dans García-Comas et al. (2016)35 :
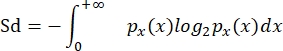
où p x(x) est la fonction de densité de probabilité de taille x, et x représente log2 (ellipVol). Cette mesure est donc l’indice de diversité de Shannon adapté à une mesure continue, comme la répartition de la taille individuelle dans une communauté.
- Calculer la diversité de taille (Sd) d’après Quintana et al. (2008)8, comme dans García-Comas et al. (2016)35 :
- Spectre granulométrique normalisé du biovolume (NBSS)
- Définir les classes de taille du NBSS, en établissant la limite inférieure du spectre comme le quantile 0,01 de la distribution de taille des macroinvertébrés dans les échantillons et en créant des classes de taille à une échelle géométrique de base 2 jusqu’à ce que le plus grand organisme des échantillons soit englobé.
Remarque : La largeur de la classe de taille augmente avec la taille pour tenir compte de la plus grande variabilité associée à des tailles plus grandes. Le NBSS des communautés de macroinvertébrés analysées ici comportait 14 classes de taille (tableau 1). - Obtenez le biovolume normalisé en divisant le biovolume total dans chaque classe de taille par la largeur de la classe de taille.
- Définir les classes de taille du NBSS, en établissant la limite inférieure du spectre comme le quantile 0,01 de la distribution de taille des macroinvertébrés dans les échantillons et en créant des classes de taille à une échelle géométrique de base 2 jusqu’à ce que le plus grand organisme des échantillons soit englobé.
- Pente du spectre de taille
- Calculer la pente linéaire du NBSS.
NOTA : La pente (μ) est calculée en fonction de la relation entre le log 2 (point médian de la classe de taille) et le log2 (biomasse normalisée) dans les classes de taille supérieures au mode, sans tenir compte des classes vides (dans la présente étude, les classes de taille de 3 à 14).
- Calculer la pente linéaire du NBSS.
| Limites de classe de taille (mm3) | Point médian de la classe de taille (mm3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
Tableau 1 : Classes de taille du spectre granulométrique normalisé de la biomasse (NBSS). Le tableau montre également les limites des 15 classes de taille et les points médians de la classe de taille des organismes.
Résultats
Acquisition d’images numériques d’échantillons de macroinvertébrés
Nuances de balayage : dépôt d’éthanol dans le bac de numérisation
Lors de l’essai du système pour les macroinvertébrés, plusieurs scans étaient de mauvaise qualité. Une zone saturée sombre en arrière-plan empêchait le traitement normal de l’image et la mesure de la taille individuelle des macroinvertébrés (Figure 2). Plusieurs raisons ont été données pour ...
Discussion
L’adaptation de la méthodologie décrite par Gorsky et al., 2010 pour les macroinvertébrés riverains permet une grande précision de classification dans l’estimation de la structure de la taille des communautés de macroinvertébrés d’eau douce. Les résultats suggèrent que le protocole peut réduire le temps d’estimation de la structure de taille individuelle dans un échantillon à environ 1 heure. Ainsi, le protocole proposé vise à promouvoir l’utilisation systématique des spectres de taille des macr...
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts potentiels.
Remerciements
Ce travail a été soutenu par le ministère espagnol de la Science, de l’Innovation et des Universités (numéro de subvention RTI2018-095363-B-I00). Nous remercions les membres du CERM-UVic-UCC Èlia Bretxa, Anna Costarrosa, Laia Jiménez, María Isabel González, Marta Jutglar, Francesc Llach et Núria Sellarès pour leur travail dans l’échantillonnage sur le terrain des macroinvertébrés et le tri en laboratoire, ainsi que David Albesa pour sa collaboration à la numérisation des échantillons. Nous remercions enfin Josep Maria Gili et l’Institut de Ciències del Mar (ICM-CSIC) pour l’utilisation des installations de laboratoire et de l’appareil scanner.
matériels
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
Références
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon