È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Elaborazione automatica delle immagini per determinare la struttura delle dimensioni della comunità dei macroinvertebrati fluviali
In questo articolo
Riepilogo
L'articolo si basa sulla creazione di un protocollo adattato per scansionare, rilevare, ordinare e identificare oggetti digitalizzati corrispondenti a macroinvertebrati fluviali bentonici utilizzando una procedura di imaging semiautomatica. Questa procedura consente l'acquisizione delle distribuzioni dimensionali individuali e delle metriche dimensionali di una comunità di macroinvertebrati in circa 1 ora.
Abstract
La dimensione corporea è un importante tratto funzionale che può essere utilizzato come bioindicatore per valutare gli impatti delle perturbazioni nelle comunità naturali. La struttura delle dimensioni della comunità risponde ai gradienti biotici e abiotici, comprese le perturbazioni antropogeniche tra taxa ed ecosistemi. Tuttavia, la misurazione manuale di organismi di piccole dimensioni come i macroinvertebrati bentonici (ad esempio, >da 500 μm a pochi centimetri di lunghezza) richiede molto tempo. Per accelerare la stima della struttura delle dimensioni della comunità, qui, abbiamo sviluppato un protocollo per misurare semi-automaticamente le dimensioni corporee individuali dei macroinvertebrati fluviali conservati, che sono uno dei bioindicatori più comunemente usati per valutare lo stato ecologico degli ecosistemi di acqua dolce. Questo protocollo è adattato da una metodologia esistente sviluppata per scansionare il mesozooplancton marino con un sistema di scansione progettato per campioni d'acqua. Il protocollo consiste in tre fasi principali: (1) scansione di sottocampioni (frazioni di dimensioni del campione fini e grossolane) di macroinvertebrati fluviali ed elaborazione delle immagini digitalizzate per individualizzare ogni oggetto rilevato in ciascuna immagine; (2) creare, valutare e convalidare un set di apprendimento attraverso l'intelligenza artificiale per separare semi-automaticamente le singole immagini di macroinvertebrati da detriti e artefatti nei campioni scansionati; e (3) raffigurando la struttura dimensionale delle comunità di macroinvertebrati. Oltre al protocollo, questo lavoro include i risultati della calibrazione ed elenca diverse sfide e raccomandazioni per adattare la procedura ai campioni di macroinvertebrati e da considerare per ulteriori miglioramenti. Nel complesso, i risultati supportano l'uso del sistema di scansione presentato per la misurazione automatica delle dimensioni corporee dei macroinvertebrati fluviali e suggeriscono che la rappresentazione del loro spettro dimensionale è uno strumento prezioso per la rapida biovalutazione degli ecosistemi di acqua dolce.
Introduzione
I macroinvertebrati bentonici sono ampiamente utilizzati come bioindicatori per determinare lo stato ecologico dei corpi idrici1. La maggior parte degli indici per descrivere le comunità di macroinvertebrati si concentra su metriche tassonomiche. Tuttavia, i nuovi strumenti di biovalutazione che integrano le dimensioni corporee sono incoraggiati a fornire una prospettiva alternativa o complementare agli approcci tassonomici 2,3.
La dimensione del corpo è considerata un metatratto correlato ad altri tratti vitali come il metabolismo, la crescita, la respirazione e il movimento4. Inoltre, le dimensioni del corpo possono determinare la posizione trofica e le interazioni5. La relazione tra le dimensioni corporee individuali e la biomassa normalizzata (o abbondanza) per classe dimensionale in una comunità è definita come lo spettro dimensionale6 e segue il modello generale di una diminuzione lineare della biomassa normalizzata all'aumentare delle dimensioni individuali su una scala logaritmica7. La pendenza di questa relazione lineare è stata ampiamente studiata teoricamente e studi empirici attraverso gli ecosistemi l'hanno utilizzata come indicatore ecologico della struttura dimensionale della comunità4. Un altro indicatore sintetico della struttura dimensionale della comunità che è stato utilizzato con successo negli studi sul funzionamento della biodiversità-ecosistema è la diversità delle dimensioni della comunità, che è rappresentata come l'indice di Shannon delle classi dimensionali dello spettro dimensionale o il suo analogo, che viene calcolato in base alle distribuzioni dimensionali individuali8.
Negli ecosistemi di acqua dolce, la struttura dimensionale di diversi gruppi faunistici viene utilizzata come indicatore atassico per valutare la risposta delle comunità biotiche ai gradienti ambientali 9,10,11 e alle perturbazioni antropogeniche 12,13,14,15,16. I macroinvertebrati non fanno eccezione, e la loro struttura dimensionale risponde anche ai cambiamenti ambientali17,18 e alle perturbazioni antropogeniche, come l'estrazione mineraria 19, l'uso del suolo 20, o l'arricchimento di azoto (N) e fosforo (P)20,21,22. Tuttavia, misurare centinaia di individui per descrivere la struttura delle dimensioni della comunità è un compito noioso e dispendioso in termini di tempo che viene spesso evitato come misurazione di routine nei laboratori a causa della mancanza di tempo. Pertanto, sono stati sviluppati diversi metodi di imaging semiautomatici o automatici per classificare e misurare i campioni23,24,25,26. Tuttavia, la maggior parte di questi metodi sono focalizzati sulla classificazione tassonomica più che sulla dimensione individuale degli organismi e non sono pronti per l'uso per tutti i tipi di macroinvertebrati. Nell'ecologia del plancton marino, un sistema di analisi delle immagini a scansione è stato ampiamente utilizzato per determinare le dimensioni e la composizione tassonomica delle comunità di zooplancton 27,28,29,30,31. Questo strumento può essere trovato in diversi istituti marini in tutto il mondo e viene utilizzato per scansionare campioni di zooplancton conservati per ottenere immagini digitali ad alta risoluzione dell'intero campione. Il presente protocollo adatta l'uso di questo strumento per stimare lo spettro dimensionale della comunità di macroinvertebrati nei fiumi in modo rapido e automatico senza investire nella creazione di un nuovo dispositivo.
Il protocollo consiste nella scansione di un campione e nell'elaborazione dell'intera immagine per ottenere automaticamente singole immagini (cioè vignette) degli oggetti nel campione. Diverse misure di forma, dimensioni e caratteristiche di livello di grigio caratterizzano ogni oggetto e consentono la classificazione automatica degli oggetti in categorie, che vengono poi convalidate da un esperto. La dimensione individuale di ciascun organismo viene calcolata utilizzando il biovolume ellissoidale (mm3), che deriva dall'area dell'organismo misurata in pixel. Ciò consente di ottenere rapidamente lo spettro dimensionale del campione. Per quanto ne sappiamo, questo sistema di imaging a scansione è stato utilizzato solo per elaborare campioni di mesozooplancton, ma il dispositivo potrebbe potenzialmente consentire di lavorare con macroinvertebrati bentonici d'acqua dolce.
L'obiettivo generale di questo studio è, quindi, quello di introdurre un metodo per ottenere rapidamente la dimensione individuale dei macroinvertebrati fluviali conservati adattando un protocollo esistente precedentemente utilizzato con mesozooplancton marino 27,32,33. La procedura consiste nell'utilizzare un approccio semi-automatico che opera con un dispositivo di scansione per scansionare campioni d'acqua e tre software aperti per elaborare le immagini scansionate. Viene qui presentato un protocollo adattato per scansionare, rilevare e identificare i macroinvertebrati fluviali digitalizzati per acquisire automaticamente la struttura delle dimensioni della comunità e le relative metriche dimensionali. La valutazione della procedura e le linee guida per migliorare l'efficienza sono presentate anche sulla base di 42 immagini scansionate di campioni di macroinvertebrati fluviali raccolti da tre bacini nella penisola iberica nord-orientale (NE) (Ter, Segre-Ebre e Besòs).
I campioni sono stati raccolti in tratti fluviali di 100 m seguendo il protocollo per il campionamento sul campo e l'analisi di laboratorio di macroinvertebrati fluviali bentonici in fiumi guadabili dal governo spagnolo34. I campioni sono stati raccolti con un campionatore surber (telaio: 0,3 m x 0,3 m, maglia: 250 μm) a seguito di un'indagine multi-habitat. In laboratorio, i campioni sono stati puliti e setacciati attraverso una maglia di 5 mm e una maglia di 500 μm per ottenere due sottocampioni: un sottocampione grossolano (maglia da 5 mm) e un sottocampione fine (maglia da 500 μm), che sono stati conservati in fiale separate e conservati in etanolo al 70%. La separazione del campione in due frazioni dimensionali consente una migliore stima della struttura dimensionale della comunità, poiché gli organismi di grandi dimensioni sono più rari e meno degli organismi piccoli. In caso contrario, il campione scansionato ha una rappresentazione distorta della frazione di grandi dimensioni.
Protocollo
NOTA: Il protocollo qui descritto si basa sul sistema sviluppato da Gorsky et al.27 per il mesozooplancton marino. Una descrizione specifica dello scanner (ZooSCAN), del software di scansione (VueScan 9x64 [9.5.09]), del software di elaborazione delle immagini (Zooprocess, ImageJ) e del software di identificazione automatica (Plankton Identifier) si trova nei riferimenti precedenti32,33. Per regolare al meglio le dimensioni dei macroinvertebrati bentonici rispetto al mesozooplancton, una volta creato il progetto seguendo il protocollo originale32,33, modificare il parametro di dimensione minima (minsizeesd_mm) a 0,3 mm e il parametro di dimensione massima (maxsizeesd_mm) a 100 mm nel file di configurazione. Per facilitare il rispetto del protocollo, questo è riassunto in un diagramma di lavoro (Figura 1). Il progetto creato viene memorizzato nella cartella C del computer ed è organizzato nelle seguenti cartelle: PID_process, Zooscan_back, Zooscan_check, Zooscan_config, Zooscan_meta, Zooscan_results e Zooscan_scan. Ogni cartella è composta da diverse sottocartelle che le diverse applicazioni software utilizzano nei passaggi successivi del protocollo.
1. Acquisizione di immagini digitali per campioni di macroinvertebrati
- Scansione ed elaborazione del pezzo vuoto
NOTA: creare due immagini vuote al giorno prima della scansione per estrarre le scansioni in background durante l'elaborazione delle immagini acquisite nello stesso giorno.- Accendere lo scanner e accendere la luce in doppia posizione per proiettare luce bianca dall'alto e dal basso.
NOTA: Quando si esegue la scansione di campioni di mesozooplancton, viene utilizzata la direzione della luce verso l'alto, ma poiché i macroinvertebrati sono più opachi, si consiglia di passare la luce in una posizione doppia. - Pulire e sciacquare il vassoio di scansione con acqua di rubinetto.
- Versare 110 ml di acqua di rubinetto conservata a temperatura ambiente (RT) nel vassoio di scansione fino a coprire il bicchiere. Posizionare la cornice grande (24,5 cm x 15,8 cm) sul vassoio di scansione nella posizione corretta (con l'angolo nella parte superiore sinistra del vassoio di scansione) e riempirla con acqua di rubinetto fino a coprire il gradino della cornice per evitare un effetto menisco, che altererebbe l'immagine acquisita. Chiudere il coperchio dello scanner.
NOTA: Utilizzare acqua in RT per evitare la formazione di condensa e bolle. Pulire il telaio senza segni o goccioline per evitare riflessi di luce. - Vai al software di elaborazione delle immagini, seleziona il progetto di lavoro e fai clic su Scansione (Converti) immagine di sfondo.
- Vai al software di scansione e fai clic su Anteprima. Assicurati di visualizzare l'anteprima dell'immagine acquisita, controlla che non ci siano linee o macchie e attendi almeno 30 secondi prima di iniziare un'altra scansione. Fare clic su Scansione e premere OK nella finestra delle istruzioni prima della seconda scansione per inviare i dati dal software di scansione al software di elaborazione delle immagini.
NOTA: eseguire la scansione due volte per ottenere le due scansioni in background che comporranno lo spazio vuoto. Questo passaggio viene eseguito una volta al giorno prima di iniziare l'elaborazione del campione e le immagini vengono archiviate nella cartella Zooscan_back. - Chiudere il software di scansione al termine della scansione.
- Accendere lo scanner e accendere la luce in doppia posizione per proiettare luce bianca dall'alto e dal basso.
- Preparazione e scansione dei campioni
ATTENZIONE: L'etanolo è un liquido infiammabile e potrebbe causare gravi danni agli occhi / irritazione.- Compilare i metadati di esempio. Vai al software di elaborazione delle immagini e seleziona Compila metadati di esempio. Immettere l'identità di esempio, fare clic su OK e compilare i metadati.
NOTA: il metafile è stato creato appositamente per i campioni di mesozooplancton, quindi non si adatta alla metodologia di campionamento dei macroinvertebrati bentonici, ma tutti i campi del file devono essere compilati prima della scansione, altrimenti verrà visualizzato un flag di errore. - Versare 110 ml di etanolo al 70% nel vassoio di scansione fino a coprire il vetro e posizionare il telaio grande (24,5 cm x 15,8 cm) con l'angolo nella parte superiore sinistra del vassoio di scansione.
NOTA: Lavorare con etanolo invece di acqua, poiché i macroinvertebrati sono conservati in etanolo. In acqua, galleggiano e vanno alla deriva nel vassoio di scansione, impedendo un'immagine nitida e, quindi, misurazioni affidabili delle dimensioni. L'etanolo deve essere conservato a RT per evitare la formazione di condensa e bolle. - Versare il campione di macroinvertebrati nel vassoio di scansione bordato dal telaio e coprire il passo del telaio con più etanolo, se necessario.
NOTA: Astenersi dall'aggiungere troppo etanolo per evitare che gli organismi fluttuino e vadano alla deriva. - Omogeneizzare il campione in tutta l'area del telaio, posizionando gli individui più grandi al centro del vassoio per una corretta elaborazione delle immagini e affondare gli organismi galleggianti usando un ago di legno.
NOTA: Se un sottocampione contiene numericamente più di 1.000 individui, dividere il sottocampione in due o più frazioni per ridurre al minimo il contatto con gli organismi nell'immagine scansionata e scansionare le frazioni separatamente. - Separare gli organismi che toccano e gli organismi che toccano i bordi del telaio usando l'ago di legno.
NOTA: questo passaggio richiede 5-20 minuti. Gli organismi che toccano sono considerati un singolo oggetto dal software; Pertanto, in questi casi, le dimensioni individuali calcolate non corrispondono a singoli organismi effettivi e possono influenzare la stima della struttura dimensionale della Comunità. C'è la possibilità di modificare l'immagine con il software di elaborazione delle immagini per separarli, ma questo passaggio aggiuntivo comporta almeno 1,5 ore di rielaborazione; Pertanto, la separazione manuale è altamente raccomandata. - Per eseguire la scansione del campione, chiudere il coperchio dello scanner, accedere al software di elaborazione delle immagini, selezionare il progetto funzionante e fare clic su SCAN Sample with Zooscan (For Archive, No Process).
- Selezionare l'esempio e seguire le istruzioni.
- Vai al software di scansione e fai clic su Anteprima. Assicurati di visualizzare l'anteprima dell'immagine acquisita, controlla che non ci siano linee o punti e attendi almeno 30 secondi prima di iniziare un'altra scansione.
- Dopo almeno 30 secondi, fare clic sul pulsante Scansione nel software di scansione.
NOTA: premere OK nel software di elaborazione delle immagini dopo aver premuto Scan nel software di scansione. Non premere alcun tasto sulla tastiera del computer ed evitare le vibrazioni della scansione durante la scansione. Nella cartella Zooscan_scan > _raw vengono generati tre file: (i) un formato di file immagine con tag (.tif) (16 bit); ii) un documento di testo standard denominato LOG (.txt) che registra le informazioni sui parametri di scansione; e iii) un documento di testo standard denominato META (.txt) con informazioni sui metodi di campionamento. - Verificare che la scansione raw sia corretta.
NOTA: se la scansione presenta strisce chiare o altri problemi visibili, è consigliabile ripetere la scansione per evitare problemi nei passaggi seguenti.
- Compilare i metadati di esempio. Vai al software di elaborazione delle immagini e seleziona Compila metadati di esempio. Immettere l'identità di esempio, fare clic su OK e compilare i metadati.
- Recupero del campione
- Rimuovere il telaio e sciacquarlo sopra il vassoio di scansione utilizzando una bottiglia di spremitura riempita con etanolo al 70% per recuperare eventuali macroinvertebrati attaccati.
- Sollevare la parte superiore dello scanner per recuperare tutti gli organismi e l'etanolo dal vassoio attraverso l'imbuto di recupero della scansione in un becher. Con la parte superiore dello scanner ancora sollevata, sciacquare il vassoio con il flacone di spremitura per spazzare via eventuali organismi rimanenti.
- Far passare i campioni e l'etanolo dal becher attraverso una maglia da 500 μm per trattenere gli invertebrati nella rete e conservarli in una fiala con etanolo al 70%.
- Una volta recuperati tutti i campioni nel flaconcino, pulire il vassoio con acqua di rubinetto.
NOTA: lavare il vassoio con acqua di rubinetto tra i campioni per ridurre al minimo la precipitazione di etanolo, che altera l'elaborazione delle immagini. Risciacquare il telaio con acqua di rubinetto per evitare potenziali danni legati all'uso di etanolo. Alla fine della giornata, pulire il vassoio con acqua di rubinetto e asciugarlo delicatamente con carta per evitare graffi.
- Elaborazione di immagini
- Vai al software di elaborazione delle immagini e seleziona Converti ed elabora immagini e organismi in modalità batch, quindi Converti ed elabora immagine E particelle (immagine nella cartella RAW). Mantenere le impostazioni predefinite e fare clic su OK. NORMAL END apparirà alla fine del processo.
NOTA: Nella cartella Zooscan_scan > _work verranno creati un file PID e le vignette corrispondenti a tutti gli oggetti rilevati nell'immagine acquisita (in un file Joint Photographic Group [.jpg]). Un file PID è un singolo file che memorizza tutti i metadati (metafile), i dati tecnici associati al file di registro e una tabella con 36 variabili misurate di tutti gli oggetti rilevati nell'immagine. Le variabili misurate corrispondono a diverse stime del livello di grigio, della dimensione frattale, della forma e delle dimensioni. Le variabili che possono essere utilizzate per la stima delle dimensioni sono l'area e gli assi maggiore e minore di un'ellisse con un'area uguale all'oggetto (vedere la sezione 3 del protocollo). Il tempo di elaborazione dipende dalla densità dell'immagine e dalle caratteristiche del computer e può essere avviato tra i campioni durante il recupero e la preparazione del campione successivo. In caso contrario, si consiglia di avviare l'elaborazione dei campioni scansionati ogni giorno in modalità batch durante la notte e verificare la corretta elaborazione delle immagini la mattina successiva. - Verificare se lo sfondo dell'immagine elaborata viene sottratto in modo appropriato dall'immagine campione utilizzando il software di elaborazione delle immagini o controllando le immagini maschera (terminate in msk1.gif) situate in Zooscan_scan > _work. Se lo sfondo contiene aree sature o molti punti, è consigliabile ripetere la scansione per garantire immagini di alta qualità.
NOTA: per evitare aree sature sullo sfondo, il vassoio di scansione deve essere risciacquato con acqua di rubinetto dopo ogni scansione con etanolo. È anche importante (1) ridurre il numero di individui scansionati (frazionando il campione e scansionando in pieghe diverse); (2) assicurarsi che i grandi organismi siano posizionati al centro del vassoio di scansione; 3) utilizzare etanolo pulito e filtrato; (4) ridurre la sporcizia sui campioni; 5) garantire che il volume di etanolo per la scansione sia adeguato; e (6) assicurarsi che il ritardo tra l'anteprima del campione e la scansione sia di almeno 30 s.
- Vai al software di elaborazione delle immagini e seleziona Converti ed elabora immagini e organismi in modalità batch, quindi Converti ed elabora immagine E particelle (immagine nella cartella RAW). Mantenere le impostazioni predefinite e fare clic su OK. NORMAL END apparirà alla fine del processo.
- Separazione degli organismi che toccano
NOTA: Quando ci sono diverse vignette con organismi che toccano, è necessario separare le immagini degli organismi che toccano da altri organismi e / o da fibre / detriti per garantire una corretta stima della struttura delle dimensioni della comunità.- Vai al software di elaborazione delle immagini per rilevare le vignette con più oggetti. Selezionare SEPARAZIONE mediante vignette e premere OK. Nella finestra di selezione della configurazione, mantenere le impostazioni predefinite e fare clic su OK.
- Nella finestra SEPARAZIONE da VIGNETTE, mantenere le impostazioni predefinite, selezionare inoltre AGGIUNGI contorni su vignette, quindi selezionare il campione da modificare.
- Separare gli organismi in contatto in ogni vignetta che si apre disegnando una linea con il mouse (premere il pulsante di rotazione per disegnare). Una volta completata la separazione in una vignetta, fare clic sul pulsante X nell'angolo superiore destro della finestra e premere SÌ per elaborare quella successiva. Premere NO per terminare e salvare le modifiche. Alla fine del processo, apparirà NORMAL END se tutto è corretto.
- Dopo la separazione, rielaborare l'immagine per ottenere i dati dell'oggetto aggiornati. Vai al software di elaborazione delle immagini, fai clic su PROCESS (Converted) Image (Process One) e seleziona Process Again Particles from Processed Images in WORK Sub-Folders. Selezionare l'esempio e, nella finestra Processo immagine singola , mantenere le impostazioni predefinite, selezionare Lavora con maschera di separazione (CREATE-MODIFY-INCLUDE), quindi fare clic su OK. Alla fine del processo, apparirà NORMAL END se tutto è corretto.
- Nella finestra Controllo separazione , premere OK per salvare l'immagine con i contorni prima dell'elaborazione; Se esiste un'immagine precedente, verrà sostituita.
- Nella finestra Maschera di controllo di separazione , se necessario, selezionare MODIFICA per aggiungere linee di separazione alla maschera utilizzando il mouse per separare gli organismi in contatto che non sono apparsi prima nel passaggio di separazione utilizzando vignette. Al termine, terminare il processo e, nella finestra Controllo maschera di separazione , selezionare SÌ per accettare la maschera. Alla fine del processo, apparirà NORMAL END se tutto è corretto.
NOTA: la rielaborazione di un campione con una maschera di separazione richiede molto tempo (potrebbe richiedere più di 1,5 ore per campione). È preferibile dedicare il tempo richiesto al punto 1.2.5 per evitare questo passaggio aggiuntivo.
2. Riconoscimento automatico degli oggetti
NOTA: Creare un set di apprendimento per prevedere automaticamente l'identità degli oggetti rilevati, separando così gli organismi dai detriti nel campione.
- Creazione di set di apprendimento
- Copiare le immagini e i file .pid associati alle immagini che verranno utilizzati per creare le categorie del set di apprendimento da Zooscan_scan > _work a PID_process > Unsorted_vignettes_pid.
NOTA: Selezionare un sottoinsieme di campioni con un'elevata diversità di taxa e diversi siti di campionamento e/o stagioni di campionamento per garantire la massima rappresentatività degli organismi nei campioni. - Nella cartella PID_process > Learning set, crea una sottocartella con il nome del nuovo set di apprendimento (ad esempio, yyyymmdd_raw_LS) e, al suo interno, crea le sottocartelle che corrisponderanno a ciascuna categoria del set di apprendimento (ad esempio, macroinvertebrati, detriti, altri invertebrati).
NOTA: Per ottenere in modo efficiente la struttura delle dimensioni della comunità dei campioni di macroinvertebrati fluviali, si consiglia di utilizzare un set di apprendimento basato su sole tre categorie: macroinvertebrati, altri invertebrati e detriti. Questo set di apprendimento separa fondamentalmente le vignette di oggetti corrispondenti agli organismi da quelli corrispondenti ai detriti (ad esempio, fibre, particelle o alghe filamentose). - Accedere al software di elaborazione delle immagini (solo modalità avanzata) e scegliere EXTRACT Vignettes for PLANKTON IDENTIFIER (unsorted vignettes for training). Mantenete le opzioni predefinite e selezionate la casella Aggiungi contorni .
- Andare al software di identificazione automatica, fare clic su Apprendimento, selezionare da PID_process > Learning_set la sottocartella creata per il nuovo set di apprendimento (passaggio 2.1.2) e premere OK.
- Nella sezione sinistra (Pollici non ordinati) della finestra aperta, selezionare la cartella Unsorted vignettes_pid. Selezionare le vignette e trascinarle con il mouse dai pollici non ordinati alla cartella della categoria corrispondente nella sezione destra, Pollici ordinati, per classificare ciascun oggetto nelle categorie definite. Le vignette spostate saranno contrassegnate con una X rossa.
NOTA: definire manualmente le categorie creando sottocartelle nella cartella dei pollici ordinati o crearle facendo clic sull'icona delle cartelle nel software. Non spostare più di 50 vignette contemporaneamente. - Una volta completate tutte le categorie con gli oggetti selezionati (circa 300 oggetti per categoria), clicca su Crea file di apprendimento e salvalo con il nome desiderato.
NOTA: il set di apprendimento verrà salvato come file .pid nella cartella PID_process > Learning set del progetto. Si consiglia di creare e testare diversi set di apprendimento con diversi livelli di categorie (dalle forme grossolane a quelle fini) e con un diverso equilibrio del numero di oggetti all'interno di ciascuna categoria. Inizia con un set di apprendimento grossolano con un numero basso di categorie e almeno 50 oggetti per categoria, quindi aumenta il numero di oggetti in ogni categoria e / o crea set di apprendimento più fini. Una categoria dovrebbe essere rappresentativa della sua variabilità nell'insieme dei campioni.
- Copiare le immagini e i file .pid associati alle immagini che verranno utilizzati per creare le categorie del set di apprendimento da Zooscan_scan > _work a PID_process > Unsorted_vignettes_pid.
- Valutazione del set di apprendimento
NOTA: Eseguire la convalida incrociata con due pieghe e cinque prove utilizzando il metodo Random Forest con il software di identificazione automatica per ottenere una matrice di confusione della classificazione risultante degli oggetti.- Vai al software di classificazione automatica e fai clic su Analisi dei dati.
- In Seleziona file di apprendimento, selezionare il file del set di apprendimento creato da PID_process > Set di apprendimento.
- In Selezionare un metodo scegliere il metodo Foresta casuale a convalida incrociata . In Variabili originali, deselezionate le variabili di posizione (X, Y, XM, YM, BX, BY e Height). In Variabili personalizzate, selezionare solo ESD.
NOTA: questo metodo utilizza una parte casuale del set di apprendimento per riconoscere l'altra parte (due pieghe) e questo viene ripetuto cinque volte per garantire che sia statisticamente robusto. - Fare clic su Avvia analisi e salvare i risultati come Analysis_name.txt nella cartella PID_process > Previsione. Quando l'analisi è stata completata correttamente, uscire dall'analisi dei dati.
- Vai alla cartella PID_process > Prediction e fai clic sul file di convalida incrociata. Apparirà una finestra con la matrice di confusione della classificazione vera (righe) rispetto alla classificazione automatica (colonne).
NOTA: Il richiamo è la percentuale di organismi appartenenti a un gruppo che è stato automaticamente ben riconosciuto, mentre 1-precisione è la percentuale di organismi classificati dall'algoritmo come gruppo che non è riconosciuto (contaminazione in un gruppo). Il richiamo dovrebbe essere superiore al 70% e la contaminazione (precisione 1) dovrebbe essere inferiore al 20%. - Ripetere i passaggi 2.1-2.5 se sono stati creati diversi set di apprendimento e si devono ottenere il richiamo e la precisione 1 di ciascuno.
NOTA: Se sono stati creati diversi set di apprendimento, scegliere quello con il maggior richiamo (buon riconoscimento) e precisione (bassa contaminazione) del gruppo di interesse (cioè macroinvertebrati) per testare la previsione automatica di un set di campioni nella fase successiva.
- Predizione dell'identificazione di macroinvertebrati
NOTA: utilizzare il set di apprendimento selezionato per stimare l'identità di tutti gli oggetti in un sottoinsieme di campioni utilizzando il software di identificazione automatica con un algoritmo di foresta casuale.- Vai al software di identificazione automatica e fai clic su Analisi dei dati.
- In Seleziona file di apprendimento selezionare il file del set di apprendimento da PID_process > set di apprendimento che deve essere utilizzato per la stima.
- In Seleziona file di esempio, selezionare dalla cartella PID_results i campioni (file PID) che verranno previsti.
NOTA: elaborare un massimo di 20 file pid contemporaneamente per evitare errori relativi a problemi di memoria. Se vengono elaborati troppi file .pid contemporaneamente, il processo mostrerà una fine corretta ma potrebbe non essere elaborato correttamente e potrebbe verificarsi un errore nei passaggi successivi durante l'elaborazione con il software di elaborazione delle immagini. - In Selezionare un metodo scegliere il metodo Foresta casuale . Selezionare Salva risultati dettagliati per ogni campione. In Variabili originali, deselezionare le variabili di posizione (X, Y, XM, YM, BX, BY e Altezza). In Variabili personalizzate, selezionare solo ESD.
- Fare clic su Avvia analisi e salvare i risultati come Analysis_name.txt nella cartella PID_process > Previsione.
- Convalida manuale
NOTA: un esperto convalida manualmente la previsione del passaggio precedente per riclassificare gli oggetti classificati erroneamente nella categoria corretta.- Copiare i file di Analysis_sample_dat1.txt da convalidare dalla cartella PID_process > Prediction alla cartella PID_process > Pid_results.
- Vai al software di elaborazione delle immagini e seleziona ESTRAI vignette in cartelle in base a PREDICTION o VALIDATION. Quindi, selezionare Usa file PREDICTED dalla cartella "pid_results". Mantenere le impostazioni predefinite e premere OK.
- Il software crea una cartella chiamata sample_yyyymmdd_hhmm_to_validate con gli oggetti previsti nella cartella PID_process > vignette ordinate.
- Passare alla cartella PID_process > Vignette ordinate e copiare la cartella sample_yyyymmdd_ hhmm_to_validate. Sostituire il nome della cartella _to convalidare con _validated.
- Per convalidare manualmente la classificazione automatica, aprire il sample_yyyymmdd_ hhmm_validated della cartella ed esaminare tutte le vignette di ogni sottocartella (categoria) per identificare se sono presenti oggetti classificati in modo errato. Quando un oggetto è classificato in modo errato, trascinare la vignetta utilizzando il mouse sulla cartella (categoria) corretta.
- Vai al software di elaborazione delle immagini e seleziona CARICA identificazioni da vignette ordinate. Mantieni le impostazioni predefinite e seleziona yyyymmdd_hhmm_name_validated da elaborare.
- Passare a PID_process > Pid_results > Dat1_validated, dove sono stati creati un file denominato Id_from_sorted_vignettes_yyyymmdd_hhmm.txt e un file di .txt per ciascuno degli esempi convalidati (sample_tot_1_dat1.txt).
NOTA: questi file .txt contengono una nuova colonna che presenta la previsione, denominata pred_valid_Id_yyyymmdd_hhmm, che specifica la classificazione di esperti di ciascun oggetto (ad esempio, la classificazione convalidata). Nuove categorie (ad esempio, categorie tassonomiche più fini) potrebbero essere create a questo punto, durante la convalida. Tuttavia, mantenere il nome della categoria originale nel nuovo nome (ad esempio, macroinvertebrate_chironomidae). Ciò consente di ripercorrere la categoria originale quando si calcola il richiamo e la precisione e di raggruppare facilmente tutti i macroinvertebrati per calcolare i parametri della struttura dimensionale della comunità (cioè lo spettro dimensionale e la diversità dimensionale). Il file di testo fornisce i dati associati a ciascun oggetto, inclusi gli assi minore e maggiore utilizzati per ottenere il volume ellissoidale di ciascun organismo come misura delle dimensioni corporee individuali. Inoltre, le ultime due colonne della tabella contengono le categorie previste e validate di ciascun oggetto (riga), che consentono di calcolare, per categoria, il richiamo e la precisione del set di apprendimento sul sottoinsieme di campioni.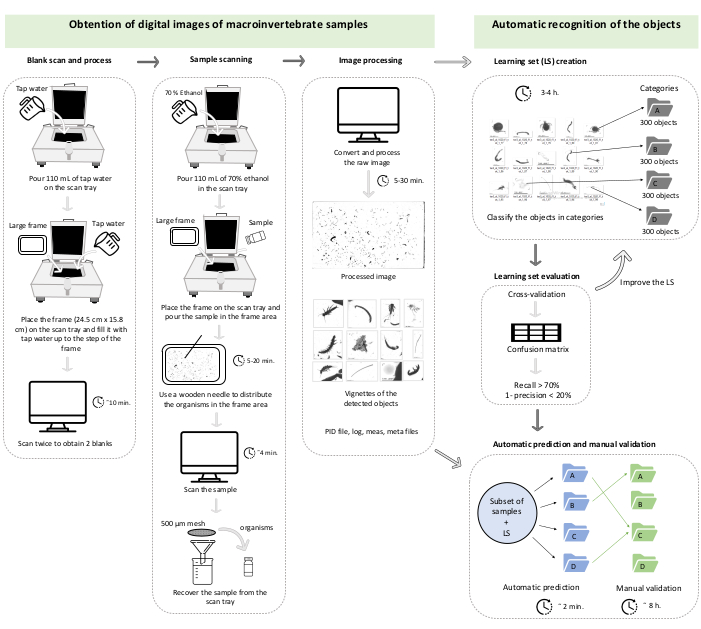
Figura 1: Diagramma di lavoro che rappresenta la sezione 1 e la sezione 2 del protocollo. I tempi sono illustrativi e potrebbero cambiare a seconda del computer, dell'abbondanza di vignette da elaborare e del numero di categorie del set di apprendimento. Questo caso corrisponde alla convalida di un set di apprendimento di tre categorie su un set di 42 sottocampioni (in totale, 47.473 vignette). Fare clic qui per visualizzare una versione ingrandita di questa figura.
3. Calcolo della distribuzione delle dimensioni individuali, degli spettri delle dimensioni e delle metriche delle dimensioni
NOTA: i calcoli menzionati in questa sezione sono stati eseguiti utilizzando Matlab (vedere lo script come file supplementare 1).
- Distribuzione dimensionale individuale
- L'ultima colonna del file Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt contiene la classificazione convalidata degli oggetti. Selezionare solo gli oggetti classificati come macroinvertebrati per rappresentare la loro distribuzione dimensionale individuale nel campione.
NOTA: La dimensione corporea individuale corrisponde al volume ellissoidale degli organismi macroinvertebrati. Il sistema fornisce misurazioni in pixel. - Concatenare i vettori con le misure dimensionali di entrambe le scansioni, perché ogni frazione ha un diverso esponente di sottocampionamento. Prima della concatenazione, correggere il frazionamento replicando i vettori di dimensione tutte le volte che il sottocampione corrispondente è stato frazionato.
NOTA: questo passaggio è necessario se una scansione corrisponde a una frazione di un campione (cioè grossolana o fine). - Calcola il volume ellissoidale dagli assi maggiore (M) e minore (m) degli ellissoidi prolati con le stesse aree di pixel degli organismi. Prima di calcolare il volume ellissoidale, convertire gli assi maggiore (M) e minore (m) da pixel a millimetri (mm) con il seguente fattore di conversione (cf):
1 pixel = 2.400 dpi
1 pollice = 25,4 mm
cf = 25.4/2400
Il volume ellissoidale (ellipVol con unità in mm3) corrisponde a: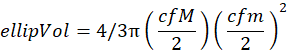
- Rappresentare la funzione di densità di probabilità della distribuzione dimensionale individuale sulla scala log2 .
- L'ultima colonna del file Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt contiene la classificazione convalidata degli oggetti. Selezionare solo gli oggetti classificati come macroinvertebrati per rappresentare la loro distribuzione dimensionale individuale nel campione.
- Diversità delle taglie
- Calcola la diversità dimensionale (Sd) seguendo Quintana et al. (2008)8, come in García-Comas et al. (2016)35:
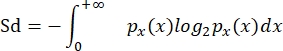
dove p x(x) è la funzione di densità di probabilità di dimensione x, e x rappresenta log2 (ellipVol). Questa misura è, quindi, l'indice di diversità di Shannon adattato a una misura continua, come la distribuzione delle dimensioni individuali in una comunità.
- Calcola la diversità dimensionale (Sd) seguendo Quintana et al. (2008)8, come in García-Comas et al. (2016)35:
- Spettro dimensionale del biovolume normalizzato (NBSS)
- Definire le classi dimensionali dell'NBSS, stabilendo il limite inferiore dello spettro come il quantile 0,01 della distribuzione dimensionale dei macroinvertebrati nei campioni e creando classi di dimensioni mediante una scala geometrica di base 2 fino a comprendere l'organismo più grande nei campioni.
NOTA: la larghezza della classe di dimensione aumenta con le dimensioni per tenere conto della maggiore variabilità associata a dimensioni maggiori. L'NBSS delle comunità di macroinvertebrati analizzate qui aveva 14 classi di dimensioni (Tabella 1). - Ottenere il biovolume normalizzato dividendo il biovolume totale in ciascuna classe di dimensione per la larghezza della classe di dimensione.
- Definire le classi dimensionali dell'NBSS, stabilendo il limite inferiore dello spettro come il quantile 0,01 della distribuzione dimensionale dei macroinvertebrati nei campioni e creando classi di dimensioni mediante una scala geometrica di base 2 fino a comprendere l'organismo più grande nei campioni.
- Pendenza dello spettro dimensionale
- Calcolare la pendenza lineare dell'NBSS.
NOTA: La pendenza (μ) viene calcolata in base alla relazione tra il log 2 (classe dimensionale mid-point) e il log2 (biomassa normalizzata) nelle classi dimensionali maggiori della modalità, ignorando quelle vuote (in questo studio, le classi di dimensione da 3 a 14).
- Calcolare la pendenza lineare dell'NBSS.
| Limiti della classe di dimensione (mm3) | Classe di dimensione punto medio (mm3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
Tabella 1: Classi dimensionali dello spettro dimensionale normalizzato della biomassa (NBSS). La tabella mostra anche i limiti delle 15 classi dimensionali e i punti medi della classe dimensionale degli organismi.
Risultati
Acquisizione di immagini digitali di campioni di macroinvertebrati
Sfumature di scansione: deposizione di etanolo nel vassoio di scansione
Durante il test del sistema per i macroinvertebrati, diverse scansioni erano di scarsa qualità. Un'area scura satura sullo sfondo ha impedito la normale elaborazione dell'immagine e la misurazione delle dimensioni individuali dei macroinvertebrati (Figura 2). Diverse ragioni sono state fornite per la comparsa di ar...
Discussione
L'adattamento della metodologia descritta da Gorsky et al. 2010 per i macroinvertebrati fluviali consente un'elevata accuratezza di classificazione nella stima della struttura dimensionale della comunità nei macroinvertebrati d'acqua dolce. I risultati suggeriscono che il protocollo può ridurre il tempo per stimare la struttura dimensionale individuale in un campione a circa 1 ora. Pertanto, il protocollo proposto ha lo scopo di promuovere l'uso di routine degli spettri dimensionali dei macroinvertebrati come bioindica...
Divulgazioni
Gli autori non dichiarano potenziali interessi concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto dal Ministero spagnolo della Scienza, dell'Innovazione e dell'Università (numero di sovvenzione RTI2018-095363-B-I00). Ringraziamo i membri del CERM-UVic-UCC Èlia Bretxa, Anna Costarrosa, Laia Jiménez, María Isabel González, Marta Jutglar, Francesc Llach e Núria Sellarès per il loro lavoro nel campionamento dei campi di macroinvertebrati e nella selezione di laboratorio e David Albesa per aver collaborato alla scansione dei campioni. Ringraziamo infine Josep Maria Gili e l'Institut de Ciències del Mar (ICM-CSIC) per l'uso delle strutture di laboratorio e del dispositivo di scansione.
Materiali
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
Riferimenti
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon