JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
강변 거대 무척추 동물의 군집 크기 구조를 결정하기위한 자동 이미지 처리
요약
이 기사는 반자동 이미징 절차를 사용하여 저서 강 거대 무척추 동물에 해당하는 디지털화 된 물체를 스캔, 감지, 정렬 및 식별하는 적응 된 프로토콜의 생성을 기반으로합니다. 이 절차를 통해 약 1 시간 내에 거대 무척추 동물 군집의 개별 크기 분포 및 크기 메트릭을 획득 할 수 있습니다.
초록
신체 크기는 자연 공동체에서 섭동의 영향을 평가하기위한 생물학적 지표로 사용될 수있는 중요한 기능적 특성입니다. 공동체 크기 구조는 분류군과 생태계 전반에 걸친 인위적 섭동을 포함하여 생물학적 및 비 생물 적 구배에 반응합니다. 그러나 저서 거대 무척추 동물과 같은 작은 몸체의 유기체 (예 : >500 μm에서 수 센티미터 길이)의 수동 측정은 시간이 많이 걸립니다. 공동체 규모 구조의 추정을 촉진하기 위해 여기에서는 담수 생태계의 생태 상태를 평가하는 데 가장 일반적으로 사용되는 생물학적 지표 중 하나 인 보존 된 강 거대 무척추 동물의 개별 신체 크기를 반자동으로 측정하는 프로토콜을 개발했습니다. 이 프로토콜은 물 샘플용으로 설계된 스캐닝 시스템으로 해양 메조동물성 플랑크톤을 스캔하기 위해 개발된 기존 방법론에서 채택되었습니다. 프로토콜은 세 가지 주요 단계로 구성됩니다 : (1) 강 거대 무척추 동물의 하위 샘플 (미세하고 거친 샘플 크기 분율)을 스캔하고 디지털화 된 이미지를 처리하여 각 이미지에서 감지 된 각 물체를 개별화합니다. (2) 인공 지능을 통해 학습 세트를 생성, 평가 및 검증하여 스캔 된 샘플의 찌꺼기 및 인공물로부터 거대 무척추 동물의 개별 이미지를 반자동으로 분리합니다. (3) 거대 무척추 동물 군집의 크기 구조를 묘사합니다. 프로토콜 외에도 이 작업에는 보정 결과가 포함되며 거대 무척추 동물 샘플에 절차를 적용하고 추가 개선을 고려하기 위한 몇 가지 과제와 권장 사항을 열거합니다. 전반적으로이 결과는 강 거대 무척추 동물의 자동 신체 크기 측정을위한 제시된 스캐닝 시스템의 사용을 뒷받침하며 크기 스펙트럼의 묘사가 담수 생태계의 신속한 생물학적 평가를위한 귀중한 도구임을 시사합니다.
서문
저서 거대 무척추 동물은 수역의 생태 상태를 결정하기위한 생물학적 지표로 널리 사용됩니다1. 거대 무척추 동물 군집을 설명하는 대부분의 지표는 분류 학적 메트릭에 중점을 둡니다. 그러나 신체 크기를 통합하는 새로운 생물학적 평가 도구는 분류 학적 접근법 2,3에 대한 대안 또는 보완적인 관점을 제공하는 것이 좋습니다.
신체 크기는 신진 대사, 성장, 호흡 및 운동과 같은 다른 중요한 특성과 관련된 메타 특성으로 간주됩니다4. 또한, 신체 크기는 영양 위치와 상호 작용을 결정할 수 있습니다5. 공동체의 크기 등급에 의한 개별 신체 크기와 정규화 된 바이오 매스 (또는 풍부) 사이의 관계는 크기 스펙트럼6 으로 정의되며 개별 크기가 로그 스케일7에서 증가함에 따라 정규화 된 바이오 매스의 선형 감소의 일반적인 패턴을 따릅니다. 이 선형 관계의 기울기는 이론적으로 광범위하게 연구되어 왔으며 생태계 전반에 걸친 경험적 연구는이를 공동체 규모 구조의 생태 학적 지표로 사용했습니다4. 생물 다양성 - 생태계 기능 연구에서 성공적으로 사용 된 공동체 크기 구조의 또 다른 합성 지표는 공동체 크기 다양성이며, 이는 크기 스펙트럼 또는 그 유사체의 크기 등급의 섀넌 지수로 표현되며, 이는 개별 크기 분포8를 기반으로 계산됩니다.
담수 생태계에서 서로 다른 동물 그룹의 크기 구조는 환경 구배 9,10,11 및 인위적 섭동 12,13,14,15,16에 대한 생물 공동체의 반응을 평가하기위한 운동 실조 지표로 사용됩니다. 거대 무척추 동물도 예외는 아니며, 그 크기 구조는 환경 변화17,18 및 채광 19, 토지 이용 20 또는 질소 (N) 및 인 (P) 농축20,21,22와 같은 인위적 섭동에도 반응합니다. 그러나 커뮤니티 규모 구조를 설명하기 위해 수백 명의 개인을 측정하는 것은 지루하고 시간이 많이 소요되는 작업이며 시간 부족으로 인해 실험실에서 일상적인 측정으로 피하는 경우가 많습니다. 따라서, 표본을 분류하고 측정하기 위한 여러 반자동 또는 자동 이미징 방법이 개발되었다 23,24,25,26. 그러나 이러한 방법의 대부분은 유기체의 개별 크기보다 분류 학적 분류에 중점을두고 있으며 모든 종류의 거대 무척추 동물에 사용할 준비가되어 있지 않습니다. 해양 플랑크톤 생태학에서, 스캐닝 이미지 분석 시스템은 동물성 플랑크톤 군집27,28,29,30,31의 크기 및 분류 학적 구성을 결정하기 위해 광범위하게 사용되었다. 이 기기는 전 세계 여러 해양 연구소에서 찾을 수 있으며 보존된 동물성 플랑크톤 샘플을 스캔하여 전체 샘플의 고해상도 디지털 이미지를 얻는 데 사용됩니다. 본 프로토콜은 새로운 장치를 만드는 데 투자하지 않고 강에서 거대 무척추 동물 군집 크기 스펙트럼을 신속하게 자동 추정하기 위해이 기기의 사용을 조정합니다.
프로토콜은 샘플을 스캔하고 전체 이미지를 처리하여 샘플에 있는 물체의 단일 이미지(즉, 비네트)를 자동으로 얻는 것으로 구성됩니다. 모양, 크기 및 그레이 레벨 피처의 여러 측정값은 각 개체의 특성을 지정하고 개체를 범주로 자동 분류한 다음 전문가가 검증할 수 있도록 합니다. 각 유기체의 개별 크기는 픽셀 단위로 측정 된 유기체의 면적에서 파생 된 타원체 생물량 (mm3)을 사용하여 계산됩니다. 이를 통해 샘플의 크기 스펙트럼을 신속하게 얻을 수 있습니다. 우리가 아는 한,이 스캐닝 이미징 시스템은 메조 동물성 플랑크톤 샘플을 처리하는 데만 사용되었지만이 장치는 잠재적으로 담수 저서 거대 무척추 동물과 함께 작업 할 수 있습니다.
따라서이 연구의 전반적인 목표는 이전에 해양 메조 동물성 플랑크톤 27,32,33과 함께 사용 된 기존 프로토콜을 적용하여 보존 된 강 거대 무척추 동물의 개별 크기를 신속하게 얻는 방법을 도입하는 것입니다. 절차는 물 샘플을 스캔하기 위해 스캐닝 장치와 스캔한 이미지를 처리하기 위한 3개의 개방형 소프트웨어로 작동하는 반자동 접근 방식을 사용하는 것으로 구성됩니다. 커뮤니티 크기 구조 및 관련 크기 메트릭을 자동으로 획득하기 위해 디지털화된 강 거대 무척추동물을 스캔, 감지 및 식별하는 적응된 프로토콜이 여기에 제시됩니다. 효율성을 높이기위한 절차 및 지침에 대한 평가는 북동부 (NE) 이베리아 반도 (Ter, Segre-Ebre 및 Besòs)의 3 개 분지에서 수집 한 강변 거대 무척추 동물 샘플의 42 스캔 이미지를 기반으로 제공됩니다.
샘플은 스페인 정부 100의 건너 가능한 강에서 저서 강 거대 무척추 동물의 현장 샘플링 및 실험실 분석을위한 프로토콜에 따라34m 강 뻗기에서 수집되었습니다. 샘플은 다중 서식지 조사 후 서버 샘플러(프레임: 0.3m x 0.3m, 메쉬: 250μm)로 수집되었습니다. 실험실에서 샘플을 5mm 및 500μm 메쉬를 통해 세척하고 체질하여 두 개의 하위 샘플을 얻었습니다 : 거친 하위 샘플 (5mm 메쉬)과 미세 하위 샘플 (500μm 메쉬), 별도의 바이알에 보관하고 70 % 에탄올에 보존했습니다. 샘플을 두 개의 크기 분수로 분리하면 큰 유기체가 작은 유기체보다 희귀하고 적기 때문에 군집 크기 구조를 더 잘 추정할 수 있습니다. 그렇지 않으면 스캔된 샘플은 큰 크기 분율의 편향된 표현을 갖습니다.
프로토콜
참고 : 여기에 설명 된 프로토콜은 Gorsky et al.27이 해양 중동물 플랑크톤에 대해 개발 한 시스템을 기반으로합니다. 스캐너(ZooSCAN), 스캐닝 소프트웨어(VueScan 9x64[9.5.09]), 이미지 처리 소프트웨어(Zooprocess, ImageJ) 및 자동 식별 소프트웨어(Plankton Identifier) 단계에 대한 구체적인 설명은 이전 참조(32, 33)에서 찾을 수 있다. 중동물성 플랑크톤에 대한 저서 거대 무척추 동물의 크기를 가장 잘 조정하려면 원래 프로토콜 32,33에 따라 프로젝트가 생성되면 구성 파일에서 최소 크기(minsizeesd_mm)의 매개변수를0.3mm로, 최대 크기(maxsizeesd_mm)의 매개변수를 100mm로 변경합니다. 프로토콜을 따르는 데 도움이 되도록 작업 차트에 요약되어 있습니다(그림 1). 생성된 프로젝트는 컴퓨터의 C 폴더에 저장되며 PID_process, Zooscan_back, Zooscan_check, Zooscan_config, Zooscan_meta, Zooscan_results 및 Zooscan_scan 폴더에 구성됩니다. 각 폴더는 서로 다른 소프트웨어 응용 프로그램이 프로토콜의 다음 단계에서 사용하는 여러 하위 폴더로 구성됩니다.
1. 거대 무척추 동물 샘플에 대한 디지털 이미지 획득
- 블랭크 스캔 및 처리
참고: 스캔하기 전에 매일 두 개의 빈 이미지를 만들어 같은 날 스캔한 이미지를 처리하는 동안 백그라운드 스캔을 추출합니다.- 스캐너를 켜고 이중 위치에서 조명을 켜서 상단과 하단에서 백색광을 투사합니다.
알림: 중동물성 플랑크톤 샘플을 스캔할 때 위쪽 조명 방향이 사용되지만 거대 무척추동물은 더 불투명하기 때문에 조명을 이중 위치로 전환하는 것이 좋습니다. - 스캔 트레이를 청소하고 수돗물로 헹굽니다.
- 유리가 덮일 때까지 실온(RT)에 보관된 수돗물 110mL를 스캔 트레이에 붓습니다. 큰 프레임(24.5cm x 15.8cm)을 스캔 트레이의 올바른 위치(스캔 트레이의 왼쪽 상단 모서리 포함)에 놓고 스캔 이미지가 변경되는 반월판 효과를 방지하기 위해 프레임 단계가 덮일 때까지 수돗물을 채웁니다. 스캐너 덮개를 닫습니다.
알림: 응결 및 기포 형성을 피하기 위해 RT에서 물을 사용하십시오. 빛 반사를 피하기 위해 자국이나 물방울 없이 프레임을 청소하십시오. - 이미지 처리 소프트웨어로 이동하여 작업 프로젝트를 선택하고 배경 이미지 스캔(변환)을 클릭합니다.
- 스캔 소프트웨어로 이동하여 미리보기를 클릭하십시오. 스캔한 이미지를 미리 보고 선이나 점이 없는지 확인한 다음 다른 스캔을 시작하기 전에 최소 30초 동안 기다리십시오. 스캔( Scan)을 클릭하고 두 번째 스캔 전에 지침 창에서 OK 를 눌러 스캔 소프트웨어에서 이미지 처리 소프트웨어로 데이터를 전송합니다.
참고: 두 번 스캔하여 공백을 구성할 두 개의 백그라운드 스캔을 얻습니다. 이 단계는 샘플 처리를 시작하기 전에 매일 한 번 수행되며 이미지는 Zooscan_back 폴더에 저장됩니다. - 스캔을 마친 후 스캔 소프트웨어를 닫습니다.
- 스캐너를 켜고 이중 위치에서 조명을 켜서 상단과 하단에서 백색광을 투사합니다.
- 시료 준비 및 스캐닝
주의 : 에탄올은 가연성 액체이며 심각한 눈 손상 / 자극을 유발할 수 있습니다.- 샘플 메타데이터를 입력합니다. 이미지 처리 소프트웨어로 이동하여 샘플 메타데이터 채우기를 선택합니다. 샘플 ID를 입력하고 확인을 클릭한 다음 메타데이터를 입력합니다.
참고 : 메타 파일은 mesozooplankton 샘플 용으로 특별히 생성되었으므로 저서 거대 무척추 동물 샘플링 방법론에 맞지 않지만 스캔 전에 파일의 모든 필드를 채워야하며 그렇지 않으면 오류 플래그가 나타납니다. - 유리가 덮일 때까지 70% 에탄올 110mL를 스캔 트레이에 붓고 큰 프레임(24.5cm x 15.8cm)을 스캔 트레이의 왼쪽 상단 모서리에 놓습니다.
참고 : 거대 무척추 동물은 에탄올에 보존되므로 물 대신 에탄올로 작업하십시오. 물에서는 스캔 트레이에 떠 있고 표류하여 선명한 이미지를 방해하여 신뢰할 수 있는 크기 측정을 할 수 있습니다. 에탄올은 응축 및 기포 형성을 피하기 위해 RT에서 보존되어야합니다. - 거대 무척추 동물 샘플을 프레임 가장자리가있는 스캔 트레이에 붓고 필요한 경우 프레임 단계를 더 많은 에탄올로 덮습니다.
알림: 유기체가 떠 다니거나 표류하는 것을 피하기 위해 너무 많은 에탄올을 첨가하지 마십시오. - 적절한 이미지 처리를 위해 트레이 중앙에 가장 큰 개체를 배치하고 프레임 영역 전체에 샘플을 균질화하고 나무 바늘을 사용하여 부유 유기체를 가라앉힙니다.
참고: 하위 샘플에 1,000명 이상의 개체가 포함된 경우 하위 샘플을 두 개 이상의 분획으로 나누어 스캔한 이미지에서 유기체가 접촉하는 것을 최소화하고 분획을 별도로 스캔합니다. - 나무 바늘을 사용하여 만지는 유기체와 프레임 가장자리에 닿는 유기체를 분리하십시오.
참고: 이 단계는 5-20분 정도 소요됩니다. 만지는 유기체는 소프트웨어에 의해 단일 개체로 간주됩니다. 따라서 이러한 경우 계산 된 개별 크기는 실제 단일 유기체와 일치하지 않으며 군집 크기 구조의 추정치를 편향시킬 수 있습니다. 이미지 처리 소프트웨어로 이미지를 편집하여 이미지를 분리 할 가능성이 있지만이 추가 단계에는 최소 1.5 시간의 재 처리가 필요합니다. 따라서 수동 분리를 적극 권장합니다. - 샘플을 스캔하려면 스캐너 덮개를 닫고 이미지 처리 소프트웨어로 이동하여 작업 프로젝트를 선택하고 Zooscan으로 샘플 스캔(보관용, 프로세스 없음)을 클릭합니다.
- 샘플을 선택하고 지침을 따릅니다.
- 스캔 소프트웨어로 이동하여 미리보기를 클릭하십시오. 스캔한 이미지를 미리 보고 선이나 점이 없는지 확인한 다음 30초 이상 기다렸다가 다른 스캔을 시작합니다.
- 최소 30초 후 스캔 소프트웨어에서 스캔 버튼을 클릭합니다.
참고: 스캔 소프트웨어에서 스캔(Scan)을 누른 후 이미지 처리 소프트웨어에서 OK를 누릅니다. 컴퓨터 키보드의 키를 누르지 말고 스캔하는 동안 스캔이 진동하지 않도록 하십시오. Zooscan_scan > _raw 폴더에는 세 개의 파일이 생성됩니다: (i) 태그가 지정된 이미지 파일 형식(.tif)(16비트); (ii) 스캐닝 파라미터에 대한 정보를 기록하는 LOG (.txt)라는 표준 텍스트 문서; (iii) 샘플링 방법에 대한 정보가 포함 된 META (.txt)라는 표준 텍스트 문서. - 원시 스캔이 올바른지 확인합니다.
참고: 스캔에 밝은 줄무늬 또는 기타 눈에 보이는 문제가 있는 경우 다음 단계에서 문제를 방지하기 위해 스캔을 반복하는 것이 좋습니다.
- 샘플 메타데이터를 입력합니다. 이미지 처리 소프트웨어로 이동하여 샘플 메타데이터 채우기를 선택합니다. 샘플 ID를 입력하고 확인을 클릭한 다음 메타데이터를 입력합니다.
- 시료 회수
- 프레임을 제거하고 부착된 거대 무척추 동물을 회수하기 위해 70% 에탄올로 채워진 스퀴즈 병을 사용하여 스캔 트레이 위로 헹굽니다.
- 스캐너의 윗부분을 들어 올려 트레이에서 스캔 검색 깔때기를 통해 비커로 모든 유기체와 에탄올을 검색합니다. 스캐너의 윗부분을 들어 올린 상태에서 스퀴즈 병으로 트레이를 헹구어 남은 유기체를 따라 쓸어냅니다.
- 비커에서 표본과 에탄올을 500μm 메쉬를 통과시켜 무척추 동물을 메쉬에 유지하고 70 % 에탄올이 함유 된 바이알에 다시 보관합니다.
- 모든 표본이 바이알에서 회수되면 수돗물로 트레이를 청소하십시오.
알림: 샘플 사이에 수돗물로 트레이를 세척하여 이미지 처리를 변경하는 에탄올 침전을 최소화하십시오. 에탄올 사용과 관련된 잠재적 인 손상을 피하기 위해 수돗물로 프레임을 헹굽니다. 하루가 끝나면 수돗물을 사용하여 트레이를 청소하고 긁힘을 방지하기 위해 종이로 부드럽게 말리십시오.
- 이미지 처리
- 이미지 처리 소프트웨어로 이동하여 배치 모드에서 이미지 및 유기체를 변환 및 처리 한 다음 변환 및 처리 이미지 및 입자 (RAW 폴더의 이미지)를 선택하십시오. 기본 설정을 유지하고 확인을 클릭하십시오. 프로세스가 끝날 때 NORMAL END 가 나타납니다.
참고: PID 파일과 스캔한 이미지에서 감지된 모든 물체에 해당하는 비네팅(공동 사진 그룹 파일 [.jpg])이 Zooscan_scan > _work 폴더에 생성됩니다. PID 파일은 모든 메타 데이터 (메타 파일), 로그 파일과 관련된 기술 데이터 및 이미지에서 감지 된 모든 객체의 36 개의 측정 변수가있는 테이블을 저장하는 단일 파일입니다. 측정된 변수는 그레이 레벨, 프랙탈 치수, 모양 및 크기의 다양한 추정치에 해당합니다. 크기 추정에 사용할 수 있는 변수는 개체와 동일한 면적을 가진 타원의 면적과 주 축과 보조 축입니다(프로토콜의 섹션 3 참조). 처리 시간은 이미지 밀도 및 컴퓨터 특성에 따라 다르며 다음 샘플을 복구하고 준비하는 동안 샘플 간에 시작할 수 있습니다. 그렇지 않으면 야간에 배치 모드로 매일 스캔 한 샘플 처리를 시작하고 다음날 아침에 적절한 이미지 처리를 확인하는 것이 좋습니다. - 이미지 처리 소프트웨어를 사용하거나 Zooscan_scan > _work에 있는 마스크 이미지(msk1.gif로 종료됨)를 확인하여 처리된 이미지의 배경이 샘플 이미지에서 적절하게 제거되었는지 확인합니다. 배경에 포화 영역이 있거나 많은 점이 있는 경우 고품질 이미지를 보장하기 위해 스캔을 반복하는 것이 좋습니다.
참고: 백그라운드에서 포화 영역을 방지하려면 에탄올로 스캔할 때마다 스캔 트레이를 수돗물로 헹구어야 합니다. 또한 (1) 스캔된 개인의 수를 줄이는 것(샘플을 분획하고 다른 접힘으로 스캔함으로써); (2) 큰 유기체가 스캔 트레이의 중앙에 배치되었는지 확인하십시오. (3) 깨끗하고 여과 된 에탄올을 사용하십시오. (4) 샘플의 더러움을 줄입니다. (5) 주사를 위한 에탄올의 부피가 적절한지 확인하십시오. (6) 샘플 미리보기와 스캔 사이의 지연이 30초 이상인지 확인합니다.
- 이미지 처리 소프트웨어로 이동하여 배치 모드에서 이미지 및 유기체를 변환 및 처리 한 다음 변환 및 처리 이미지 및 입자 (RAW 폴더의 이미지)를 선택하십시오. 기본 설정을 유지하고 확인을 클릭하십시오. 프로세스가 끝날 때 NORMAL END 가 나타납니다.
- 만지는 유기체의 분리
참고: 접촉하는 유기체가 있는 비네트가 여러 개 있는 경우 커뮤니티 크기 구조의 적절한 추정을 보장하기 위해 접촉하는 유기체의 이미지를 다른 유기체 및/또는 섬유/파편과 분리해야 합니다.- 이미지 처리 소프트웨어로 이동하여 여러 개체가 있는 비네팅을 감지합니다. 비네팅을 사용하여 분리를 선택하고 OK를 누릅니다. 구성 선택 창에서 기본 설정을 유지하고 확인을 클릭합니다.
- 비네팅에서 분리 창에서 기본 설정을 유지하고 비네팅에 윤곽선 추가를 추가로 선택한 다음 편집할 샘플을 선택합니다.
- 마우스로 선을 그려 팝업되는 각 비네트에서 만지는 유기체를 분리합니다(롤 버튼을 눌러 그립니다). 비네트의 분리가 완료되면 창의 오른쪽 상단 모서리에 있는 X 버튼을 클릭하고 YES 를 눌러 다음 항목을 처리합니다. NO 를 눌러 변경 사항을 종료하고 저장합니다. 프로세스가 끝나면 모든 것이 정확하면 NORMAL END 가 나타납니다.
- 분리 후 이미지를 다시 처리하여 업데이트된 개체 데이터를 가져옵니다. 이미지 처리 소프트웨어로 이동하여 PROCESS(변환된) 이미지(Process One)를 클릭하고 WORK 하위 폴더의 처리된 이미지에서 파티클 다시 처리를 선택합니다. 샘플을 선택하고 단일 이미지 프로세스 창에서 기본 설정을 유지하고 분리 마스크 작업(CREATE-MODIFY-INCLUDE)을 선택한 다음 확인을 클릭합니다. 프로세스가 끝나면 모든 것이 정확하면 NORMAL END 가 나타납니다.
- 분리 제어 창에서 OK를 눌러 처리하기 전에 윤곽선이있는 이미지를 저장합니다. 이전 이미지가 있으면 대체됩니다.
- 분리 제어 마스크 창에서 필요한 경우 편집을 선택하여 마우스를 사용하여 마스크에 분리 선을 추가하여 비네팅을 사용하여 분리 단계에서 이전에 나타나지 않은 접촉 유기체를 분리합니다. 완료되면 프로세스를 종료하고 분리 마스크 제어 창에서 예를 선택하여 마스크를 적용합니다. 프로세스가 끝나면 모든 것이 정확하면 NORMAL END가 나타납니다.
참고: 분리 마스크로 샘플을 재처리하는 것은 시간이 많이 걸립니다(샘플당 1.5시간 이상 걸릴 수 있음). 이 추가 단계를 피하기 위해 1.2.5단계에서 필요한 시간을 할애하는 것이 좋습니다.
2. 물체 자동 인식
참고: 검출된 물체의 신원을 자동으로 예측하는 학습 세트를 생성하여 샘플의 파편에서 유기체를 분리합니다.
- 학습 집합 생성
- 학습 세트의 범주를 Zooscan_scan > _work 에서 PID_process > Unsorted_vignettes_pid로 만드는 데 사용할 이미지와 연결된 이미지 및 .pid 파일을 복사합니다.
참고: 분류군 다양성이 높고 샘플링 부위 및/또는 샘플링 계절이 다른 샘플의 하위 집합을 선택하여 샘플에서 유기체의 최대 대표성을 보장합니다. - PID_process > 학습 집합 폴더에서 새 학습 집합의 이름(예: yyyymmdd_raw_LS)으로 하위 폴더를 만들고 그 안에 학습 집합의 각 범주(예: 거대 무척추 동물, 파편, 기타 무척추 동물)에 해당하는 하위 폴더를 만듭니다.
참고: 강 거대 무척추 동물 샘플의 군집 크기 구조를 효율적으로 얻으려면 거대 무척추 동물, 기타 무척추 동물 및 파편의 세 가지 범주를 기반으로 한 학습 세트를 사용하는 것이 좋습니다. 이 학습 세트는 기본적으로 유기체에 해당하는 물체의 비 네트와 파편 (예 : 섬유, 입자 또는 사상 조류)에 해당하는 물체를 분리합니다. - 이미지 처리 소프트웨어(고급 모드만 해당)로 이동하여 PLANKTON 식별자에 대한 비네트 추출(훈련용 정렬되지 않은 비네트)을 선택합니다. 기본 옵션을 유지하고 윤곽선 추가 상자를 선택합니다.
- 자동 식별 소프트웨어로 이동하여 학습을 클릭하고 새 학습 세트에 대해 생성된 하위 폴더 PID_process > Learning_set 선택한 다음(2.1.2단계) 확인을 누릅니다.
- 열린 창의 왼쪽 섹션 (정렬되지 않은 엄지 손가락)에서 정렬되지 않은 vignettes_pid 폴더를 선택하십시오. 비네팅을 선택하고 정렬되지 않은 엄지 단추에서 오른쪽 섹션인 Sorted Thumbs에 있는 해당 범주의 폴더로 마우스로 드래그하여 각 개체를 정의된 범주로 분류합니다. 이동된 비네팅은 빨간색 X로 표시됩니다.
알림: 정렬된 thumbs 폴더에 하위 폴더를 만들어 범주를 수동으로 정의하거나 소프트웨어에서 폴더 아이콘을 클릭하여 범주를 만듭니다. 동시에 50개 이상의 비네팅을 이동하지 마십시오. - 선택한 객체(카테고리당 약 300개 객체)로 모든 카테고리가 완료되면 학습 파일 생성을 클릭하고 원하는 이름으로 저장합니다.
참고: 학습 집합은 프로젝트의 PID_process > 학습 집합 폴더에 .pid 파일로 저장됩니다. 다양한 범주 수준(거친 형식에서 미세 형식까지)과 각 범주 내의 개체 수의 균형이 다른 여러 학습 집합을 만들고 테스트하는 것이 좋습니다. 범주 수가 적고 범주당 50개 이상의 개체가 있는 대략적인 학습 집합으로 시작한 다음 각 범주의 개체 수를 늘리거나 더 세부적인 학습 집합을 만듭니다. 범주는 표본 집합의 변동성을 나타내야 합니다.
- 학습 세트의 범주를 Zooscan_scan > _work 에서 PID_process > Unsorted_vignettes_pid로 만드는 데 사용할 이미지와 연결된 이미지 및 .pid 파일을 복사합니다.
- 학습 집합 평가
참고: 자동 식별 소프트웨어와 함께 Random Forest 방법을 사용하여 두 번 접고 다섯 번의 시도로 교차 검증을 수행하여 개체 분류의 혼동 행렬을 얻습니다.- 자동 분류 소프트웨어로 이동하여 데이터 분석을 클릭합니다.
- 학습 파일 선택의 학습 세트에서 생성된 학습 세트 파일을 선택합니다 PID_process >.
- 메서드 선택에서 교차 검증 랜덤 포리스트 메서드를 선택합니다. 원래 변수에서 위치 변수(X, Y, XM, YM, BX, BY, 높이 등)의 선택을 취소합니다. 사용자 정의 변수에서 ESD만 선택합니다.
참고: 이 방법은 학습 집합의 한 무작위 부분을 사용하여 다른 부분(두 겹)을 인식하며, 통계적으로 견고한지 확인하기 위해 5번 반복됩니다. - 분석 시작을 클릭하고 결과를 PID_process > 예측 폴더에 Analysis_name.txt로 저장합니다. 분석이 성공적으로 완료되면 데이터 분석을 종료합니다.
- PID_process > 예측 폴더로 이동하여 교차 유효성 검사 파일을 클릭합니다. 실제 분류(행)와 자동 분류(열)의 혼동 행렬이 있는 창이 나타납니다.
참고: 리콜은 자동으로 잘 인식된 그룹에 속하는 유기체의 백분율인 반면, 1-정밀도는 알고리즘에 의해 인식되지 않는 그룹(그룹의 오염)으로 분류된 유기체의 백분율입니다. 리콜은 70% 이상이어야 하며 오염(1정밀도)은 20% 미만이어야 합니다. - 여러 학습 세트가 생성되고 각 학습 세트의 재현율 및 1-정밀도를 얻어야 하는 경우 2.1-2.5단계를 반복합니다.
참고: 여러 학습 세트가 생성된 경우 관심 그룹(즉, 거대 무척추동물)의 재현율(양호한 인식) 및 정밀도(낮은 오염)가 가장 큰 학습 세트를 선택하여 다음 단계에서 샘플 세트의 자동 예측을 테스트합니다.
- 거대 무척추 동물의 식별 예측
참고: 선택한 학습 세트를 사용하여 랜덤 포리스트 알고리즘이 있는 자동 식별 소프트웨어를 사용하여 샘플의 하위 집합에 있는 모든 객체의 ID를 예측합니다.- 자동 식별 소프트웨어로 이동하여 데이터 분석을 클릭하십시오.
- 학습 파일 선택에서 예측에 사용해야 하는 학습 집합PID_process > 학습 집합 의 학습 집합 파일을 선택합니다.
- 샘플 파일 선택의 PID_results 폴더에서 예측할 샘플(PID 파일)을 선택합니다.
참고: 메모리 문제와 관련된 오류를 방지하기 위해 최대 20개의 .pid 파일을 동시에 처리합니다. 너무 많은 .pid 파일을 동시에 처리하면 프로세스가 올바르게 종료되지만 제대로 처리되지 않을 수 있으며 이미지 처리 소프트웨어로 처리할 때 다음 단계에서 오류가 발생할 수 있습니다. - 메서드 선택에서 랜덤 포리스트 메서드를 선택합니다. 각 샘플에 대한 자세한 결과 저장을 선택합니다. 원래 변수에서 위치 변수(X, Y, XM, YM, BX, BY 및 높이)의 선택을 취소합니다. 사용자 지정 변수에서 ESD만 선택합니다.
- 분석 시작을 클릭하고 결과를 PID_process > 예측 폴더에 Analysis_name.txt로 저장합니다.
- 수동 검증
참고: 전문가는 이전 단계의 예측을 수동으로 검증하여 잘못 분류된 개체를 올바른 범주로 재분류합니다.- 유효성을 검사할 Analysis_sample_dat1.txt 파일을 PID_process > 예측 폴더에서 PID_process > Pid_results 폴더로 복사합니다.
- 이미지 처리 소프트웨어로 이동하여 예측 또는 검증에 따라 폴더에서 비네트 추출을 선택합니다. 그런 다음 " pid_results" 폴더의 예측 파일 사용을 선택합니다. 기본 설정을 유지하고 OK를 누릅니다.
- 소프트웨어는 PID_process > 정렬된 비네트 폴더에 예측된 개체가 있는 sample_yyyymmdd_hhmm_to_validate라는 폴더를 만듭니다.
- PID_process > 정렬된 비네트 폴더로 이동하여 폴더를 sample_yyyymmdd_ hhmm_to_validate 복사합니다. 유효성을 검사_to 폴더 이름을 _validated로 바꿉니다.
- 자동 분류의 유효성을 수동으로 검사하려면 폴더 sample_yyyymmdd_ hhmm_validated 열고 각 하위 폴더(범주)의 모든 비네팅을 검토하여 잘못 분류된 개체가 있는지 확인합니다. 한 개체가 잘못 분류되면 마우스를 사용하여 비네팅을 올바른 폴더(범주)로 끕니다.
- 이미지 처리 소프트웨어로 이동하여 정렬된 비네팅에서 LOAD 식별을 선택합니다. 기본 설정을 유지하고 처리할 yyyymmdd_hhmm_name_validated 선택합니다.
- Id_from_sorted_vignettes_yyyymmdd_hhmm.txt라는 파일과 유효성이 검사된 각 샘플(sample_tot_1_dat1.txt)에 대해 하나의 .txt 파일이 작성된 PID_process > Pid_results > Dat1_validated로 이동합니다.
참고: 이러한 .txt 파일에는 각 개체의 전문가 분류(즉, 검증된 분류)를 지정하는 pred_valid_Id_yyyymmdd_hhmm라는 예측을 제공하는 새 열이 포함되어 있습니다. 새로운 범주(예: 더 미세한 분류학적 범주)는 유효성 검사 중에 이 시점에서 생성될 수 있습니다. 그러나 원래 범주의 이름은 새 이름(예: macroinvertebrate_chironomidae)으로 유지합니다. 이를 통해 재현율 및 정밀도를 계산할 때 원래 범주를 다시 추적하고 모든 거대 무척추 동물을 쉽게 그룹화하여 군집 크기 구조 매개 변수 (즉, 크기 스펙트럼 및 크기 다양성)를 계산할 수 있습니다. 텍스트 파일은 개별 신체 크기의 척도로 각 유기체의 타원체 부피를 얻는 데 사용되는 부 및 주 축을 포함하여 각 개체와 관련된 데이터를 제공합니다. 또한 테이블의 마지막 두 열에는 각 개체(행)의 예측 및 유효성이 검사된 범주가 포함되어 있어 샘플의 하위 집합에 대한 학습 집합의 재현율과 정밀도를 범주별로 계산할 수 있습니다.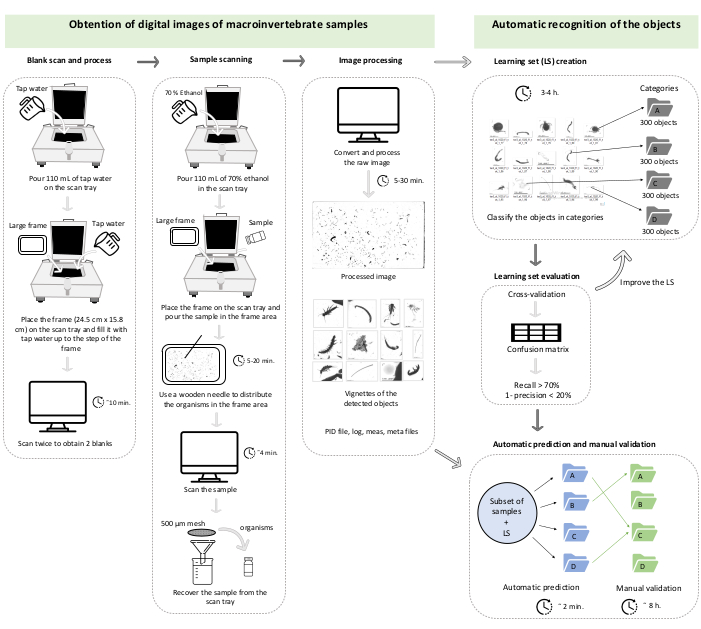
그림 1: 프로토콜의 섹션 1과 섹션 2를 나타내는 작업 차트. 시간은 예시이며 컴퓨터, 처리할 비네트의 풍부함, 학습 세트의 범주 수에 따라 변경될 수 있습니다. 이 경우는 42개의 하위 샘플 집합(총 47,473개의 비네트)에 대한 세 가지 범주의 학습 집합의 유효성 검사에 해당합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 개별 크기 분포, 크기 스펙트럼 및 크기 메트릭 계산
참고: 이 섹션에 언급된 계산은 Matlab을 사용하여 수행되었습니다(보조 파일 1의 스크립트 참조).
- 개별 크기 분포
- Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt 파일의 마지막 열에는 유효성이 검사된 개체 분류가 포함됩니다. 거대 무척추 동물로 분류된 개체만 선택하여 표본에서 개별 크기 분포를 나타냅니다.
알림: 개별 신체 크기는 거대 무척추 동물의 타원체 부피에 해당합니다. 시스템은 픽셀 단위로 측정값을 제공합니다. - 벡터를 두 스캔의 크기 측정값과 연결하면 각 분수가 서로 다른 서브샘플링 지수를 갖기 때문입니다. 연결하기 전에 크기 벡터를 해당 하위 표본이 분획된 횟수만큼 복제하여 분획을 수정합니다.
참고: 이 단계는 스캔이 샘플의 일부(즉, 거칠거나 미세)에 해당하는 경우에 필요합니다. - 유기체와 동일한 픽셀 영역을 가진 prolate 타원체의 주 (M) 및 부 (m) 축에서 타원체 부피를 계산하십시오. 타원체 부피를 계산하기 전에 다음 변환 계수(cf)를 사용하여 주축(M) 및 부축(m) 축을 픽셀에서 밀리미터(mm)로 변환합니다.
1픽셀 = 2,400dpi
1인치 = 25.4mm
참조 = 25.4/2400
타원체 부피 (mm3 단위의 단위를 갖는 ellipVol)는 다음에 해당합니다.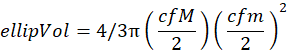
- 로그2 척도에서 개별 크기 분포의 확률 밀도 함수를 나타냅니다.
- Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt 파일의 마지막 열에는 유효성이 검사된 개체 분류가 포함됩니다. 거대 무척추 동물로 분류된 개체만 선택하여 표본에서 개별 크기 분포를 나타냅니다.
- 크기 다양성
- García-Comas et al. (2016) 35에서와 같이 Quintana et al. (2008) 8 :
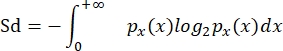
여기서 p x(x)는 크기 x의 확률 밀도 함수이고 x는 log2(ellipVol)를 나타냅니다. 따라서이 측정은 커뮤니티의 개별 크기 분포와 같은 연속 측정에 적합한 Shannon 다양성 지수입니다.
- García-Comas et al. (2016) 35에서와 같이 Quintana et al. (2008) 8 :
- 정규화된 생체부피 크기 스펙트럼(NBSS)
- NBSS의 크기 클래스를 정의하여 스펙트럼의 하한을 샘플의 거대 무척추 동물 크기 분포의 0.01 분위수로 설정하고 샘플에서 가장 큰 유기체가 포함될 때까지 밑이 2인 기하학적 척도로 크기 클래스를 만듭니다.
참고: 크기 클래스 너비는 더 큰 크기와 관련된 더 큰 가변성을 고려하기 위해 크기에 따라 증가합니다. 여기에서 분석 된 거대 무척추 동물 군집의 NBSS는 14 가지 크기 등급을 가졌습니다 (표 1). - 각 크기 클래스의 총 바이오볼륨을 크기 클래스 너비로 나누어 정규화된 바이오볼륨을 얻습니다.
- NBSS의 크기 클래스를 정의하여 스펙트럼의 하한을 샘플의 거대 무척추 동물 크기 분포의 0.01 분위수로 설정하고 샘플에서 가장 큰 유기체가 포함될 때까지 밑이 2인 기하학적 척도로 크기 클래스를 만듭니다.
- 크기 스펙트럼 기울기
- NBSS의 선형 기울기를 계산합니다.
참고: 기울기(μ)는 빈 크기 클래스(이 연구에서 크기 클래스 3에서 14까지의 크기 클래스)를 무시하고 모드보다 큰 크기 클래스에서 log2 (크기 클래스 중간 지점)와 log2 (정규화된 바이오매스) 간의 관계를 기반으로 계산됩니다.
- NBSS의 선형 기울기를 계산합니다.
| 크기 등급 제한 (mm3) | 크기 클래스 중간 지점 (mm3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
표 1: 정규화된 바이오매스 크기 스펙트럼(NBSS)의 크기 클래스. 이 표는 또한 유기체의 15 크기 등급 제한과 크기 등급 중간 지점을 보여줍니다.
결과
거대 무척추 동물 샘플의 디지털 이미지 수집
스캔 뉘앙스: 스캔 트레이에 에탄올 침착
거대 무척추 동물에 대한 시스템을 테스트하는 동안 여러 스캔의 품질이 좋지 않았습니다. 배경의 어두운 포화 영역은 이미지의 정상적인 처리와 거대 무척추 동물의 개별 크기 측정을 방해했습니다 (그림 2). 배경에 포화 영역이 나타나거나 고도로 픽셀 화 ...
토론
강변 거대 무척추 동물에 대한 Gorsky et al. 2010에 의해 설명 된 방법론의 적용은 담수 거대 무척추 동물의 공동체 크기 구조를 추정 할 때 높은 분류 정확도를 허용합니다. 결과는 프로토콜이 샘플에서 개별 크기 구조를 추정하는 시간을 약 1시간으로 줄일 수 있음을 시사합니다. 따라서, 제안 된 프로토콜은 담수 생태계에서 섭동의 영향을 평가하기위한 빠르고 통합 된 생물학적 지표로서 거대 무척...
공개
저자는 잠재적 인 경쟁 이익이 없다고 선언합니다.
감사의 말
이 작업은 스페인 과학 혁신 및 대학부(보조금 번호 RTI2018-095363-B-I00)의 지원을 받았습니다. CERM-UVic-UCC 회원인 Èlia Bretxa, Anna Costarrosa, Laia Jiménez, María Isabel González, Marta Jutglar, Francesc Llach 및 Núria Sellarès에게 거대 무척추동물 현장 샘플링 및 실험실 분류 작업과 샘플 스캐닝에 협력한 David Albesa에게 감사드립니다. 마지막으로 Josep Maria Gili와 Institut de Ciències del Mar (ICM-CSIC)에 실험실 시설 및 스캐너 장치 사용에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
참고문헌
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유