É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Processamento Automático de Imagem para Determinar a Estrutura do Tamanho da Comunidade de Macroinvertebrados Ribeirinhos
Neste Artigo
Resumo
O artigo baseia-se na criação de um protocolo adaptado para escanear, detectar, classificar e identificar objetos digitalizados correspondentes a macroinvertebrados de rios bentônicos usando um procedimento de imagem semiautomático. Este procedimento permite a aquisição das distribuições de tamanho individual e métricas de tamanho de uma comunidade de macroinvertebrados em cerca de 1 h.
Resumo
O tamanho do corpo é um traço funcional importante que pode ser usado como um bioindicador para avaliar os impactos das perturbações nas comunidades naturais. A estrutura do tamanho da comunidade responde a gradientes bióticos e abióticos, incluindo perturbações antropogênicas entre táxons e ecossistemas. No entanto, a medição manual de organismos de corpo pequeno, como macroinvertebrados bentônicos (por exemplo, >500 μm a alguns centímetros de comprimento) é demorada. Para agilizar a estimativa da estrutura do tamanho da comunidade, desenvolvemos um protocolo para medir semi-automaticamente o tamanho corporal individual de macroinvertebrados de rios preservados, que são um dos bioindicadores mais comumente usados para avaliar o estado ecológico dos ecossistemas de água doce. Este protocolo é adaptado de uma metodologia existente desenvolvida para escanear o mesozooplâncton marinho com um sistema de varredura projetado para amostras de água. O protocolo consiste em três etapas principais: (1) varredura de subamostras (frações amostrais finas e grosseiras) de macroinvertebrados fluviais e processamento das imagens digitalizadas para individualizar cada objeto detectado em cada imagem; (2) criar, avaliar e validar um conjunto de aprendizagem através de inteligência artificial para separar semi-automaticamente as imagens individuais de macroinvertebrados de detritos e artefatos nas amostras digitalizadas; e (3) representando a estrutura de tamanho das comunidades de macroinvertebrados. Além do protocolo, este trabalho inclui os resultados da calibração e enumera vários desafios e recomendações para adaptar o procedimento a amostras de macroinvertebrados e considerar novas melhorias. No geral, os resultados apoiam o uso do sistema de varredura apresentado para a medição automática do tamanho corporal de macroinvertebrados fluviais e sugerem que a representação de seu espectro de tamanho é uma ferramenta valiosa para a rápida bioavaliação de ecossistemas de água doce.
Introdução
Os macroinvertebrados bentônicos são amplamente utilizados como bioindicadores para determinar o estado ecológico dos corpos d'água1. A maioria dos índices para descrever comunidades de macroinvertebrados se concentra em métricas taxonômicas. No entanto, novas ferramentas de bioavaliação que integrem o tamanho corporal são encorajadas a fornecer uma perspectiva alternativa ou complementar às abordagens taxonômicas 2,3.
O tamanho corporal é considerado um metatraço relacionado a outros traços vitais, como metabolismo, crescimento, respiração e movimento4. Além disso, o tamanho do corpo pode determinar a posição trófica e as interações5. A relação entre o tamanho corporal individual e a biomassa normalizada (ou abundância) por classe de tamanho em uma comunidade é definida como o espectro de tamanho6 e segue o padrão geral de uma diminuição linear da biomassa normalizada à medida que o tamanho individual aumenta em uma escala logarítmica7. A inclinação dessa relação linear tem sido extensivamente estudada teoricamente, e estudos empíricos em ecossistemas a têm utilizado como um indicador ecológico da estrutura de tamanho da comunidade4. Outro indicador sintético da estrutura de tamanho da comunidade que tem sido usado com sucesso em estudos de funcionamento da biodiversidade-ecossistema é a diversidade de tamanho da comunidade, que é representada como o índice de Shannon das classes de tamanho do espectro de tamanho ou seu analógico, que é calculado com base nas distribuições de tamanho individuais8.
Em ecossistemas de água doce, a estrutura de tamanho de diferentes grupos faunísticos é utilizada como indicador atáxico para avaliar a resposta de comunidades bióticas a gradientes ambientais 9,10,11 e a perturbações antrópicas 12,13,14,15,16. Os macroinvertebrados não são uma exceção, e sua estrutura de tamanho também responde a mudanças ambientais17,18 e perturbações antropogênicas, como mineração 19, uso da terra 20 ou enriquecimento de nitrogênio (N) e fósforo (P)20,21,22. No entanto, medir centenas de indivíduos para descrever a estrutura do tamanho da comunidade é uma tarefa tediosa e demorada que muitas vezes é evitada como uma medição de rotina em laboratórios devido à falta de tempo. Assim, vários métodos de imagem semiautomáticos ou automáticos para classificar e medir espécimes têm sido desenvolvidos23,24,25,26. No entanto, a maioria desses métodos está focada na classificação taxonômica mais do que no tamanho individual dos organismos e não está pronta para uso em todos os tipos de macroinvertebrados. Na ecologia do plâncton marinho, um sistema de análise de imagens de varredura tem sido amplamente utilizado para determinar o tamanho e a composição taxonômica de comunidades de zooplâncton 27,28,29,30,31. Este instrumento pode ser encontrado em vários institutos marinhos em todo o mundo, e é usado para digitalizar amostras de zooplâncton preservadas para obter imagens digitais de alta resolução de toda a amostra. O presente protocolo adapta o uso deste instrumento para estimar o tamanho do espectro da comunidade de macroinvertebrados em rios de forma rápida e automática, sem investir na criação de um novo dispositivo.
O protocolo consiste em digitalizar uma amostra e processar toda a imagem para obter automaticamente imagens únicas (ou seja, vinhetas) dos objetos na amostra. Várias medidas de forma, tamanho e características de nível de cinza caracterizam cada objeto e permitem a classificação automática dos objetos em categorias, que são então validadas por um especialista. O tamanho individual de cada organismo é calculado usando o biovolume elipsoidal (mm3), que é derivado da área do organismo medida em pixels. Isso permite obter o espectro de tamanho da amostra de maneira rápida. Até onde sabemos, este sistema de imagem de varredura só foi usado para processar amostras de mesozooplâncton, mas o dispositivo pode potencialmente permitir o trabalho com macroinvertebrados bentônicos de água doce.
O objetivo geral deste estudo é, portanto, introduzir um método para obter rapidamente o tamanho individual de macroinvertebrados fluviais preservados, adaptando um protocolo existente anteriormente utilizado com mesozooplâncton marinho 27,32,33. O procedimento consiste em usar uma abordagem semiautomática que opera com um dispositivo de digitalização para digitalizar amostras de água e três softwares abertos para processar as imagens digitalizadas. Um protocolo adaptado para digitalizar, detectar e identificar macroinvertebrados fluviais digitalizados para adquirir automaticamente a estrutura de tamanho da comunidade e as métricas de tamanho relacionadas é aqui apresentado. A avaliação do procedimento e as orientações para aumentar a eficiência também são apresentadas com base em 42 imagens digitalizadas de amostras de macroinvertebrados ribeirinhos coletadas de três bacias do Nordeste (NE) da Península Ibérica (Ter, Segre-Ebre e Besòs).
As amostras foram coletadas em trechos de rios de 100 m seguindo o protocolo de amostragem de campo e análise laboratorial de macroinvertebrados de rios bentônicos em rios transbordáveis do Governo Espanhol34. As amostras foram coletadas com um amostrador de surber (quadro: 0,3 m x 0,3 m, malha: 250 μm) após um levantamento multi-habitat. No laboratório, as amostras foram limpas e peneiradas através de uma malha de 5 mm e 500 μm para obtenção de duas subamostras: uma subamostra grossa (malha de 5 mm) e uma subamostra fina (malha de 500 μm), que foram armazenadas em frascos separados e preservadas em etanol a 70%. Separar a amostra em duas frações de tamanho permite uma melhor estimativa da estrutura de tamanho da comunidade, uma vez que os organismos grandes são mais raros e menores do que os organismos pequenos. Caso contrário, a amostra digitalizada tem uma representação tendenciosa da fração de tamanho grande.
Protocolo
NOTA: O protocolo aqui descrito baseia-se no sistema desenvolvido por Gorsky et al.27 para o mesozooplâncton marinho. Uma descrição específica das etapas do scanner (ZooSCAN), do software de digitalização (VueScan 9x64 [9.5.09]), do software de processamento de imagens (Zooprocess, ImageJ) e do software de identificação automática (Plankton Identifier) pode ser encontrada nas referências anteriores32,33. Para melhor ajustar os tamanhos dos macroinvertebrados bentônicos em relação ao mesozooplâncton, uma vez que o projeto é criado seguindo o protocolo original32,33, altere o parâmetro de tamanho mínimo (minsizeesd_mm) para 0,3 mm e o parâmetro de tamanho máximo (maxsizeesd_mm) para 100 mm no arquivo de configuração. Para ajudar a seguir o protocolo, isso é resumido em um gráfico de trabalho (Figura 1). O projeto criado é armazenado na pasta C do computador e é organizado nas seguintes pastas: PID_process, Zooscan_back, Zooscan_check, Zooscan_config, Zooscan_meta, Zooscan_results e Zooscan_scan. Cada pasta é composta de várias subpastas que os diferentes aplicativos de software usam nas etapas a seguir do protocolo.
1. Aquisição de imagens digitais para amostras de macroinvertebrados
- Digitalizando e processando o espaço em branco
NOTA: Crie duas imagens em branco diariamente antes da digitalização para extrair as digitalizações de fundo enquanto processa as imagens digitalizadas no mesmo dia.- Ligue o scanner e ligue a luz na posição dupla para projetar a luz branca da parte superior e inferior.
NOTA: Ao escanear amostras de mesozooplâncton, a direção da luz para cima é usada, mas como os macroinvertebrados são mais opacos, recomenda-se alternar a luz para uma posição dupla. - Limpe e lave a bandeja de digitalização com água da torneira.
- Deite 110 ml de água da torneira armazenada à temperatura ambiente (RT) no tabuleiro de digitalização até que o vidro esteja coberto. Coloque o quadro grande (24,5 cm x 15,8 cm) na bandeja de digitalização na posição correta (com o canto na parte superior esquerda da bandeja de digitalização) e preencha-o com água da torneira até que o degrau do quadro seja coberto para evitar um efeito de menisco, o que alteraria a imagem digitalizada. Feche a tampa do scanner.
NOTA: Use água no RT para evitar condensação e formação de bolhas. Limpe o quadro sem marcas ou gotículas para evitar a reflexão da luz. - Vá para o software de processamento de imagem, selecione o projeto de trabalho e clique em Digitalizar (Converter) Imagem de fundo.
- Vá para o software de digitalização e clique em Preview. Certifique-se de visualizar a imagem digitalizada, verifique se não há linhas ou pontos e aguarde pelo menos 30 s antes de iniciar outra varredura. Clique em Digitalizar e pressione OK na janela de instruções antes da segunda digitalização para enviar os dados do software de digitalização para o software de processamento de imagem.
Observação : varredura duas vezes para obter as duas varreduras em segundo plano que irão compor o espaço em branco. Essa etapa é feita uma vez por dia antes de iniciar o processamento da amostra e as imagens são armazenadas na pasta Zooscan_back. - Feche o software de digitalização depois de concluir a verificação.
- Ligue o scanner e ligue a luz na posição dupla para projetar a luz branca da parte superior e inferior.
- Preparação e digitalização de amostras
CUIDADO: O etanol é um líquido inflamável e pode causar sérios danos / irritação ocular.- Preencha os metadados de exemplo. Vá para o software de processamento de imagem e selecione Preencher metadados de amostra. Insira a identidade do exemplo, clique em OK e preencha os metadados.
NOTA: O metarquivo é criado especificamente para amostras de mesozooplâncton, portanto, ele não se encaixa na metodologia de amostragem de macroinvertebrados bentônicos, mas todos os campos do arquivo precisam ser preenchidos antes da varredura ou um sinalizador de erro aparecerá. - Despeje 110 mL de etanol a 70% na bandeja de digitalização até que o vidro esteja coberto e coloque a estrutura grande (24,5 cm x 15,8 cm) com o canto na parte superior esquerda da bandeja de digitalização.
NOTA: Trabalhe com etanol em vez de água, pois os macroinvertebrados são preservados em etanol. Na água, eles flutuam e flutuam na bandeja de digitalização, impedindo uma imagem nítida e, portanto, medições de tamanho confiáveis. O etanol deve ser preservado no RT para evitar condensação e formação de bolhas. - Despeje a amostra de macroinvertebrados na bandeja de varredura bordada pelo quadro e cubra a etapa do quadro com mais etanol, se necessário.
NOTA: Abster-se de adicionar muito etanol para evitar que os organismos flutuem e fiquem à deriva. - Homogeneize a amostra em toda a área do quadro, colocando os maiores indivíduos no centro da bandeja para o processamento adequado da imagem, e afunde os organismos flutuantes usando uma agulha de madeira.
NOTA: Se uma subamostra contiver numericamente mais de 1.000 indivíduos, divida a subamostra em duas ou mais frações para minimizar o toque de organismos na imagem digitalizada e digitalize as frações separadamente. - Separe os organismos que tocam e os organismos que tocam as bordas da estrutura usando a agulha de madeira.
NOTA: Este passo requer 5-20 min. Os organismos tocantes são considerados um único objeto pelo software; assim, nesses casos, os tamanhos individuais calculados não correspondem a organismos únicos reais e podem enviesar a estimativa da estrutura de tamanho da comunidade. Existe a possibilidade de editar a imagem com o software de processamento de imagem para separá-las, mas essa etapa adicional envolve pelo menos 1,5 h de reprocessamento; portanto, a separação manual é altamente recomendada. - Para digitalizar a amostra, feche a tampa do scanner, vá para o software de processamento de imagem, selecione o projeto de trabalho e clique em SCAN Sample with Zooscan (For Archive, No Process).
- Selecione a amostra e siga as instruções.
- Vá para o software de digitalização e clique em Preview. Certifique-se de visualizar a imagem digitalizada, verifique se não há linhas ou pontos e aguarde pelo menos 30 s antes de iniciar outra varredura.
- Após pelo menos 30 s, clique no botão Digitalizar no software de digitalização.
NOTA: Pressione OK no software de processamento de imagem depois de pressionar Digitalizar no software de digitalização. Não pressione nenhuma tecla no teclado do computador e evite vibrações da varredura durante a digitalização. Três arquivos são gerados na pasta Zooscan_scan > _raw : (i) um formato de arquivo de imagem marcado (.tif) (16 bits); (ii) um documento de texto padrão chamado LOG (.txt) que registra informações sobre os parâmetros de digitalização; e (iii) um documento de texto padrão denominado META (.txt) com informações sobre os métodos de amostragem. - Verifique se a varredura bruta está correta.
Observação : se a varredura tiver listras de luz ou outros problemas visíveis, considere repetir a varredura para evitar problemas nas etapas a seguir.
- Preencha os metadados de exemplo. Vá para o software de processamento de imagem e selecione Preencher metadados de amostra. Insira a identidade do exemplo, clique em OK e preencha os metadados.
- Recuperação de amostra
- Remova o quadro e enxágue-o acima da bandeja de digitalização usando um frasco de compressão cheio de etanol a 70% para recuperar quaisquer macroinvertebrados ligados.
- Levante a parte superior do scanner para recuperar todos os organismos e etanol da bandeja através do funil de recuperação de varredura em um copo. Com a parte superior do scanner ainda levantada, lave a bandeja com o frasco de aperto para varrer os organismos restantes.
- Passe os espécimes e o etanol do copo através de uma malha de 500 μm para reter os invertebrados na malha e guarde-os novamente em um frasco para injetáveis com etanol a 70%.
- Uma vez que todos os espécimes estejam recuperados no frasco para injetáveis, limpe a bandeja com água da torneira.
NOTA: Lave a bandeja com água da torneira entre as amostras para minimizar a precipitação de etanol, o que altera o processamento da imagem. Lave o quadro com água da torneira para evitar possíveis danos relacionados ao uso de etanol. No final do dia, limpe a bandeja usando água da torneira e seque-a suavemente com papel para evitar arranhões.
- Processamento de imagens
- Vá para o software de processamento de imagem e selecione CONVERTER e PROCESSAR Imagens e Organismos no Modo de Lote e, em seguida, Converter E Processar Imagem E Partículas (Imagem na Pasta RAW). Mantenha as configurações padrão e clique em OK. NORMAL END aparecerá no final do processo.
OBSERVAÇÃO: Um arquivo PID e as vinhetas correspondentes a todos os objetos detectados na imagem digitalizada (em um arquivo de Grupo Fotográfico Conjunto [.jpg]) serão criados na pasta Zooscan_scan > _work. Um arquivo PID é um único arquivo que armazena todos os metadados (metarquivo), os dados técnicos associados ao arquivo de log e uma tabela com 36 variáveis medidas de todos os objetos detectados na imagem. As variáveis medidas correspondem a diferentes estimativas de nível de cinza, dimensão fractal, forma e tamanho. As variáveis que podem ser usadas para estimativa de tamanho são a área e os eixos maior e menor de uma elipse com uma área igual ao objeto (ver seção 3 do protocolo). O tempo de processamento depende da densidade da imagem e das características do computador, e pode ser iniciado entre as amostras durante a recuperação e preparação da próxima amostra. Caso contrário, recomenda-se iniciar o processamento das amostras digitalizadas todos os dias no modo de lote durante a noite e verificar se há processamento de imagem adequado na manhã seguinte. - Verifique se o plano de fundo na imagem processada é adequadamente subtraído da imagem de amostra usando o software de processamento de imagem ou verificando as imagens de máscara (terminadas em msk1.gif) localizadas em Zooscan_scan > _work. Se o plano de fundo contiver áreas saturadas ou muitos pontos, considere repetir a varredura para garantir imagens de alta qualidade.
NOTA: Para evitar áreas saturadas no fundo, a bandeja de digitalização deve ser enxaguada com água da torneira após cada varredura com etanol. Também é importante (1) reduzir o número de indivíduos escaneados (fracionando a amostra e digitalizando em diferentes dobras); (2) garantir que os grandes organismos sejam colocados no centro da bandeja de digitalização; (3) usar etanol limpo e filtrado; (4) reduzir a sujeira nas amostras; (5) assegurar que o volume de etanol para a varredura seja adequado; e (6) garantir que o atraso entre a visualização da amostra e a varredura seja de pelo menos 30 s.
- Vá para o software de processamento de imagem e selecione CONVERTER e PROCESSAR Imagens e Organismos no Modo de Lote e, em seguida, Converter E Processar Imagem E Partículas (Imagem na Pasta RAW). Mantenha as configurações padrão e clique em OK. NORMAL END aparecerá no final do processo.
- Separação de organismos tocantes
NOTA: Quando há várias vinhetas com organismos tocantes, é necessário separar as imagens dos organismos tocantes de outros organismos e/ou de fibras/detritos para garantir uma estimativa adequada da estrutura do tamanho da comunidade.- Vá para o software de processamento de imagem para detectar as vinhetas com vários objetos. Selecione SEPARAÇÃO Usando Vinhetas e pressione OK. Na janela de seleção de configuração, mantenha as configurações padrão e clique em OK.
- Na janela SEPARAÇÃO de VINHETAS , mantenha as configurações padrão, selecione adicionalmente ADICIONAR Contornos em Vinhetas e, em seguida, selecione o exemplo a ser editado.
- Separe os organismos que tocam em cada vinheta que aparece desenhando uma linha com o mouse (pressione o botão de rolo para desenhar). Quando a separação em uma vinheta estiver concluída, clique no botão X no canto superior direito da janela e pressione SIM para processar a próxima. Pressione NÃO para terminar e salvar as alterações. No final do processo, NORMAL END aparecerá se tudo estiver correto.
- Após a separação, reprocesse a imagem para obter os dados de objeto atualizados. Vá para o software de processamento de imagem, clique em PROCESS (Converted) Image (Process One) e selecione Process Again Particles from Processed Images in WORK Sub-Folders. Selecione o exemplo e, na janela Processo de Imagem Única , mantenha as configurações padrão, marque Trabalhar com Máscara de Separação (CREATE-MODIFY-INCLUDE) e clique em OK. No final do processo, NORMAL END aparecerá se tudo estiver correto.
- Na janela Controle de Separação , pressione OK para salvar a imagem com os contornos antes do processamento; se uma imagem anterior existir, ela será substituída.
- Na janela Máscara de Controle de Separação , se necessário, selecione EDITAR para adicionar linhas de separação à máscara usando o mouse para separar os organismos que tocam que não apareceram antes na etapa de separação usando vinhetas. Quando terminar, encerre o processo e, na janela Controle de Máscara de Separação , selecione SIM para aceitar a máscara. No final do processo, NORMAL END aparecerá se tudo estiver correto.
NOTA: O reprocessamento de uma amostra com uma máscara de separação é demorado (isso pode levar mais de 1,5 h por amostra). É preferível dedicar o tempo necessário na etapa 1.2.5 para evitar essa etapa adicional.
2. Reconhecimento automático dos objetos
NOTA: Crie um conjunto de aprendizagem para prever automaticamente a identidade dos objetos detectados, separando assim os organismos dos detritos na amostra.
- Criação de conjunto de aprendizagem
- Copie as imagens e os arquivos .pid associados às imagens que serão usadas para criar as categorias do conjunto de aprendizado de Zooscan_scan > _work para PID_process > Unsorted_vignettes_pid.
NOTA: Seleccione um subconjunto de amostras com elevada diversidade de táxons e diferentes locais de amostragem e/ou épocas de amostragem para garantir a máxima representatividade dos organismos nas amostras. - Na pasta PID_process > conjunto de aprendizado, crie uma subpasta com o nome do novo conjunto de aprendizado (ou seja, yyyymmdd_raw_LS) e, dentro dela, crie as subpastas que corresponderão a cada categoria do conjunto de aprendizado (ou seja, macroinvertebrados, detritos, outros invertebrados).
NOTA: Para obter de forma eficiente a estrutura de tamanho da comunidade de amostras de macroinvertebrados de rios, recomenda-se usar um conjunto de aprendizado baseado em apenas três categorias: macroinvertebrados, outros invertebrados e detritos. Esse conjunto de aprendizado basicamente separa as vinhetas de objetos correspondentes a organismos daquelas correspondentes a detritos (por exemplo, fibras, partículas ou algas filamentosas). - Vá para o software de processamento de imagem (somente modo Avançado) e escolha Vinhetas EXTRACT para PLANKTON IDENTIFIER (vinhetas não classificadas para treinamento). Mantenha as opções padrão e marque a caixa Adicionar Contornos .
- Vá para o software de identificação automática, clique em Aprendizagem, selecione PID_process > Learning_set a subpasta criada para o novo conjunto de aprendizagem (etapa 2.1.2) e pressione OK.
- Na seção esquerda (Unsorted Thumbs) da janela aberta, selecione a pasta Unsorted vignettes_pid. Selecione as vinhetas e arraste-as com o mouse dos polegares não classificados para a pasta de sua categoria correspondente na seção direita, Polegares Classificados, para classificar cada objeto nas categorias definidas. As vinhetas movidas serão marcadas com um X vermelho.
Observação : defina as categorias manualmente criando subpastas na pasta polegares classificados ou crie-as clicando no ícone de pastas no software. Não mova mais de 50 vinhetas ao mesmo tempo. - Depois que todas as categorias forem concluídas com os objetos selecionados (cerca de 300 objetos por categoria), clique em Criar arquivo de aprendizado e salve-o com o nome desejado.
Observação : O conjunto de aprendizado será salvo como um arquivo .pid na pasta PID_process > conjunto de aprendizado do projeto. Recomenda-se criar e testar vários conjuntos de aprendizagem com diferentes níveis de categorias (de formas grosseiras a finas) e com um equilíbrio diferente do número de objetos dentro de cada categoria. Comece com um conjunto de aprendizado grosseiro com um número baixo de categorias e pelo menos 50 objetos por categoria e, em seguida, aumente o número de objetos em cada categoria e/ou crie conjuntos de aprendizado mais refinados. Uma categoria deve ser representativa de sua variabilidade no conjunto de amostras.
- Copie as imagens e os arquivos .pid associados às imagens que serão usadas para criar as categorias do conjunto de aprendizado de Zooscan_scan > _work para PID_process > Unsorted_vignettes_pid.
- Avaliação do conjunto de aprendizagem
NOTA: Execute a validação cruzada com duas dobras e cinco ensaios usando o método Random Forest com o software de identificação automática para obter uma matriz de confusão da classificação resultante dos objetos.- Vá para o software de classificação automática e clique em Análise de Dados.
- Em Selecionar arquivo de aprendizado, selecione o arquivo de conjunto de aprendizado criado PID_process > conjunto de aprendizado.
- Em Selecionar um método, escolha o método Floresta aleatória de validação cruzada. Em Variáveis Originais, desmarque as variáveis de posição (X, Y, XM, YM, BX, BY e Altura). Em Variáveis Personalizadas, marque somente ESD.
Observação : esse método usa uma parte aleatória do conjunto de aprendizado para reconhecer a outra parte (duas vezes), e isso é repetido cinco vezes para garantir que seja estatisticamente robusto. - Clique em Iniciar análise e salve os resultados como Analysis_name.txt na pasta Previsão PID_process >. Quando a análise tiver sido concluída com êxito, encerre a análise de dados.
- Vá para a pasta PID_process > Prediction e clique no arquivo de validação cruzada. Uma janela aparecerá com a matriz de confusão da classificação verdadeira (linhas) versus a classificação automática (colunas).
NOTA: A recordação é a porcentagem de organismos pertencentes a um grupo que foi automaticamente bem reconhecido, enquanto 1-precisão é a porcentagem de organismos classificados pelo algoritmo como um grupo que não é reconhecido (contaminação em um grupo). O recall deve ser superior a 70% e a contaminação (precisão 1) deve ser inferior a 20%. - Repita as etapas 2.1-2.5 se vários conjuntos de aprendizagem foram criados e a recordação e a precisão 1 de cada um precisam ser obtidas.
NOTA: Se vários conjuntos de aprendizagem tiverem sido criados, escolha aquele com maior recordação (bom reconhecimento) e precisão (baixa contaminação) do grupo de interesse (ou seja, macroinvertebrados) para testar a previsão automática de um conjunto de amostras na próxima etapa.
- Predição da identificação de macroinvertebrados
Observação : use o conjunto de aprendizado selecionado para prever a identidade de todos os objetos em um subconjunto de amostras usando o software de identificação automática com um algoritmo de floresta aleatória.- Vá para o software de identificação automática e clique em Análise de Dados.
- Em Selecionar arquivo de aprendizado, selecione o arquivo de conjunto de aprendizado PID_process > conjunto de aprendizado que deve ser usado para a previsão.
- Em Selecionar arquivo(s) de exemplo, selecione na pasta PID_results as amostras (arquivos PID) que serão previstas.
Observação : processar um máximo de 20 arquivos .pid ao mesmo tempo para evitar erros relacionados a problemas de memória. Se muitos arquivos .pid forem processados ao mesmo tempo, o processo mostrará um final correto, mas pode não ser bem processado, e um erro pode ocorrer nas próximas etapas ao processar com o software de processamento de imagem. - Em Select a Method, escolha o método Random Forest . Marque Salvar resultados detalhados para cada amostra. Em Variáveis Originais, desmarque as variáveis de posição (X, Y, XM, YM, BX, BY e Altura). Em Variáveis Personalizadas, marque somente ESD.
- Clique em Iniciar análise e salve os resultados como Analysis_name.txt na pasta Previsão PID_process >.
- Validação manual
Observação : um especialista valida manualmente a previsão da etapa anterior para reclassificar objetos classificados incorretamente na categoria correta.- Copie os arquivos Analysis_sample_dat1.txt a serem validados da pasta Previsão do PID_process > para a pasta PID_process > Pid_results.
- Vá para o software de processamento de imagem e selecione Vinhetas EXTRACT em Pastas de acordo com PREVISÃO ou VALIDAÇÃO. Em seguida, selecione Usar arquivos previstos da pasta "pid_results". Mantenha as configurações padrão e pressione OK.
- O software cria uma pasta chamada sample_yyyymmdd_hhmm_to_validate com os objetos previstos na pasta PID_process > vinhetas classificadas.
- Vá para a pasta PID_process > Vinhetas Ordenadas e copie a pasta sample_yyyymmdd_ hhmm_to_validate. Substitua o nome da pasta _to validar por _validated.
- Para validar manualmente a classificação automática, abra a pasta sample_yyyymmdd_ hhmm_validated e revise todas as vinhetas de cada subpasta (categoria) para identificar se há objetos classificados incorretamente. Quando um objeto for classificado incorretamente, arraste a vinheta usando o mouse para a pasta (categoria) correta.
- Vá para o software de processamento de imagem e selecione LOAD Identifications from Sorted Vignettes. Mantenha as configurações padrão e selecione yyyymmdd_hhmm_name_validated a serem processadas.
- Vá para PID_process > Pid_results > Dat1_validated, onde um arquivo chamado Id_from_sorted_vignettes_yyyymmdd_hhmm.txt e um arquivo .txt para cada uma das amostras validadas (sample_tot_1_dat1.txt) foram criados.
Observação : esses arquivos de .txt contêm uma nova coluna que apresenta a previsão, chamada pred_valid_Id_yyyymmdd_hhmm, que especifica a classificação de especialista de cada objeto (ou seja, a classificação validada). Novas categorias (por exemplo, categorias taxonômicas mais finas) poderiam ser criadas neste ponto, durante a validação. No entanto, mantenha o nome da categoria original no novo nome (por exemplo, macroinvertebrate_chironomidae). Isso permite refazer a categoria original ao calcular a recordação e a precisão e agrupar facilmente todos os macroinvertebrados para calcular os parâmetros da estrutura do tamanho da comunidade (ou seja, o espectro de tamanho e a diversidade de tamanho). O arquivo de texto fornece os dados associados a cada objeto, incluindo os eixos menor e maior que são usados para obter o volume elipsoidal de cada organismo como uma medida do tamanho do corpo individual. Além disso, as duas últimas colunas da tabela contêm as categorias previstas e validadas de cada objeto (linha), que permitem calcular, por categoria, a recordação e a precisão do conjunto de aprendizagem no subconjunto de amostras.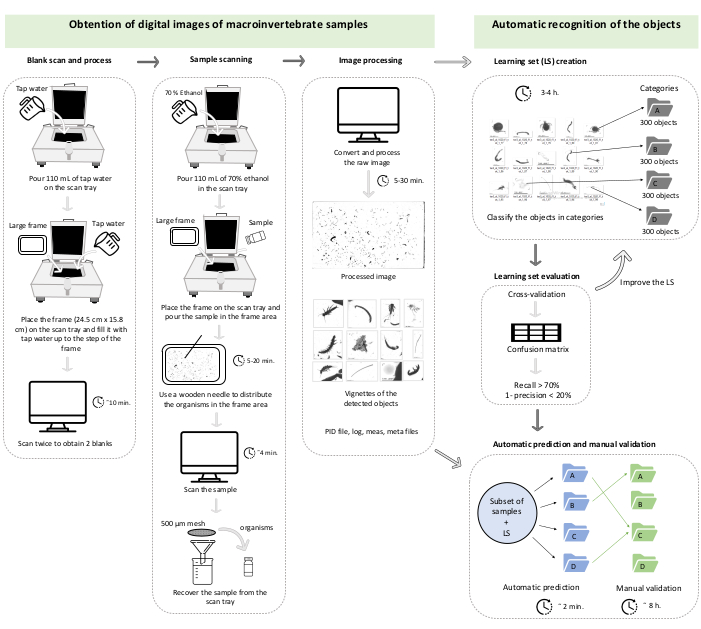
Figura 1: Gráfico de trabalho representando a seção 1 e a seção 2 do protocolo. Os tempos são ilustrativos e podem mudar dependendo do computador, da abundância de vinhetas para processar e do número de categorias do conjunto de aprendizado. Este caso corresponde à validação de um conjunto de aprendizagem de três categorias em um conjunto de 42 subamostras (no total, 47.473 vinhetas). Por favor, clique aqui para ver uma versão maior desta figura.
3. Calculando a distribuição de tamanho individual, espectros de tamanho e métricas de tamanho
Observação : os cálculos mencionados nesta seção foram executados usando o Matlab (consulte o script como arquivo suplementar 1).
- Distribuição de tamanho individual
- A última coluna do arquivo Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt contém a classificação validada dos objetos. Selecione apenas os objetos classificados como macroinvertebrados para representar sua distribuição de tamanho individual na amostra.
NOTA: O tamanho individual do corpo corresponde ao volume elipsoidal dos organismos macroinvertebrados. O sistema fornece medições em pixels. - Concatene os vetores com as medidas de tamanho de ambas as varreduras, porque cada fração tem um expoente de subamostragem diferente. Antes da concatenação, corrija o fracionamento replicando os vetores de tamanho quantas vezes a subamostra correspondente tiver sido fracionada.
NOTA: Esta etapa é necessária se uma varredura corresponder a uma fração de uma amostra (ou seja, grossa ou fina). - Calcule o volume elipsoidal a partir dos eixos maior (M) e menor (m) dos elipsoides prolatos com as mesmas áreas de pixel que os organismos. Antes de calcular o volume elipsoidal, converta os eixos maior (M) e menor (m) de pixels para milímetros (mm) com o seguinte fator de conversão (cf):
1 pixel = 2.400 dpi
1 pol = 25,4 mm
cf = 25,4/2400
O volume elipsoidal (elipse com unidades em mm3) corresponde a: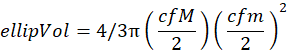
- Descreva a função densidade de probabilidade da distribuição de tamanho individual na escala log2 .
- A última coluna do arquivo Id_from_sorted_vignettes_YYYYMMDD_HHHH.txt contém a classificação validada dos objetos. Selecione apenas os objetos classificados como macroinvertebrados para representar sua distribuição de tamanho individual na amostra.
- Diversidade de tamanho
- Calcule a diversidade de tamanho (Sd) seguindo Quintana et al. (2008)8, como em García-Comas et al. (2016)35:
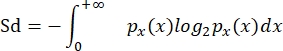
onde p x(x) é a função de densidade de probabilidade do tamanho x, e x representa o log2 (ellipVol). Esta medida é, portanto, o índice de diversidade de Shannon adaptado a uma medida contínua, como a distribuição de tamanho individual em uma comunidade.
- Calcule a diversidade de tamanho (Sd) seguindo Quintana et al. (2008)8, como em García-Comas et al. (2016)35:
- Espectro de tamanho de biovolume normalizado (NBSS)
- Definir as classes de tamanho do NBSS, estabelecendo o limite inferior do espectro como o quantil 0,01 da distribuição de tamanho dos macroinvertebrados nas amostras e criando classes de tamanho por uma escala geométrica de base 2 até que o maior organismo nas amostras seja englobado.
Observação : A largura da classe de tamanho aumenta com o tamanho para levar em conta a maior variabilidade associada a tamanhos maiores. O NBSS das comunidades de macroinvertebrados aqui analisadas apresentou 14 classes de tamanho (Tabela 1). - Obter o biovolume normalizado dividindo o biovolume total em cada classe de tamanho pela largura da classe de tamanho.
- Definir as classes de tamanho do NBSS, estabelecendo o limite inferior do espectro como o quantil 0,01 da distribuição de tamanho dos macroinvertebrados nas amostras e criando classes de tamanho por uma escala geométrica de base 2 até que o maior organismo nas amostras seja englobado.
- Inclinação do espectro de tamanho
- Calcule a inclinação linear do NBSS.
NOTA: A inclinação (μ) é calculada com base na relação entre o log 2 (ponto médio da classe de tamanho) e o log2 (biomassa normalizada) nas classes de tamanho maiores que o modo, ignorando quaisquer vazias (neste estudo, as classes de tamanho de 3 a 14).
- Calcule a inclinação linear do NBSS.
| Limites de classe de tamanho (mm3) | Ponto médio da classe de tamanho (mm3) |
| 0,1236 | 0,1855 |
| 0,2473 | 0,3709 |
| 0,4946 | 0,7418 |
| 0,9891 | 1,4837 |
| 1,9783 | 1,4837 |
| 3,9560 | 5,9348 |
| 7,9131 | 11,8696 |
| 15,8261 | 23,7392 |
| 31,6522 | 47,4783 |
| 63,3044 | 94,9567 |
| 126,6089 | 189,9133 |
| 253,2178 | 379,8267 |
| 506,4300 | 7597,7000 |
| 1012,9000 | 15193,0000 |
| 2025,7000 |
Tabela 1: Classes de tamanho do espectro de tamanho de biomassa normalizado (NBSS). A tabela também mostra os limites de classe de tamanho 15 e os pontos médios da classe de tamanho dos organismos.
Resultados
Aquisição de imagens digitais de amostras de macroinvertebrados
Nuances de varredura: Deposição de etanol na bandeja de varredura
Ao testar o sistema para macroinvertebrados, vários exames foram de baixa qualidade. Uma área saturada escura no fundo impediu o processamento normal da imagem e a medição dos tamanhos individuais dos macroinvertebrados (Figura 2). Várias razões foram dadas para o aparecimento de áreas saturadas no fundo ou imagen...
Discussão
A adaptação da metodologia descrita por Gorsky et al. 2010 para macroinvertebrados ribeirinhos permite alta acurácia de classificação na estimativa da estrutura do tamanho da comunidade em macroinvertebrados de água doce. Os resultados sugerem que o protocolo pode reduzir o tempo para estimar a estrutura de tamanho individual em uma amostra para cerca de 1 hora. Assim, o protocolo proposto pretende promover o uso rotineiro de espectros de tamanho de macroinvertebrados como um bioindicador rápido e integrador para ...
Divulgações
Os autores declaram não haver potenciais interesses concorrentes.
Agradecimentos
Este trabalho foi apoiado pelo Ministério da Ciência, Inovação e Universidades espanholas (número de intervenção RTI2018-095363-B-I00). Agradecemos aos membros do CERM-UVic-UCC Èlia Bretxa, Anna Costarrosa, Laia Jiménez, María Isabel González, Marta Jutglar, Francesc Llach e Núria Sellarès por seu trabalho em amostragem de campo de macroinvertebrados e triagem laboratorial e a David Albesa por colaborar na digitalização de amostras. Finalmente, agradecemos a Josep Maria Gili e ao Institut de Ciències del Mar (ICM-CSIC) pelo uso das instalações laboratoriais e do dispositivo de scanner.
Materiais
| Name | Company | Catalog Number | Comments |
| Beaker | Labbox | Other containers could be used | |
| Dionized water | Icopresa | 8420239600123 | To dilute the ethanol |
| Funnel | Vitlab | 41094 | |
| Glass vials 8 ml | Labbox | SVSN-C10-195 | 1 vial/subsample |
| ImageJ Software | Free access | Version 4.41o/ Image processing software | |
| Large frame | Hydroptic | Provided by ZooScan | 24.5 cm x 15.8 cm |
| Monalcol 96 (Ethanol 96) | Montplet | 1050JE001 | |
| Plankton Identifier Software | Free access | Version 1.2.6/ Automatic identification software | |
| Sieve | Cisa | 26852.2 | Nominal aperture 500µ and nominal aperture 0,5 cm |
| Tweezers | Bondline | B5SA | Stainless, anti-magnetic, anti-acid |
| VueScan 9 x 64 (9.5.09) Software | Hydroptic | Version 9.0.51/ Sacn software | |
| Wooden needle | Any plastic or wood needle can be used | ||
| Zooprocess Software | Free access | Version 7.14/Image processing software | |
| ZooScan | Hydroptic | 54 | Version III/ Scanner |
Referências
- Birk, S., et al. Three hundred ways to assess Europe's surface waters: An almost complete overview of biological methods to implement the Water Framework Directive. Ecological Indicators. 18, 31-41 (2012).
- Basset, A., Sangiorgio, F., Pinna, M. Monitoring with benthic macroinvertebrates: advantages and disadvantages of body size descriptors. Aquatic Conservation: Marine and Freshwater Ecosystems. 14, S43-S58 (2004).
- Reyjol, Y., et al. Assessing the ecological status in the context of the European Water Framework Directive: Where do we go now. Science of the Total Environment. 497-498, 332-344 (2014).
- Brown, J. H., Gillooly, J. F., Allen, A. P., Savage, V. M., West, G. B. Toward a metabolic theory of ecology. Ecology. 85 (7), 1771-1789 (2004).
- Woodward, G., et al. Body size in ecological networks. Trends in Ecology & Evolution. 20 (7), 402-409 (2005).
- Sprules, W. G., Barth, L. E. Surfing the biomass size spectrum: Some remarks on history, theory, and application. Canadian Journal of Fisheries and Aquatic Sciences. 73 (4), 477-495 (2016).
- White, E. P., Ernest, S. K. M., Kerkhoff, A. J., Enquist, B. J. Relationships between body size and abundance in ecology. Trends in Ecology & Evolution. 22 (6), 323-330 (2007).
- Quintana, X. D., et al. A nonparametric method for the measurement of size diversity with emphasis on data standardization. Limnology and Oceanography - Methods. 6 (1), 75-86 (2008).
- Blanchard, J. L., Heneghan, R. F., Everett, J. D., Trebilco, R., Richardson, A. J. From bacteria to whales: Using functional size spectra to model marine ecosystems. Trends in Ecology & Evolution. 32 (3), 174-186 (2017).
- Petchey, O. L., Belgrano, A. Body-size distributions and size-spectra: Universal indicators of ecological status. Biology Letters. 6 (4), 434-437 (2010).
- Emmrich, M., et al. Geographical patterns in the body-size structure of European lake fish assemblages along abiotic and biotic gradients. Journal of Biogeography. 41 (12), 2221-2233 (2014).
- Arranz, I., Brucet, S., Bartrons, M., García-Comas, C., Benejam, L. Fish size spectra are affected by nutrient concentration and relative abundance of non-native species across streams on the NE Iberian Peninsula. Science of the Total Environment. 795, 148792 (2021).
- Vila-Martínez, N., Caiola, N., Ibáñez, C., Benejam, L. l., Brucet, S. Normalized abundance spectra of the fish community reflect hydropeaking on a Mediterranean large river. Ecological Indicators. 97, 280-289 (2019).
- Benejam, L. l., Tobes, I., Brucet, S., Miranda, R. Size spectra and other size-related variables of river fish communities: systematic changes along the altitudinal gradient on pristine Andean streams. Ecological Indicators. 90, 366-378 (2018).
- Sutton, I. A., Jones, N. E. Measures of fish community size structure as indicators for stream monitoring programs. Canadian Journal of Fisheries and Aquatic Sciences. 77 (5), 824-835 (2019).
- Murry, B. A., Farrell, J. M. Resistance of the size structure of the fish community to ecological perturbations in a large river ecosystem. Freshwater Biology. 59, 155-167 (2014).
- Townsend, C. R., Thompson, R. M., Hildrew, A. G., Raffaelli, D. G., Edmonds-Brown, R. Body size in streams: Macroinvertebrate community size composition along natural and human-induced environmental gradients. In Body Size: The Structure and Function of Aquatic Ecosystems. , (2007).
- Gjoni, V., et al. Patterns of functional diversity of macroinvertebrates across three aquatic ecosystem types, NE Mediterranean. Mediterranean Marine Science. 20 (4), 703-717 (2019).
- Pomeranz, J. P. F., Warburton, H. J., Harding, J. S. Anthropogenic mining alters macroinvertebrate size spectra in streams. Freshwater Biology. 64 (1), 81-92 (2019).
- García-Girón, J., et al. Anthropogenic land-use impacts on the size structure of macroinvertebrate assemblages are jointly modulated by local conditions and spatial processes. Environmental Research. 204, 112055 (2022).
- Demi, L. M., Benstead, J. P., Rosemond, A. D., Maerz, J. C. Experimental N and P additions alter stream macroinvertebrate community composition via taxon-level responses to shifts in detrital resource stoichiometry. Functional Ecology. 33 (5), 855-867 (2019).
- Basset, A., et al. A benthic macroinvertebrate size spectra index for implementing the Water Framework Directive in coastal lagoons in Mediterranean and Black Sea ecoregions. Ecological Indicators. 12 (1), 72-83 (2012).
- Ärje, J., et al. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution. 11 (8), 922-931 (2020).
- Raitoharju, J., et al. Benchmark database for fine-grained image classification of benthic macroinvertebrates. Image and Vision Computing. 78, 73-83 (2018).
- Lytle, D. A., et al. Automated processing and identification of benthic invertebrate samples. Journal of the North American Benthological Society. 29 (3), 867-874 (2010).
- Serna, J. P., Fernández, D. S., Vélez, F. J., Aguirre, N. J. An image processing method for recognition of four aquatic macroinvertebrates genera in freshwater environments in the Andean region of Colombia. Environmental Monitoring and Assessment. 192, 617 (2020).
- Gorsky, G., et al. Digital zooplankton image analysis using the ZooScan integrated system. Journal of Plankton Research. 32 (3), 285-303 (2010).
- Marcolin, C. R., Schultes, S., Jackson, G. A., Lopes, R. M. Plankton and seston size spectra estimated by the LOPC and ZooScan in the Abrolhos Bank ecosystem (SE Atlantic). Continental Shelf Research. 70, 74-87 (2013).
- Silva, N., Marcolin, C. R., Schwamborn, R. Using image analysis to assess the contributions of plankton and particles to tropical coastal ecosystems. Estuarine, Coast and Shelf Science. 219, 252-261 (2019).
- Vandromme, P., et al. Assessing biases in computing size spectra of automatically classified zooplankton from imaging systems: A case study with the ZooScan integrated system. Methods in Oceanography. 1-2, 3-21 (2012).
- Naito, A., et al. Surface zooplankton size and taxonomic composition in Bowdoin Fjord, north-western Greenland: A comparison of ZooScan, OPC and microscopic analyses. Polar Science. 19, 120-129 (2019).
- . Zooprocess/Plankton Identifier protocol for computer assisted zooplankton sorting Available from: https://manualzz.com/doc/43116355/zooprocess—plankton-identifier-protocol-for (2013)
- Protocolo de muestreo y laboratorio de fauna bentónica de invertebrados en ríos vadeables. CÓDIGO: ML-Rv-I-2013. Ministerio de Agricultura, Alimentación y Medio Ambiente Available from: https://www.miteco.gob.es/es/agua/temas/estado-y-calidad-de-las-aguas/ML-Rv-I-2013_Muestreo%20y%20laboratorio_Fauna%20bent%C3%B3nica%20de%20de%20invertebrado_%20R%C3%Ados%20vadeables_24_05_2013_tcm30-175284.pdf (2013)
- García-Comas, C., et al. Prey size diversity hinders biomass trophic transfer and predator size diversity promotes it in planktonic communities. Proceedings of the Royal Society Biological Sciences. 283 (1824), 20152129 (2016).
- García-Comas, C., et al. Mesozooplankton size structure in response to environmental conditions in the East China Sea: How much does size spectra theory fit empirical data of a dynamic coastal area. Progress in Oceanography. 121, 141-157 (2014).
- Marquina, D., Buczek, M., Ronquist, F., Lukasik, P. The effect of ethanol concentration on the morphological and molecular preservation of insects for biodiversity studies. PeerJ. 9, 10799 (2021).
- Bell, J. L., Hopcroft, R. R. Assessment of ZooImage as a tool for the classification of zooplankton. Journal of Plankton Research. 30 (12), 1351-1367 (2008).
- Colas, F., et al. The ZooCAM, a new in-flow imaging system for fast onboard counting, sizing and classification of fish eggs and metazooplankton. Progress in Oceanography. 166, 54-65 (2018).
- Bachiller, E., Fernandes, J. A., Irigoien, X. Improving semiautomated zooplankton classification using an internal control and different imaging devices. Limnology and Oceanography Methods. 10 (1), 1-9 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados