A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
قياس طول ذيل بولي أ من دماغ يرقة ذبابة الفاكهة وخط الخلية
In This Article
Summary
يصف البروتوكول طريقة فعالة وموثوقة لتحديد طول poly(A) للجين محل الاهتمام من الجهاز العصبي لذبابة الفاكهة ، والذي يمكن تكييفه بسهولة مع الأنسجة أو أنواع الخلايا من الأنواع الأخرى.
Abstract
Polyadenylation هو تعديل حاسم بعد النسخ يضيف ذيول poly (A) إلى الطرف 3 'من جزيئات mRNA. يتم تنظيم طول ذيل poly (A) بإحكام بواسطة العمليات الخلوية. ارتبط عدم تنظيم polyadenylation mRNA بالتعبير الجيني غير الطبيعي وأمراض مختلفة ، بما في ذلك السرطان والاضطرابات العصبية وتشوهات النمو. لذلك ، فإن فهم ديناميكيات polyadenylation أمر حيوي لكشف تعقيدات معالجة mRNA وتنظيم الجينات بعد النسخ.
يقدم هذا البحث طريقة لقياس أطوال ذيل poly (A) في عينات الحمض النووي الريبي المعزولة من أدمغة يرقات ذبابة الفاكهة وخلايا ذبابة الفاكهة شنايدر S2. استخدمنا نهج مخلفات جوانوزين / إينوزين (G / I) ، والذي يتضمن الإضافة الأنزيمية لبقايا G / I في نهاية 3 'من mRNA باستخدام بوليميراز الخميرة بولي (A). يحمي هذا التعديل نهاية الحمض النووي الريبي 3 'من التدهور الأنزيمي. ثم يتم نسخ ذيول poly (A) المحمية كاملة الطول بشكل عكسي باستخدام طبقة أولية عالمية مضادة للحساسية. بعد ذلك ، يتم إجراء تضخيم تفاعل البوليميراز المتسلسل باستخدام oligo خاص بالجين يستهدف الجين محل الاهتمام ، جنبا إلى جنب مع oligo تسلسل عالمي يستخدم للنسخ العكسي.
هذا يولد منتجات تفاعل البوليميراز المتسلسل التي تشمل ذيول بولي (أ) للجين محل الاهتمام. نظرا لأن polyadenylation ليس تعديلا موحدا وينتج عنه ذيول بأطوال متفاوتة ، فإن منتجات PCR تعرض مجموعة من الأحجام ، مما يؤدي إلى نمط تشويه على هلام الأغاروز. أخيرا ، تخضع منتجات تفاعل البوليميراز المتسلسل للرحلان الكهربائي للهلام الشعري عالي الدقة ، يليه القياس الكمي باستخدام أحجام منتجات poly (A) PCR ومنتج تفاعل البوليميراز المتسلسل الخاص بالجينات. توفر هذه التقنية أداة مباشرة وموثوقة لتحليل أطوال ذيل poly (A) ، مما يمكننا من اكتساب رؤى أعمق حول الآليات المعقدة التي تحكم تنظيم mRNA.
Introduction
معظم mRNAs حقيقية النواة هي polyadenylated بعد النسخ في نهايتها 3 ′ في النواة عن طريق إضافة الأدينوزينات غير المقولبة بواسطة بوليميراز بولي (A) الكنسي. يعد ذيل poly (A) السليم محوريا طوال دورة حياة mRNA ، لأنه ضروري لتصدير mRNAالنووي 1 ، ويسهل التفاعل مع البروتينات المرتبطة ب poly (A) لتعزيز الكفاءة الانتقالية2 ، ويضفي مقاومة ضد التدهور3. في بعض الحالات، يمكن أن يخضع ذيل بولي (أ) أيضا للتمدد في السيتوبلازم، الذي تسهله بوليميراز بولي (أ) غير المتعارف عليه4. في السيتوبلازم ، يتغير طول ذيل بولي (A) ديناميكيا ويؤثر على العمر الافتراضي لجزيء mRNA. تشتهر العديد من البلمرة و deadenylases بتعديل طول الذيل5،6،7. على سبيل المثال ، يرتبط تقصير ذيول poly (A) بالقمع الانتقالي ، في حين أن إطالة ذيول poly (A) تعزز الترجمة 8,9.
أظهرت الدراسات الجينومية المتراكمة الأهمية الأساسية لطول ذيل poly (A) عبر جوانب مختلفة من بيولوجيا حقيقية النواة. وهذا يشمل الأدوار في تطوير الخلايا الجرثومية ، والتطور الجنيني المبكر ، واللدونة العصبية المشبكية للتعلم والذاكرة ، والاستجابة الالتهابية10. تم تطوير العديد من الطرق والمقايسات لقياس أطوال ذيل poly (A). على سبيل المثال ، يستفيد اختبار RNase H / oligo (dT) من RNase H في وجود أو عدم وجود oligo (dT) لدراسة طول ذيل poly (A)11,12. تشمل الطرق الأخرى لدراسة ذيل poly (A) تضخيم تفاعل البوليميراز المتسلسل لنهايات 3 بوصات مثل التضخيم السريع لنهايات cDNA اختبار poly (A) (RACE-PAT) 12،13 واختبار poly (A) بوساطة ligase (LM-PAT) 14. تشمل التعديلات الإضافية لمقايسة PAT ePAT15 و sPAT16. الأنزيمية G-tailing17,18 أو G / I-tailing من نهاية 3 'هي اختلافات أخرى في فحص PAT. يتضمن التعديل الإضافي لهذه التقنيات استخدام البادئات ذات العلامات الفلورية جنبا إلى جنب مع الرحلان الكهربائي للهلام الشعري للتحليل عالي الدقة ، والذي يشار إليه باسم اختبار poly (A) عالي الدقة (Hire-PAT) 19. تسمح هذه المقايسات التي تعتمد على تفاعل البوليميراز المتسلسل بتحديد كمية طول بولي (أ) بسرعة وعالية الحساسية.
مع تطوير تسلسل الجيل التالي ، تسمح طريقة التسلسل عالية الإنتاجية ، مثل PAL-seq20 و TAIL-seq21 ، بتحليلات polyadenylation على نطاق واسع للنسخ. ومع ذلك ، توفر هذه الطرق قراءات تسلسل قصيرة فقط من 36-51 نيوكليوتيدات. لذلك ، تم تطوير FLAM-Seq22 لتحديد طول الذيل العالمي ل mRNA كامل الطول ويوفر قراءات طويلة. توفر تقنية Nanopore23 تسلسل الحمض النووي الريبي المباشر المستقل عن تفاعل البوليميراز المتسلسل أو تسلسل cDNA المباشر لتقديرات طول الذيل poly (A). ومع ذلك ، فإن هذه الأساليب عالية الإنتاجية لا تخلو من القيود. إنها تتطلب كميات كبيرة من المواد الأولية ، وهي باهظة الثمن وتستغرق وقتا طويلا. علاوة على ذلك ، يمكن أن يكون تحليل النصوص النادرة أمرا صعبا للغاية مع الطرق عالية الإنتاجية ، ولا تزال الطرق منخفضة الإنتاجية القائمة على تفاعل البوليميراز المتسلسل توفر ميزة عندما يحتاج عدد صغير من النصوص إلى التحليل ، لإجراء تجارب تجريبية ، والتحقق من صحة الطرق الأخرى.
لقد أثبتنا مؤخرا أن Dscam1 mRNAs تحتوي على ذيول بولي (A) قصيرة في ذبابة الفاكهة ، مما يستلزم ارتباطا غير قانوني للبروتين المرتبط بالسيتوبلازم poly (A) على Dscam1 3'UTR باستخدام طريقة المخلفات G / I24. نقدم هنا إجراء مبسطا لإعداد الأنسجة وتحديد طول poly (A) من mRNAs من الجهاز العصبي لذبابة الفاكهة وخلايا ذبابة الفاكهة S2.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. تربية واختيار يرقات ذبابة الفاكهة
- الحفاظ على / استزراع سلالة الذباب (w1118 ، wildtype) على وسط طعام الذباب القياسي عند 25 درجة مئوية في حاضنة مرطبة.
- حدد 10 تجول 3 يرقاتداخلية بعد 72 ساعة من وضع البيض.
- ضع اليرقات في طبق بتري فارغ 35 مم واغسلها برفق عن طريق نقل اليرقات إلى الطبق الجديد الذي يحتوي على ماء الصنبور باستخدام ملقط. افعل هذا 2x لإزالة أي طعام متبقي.
2. عزل الدماغ من يرقات ذبابة الفاكهة (الشكل 1)
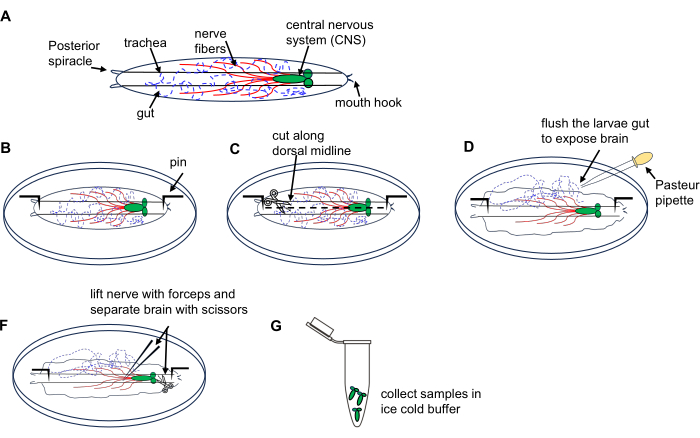
الشكل 1: تشريح دماغ يرقات ذبابة الفاكهة من مرحلة تجول 3 الثالثة. (أ) رسومات تخطيطية ليرقة ذبابة الفاكهة. (ب-ز) تشريح اليرقة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
- ضع 10 يرقات على طبق تشريح يحتوي على برنامج تلفزيوني بارد.
- ضع الجانب الظهري لليرقة لأعلى (تم تحديده بواسطة أنابيب القصبة الهوائية التي تمتد على طوله) وقم بتثبيت كل طرف في قاع الطبق متبوعا بعمل شق صغير على جدار الجسم في الطرف الخلفي.
- اقطع جدار الجسم على طول خط الوسط الظهري باتجاه الطرف الأمامي باستخدام مقص التشريح المجهري.
- اغسل الجزء الداخلي من اليرقة لفترة وجيزة باستخدام ماصة باستور 3x مع برنامج تلفزيوني في الطبق لفضح الدماغ.
- حدد موقع الدماغ وارفعه باستخدام الملقط واعزله بعناية باستخدام مقص التشريح المجهري.
- نقل الأدمغة المشرحة إلى أنبوب طرد مركزي دقيق سعة 1.5 مل مملوء ببرنامج تلفزيوني بارد على الجليد ؛ جمع كل أدمغة اليرقة. تابع استخراج الحمض النووي الريبي باستخدام مجموعة microprep من الحمض النووي الريبي كما هو موضح في القسم 4.
ملاحظة: قم بتشريح 10 يرقات في غضون 15 دقيقة لمنع تلف الأنسجة وتدهور الحمض النووي الريبي.
3. ذبابة الفاكهة S2 خلايا شنايدر
- تنمو خلايا ذبابة الفاكهة S2 في وسط ذبابة الفاكهة شنايدر مع 10٪ مصل بقري جنيني (FBS) عند 25 درجة مئوية في حاضنة مرطبة بكثافة 8 × 106 إلى 10 × 106 خلايا / مل مع بقاء 90٪ كحد أدنى.
- في أنبوب مخروطي معقم سعة 50 مل ، قم بتخفيف الخلايا إلى 2.5 × 106 خلايا / مل مع وسط ذبابة الفاكهة من شنايدر المكمل ب 10٪ FBS الذي تم تسخينه مسبقا إلى 25 درجة مئوية.
- انقل 8 مل من معلق الخلية (20 × 106 خلايا) إلى لوحة استزراع 100 مم وأضف 4 مل من الوسط لتصل إلى 12 مل (اليوم الأول).
- احتضان الخلايا المستزرعة عند 25 درجة مئوية في حاضنة مرطبة.
ملاحظة: تلتصق الخلايا بشكل فضفاض باللوحة بعد 12-16 ساعة (اليوم 2). - نقل الخلايا مع بلازميدات الحمض النووي المناسبة24.
- احتضان لمدة 48 ساعة في حاضنة مرطبة.
- بعد الحضانة ، اجمع الخلايا بإضافة 5 مل من برنامج تلفزيوني بارد عن طريق سحب لطيف (اليوم 4).
- انقل الخلايا إلى أنبوب سعة 15 مل.
- قم بإذابة الخلايا عن طريق الطرد المركزي عند 1000 × جم لمدة 5 دقائق عند 4 درجات مئوية.
- شطف الخلايا 2x مع برنامج تلفزيوني بارد عن طريق ماصة لطيفة وجمع الخلايا عن طريق الطرد المركزي لها في 1000 × غرام لمدة 5 دقائق عند 4 درجات مئوية.
- قم بإجراء استخراج الحمض النووي الريبي باستخدام مجموعة أدوات تحضير الحمض النووي الريبي.
ملاحظة: قم بتنفيذ الخطوات التالية داخل غطاء التدفق الصفحي المعقم.
4. استخراج الحمض النووي الريبي الكلي من دماغ يرقات ذبابة الفاكهة وخلايا S2
- دماغ اليرقات: قم بإزالة PBS عن طريق الطرد المركزي القصير (8 ثوان تدور قصيرة عند 5000 × جم).
- أضف 600 ميكرولتر من محلول تحلل الحمض النووي الريبي وقم بتجانس 10x باستخدام مدقة بلاستيكية. افحص الأنبوب بصريا تحت مجهر ستيريو لضمان التحلل الكامل.
- جهاز طرد مركزي عند 1000 × جم لمدة 5 دقائق عند 4 درجات مئوية لإزالة حطام الأنسجة. انقل المادة الطافية التي تم تطهيرها إلى أنبوب طرد مركزي خال من النيوكلياز.
- اعزل الحمض النووي الريبي باستخدام مجموعة تحضير دقيق للحمض النووي الريبي وفقا لتعليمات الشركة المصنعة.
ملاحظة: يعد استخدام مجموعة microprep من الحمض النووي الريبي ضروريا لعينات دماغ اليرقات بسبب الكمية الصغيرة من الحمض النووي الريبي الموجودة في العينات. - خلايا S2: قم بإزالة PBS وعزل الحمض النووي الريبي وفقا لتعليمات الشركة الصانعة.
- قياس إنتاجية الحمض النووي الريبي وجودته عن طريق القياس الطيفي وهلام الأغاروز الكهربائي.
- تحديد نقاء وكمية الحمض النووي الريبي المستخرج عن طريق قياس الكثافة البصرية للحمض النووي الريبي المستخرج عند A260 نانومتر وA 280 نانومتر على التوالي. تأكد من أن نسبة A260 نانومتر / A280 نانومتر هي ≥2.0 وتركيز الحمض النووي الريبي هو >350 نانوغرام / ميكرولتر للتطبيقات النهائية.
ملاحظة: العائد النموذجي للحمض النووي الريبي من 10 أدمغة يرقة ذبابة الفاكهة هو ~ 500-800 نانوغرام / ميكرولتر أو 2.5-4 ميكروغرام في 5 ميكرولتر. بالنسبة لخلايا S2 ، يكون العائد ~ 2-3 ميكروغرام / ميكرولتر (15-30 ميكروغرام في 15 ميكرولتر). يمكن تخزين الحمض النووي الريبي المعزول عند -80 درجة مئوية للتخزين طويل الأجل.
- تحديد نقاء وكمية الحمض النووي الريبي المستخرج عن طريق قياس الكثافة البصرية للحمض النووي الريبي المستخرج عند A260 نانومتر وA 280 نانومتر على التوالي. تأكد من أن نسبة A260 نانومتر / A280 نانومتر هي ≥2.0 وتركيز الحمض النووي الريبي هو >350 نانوغرام / ميكرولتر للتطبيقات النهائية.
5. إعداد هلام الحمض النووي الريبي والكهربائي
- 1.5٪ جل الحمض النووي الريبي (100 مل)
ملاحظة: الفورمالديهايد سام من خلال ملامسة الجلد واستنشاق الأبخرة. تعامل معها في غطاء دخان كيميائي.- قم بإذابة ثلاثة أقراص أغاروز (1.5 جم) في 82 مل من مخزن MOPS المؤقت (الملف التكميلي 1) حتى تتفكك الأقراص تماما لتشكل جزيئات دقيقة.
- سخني ملاط الأغاروز في الميكروويف حتى يصبح المحلول صافيا وتذوب جميع الجزيئات تماما.
- تبريد الحل إلى ~ 60 °C.
- أضف 18 مل من 37٪ فورمالديهايد ، ثم اخلطها بتحريك لطيف. صب المحلول في صينية الصب واتركه يتجمد في غطاء الدخان.
- تحضير عينة الحمض النووي الريبي والرحلان الكهربائي
- قم بتخفيف عينة الحمض النووي الريبي إلى 200 نانوغرام (في 5 ميكرولتر) وأضف 5 ميكرولتر من صبغة تحميل الحمض النووي الريبي 2x.
- سخني العينات على حرارة 70 درجة مئوية لمدة 5 دقائق في حمام جاف.
- قم بتحميل 2 ميكرولتر من سلم الحمض النووي الريبي في الممر الأول و 10 ميكرولتر من العينات في الممرات المجاورة.
- إجراء الكهربائي في المخزن المؤقت MOPS عند 100 فولت لمدة 60 دقيقة لهلام 5 × 6 سم.
ملاحظة: اضبط ظروف الرحلان الكهربائي حسب أحجام الأمبليكون. - تصور الجل على جهاز نقل الأشعة فوق البنفسجية.
ملاحظة: يشير وجود نطاق واحد ~ 600 حجم النوكليوتيدات إلى تحضير الحمض النووي الريبي السليم (انظر الشكل 2 أ).
6. بولي (أ) قياس طول الذيل
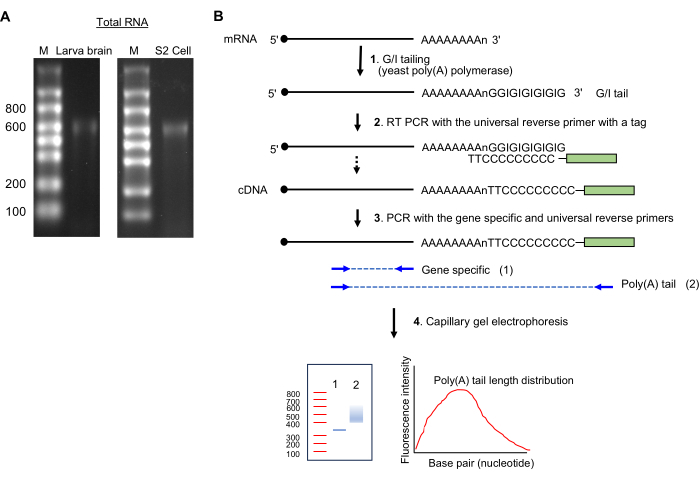
الشكل 2: تحضير عينة الحمض النووي الريبوزي (RNA) ومقايسة ذيل poly(A). (أ) تظهر صور هلام الحمض النووي الريبي إجمالي الحمض النووي الريبي من دماغ يرقة ذبابة الفاكهة (يسار) وخلايا S2 (يمين) على هلام أغاروز فورمالديهايد بنسبة 1.5٪. تظهر أحجام سلم الحمض النووي الريبي أحادي الشريط في النيوكليوتيدات على الحارة M. لاحظ نطاقات RNA رئيسية عند ~ 600 nt ، وهي من rRNA. (ب) مخططات مقايسة الذيل بولي (أ). اختصار: G / I = جوانوزين / إينوزين. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
- مخلفات GI (الشكل 2 ب)
- الحفاظ على الكواشف على الجليد ، وإعداد الخليط التالي (20 ميكرولتر): ما يصل إلى 14 ميكرولتر من إجمالي عينة الحمض النووي الريبي (1 ميكروغرام) ، 4 ميكرولتر من مزيج عازلة 5x الذيل ، و 2 ميكرولتر من مزيج إنزيم الذيل 10x.
- احتضان في 37 درجة مئوية لمدة 60 دقيقة في دورة حرارية.
- أضف 1.5 ميكرولتر من محلول توقف الذيل ؛ يحفظ على الثلج لمدة 2 دقيقة.
ملاحظة: تابع النسخ العكسي أو قم بتخزين عينات الحمض النووي الريبي GI-tailed عند -80 درجة مئوية حتى تصبح جاهزة للمتابعة في النسخ العكسي.
- النسخ العكسي وتضخيم PCR
- توليف cDNA عن طريق تحضير المزيج واحتضانه وفقا للشروط الموضحة في الملف التكميلي 1.
- قم بتخفيف عينات cDNA وإجراء تفاعل البوليميراز المتسلسل لتضخيم الحمض النووي كما هو موضح في الملف التكميلي 1.
7. تحليل منتج PCR بواسطة هلام الأغاروز الكهربائي
- قم بتحليل جزء صغير (2-5 ميكرولتر) من منتجات تفاعل البوليميراز المتسلسل من الخطوة 6.2.2 على هلام أغاروز 2.5٪ عن طريق الرحلان الكهربائي عند 100 فولت لمدة 45 دقيقة لمراقبة الجودة.
- تحقق من خصوصية تفاعل البوليميراز المتسلسل للتفاعلات الخاصة بالجينات والذيل عن طريق تسلسل نطاقات تفاعل البوليميراز المتسلسل المستخرجة بالهلام.
8. الرحلان الكهربائي الشعري
- قم بإجراء رحلان كهربائي جل عالي الدقة على 1 ميكرولتر من منتجات تفاعل البوليميراز المتسلسل (0.5-5 نانوغرام / ميكرولتر) من تفاعل البوليميراز المتسلسل الخاص بالجينات والمتعدد (A) باستخدام المحلل الحيوي مع مجموعة الحمض النووي عالية الحساسية. ابحث عن قمم تم حلها جيدا تشير إلى تشغيل ناجح.
9. تحليل البيانات: بولي (A) قياس طول الذيل (الشكل 3)
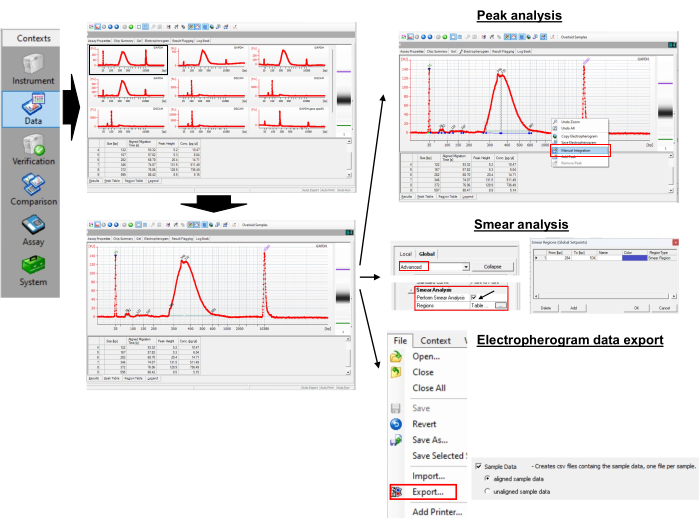
الشكل 3: طول ذيل Poly (A) وقياس قيمة الذروة. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
- الوصول إلى البيانات
- للوصول إلى البيانات ، افتح ملف xad في البرنامج.
- حدد اسم العينة أو السلم في لوحة عرض الشجرة.
ملاحظة: سيظهر هذا النتيجة كمخططات كهربائية أو صور تشبه الهلام للعينات المحددة. العلامة السفلية 35 زوج القاعدة (bp) والعلامة العليا 10,380 bp هي المعايير الداخلية المستخدمة لمواءمة بيانات السلم (50-7,000 bp) مع البيانات من آبار العينة. - قم بتكبير وتصغير المخططات الكهربائية والصور الشبيهة بالهلام لعرض التفاصيل.
- إجراء تحليل الذروة واللطاخة
- للحصول على أحجام الذروة ، افتح مخطط كهربية العينة المحددة.
- انقر بزر الماوس الأيمن على مخطط كهربية الفيروجرام وحدد التكامل اليدوي لتحديد القمم يدويا عن طريق سحب الخط الأفقي.
- لاحظ قيم الذروة في جدول الذروة. حدد القمة ذات الارتفاع الأكبر. هذه هي قمة طول ذيل poly (A) لعينة فردية. في المثال الموضح ، هو 346 bp.
- قم بتمكين أيقونة إظهار / إخفاء نقاط التعيين في القائمة العلوية وانتظر ظهور لوحة جديدة على الجانب الأيمن.
- حدد خيارات متقدمة، وقم بالتمرير لأسفل للعثور على إجراء تحليل اللطاخة، وحدد خانة الاختيار. سيؤدي هذا إلى إضافة جدول المنطقة إلى علامة التبويب مخطط كهربية الفيروجرام.
- حدد مخطط كهربية من عينة وانتقل إلى جدول المنطقة، الذي يعرض القائمة من [bp] وإلى [bp]. لتعيين البداية والنهاية [bp] ، انقر بزر الماوس الأيمن على مخطط كهربية الفيروجرام وحدد المنطقة لإضافة إضافة منطقة.
- انقر بزر الماوس الأيمن فوق أي خلايا في جدول المنطقة وحدد تعديل المناطق لإنشاء نافذة صغيرة جديدة منبثقة حيث يمكن تعيين مناطق مخصصة.
ملاحظة: على سبيل المثال، استخدمنا منطقة من 300 نقطة أساس إلى 550 نقطة أساس ل GAPDH. أسفر GAPDH PCR الخاص بالجينات عن ذروة عند 265 نقطة أساس. يمتد التمهيدي العالمي (الجدول 1) طول poly (A) PCR بمقدار 35 bp عن طريق التلدين إلى الحمض النووي الريبي G / I-tailed. وهكذا ، تبدأ أول نيوكليوتيدات الأدينين على GAPDH RNA عند 300 bp (265 + 35). لقد حددنا بشكل تعسفي الحد الأقصى لطول ذيل poly (A) إلى 250 (300 + 250 = 550). من جدول المنطقة، يرجع البرنامج متوسط الحجم داخل المنطقة ك 387 نقطة أساس. - استخدم المعادلة (1) لحساب طول ذيل poly (A) على mRNA محل الاهتمام:
بولي (أ) طول الذيل = (أ - ب - 35) (1)
حيث A هو متوسط bp لمنتج تفاعل البوليميراز المتسلسل الخاص ببولي (A) من مخطط كهربي (أي 387 bp ل GAPDH) ، B هو ذروة bp لمنتج تفاعل البوليميراز المتسلسل الخاص بالجينات من مخطط كهربي (أي 265 bp ل GAPDH) ، و "35" هو طول علامة التمهيدي العكسي العالمية.
ملاحظة: من الحساب أعلاه ، فإن متوسط طول ذيل بولي (A) ل GAPDH هو 387 - 265 - 35 = 87 نقطة أساس.
10. تصور توزيع طول الذيل بولي (أ)
- تصدير البيانات كملف csv ضمن ملف | تصدير | عينة بيانات لاسترداد عينة البيانات.
ملاحظة: يعرض ملف csv المصدر وقت التشغيل بدلا من bp على المحور X. - انتقل إلى مخطط كهربية لعينة وتحقق من إظهار الأحجام في علامة التبويب مخطط كهربية الفيروجرام لتحويل جدول المنطقة تلقائيا في جدول المنطقة من bp إلى وقت التشغيل. في المثال ، 70.39 ثانية إلى 86.28 ثانية تتوافق مع 300 نقطة أساس إلى 550 نقطة أساس.
- افتح ملف csv وحدد القيم من 70.39 ثانية إلى 86.28 ثانية من وقت التشغيل لإنشاء رسم بياني. لتصور أحجام bp إلى المحور X على الرسم البياني ، قم بتصدير مخطط كهربية الفيروجرام بأحجام bp كملف صورة وقم بتراكبه على الرسم البياني الذي تم إنشاؤه في جدول البيانات. سيتطابق هذا بشكل مناسب مع أحجام bp على توزيع ذيل poly (A).
Access restricted. Please log in or start a trial to view this content.
النتائج
هنا ، قمنا بتحليل طول ذيل poly (A) ل Dscam1 و GAPDH من أدمغة يرقات ذبابة الفاكهة (الشكل 4). تم تصور الحمض النووي الريبي المعزول على هلام الأغاروز لمراقبة الجودة. يشير نطاق الحمض النووي الريبي الواحد بحجم 600 نيوكليوتيد تقريبا إلى تحضير الحمض النووي الريبي السليم (
Access restricted. Please log in or start a trial to view this content.
Discussion
في هذا البروتوكول ، نصف تقنية تشريح دماغ يرقات ذبابة الفاكهة من تجول3 المرحلة الداخلية بالإضافة إلى تحضير العينة من خلايا ذبابة الفاكهة S2. نظرا للطبيعة المرنة ل mRNAs ، يتطلب جمع العينات مزيدا من الحذر. بالنسبة لتشريح دماغ اليرقات ، يجب عدم تلف الأدمغة أثناء العزلة ويجب عدم ال...
Access restricted. Please log in or start a trial to view this content.
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للكشف عنه.
Acknowledgements
تم دعم هذه الدراسة من قبل المعهد الوطني للاضطرابات العصبية والسكتة الدماغية R01NS116463 إلى JK ، ومرفق التصوير الخلوي والجزيئي الأساسي في جامعة نيفادا ، رينو ، والذي تم دعمه من قبل المعاهد الوطنية للصحة Grant P20GM103650 واستخدامها للبحث المذكور في هذه الدراسة.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
References
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435(2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622(2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57(2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294(2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved