É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Medição do comprimento da cauda de Poly A do cérebro da larva de Drosophila e da linha celular
Neste Artigo
Resumo
O protocolo descreve um método eficiente e confiável para quantificar o comprimento poli(A) do gene de interesse do sistema nervoso de Drosophila , que pode ser facilmente adaptado a tecidos ou tipos celulares de outras espécies.
Resumo
A poliadenilação é uma modificação pós-transcricional crucial que adiciona caudas de poli(A) à extremidade 3' das moléculas de RNAm. O comprimento da cauda poli(A) é fortemente regulado por processos celulares. A desregulação da poliadenilação de RNAm tem sido associada à expressão gênica anormal e a várias doenças, incluindo câncer, distúrbios neurológicos e anormalidades do desenvolvimento. Portanto, compreender a dinâmica da poliadenilação é vital para desvendar as complexidades do processamento de RNAm e da regulação gênica pós-transcricional.
Este trabalho apresenta um método para medir o comprimento da cauda de poli(A) em amostras de RNA isoladas de cérebros de larvas de Drosophila e células S2 de Drosophila Schneider. Empregamos a abordagem de rejeito de guanosina/inosina (G/I), que envolve a adição enzimática de resíduos de G/I na extremidade 3' do RNAm usando poli(A) polimerase de levedura. Esta modificação protege a extremidade 3' do RNA da degradação enzimática. As caudas poli(A) de comprimento total protegidas são então transcritas reversamente usando um primer antisenso universal. Posteriormente, a amplificação por PCR é realizada usando um oligo gene-específico que tem como alvo o gene de interesse, juntamente com uma sequência universal oligo usada para transcrição reversa.
Isso gera produtos de PCR englobando as caudas poli(A) do gene de interesse. Como a poliadenilação não é uma modificação uniforme e resulta em caudas de comprimentos variados, os produtos de PCR exibem uma variedade de tamanhos, levando a um padrão de esfregaço no gel de agarose. Finalmente, os produtos de PCR são submetidos à eletroforese em gel capilar de alta resolução, seguida de quantificação usando os tamanhos dos produtos de poli(A) PCR e do produto de PCR gene-específico. Esta técnica oferece uma ferramenta simples e confiável para analisar comprimentos de cauda poli(A), permitindo-nos obter insights mais profundos sobre os intrincados mecanismos que governam a regulação do RNAm.
Introdução
A maioria dos mRNAs eucarióticos são pós-transcricionalmente poliadenilados, em seu término 3′ no núcleo, pela adição de adenosinas não moldadas por poli(A) polimerases canônicas. Uma cauda poli(A) intacta é fundamental durante todo o ciclo de vida do mRNA, pois é essencial para a exportação nuclear de mRNA1, facilita a interação com proteínas ligadoras de poli(A) para aumentar a eficiência translacional2 e confere resistência contra a degradação3. Em certos casos, a cauda de poli(A) também pode sofrer extensão no citoplasma, facilitada por poli(A) polimerases nãocanônicas4. No citoplasma, o comprimento da cauda do poli(A) muda dinamicamente e influencia o tempo de vida da molécula de RNAm. Numerosas polimerases e deadenilases são conhecidas por modular o comprimento da cauda 5,6,7. Por exemplo, o encurtamento das caudas poli(A) correlaciona-se com a repressão translacional, enquanto o alongamento das caudas poli(A) aumenta a tradução 8,9.
Estudos genômicos acumulados têm demonstrado a importância fundamental do comprimento da cauda poli(A) em várias facetas da biologia eucariótica. Isso inclui papéis no desenvolvimento de células germinativas, desenvolvimento embrionário inicial, plasticidade sináptica neuronal para aprendizagem e memória, e a resposta inflamatória10. Existem inúmeros métodos e ensaios desenvolvidos para medir o comprimento da cauda do poli(A). Por exemplo, o ensaio RNase H/oligo(dT) aproveita a RNase H na presença ou ausência de oligo(dT) para estudar o comprimento da cauda do poli(A)11,12. Outros métodos para estudar a cauda poli(A) incluem a amplificação por PCR das extremidades 3', como o teste de amplificação rápida das extremidades do cDNA poli(A) (RACE-PAT)12,13 e o teste poli(A) mediado por ligase (LM-PAT)14. Outras modificações do ensaio PAT incluem ePAT15 e sPAT16. A cauda G enzimática17,18 ou a cauda G/I da extremidade 3' são outras variações do ensaio PAT. Outras modificações dessas técnicas incluem o uso de primers fluorescentemente marcados juntamente com eletroforese em gel capilar para análise de alta resolução, conhecido como teste de poli(A) de alta resolução (Hire-PAT)19. Estes ensaios conduzidos por PCR permitem a quantificação rápida e de alta sensibilidade do comprimento poli(A).
Com o desenvolvimento do sequenciamento de última geração, um método de sequenciamento de alto rendimento, como o PAL-seq20 e o TAIL-seq21, permite análises de poliadenilação em escala de transcriptoma. No entanto, esses métodos fornecem apenas leituras curtas de sequenciamento de 36-51 nucleotídeos. Portanto, o FLAM-Seq22 foi desenvolvido para o perfil global do comprimento da cauda do mRNA de comprimento total e fornece leituras longas. A tecnologia de nanoporos23 fornece sequenciamento direto de RNA ou cDNA independente de PCR para estimativas do comprimento da cauda de poli(A). No entanto, esses métodos de alto rendimento não são isentos de limitações. Eles exigem grandes quantidades de materiais de partida, são caros e demorados. Além disso, a análise de transcrições raras pode ser extremamente desafiadora com métodos de alto rendimento, e métodos baseados em PCR de baixo rendimento ainda fornecem uma vantagem quando um pequeno número de transcritos precisa ser analisado, para experimentos piloto e validação de outros métodos.
Recentemente, demonstramos que os mRNAs de Dscam1 contêm caudas curtas de poli(A) em Drosophila, o que requer uma ligação não canônica da proteína de ligação citoplasmática poli(A) em Dscam1 3'UTR usando o método de rejeito G/I24. Aqui nós fornecemos um procedimento simplificado para a preparação de tecidos e quantificação do comprimento poli(A) de mRNAs do sistema nervoso Drosophila e células Drosophila S2.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Criação e seleção de larvas de Drosophila
- Manter/cultivar a estirpe de mosca (w1118, wildtype) em meio alimentar padrão a 25 °C numa incubadora humidificada.
- Selecione 10 larvas errantes de3º ínstar 72 h após a postura dos ovos.
- Coloque as larvas em uma placa de Petri vazia de 35 mm e lave-as suavemente, transferindo as larvas para a nova placa contendo água da torneira usando pinças. Faça isso 2x para remover qualquer alimento restante.
2. Isolamento cerebral de larvas de Drosophila (Figura 1)
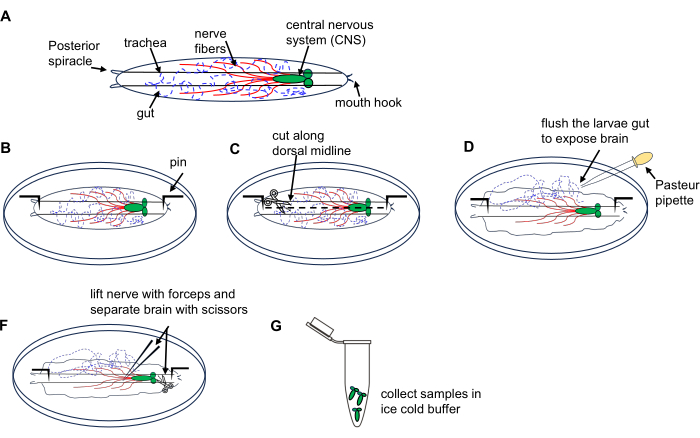
Figura 1: Dissecção do cérebro de larvas de Drosophila do estágio errante de3º ínstar. (A) Desenhos esquemáticos da larva de Drosophila. (B-G) Dissecção larva. Clique aqui para ver uma versão maior desta figura.
- Coloque 10 larvas em uma placa de dissecção contendo PBS gelado.
- Coloque a larva dorsal para cima (identificada por tubos traqueais que percorrem todo o seu comprimento) e fixe cada extremidade no fundo da placa, fazendo uma pequena incisão na parede do corpo na extremidade posterior.
- Cortar a parede do corpo ao longo da linha média dorsal em direção à extremidade anterior com tesoura de microdissecção.
- Lave brevemente o interior da larva usando uma pipeta de Pasteur 3x com PBS no prato para expor o cérebro.
- Localize e levante o cérebro usando a pinça e isole-o cuidadosamente usando uma tesoura de microdissecção.
- Transferir os cérebros dissecados para um tubo de microcentrífuga de 1,5 mL preenchido com PBS gelado sobre gelo; coletar todos os cérebros de larvas. Prossiga com a extração de RNA usando um kit de micropreparação de RNA, conforme descrito na seção 4.
NOTA: Faça uma dissecção de 10 larvas dentro de 15 min para evitar danos teciduais e degradação do RNA.
3. Drosophila S2 células de Schneider
- Cultivar células de Drosophila S2 em meio de Drosophila Schneider suplementado com 10% de soro fetal bovino (SFB) a 25 °C em estufa umidificada a uma densidade de 8 × 106 a 10 × 106 células/mL com viabilidade mínima de 90%.
- Em um tubo cônico estéril de 50 mL, diluir as células para 2,5 × 106 células/mL com meio Drosophila de Schneider suplementado com FBS a 10% que foi pré-aquecido a 25 °C.
- Transferir 8 mL da suspensão celular (20 × 106 células) para uma placa de cultura de 100 mm e adicionar 4 mL de meio para chegar a 12 mL (dia 1).
- Incubar as células cultivadas a 25 °C numa estufa humidificada.
NOTA: As células aderem frouxamente à placa após 12-16 h (dia 2). - Transfectar as células com plasmídeos de DNA apropriados24.
- Incubar por 48 h em incubadora umedecida.
- Após a incubação, coletar células adicionando 5 mL de PBS gelado por pipetagem suave (dia 4).
- Transfira as células para um tubo de 15 mL.
- Granular as células centrifugando a 1.000 × g por 5 min a 4 °C.
- Enxaguar as células 2x com PBS gelado por pipetagem suave e coletar as células centrifugando-as a 1.000 × g por 5 min a 4 °C.
- Realize a extração de RNA usando um kit de miniprep de RNA.
Observação : execute as seguintes etapas dentro de uma capela de fluxo laminar estéril.
4. Extração de RNA total do cérebro de larvas de Drosophila e células S2
- Cérebro larval: Remova o PBS por centrifugação breve (giro curto de 8 s a 5.000 × g).
- Adicionar 600 μL de tampão de lise de RNA e homogeneizar 10x com um pilão plástico. Inspecione visualmente o tubo sob um microscópio estéreo para garantir a lise completa.
- Centrifugar a 1.000 × g por 5 min a 4 °C para remover detritos teciduais. Transfira o sobrenadante limpo para um tubo de microcentrífuga livre de nucleases.
- Isole o RNA usando um kit de micropreparação de RNA de acordo com as instruções do fabricante.
NOTA: O uso de um kit de micropreparação de RNA é essencial para as amostras de cérebro de larvas devido à pequena quantidade de RNA presente nas amostras. - Células S2: Remova o PBS e isole o RNA de acordo com as instruções do fabricante.
- Medir o rendimento e a qualidade do RNA por espectrofotometria e eletroforese em gel de agarose.
- Determinar a pureza e a quantidade de RNA extraído medindo a densidade óptica do RNA extraído em A260 nm e A280 nm, respectivamente. Certifique-se de que a relação A260 nm/A280 nm é ≥2,0 e a concentração de RNA é >350 ng/μL para aplicações a jusante.
NOTA: Um rendimento típico de RNA de 10 cérebros de larvas de Drosophila é de ~500-800 ng/μL ou 2,5-4 μg em 5 μL. Para células S2, o rendimento é de ~2-3 μg/μL (15-30 μg em 15 μL). O RNA isolado pode ser armazenado a -80 °C para armazenamento a longo prazo.
- Determinar a pureza e a quantidade de RNA extraído medindo a densidade óptica do RNA extraído em A260 nm e A280 nm, respectivamente. Certifique-se de que a relação A260 nm/A280 nm é ≥2,0 e a concentração de RNA é >350 ng/μL para aplicações a jusante.
5. Preparação do gel de RNA e eletroforese
- 1,5% de RNA desnaturante gel (100 mL)
OBS: O formaldeído é tóxico pelo contato com a pele e inalação de vapores; manuseá-lo em um exaustor de fumaça química.- Dissolver três comprimidos de agarose (1,5 g) em 82 ml de tampão MOPS (Ficheiro Suplementar 1) até que os comprimidos se partam completamente para formar partículas finas.
- Aqueça a pasta de agarose no micro-ondas até que a solução esteja límpida e todas as partículas estejam completamente dissolvidas.
- Arrefecer a solução a ~60 °C.
- Adicione 18 mL de formol a 37% e, em seguida, misture com um redemoinho suave. Despeje a solução na bandeja de fundição e deixe solidificar em uma exausta de fumaça.
- Preparação de amostras de RNA e eletroforese
- Diluir a amostra de ARN para 200 ng (em 5 μL) e adicionar 5 μL de corante de carregamento de 2x ARN.
- Aquecer as amostras a 70 °C durante 5 minutos em banho seco.
- Carregar 2 μL de escada de RNA na primeira faixa e 10 μL de amostras nas pistas adjacentes.
- Realizar eletroforese em tampão MOPS a 100 V por 60 min para um gel de 5 x 6 cm.
NOTA: Ajuste as condições de eletroforese dependendo dos tamanhos dos amplicones. - Visualize o gel em um transiluminador UV.
NOTA: A presença de uma única banda ~600 tamanho de nucleotídeo indica preparação de RNA intacta (ver Figura 2A).
6. Medição do comprimento da cauda Poly(A)
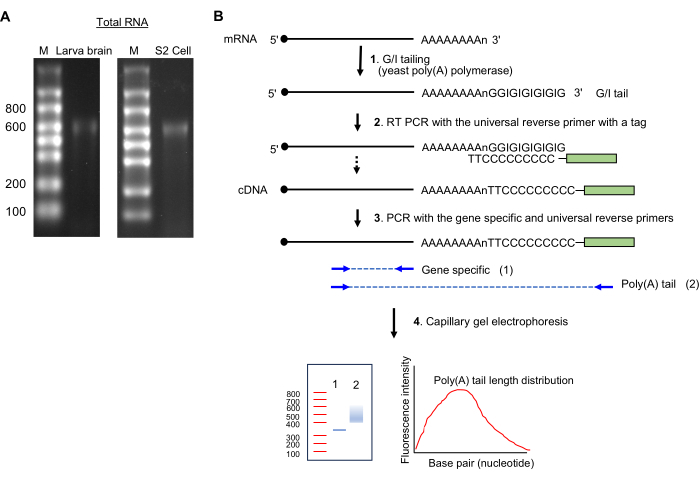
Figura 2: Preparação da amostra de RNA e ensaio de poli(A)-cauda . (A) As imagens de RNA gel mostram RNA total do cérebro da larva de Drosophila (esquerda) e das células S2 (direita) em um gel de agarose de formaldeído a 1,5%. Os tamanhos de escada de RNA de fita simples são mostrados em nucleotídeos na faixa M. Observe uma banda de RNA principal em ~600 nt, que é de rRNA. (B) Esquemas do ensaio poli(A)-cauda .Abreviação: G/I = guanosina/inosina. Clique aqui para ver uma versão maior desta figura.
- Rejeito GI (Figura 2B)
- Mantendo os reagentes no gelo, preparar a seguinte mistura (20 μL): até 14 μL de amostra de RNA total (1 μg), 4 μL de 5x mistura tampão Tail e 2 μL de 10x Tail enzyme mix.
- Incubar a 37 °C durante 60 minutos num termociclador.
- Adicionar 1,5 μL da solução de parada de cauda; Mantenha no gelo por 2 min.
NOTA: Prossiga para a transcrição reversa ou armazene as amostras de RNA de cauda GI a -80 °C até que esteja pronto para prosseguir para a transcrição reversa.
- Transcrição reversa e amplificação por PCR
- Sintetizar cDNA preparando a mistura e incubando-a nas condições descritas no Arquivo Suplementar 1.
- Diluir as amostras de cDNA e realizar PCR para amplificar o DNA, conforme indicado no Arquivo Suplementar 1.
7. Análise do produto de PCR por eletroforese em gel de agarose
- Analisar uma pequena porção (2-5 μL) dos produtos de PCR da etapa 6.2.2 em gel de agarose a 2,5% por eletroforese a 100 V por 45 min para controle de qualidade.
- Verificar a especificidade da PCR para reações gene-específicas e cauda-específicas através do sequenciamento das bandas de PCR extraídas em gel.
8. Eletroforese capilar
- Realizar eletroforese em gel de alta resolução em 1 μL de produtos de PCR (0,5-5 ng/μL) a partir de PCR gene-específico e poli(A)-específico usando o bioanalisador com um kit de DNA de alta sensibilidade. Procure picos bem resolvidos indicando uma execução bem-sucedida.
9. Análise dos dados: medida do comprimento da cauda poli(A) (Figura 3)
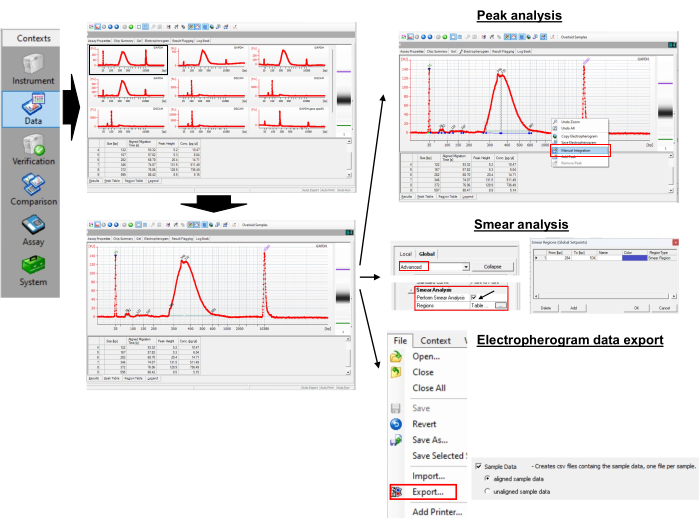
Figura 3: Medição do comprimento da cauda de Poly(A) e do valor de pico. Clique aqui para ver uma versão maior desta figura.
- Acessando dados
- Para acessar os dados, abra o arquivo xad no software.
- Selecione o nome da amostra ou a escada no painel de visualização em árvore.
NOTA: Isso mostrará o resultado como eletroferogramas ou imagens semelhantes a gel para as amostras selecionadas. O marcador inferior 35 pares de bases (pb) e o marcador superior 10.380 pb são os padrões internos utilizados para alinhar os dados da escada (50-7.000 pb) com os dados dos poços amostrais. - Aumente e diminua o zoom dos eletroferogramas e imagens em forma de gel para exibir os detalhes.
- Realização de análises de pico e esfregaço
- Para obter os tamanhos dos picos, abra o eletroferograma de uma amostra selecionada.
- Clique com o botão direito do mouse no eletroferograma e selecione a integração manual para selecionar manualmente os picos arrastando a linha horizontal.
- Observe os valores de pico na tabela Pico. Identifique o pico com a maior altura do pico. Este é um pico do comprimento da cauda poli(A) para uma amostra individual. No exemplo mostrado, é de 346 pb.
- Ative o ícone Mostrar/Ocultar Setpoints no menu superior e aguarde até que um novo painel apareça no lado direito.
- Selecione Avançado, role para baixo até encontrar Executar análise de esfregaço e marque a caixa de seleção. Isso adicionará a tabela Região à guia eletroferograma.
- Selecione um eletroferograma de uma amostra e vá para a tabela Região, que mostra o menu De [bp] e Para [bp]. Para definir o início e o fim [bp], clique com o botão direito do mouse no eletroferograma e selecione Região para adicionar Adicionar Região.
- Clique com o botão direito do mouse em qualquer célula na tabela Região e selecione Modificar Regiões para criar uma pequena nova janela onde as regiões personalizadas podem ser definidas.
NOTA: Por exemplo, usamos uma região de 300 pb a 550 pb para GAPDH. A PCR GPD GAPDH gene-específica produziu um pico em 265 pb. O primer universal (Tabela 1) estende o comprimento da poli(A) PCR em 35 pb via anelamento para RNAs de cauda G/I. Assim, o primeiro nucleotídeo de adenina no RNA do GAPDH começa em 300 pb (265 + 35). Arbitrariamente limitamos o comprimento máximo da cauda poli(A) para 250 (300 + 250 = 550). A partir da tabela de regiões, o programa retorna o tamanho médio dentro da região como 387 pb. - Use a equação (1) para calcular o comprimento da cauda poli(A) no mRNA de interesse:
Comprimento da cauda de Poly(A) = (A - B - 35) (1)
Onde A é a pb média do produto da PCR poli(A) específica do eletroferograma (i.e., 387 pb para GAPDH), B é o pico pb do produto da PCR gene-específico do eletroferograma (i.e., 265 pb para GAPDH), e "35" é o comprimento da etiqueta universal do primer reverso.
NOTA: A partir do cálculo acima, o comprimento médio da cauda poli(A) do GAPDH é de 387 - 265 - 35= 87 pb.
10. Visualizando a distribuição do comprimento da cauda poli(A)
- Exportar dados como um arquivo csv em Arquivo | Exportação | Dados de exemplo para recuperar dados de exemplo.
Observação : O arquivo csv exportado exibe tempo de execução em vez de bp no eixo X. - Vá para um eletroferograma de uma amostra e marque Mostrar tamanhos na guia Eletroferograma para converter automaticamente a tabela de regiões na Tabela de regiões de bp para tempo de execução. No exemplo, 70,39 s a 86,28 s correspondem a 300 pb a 550 pb.
- Abra o arquivo csv e selecione os valores de 70,39 s a 86,28 s de tempo de execução para gerar um gráfico. Para visualizar os tamanhos de bp para o eixo X no gráfico, exporte o eletroferograma com tamanhos de pb como um arquivo de imagem e sobreponha-o no gráfico gerado na planilha. Isso corresponderá adequadamente aos tamanhos de pb na distribuição da cauda poli(A).
Access restricted. Please log in or start a trial to view this content.
Resultados
Aqui, analisamos o comprimento da cauda poli(A) de Dscam1 e GAPDH de cérebros de larvas de Drosophila (Figura 4). RNAs isolados foram visualizados em gel de agarose para controle de qualidade. Uma única banda de RNA com cerca de 600 nucleotídeos indica preparação de RNA intacta (Figura 2A). RNAs foram submetidos à eletroforese capilar de rejeito G/I e alta resolução utilizando um bioanalisador Agilent 2100. As imagens em gel fo...
Access restricted. Please log in or start a trial to view this content.
Discussão
Neste protocolo, descrevemos a técnica para dissecar o cérebro de larvas de Drosophila do estágio errante de3º ínstar, bem como a preparação de amostras de células de Drosophila S2. Devido à natureza lábil dos mRNAs, a coleta de amostras requer cuidado extra. Para a dissecção cerebral larval, os cérebros não devem ser danificados durante o isolamento e não devem ser mantidos em solução por um período prolongado. Manter o tempo de dissecção para 8-10 min para uma rodada de ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
Este estudo foi apoiado pelo Instituto Nacional de Distúrbios Neurológicos e Stroke Grant R01NS116463 a J.K., e a instalação do núcleo de imagem celular e molecular na Universidade de Nevada, Reno, que foi apoiado pelo National Institutes of Health Grant P20GM103650 e usado para a pesquisa relatada neste estudo.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
Referências
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435(2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622(2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57(2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294(2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados