JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Drosophila Larva Brain and Cell Line의 Poly A 꼬리 길이 측정
요약
이 프로토콜은 다른 종의 조직이나 세포 유형에 쉽게 적응할 수 있는 초파리 신경계의 관심 유전자의 폴리(A) 길이를 정량화하기 위한 효율적이고 신뢰할 수 있는 방법을 설명합니다.
초록
폴리아데닐화(Polyadenylation)는 mRNA 분자의 3' 말단에 폴리(A) 꼬리를 추가하는 중요한 전사 후 변형입니다. 폴리(A) 꼬리의 길이는 세포 과정에 의해 엄격하게 조절됩니다. mRNA 폴리아데닐화의 조절 장애는 비정상적인 유전자 발현 및 암, 신경 장애 및 발달 이상을 포함한 다양한 질병과 관련이 있습니다. 따라서 폴리아데닐화의 역학을 이해하는 것은 mRNA 처리 및 전사 후 유전자 조절의 복잡성을 밝히는 데 매우 중요합니다.
이 논문은 초파리 유충 뇌와 초파리 슈나이더 S2 세포에서 분리된 RNA 샘플에서 폴리(A) 꼬리 길이를 측정하는 방법을 제시합니다. 우리는 효모 폴리(A) 중합효소를 사용하여 mRNA의 3' 말단에 G/I 잔기를 효소로 추가하는 구아노신/이노신(G/I) 테일링 접근법을 사용했습니다. 이 변형은 RNA의 3' 말단을 효소 분해로부터 보호합니다. 그런 다음 보호된 전체 길이 폴리(A) 꼬리는 범용 안티센스 프라이머를 사용하여 역전사됩니다. 이어서, PCR 증폭은 역전사에 사용되는 범용 서열 올리고와 함께 관심 유전자를 표적으로 하는 유전자 특이적 올리고를 사용하여 수행됩니다.
이것은 관심 유전자의 폴리(A) 꼬리를 포함하는 PCR 산물을 생성합니다. 폴리아데닐화는 균일한 변형이 아니고 다양한 길이의 꼬리가 생기기 때문에 PCR 산물은 다양한 크기를 표시하여 아가로스 겔에 얼룩 패턴을 유발합니다. 마지막으로, PCR 산물은 고분해능 모세관 겔 전기영동을 거친 후 폴리(A) PCR 산물 및 유전자 특이적 PCR 산물의 크기를 사용하여 정량화합니다. 이 기술은 폴리(A) 꼬리 길이를 분석하기 위한 간단하고 신뢰할 수 있는 도구를 제공하여 mRNA 조절을 지배하는 복잡한 메커니즘에 대한 더 깊은 통찰력을 얻을 수 있도록 합니다.
서문
대부분의 진핵생물 mRNA는 canonical poly(A) polymerases에 의한 non-templated 아데노신의 첨가에 의해 핵의 3′ 말단에서 전사 후 폴리아데닐화됩니다. 온전한 poly(A) tail은 mRNA핵 내보내기에 필수적이고1, poly(A)-결합 단백질과의 상호작용을 촉진하여 번역 효율을 높이고2, 분해에 대한 저항성을 부여하기 때문에 mRNA의 수명 주기 전반에 걸쳐 중추적인 역할을합니다3. 어떤 경우에는 poly(A) 꼬리가 세포질에서 확장될 수도 있는데, 이는 noncanonical poly(A) polymerases4에 의해 촉진된다. 세포질에서 폴리(A) 꼬리 길이는 동적으로 변화하고 mRNA 분자의 수명에 영향을 미칩니다. 수많은 폴리메라이제와 데데닐라아제는 꼬리 길이 5,6,7을 조절하는 것으로 알려져 있습니다. 예를 들어, poly(A) 꼬리의 짧아짐은 translational repression과 상관관계가 있는 반면, poly(A) tails의 길어지는 것은 translation 8,9를 향상시킵니다.
축적된 게놈 연구는 진핵생물 생물학의 다양한 측면에 걸쳐 폴리(A) 꼬리 길이의 근본적인 중요성을 입증했습니다. 여기에는 생식세포 발달, 초기 배아 발달, 학습 및 기억을 위한 신경 시냅스 가소성, 염증 반응10에서의 역할이 포함된다. 폴리(A) 꼬리 길이를 측정하기 위해 개발된 수많은 방법과 분석법이 있습니다. 예를 들어, RNase H/oligo(dT) 분석은 oligo(dT)의 존재 또는 부재에서 RNase H를 활용하여 poly(A) 꼬리 길이11,12를 연구합니다. 폴리(A) 꼬리를 연구하는 다른 방법으로는 cDNA 말단의 빠른 증폭과 같은 3' 말단의 PCR 증폭, 폴리(A) 테스트(RACE-PAT)12,13 및 리가아제 매개 폴리(A) 테스트(LM-PAT)14가 있습니다. PAT 분석의 추가 변형에는 ePAT15 및 sPAT16이 포함됩니다. 효소 G-테일링17,18 또는 3' 말단의 G/I-테일링은 PAT 분석의 다른 변형입니다. 이러한 기법의 추가 수정에는 고분해능 폴리(A) 테스트(Hire-PAT)19라고 하는 고분해능 분석을 위한 모세관 겔 전기영동과 함께 형광 표지 프라이머의 사용이 포함됩니다. 이러한 PCR 기반 분석은 빠르고 감도가 높은 폴리(A) 길이 정량을 가능하게 합니다.
차세대 염기서열분석법의 개발로 PAL-seq20 및 TAIL-seq21과 같은 고처리량 염기서열분석법을 통해 전사체 전체 규모에서 폴리아데닐화 분석이 가능합니다. 그러나 이러한 방법은 36-51 뉴클레오티드의 짧은 염기서열분석 판독만을 제공합니다. 따라서 FLAM-Seq22 는 full-length mRNA의 global tail length profiling을 위해 개발되었으며 긴 판독을 제공합니다. 나노포어 기술(23 )은 폴리(A) 꼬리 길이 추정을 위해 PCR 독립적, 직접 RNA 또는 직접 cDNA 시퀀싱을 제공합니다. 그러나 이러한 고처리량 방법에 제한이 없는 것은 아닙니다. 많은 양의 출발 물질이 필요하고 비용이 많이 들고 시간이 많이 걸립니다. 또한, 고처리량 분석법에서는 희귀 전사체 분석이 매우 어려울 수 있으며, 저처리량 PCR 기반 분석법은 파일럿 실험 및 다른 분석법의 검증을 위해 소수의 전사체를 분석해야 하는 경우에도 여전히 이점을 제공합니다.
우리는 최근 Dscam1 mRNA가 초파리에 짧은 폴리(A) 꼬리를 포함한다는 것을 입증했으며, 이는 G/I 테일링 방법24를 사용하여 Dscam1 3'UTR에서 세포질 폴리(A) 결합 단백질의 비표준 결합을 필요로 합니다. 여기서는 조직 준비 및 초파리 신경계 및 초파리 S2 세포의 mRNA의 폴리(A) 길이를 정량화하기 위한 간소화된 절차를 제공합니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 초파리 유충 사육 및 선택
- 가습 인큐베이터에서 25°C의 표준 플라이 식품 배지에서 플라이 균주(w1118, 야생형)를 유지/배양합니다.
- 알을 낳은 후 72시간 동안 방황하는 3번째 인스타 유충 10마리를 선택합니다.
- 유충을 35mm의 빈 페트리 접시에 넣고 집게를 사용하여 수돗물이 담긴 새 접시에 유충을 옮겨 부드럽게 씻습니다. 남은 음식을 제거하려면 이 작업을 2번 수행합니다.
2. 초파리 유충으로부터의 뇌 분리(그림 1)
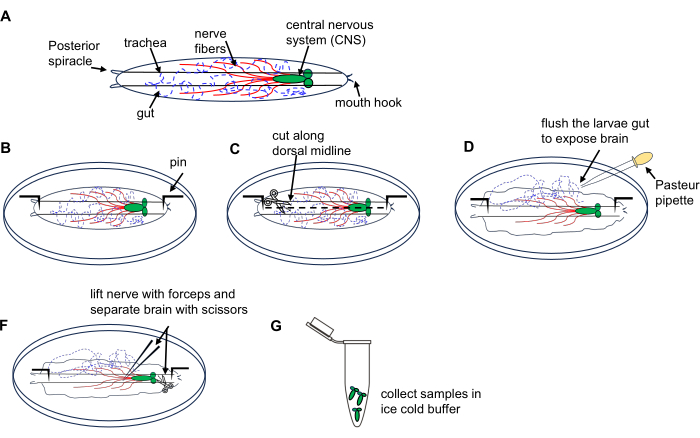
그림 1: 3차 방랑 단계에서 초파리 유충 뇌의 해부. (A) 초파리 유충의 개략도. (비지) 유충 해부. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 얼음처럼 차가운 PBS가 들어 있는 해부 접시에 유충 10마리를 놓습니다.
- 유충의 등쪽을 위로 향하게 하고(길이를 따라 달리는 기관관으로 식별됨) 각 끝을 접시 바닥에 고정한 다음 뒤쪽 끝의 체벽을 작게 절개합니다.
- 미세 해부 가위를 사용하여 등쪽 정중선을 따라 앞쪽 끝을 향해 체벽을 자릅니다.
- 접시에 PBS가 있는 파스퇴르 피펫을 3번 사용하여 유충 내부를 잠시 씻어내어 뇌를 노출시킵니다.
- 집게를 사용하여 뇌를 찾아 들어 올리고 미세 해부 가위를 사용하여 조심스럽게 분리합니다.
- 절개된 뇌를 얼음 위에 얼음처럼 차가운 PBS로 채워진 1.5mL 미세 원심분리기 튜브로 옮깁니다. 모든 애벌레 뇌를 수집하십시오. 섹션 4에 설명된 대로 RNA 마이크로프렙 키트를 사용하여 RNA 추출을 진행합니다.
참고: 조직 손상과 RNA 분해를 방지하기 위해 15분 이내에 10마리의 유충을 해부하십시오.
3. 초파리 S2 슈나이더 세포
- 가습 인큐베이터에서 25°C에서 10% 소 태아 혈청(FBS)이 보충된 Drosophila Schneider의 배지에서 최소 90%의 생존율로 8 × 106 - 10 ×10 6 cells/mL의 밀도로 성장시킵니다.
- 50mL 멸균 원뿔형 튜브에서 25°C로 예열된 10% FBS가 보충된 슈나이더의 Drosophila 배지를 사용하여 세포를 2.5 × 106 cells/mL로 희석합니다.
- 세포 현탁액 8mL(20 ×10 6 개 세포)를 100mm 배양 플레이트에 옮기고 배지 4mL를 추가하여 12mL가 됩니다(1일차).
- 25°C에서 가습된 인큐베이터에서 배양된 세포를 배양합니다.
알림: 세포는 12-16시간(2일) 후에 플레이트에 느슨하게 부착됩니다. - 적절한 DNA 플라스미드로 세포를 transfection(형질주입)24.
- 가습 인큐베이터에서 48시간 동안 배양합니다.
- 배양 후 부드러운 피펫팅으로 5mL의 얼음처럼 차가운 PBS를 추가하여 세포를 수집합니다(4일차).
- 세포를 15mL 튜브에 옮깁니다.
- 4°C에서 5분 동안 1,000× g 에서 원심분리하여 세포를 펠렛화합니다.
- 부드러운 피펫팅으로 얼음처럼 차가운 PBS로 세포를 2배 헹구고 4°C에서 5분 동안 1,000× g 에서 원심분리하여 세포를 수집합니다.
- RNA miniprep 키트를 사용하여 RNA 추출을 수행합니다.
알림: 멸균 층류 후드 내부에서 다음 단계를 수행합니다.
4. 초파리 유충 뇌 및 S2 세포에서 총 RNA 추출
- 애벌레 뇌: 간단한 원심분리(5,000× g에서 8초 짧은 회전)로 PBS를 제거합니다.
- 600μL의 RNA 용해 완충액을 추가하고 플라스틱 유봉으로 10배 균질화합니다. 실체 현미경으로 튜브를 육안으로 검사하여 완전한 용해를 확인합니다.
- 1,000× g 에서 4°C에서 5분 동안 원심분리하여 조직 파편을 제거합니다. 세척된 상층액을 뉴클레아제가 없는 마이크로 원심분리기 튜브로 옮깁니다.
- 제조업체의 지침에 따라 RNA 마이크로프렙 키트를 사용하여 RNA를 분리합니다.
참고: RNA 마이크로프렙 키트의 사용은 샘플에 존재하는 소량의 RNA 때문에 유충 뇌 샘플에 필수적입니다. - S2 세포: PBS를 제거하고 제조업체의 지침에 따라 RNA를 분리합니다.
- 분광광도법과 아가로스 겔 전기영동으로 RNA 수율과 품질을 측정합니다.
- A260 nm 및 A 280 nm에서 각각 A 260 nm 및 A280 nm에서 추출된 RNA의 광학 밀도를 측정하여 추출된 RNA의 순도와 양을 측정합니다. 다운스트림 애플리케이션의 경우 A260nm/A280nm 비율이 ≥2.0이고 RNA 농도가 >350ng/μL인지 확인하십시오.
참고: 10개의 초파리 유충 뇌에서 얻은 일반적인 RNA 수율은 5μL에서 ~500-800ng/μL 또는 2.5-4μg입니다. S2 셀의 경우 수율은 ~2-3μg/μL(15μL에서 15-30μg)입니다. 분리된 RNA는 장기 보관을 위해 -80°C에서 보관할 수 있습니다.
- A260 nm 및 A 280 nm에서 각각 A 260 nm 및 A280 nm에서 추출된 RNA의 광학 밀도를 측정하여 추출된 RNA의 순도와 양을 측정합니다. 다운스트림 애플리케이션의 경우 A260nm/A280nm 비율이 ≥2.0이고 RNA 농도가 >350ng/μL인지 확인하십시오.
5. RNA 겔 및 전기영동 준비
- 1.5% 변성 RNA 겔(100mL)
알림: 포름알데히드는 피부 접촉과 증기 흡입을 통해 독성이 있습니다. 화학 흄 후드에서 취급하십시오.- 아가로스 정제 3정(1.5g)을 82mL의 MOPS 완충액(보충 파일 1)에 정제가 완전히 분해되어 미세 입자를 형성할 때까지 녹입니다.
- 용액이 맑아지고 모든 입자가 완전히 용해될 때까지 전자레인지에서 아가로스 슬러리를 가열합니다.
- 용액을 ~60°C로 냉각합니다.
- 37% 포름알데히드 18mL를 넣고 부드럽게 휘저으며 섞습니다. 용액을 주조 트레이에 붓고 흄 후드에서 응고되도록 합니다.
- RNA 시료 전처리 및 전기영동
- RNA 샘플을 200ng(5μL 단위)로 희석하고 5μL의 2x RNA 로딩 염료를 추가합니다.
- 샘플을 건식 수조에서 70°C에서 5분 동안 가열합니다.
- 첫 번째 레인에 2μL의 RNA 래더를 로드하고 인접 레인에 10μL의 샘플을 로드합니다.
- 5 x 6cm 겔에 대해 100V의 MOPS 버퍼에서 60분 동안 전기영동을 수행합니다.
알림: 전기영동 조건을 조정하십시오 amplicon 크기. - UV 투광기에서 겔을 시각화합니다.
참고: 단일 밴드 ~600 뉴클레오티드 크기의 존재는 온전한 RNA 준비를 나타냅니다( 그림 2A 참조).
6. 폴리(A) 꼬리 길이 측정
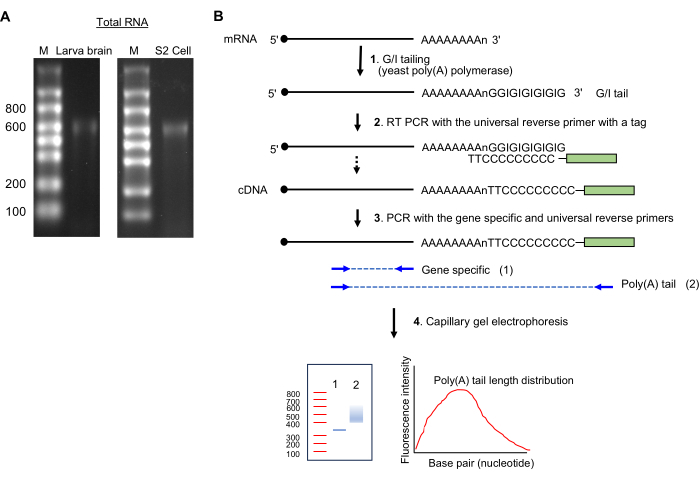
그림 2: RNA 시료 전처리 및 폴리(A)-꼬리 분석. (A) RNA 겔 이미지는 1.5% 포름알데히드 아가로스 겔에서 초파리 유충 뇌(왼쪽)와 S2 세포(오른쪽)의 총 RNA를 보여줍니다. 단일 가닥 RNA 사다리 크기는 레인 M의 뉴클레오티드에 표시되며, rRNA에서 온 ~600nt의 주요 RNA 밴딩에 주목하십시오. (B) 폴리(A)-테일 분석의 개략도. 약어: G/I = 구아노신/이노신. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- GI 테일링(그림 2B)
- 시약을 얼음 위에 두고 최대 14μL의 총 RNA 샘플(1μg), 4μL의 5x Tail 완충액 혼합물 및 2μL의 10x Tail 효소 혼합물을 준비합니다.
- 37°C에서 thermocycler에서 60분 동안 배양합니다.
- 테일 스톱 용액 1.5μL를 추가합니다. 2분 동안 얼음 위에 두십시오.
참고: 역전사를 진행하거나 역전사를 진행할 준비가 될 때까지 GI 꼬리 RNA 샘플을 -80°C에 보관합니다.
- 역전사 및 PCR 증폭
- 혼합물을 준비하고 보충 파일 1에 기재된 조건 하에서 배양하여 cDNA를 합성한다.
- cDNA 샘플을 희석하고 PCR을 수행하여 보충 파일 1에 표시된 대로 DNA를 증폭합니다.
7. 아가로스 겔 전기영동에 의한 PCR 산물 분석
- 품질 관리를 위해 45분 동안 100V에서 전기영동으로 2.5% 아가로스 겔에서 6.2.2단계 PCR 산물의 작은 부분(2-5μL)을 분석합니다.
- 겔 추출 PCR 밴드의 염기서열분석을 통해 유전자 특이적 및 꼬리 특이적 반응에 대한 PCR의 특이성을 검증합니다.
8. 모세관 전기영동
- 고감도 DNA 키트가 포함된 바이오분석기를 사용하여 유전자 특이적 및 폴리(A) 특이적 PCR의 PCR 산물 1μL(0.5-5ng/μL)에 대해 고분해능 겔 전기영동을 수행합니다. 성공적인 실행을 나타내는 잘 해결된 피크를 찾습니다.
9. 데이터 분석: 폴리(A) 꼬리 길이 측정(그림 3)
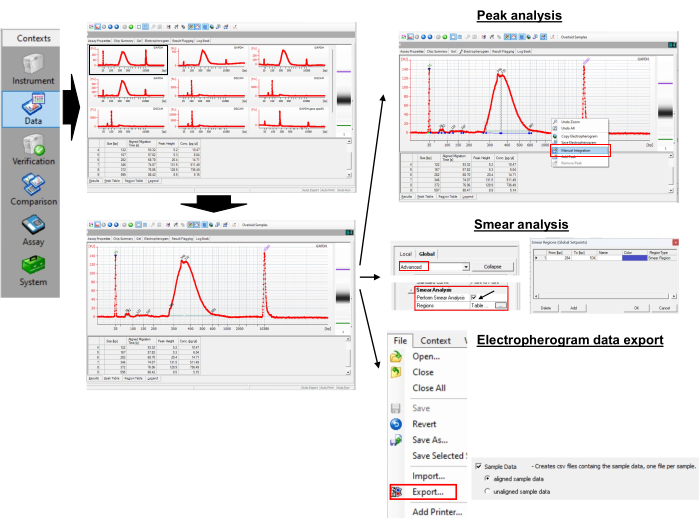
그림 3: Poly(A) 꼬리 길이 및 피크값 측정. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 데이터 액세스
- 데이터에 액세스하려면 소프트웨어에서 XAD 파일을 엽니다.
- 트리 뷰 패널에서 샘플 이름 또는 래더를 선택합니다.
알림: 이렇게 하면 선택한 샘플에 대한 전기 페로그램 또는 젤 같은 이미지로 결과가 표시됩니다. 하부 마커 35 염기쌍(bp)과 상부 마커 10,380 bp는 래더 데이터(50-7,000 bp)를 샘플 웰의 데이터와 정렬하는 데 사용되는 내부 표준물질입니다. - 일렉트로페로그램과 젤 같은 이미지를 확대 및 축소하여 세부 정보를 표시합니다.
- 피크 및 스미어 분석 수행
- 피크 크기를 얻으려면 선택한 샘플의 전기 페로그램을 엽니다.
- 일렉트로페로그램을 마우스 오른쪽 버튼으로 클릭하고 수동 적분을 선택하면 수평선을 드래그하여 피크를 수동으로 선택할 수 있습니다.
- 피크 테이블에서 피크 값을 관찰합니다. 피크 높이가 가장 큰 피크를 식별합니다. 이것은 개별 샘플에 대한 폴리(A) 꼬리 길이의 피크입니다. 표시된 예에서는 346bp입니다.
- 상단 메뉴에서 설정값 표시/숨기 기 아이콘을 활성화하고 오른쪽에 새 패널이 나타날 때까지 기다립니다.
- Advanced(고급)를 선택하고 아래로 스크롤하여 Perform Smear Analysis(스미어 분석 수행)를 찾은 다음 확인란을 선택합니다. 이렇게 하면 지역 테이블이 전기 페로그램 탭에 추가됩니다.
- 샘플에서 전기 페로그램을 선택하고 From [bp] 및 To [bp] 메뉴를 보여주는 Region 테이블로 이동합니다. 시작과 끝 [bp]를 설정하려면 일렉트로페로그램을 마우스 오른쪽 버튼으로 클릭하고 Region을 선택하여 Add Region을 추가합니다.
- 지역 테이블의 셀을 마우스 오른쪽 버튼으로 클릭하고 지역 수정을 선택하여 사용자 지정 영역을 설정할 수 있는 작은 새 창이 팝업되도록 합니다.
참고: 예를 들어 GAPDH에 300bp에서 550bp 사이의 영역을 사용했습니다. 유전자 특이적 GAPDH PCR은 265bp에서 피크를 생성했습니다. 범용 프라이머(표 1)는 G/I-tailed RNA에 대한 어닐링을 통해 poly(A) PCR의 길이를 35bp 연장합니다. 따라서, GAPDH RNA의 첫 번째 아데닌 뉴클레오티드는 300 bp (265 + 35)에서 시작한다. 최대 폴리(A) 꼬리 길이를 임의로 250(300 + 250 = 550)으로 제한했습니다. 영역 테이블에서 프로그램은 영역 내의 평균 크기를 387bp로 반환합니다. - 방정식 (1)을 사용하여 관심 mRNA의 poly(A) 꼬리 길이를 계산합니다.
폴리(A) 꼬리 길이 = (A - B - 35) (1)
여기서 A는 전기페로그램에서 poly(A)-특이적 PCR 산물의 평균 bp(즉, GAPDH의 경우 387 bp)이고, B는 electropherogram에서 유전자 특이적 PCR 산물의 피크 bp(즉, GAPDH의 경우 265 bp)이며, "35"는 범용 역방향 프라이머 태그의 길이입니다.
참고: 위의 계산에서 GAPDH의 평균 폴리(A) 꼬리 길이는 387 - 265 - 35= 87bp입니다.
10. poly(A) 꼬리 길이 분포 시각화
- File(파일) | 수출 | 샘플 데이터 - 샘플 데이터를 검색합니다.
참고: 내보낸 csv 파일은 X축에 bp가 아닌 런타임을 표시합니다. - 샘플의 전기 페로그램으로 이동하여 Electropherogram 탭의 Show Sizes를 확인하여 Region Table의 영역 테이블을 bp에서 실행 시간으로 자동 변환합니다. 이 예에서 70.39초에서 86.28초는 300bp에서 550bp에 해당합니다.
- csv 파일을 열고 실행 시간의 70.39초 에서 86.28초 사이의 값을 선택하여 그래프를 생성합니다. 그래프에서 bp 크기를 X축으로 시각화하려면 bp 크기의 전기 페로그램을 이미지 파일로 내보내고 스프레드시트에 생성된 그래프에 오버레이합니다. 이것은 poly(A) 꼬리 분포의 bp 크기와 적절하게 일치합니다.
Access restricted. Please log in or start a trial to view this content.
결과
여기에서 우리는 초파리 유충 뇌에서 Dscam1과 GAPDH의 poly(A) 꼬리 길이를 분석했습니다(그림 4). 분리된 RNA는 품질 관리를 위해 아가로스 겔에서 시각화되었습니다. 약 600 뉴클레오티드 크기의 단일 RNA 밴드는 온전한 RNA 준비를 나타냅니다(그림 2A). RNA는 Agilent 2100 바이오분석기를 사용하여 G/I 테일링 및 고분해능 모세관 전기영동을 수...
Access restricted. Please log in or start a trial to view this content.
토론
이 프로토콜에서는 방황하는 3번째 instar 단계에서 초파리 유충 뇌를 해부하는 기술과 초파리 S2 세포의 샘플 준비에 대해 설명합니다. mRNA의 불안정한 특성으로 인해 검체 채취에는 각별한 주의가 필요합니다. 유충 뇌 해부의 경우 격리 중에 뇌가 손상되어서는 안 되며 장기간 용액에 보관해서는 안 됩니다. 해부 라운드를 위해 해부 시간을 8-10분으로 유지하는 것이 필수적입?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
이 연구는 J.K.에 R01NS116463 국립 신경 장애 및 뇌졸중 연구소(National Institute of Neurological Disorders and Stroke Grant )와 국립 보건원(National Institutes of Health)의 P20GM103650보조금을 지원받아 이 연구에서 보고된 연구에 사용된 리노 네바다 대학교(University of Nevada, Reno)의 세포 및 분자 이미징 핵심 시설(Cellular and Molecular Imaging Core facility)의 지원을 받았습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
참고문헌
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435(2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622(2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57(2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294(2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유