Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesure de la longueur de la queue Poly A à partir du cerveau et de la lignée cellulaire des larves de drosophile
Dans cet article
Résumé
Le protocole décrit une méthode efficace et fiable pour quantifier la longueur poly(A) du gène d’intérêt du système nerveux de la drosophile , qui peut être facilement adaptée aux tissus ou aux types de cellules d’autres espèces.
Résumé
La polyadénylation est une modification post-transcriptionnelle cruciale qui ajoute des queues poly(A) à l’extrémité 3' des molécules d’ARNm. La longueur de la queue poly(A) est étroitement régulée par les processus cellulaires. La dérégulation de la polyadénylation de l’ARNm a été associée à une expression génique anormale et à diverses maladies, notamment le cancer, les troubles neurologiques et les anomalies du développement. Par conséquent, la compréhension de la dynamique de la polyadénylation est essentielle pour démêler les complexités du traitement de l’ARNm et de la régulation post-transcriptionnelle des gènes.
Cet article présente une méthode de mesure de la longueur de la queue poly(A) dans des échantillons d’ARN isolés à partir de cerveaux larvaires de drosophile et de cellules S2 de Drosophila Schneider. Nous avons utilisé l’approche de guanosine/inosine (G/I), qui implique l’ajout enzymatique de résidus G/I à l’extrémité 3' de l’ARNm à l’aide de la poly(A) polymérase de levure. Cette modification protège l’extrémité 3' de l’ARN de la dégradation enzymatique. Les queues de poly(A) protégées sur toute la longueur sont ensuite transcrites à l’envers à l’aide d’une amorce antisens universelle. Par la suite, l’amplification par PCR est réalisée à l’aide d’un oligo spécifique au gène qui cible le gène d’intérêt, ainsi que d’un oligo de séquence universel utilisé pour la transcription inverse.
Cela génère des produits de PCR englobant les queues poly(A) du gène d’intérêt. Étant donné que la polyadénylation n’est pas une modification uniforme et qu’elle donne des queues de longueurs variables, les produits PCR présentent une gamme de tailles, ce qui conduit à un motif de frottis sur le gel d’agarose. Enfin, les produits PCR sont soumis à une électrophorèse sur gel capillaire à haute résolution, suivie d’une quantification à l’aide des tailles des produits de PCR poly(A) et du produit PCR spécifique au gène. Cette technique offre un outil simple et fiable pour analyser les longueurs de queue poly(A), ce qui nous permet de mieux comprendre les mécanismes complexes régissant la régulation de l’ARNm.
Introduction
La plupart des ARNm eucaryotes sont polyadénylés post-transcriptionnel à leur extrémité 3′ dans le noyau par l’ajout d’adénosines non matricielles par des poly(A) polymérases canoniques. Une queue poly(A) intacte est essentielle tout au long du cycle de vie de l’ARNm, car elle est essentielle à l’exportation nucléaire de l’ARNm1, facilite l’interaction avec les protéines de liaison poly(A) pour améliorer l’efficacité de la traduction2 et confère une résistance à la dégradation3. Dans certains cas, la queue poly(A) peut également subir une extension dans le cytoplasme, facilitée par des poly(A) polymérases non canoniques4. Dans le cytoplasme, la longueur de la queue poly (A) change dynamiquement et influence la durée de vie de la molécule d’ARNm. De nombreuses polymérases et deadenylases sont connues pour moduler la longueur de la queue 5,6,7. Par exemple, le raccourcissement des queues poly(A) est corrélé à la répression traductionnelle, tandis que l’allongement des queues poly(A) améliore la traduction 8,9.
De plus en plus d’études génomiques ont démontré l’importance fondamentale de la longueur de la queue poly(A) dans diverses facettes de la biologie eucaryote. Cela inclut les rôles dans le développement des cellules germinales, le développement embryonnaire précoce, la plasticité synaptique neuronale pour l’apprentissage et la mémoire, et la réponse inflammatoire10. De nombreuses méthodes et tests ont été développés pour mesurer les longueurs de queue poly(A). Par exemple, le test RNase H/oligo(dT) tire parti de la RNase H en présence ou en absence d’oligo(dT) pour étudier la longueur de la queue poly(A)11,12. D’autres méthodes d’étude de la queue poly(A) comprennent l’amplification par PCR des extrémités 3', comme l’amplification rapide des extrémités de l’ADNc, le test poly(A) (RACE-PAT)12,13 et le test poly(A) médié par la ligase (LM-PAT)14. D’autres modifications du test PAT incluent ePAT15 et sPAT16. La queue enzymatiqueG 17,18 ou la queue G/I de l’extrémité 3' sont d’autres variantes du test PAT. D’autres modifications de ces techniques comprennent l’utilisation d’amorces marquées par fluorescence ainsi que l’électrophorèse sur gel capillaire pour l’analyse à haute résolution, appelée test poly(A) à haute résolution (Hire-PAT)19. Ces tests basés sur la PCR permettent une quantification rapide et haute sensibilité de la longueur du poly(A).
Avec le développement du séquençage de nouvelle génération, une méthode de séquençage à haut débit, telle que PAL-seq20 et TAIL-seq21, permet des analyses de polyadénylation à l’échelle du transcriptome. Cependant, ces méthodes ne fournissent que de courtes lectures de séquençage de 36 à 51 nucléotides. Par conséquent, FLAM-Seq22 a été développé pour le profilage global de la longueur de la queue de l’ARNm pleine longueur et fournit des lectures longues. La technologie Nanopore23 fournit un séquençage direct de l’ARN ou de l’ADNc indépendant de la PCR pour les estimations de la longueur de la queue poly(A). Cependant, ces méthodes à haut débit ne sont pas sans limites. Ils nécessitent de grandes quantités de matières premières, sont coûteux et prennent du temps. De plus, l’analyse de transcrits rares peut être extrêmement difficile avec les méthodes à haut débit, et les méthodes basées sur la PCR à faible débit offrent toujours un avantage lorsqu’un petit nombre de transcrits doivent être analysés, pour des expériences pilotes et la validation d’autres méthodes.
Nous avons récemment démontré que les ARNm de Dscam1 contiennent de courtes queues poly(A) chez la drosophile, ce qui nécessite une liaison non canonique de la protéine cytoplasmique de liaison poly(A) sur Dscam1 3'UTR en utilisant la méthode de queue G/I24. Nous proposons ici une procédure simplifiée pour la préparation des tissus et la quantification de la longueur poly(A) des ARNm du système nerveux de la drosophile et des cellules S2 de la drosophile .
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Élevage et sélection des larves de drosophile
- Maintenir/cultiver la souche de mouche (w1118, type sauvage) sur un milieu alimentaire standard à 25 °C dans un incubateur humidifié.
- Sélectionnez 10 larves errantes du 3e stade 72 h après la ponte.
- Placez les larves dans une boîte de Pétri vide de 35 mm et lavez-les délicatement en transférant les larves dans la nouvelle boîte contenant l’eau du robinet à l’aide d’une pince. Faites-le 2 fois pour enlever toute nourriture restante.
2. Isolement du cerveau des larves de drosophile (Figure 1)
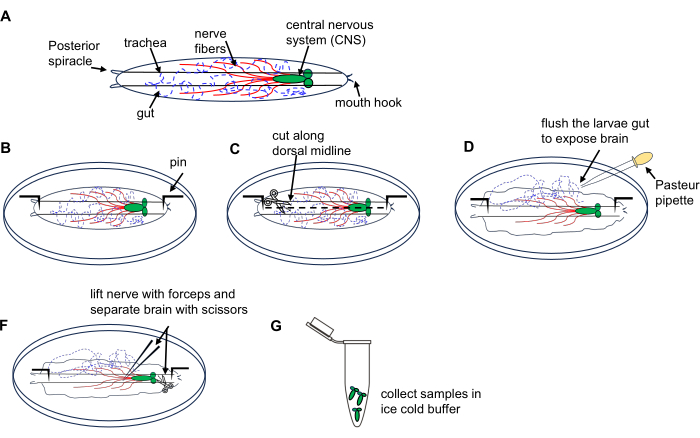
Figure 1 : Dissection du cerveau des larves de drosophile au stade d’errance du 3e stade. (A) Dessins schématiques des larves de drosophile. (B-G) Dissection larvaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Placer 10 larves sur une boîte de dissection contenant du PBS glacé.
- Placez la larve côté dorsal vers le haut (identifiée par des tubes trachéaux sur toute sa longueur) et épinglez chaque extrémité au fond de la boîte, puis faites une petite incision sur la paroi du corps à l’extrémité postérieure.
- Coupez la paroi du corps le long de la ligne médiane dorsale vers l’extrémité antérieure à l’aide de ciseaux de microdissection.
- Rincer brièvement l’intérieur de la larve à l’aide d’une pipette Pasteur 3x avec du PBS dans la boîte pour exposer le cerveau.
- Localisez et soulevez le cerveau à l’aide de la pince et isolez-le soigneusement à l’aide de ciseaux de microdissection.
- Transférer les cerveaux disséqués dans un tube de microcentrifugation de 1,5 mL rempli de PBS glacé sur de la glace ; Recueillir tous les cerveaux des larves. Procéder à l’extraction de l’ARN à l’aide d’un kit de micropréparation d’ARN comme décrit dans la rubrique 4.
REMARQUE : Effectuez une dissection de 10 larves en 15 minutes pour éviter les dommages tissulaires et la dégradation de l’ARN.
3. Cellules de Schneider de la drosophile S2
- Cultiver des cellules de drosophile S2 dans le milieu de Drosophila Schneider complété par 10 % de sérum de veau fœtal (FBS) à 25 °C dans un incubateur humidifié à une densité de 8 × 106 à 10 × 106 cellules/mL avec une viabilité minimale de 90 %.
- Dans un tube conique stérile de 50 mL, diluer les cellules à 2,5 × 10à 6 cellules/mL avec un milieu de drosophile de Schneider complété par 10 % de FBS préchauffé à 25 °C.
- Transvaser 8 mL de suspension cellulaire (20 × 106 cellules) dans une plaque de culture de 100 mm et ajouter 4 mL de milieu pour atteindre 12 mL (jour 1).
- Incuber les cellules cultivées à 25 °C dans un incubateur humidifié.
REMARQUE : Les cellules adhèrent lâchement à la plaque après 12-16 h (jour 2). - Transfectez les cellules avec les plasmides d’ADN appropriés24.
- Incuber pendant 48 h dans un incubateur humidifié.
- Après l’incubation, prélever les cellules en ajoutant 5 mL de PBS glacé par pipetage doux (jour 4).
- Transférer les cellules dans un tube de 15 ml.
- Granuler les cellules par centrifugation à 1 000 × g pendant 5 min à 4 °C.
- Rincer les cellules 2 fois avec du PBS glacé par pipetage doux et prélever les cellules en les centrifugeant à 1 000 × g pendant 5 min à 4 °C.
- Effectuez l’extraction de l’ARN à l’aide d’un kit de mini-préparation de l’ARN.
REMARQUE : Effectuez les étapes suivantes à l’intérieur d’une hotte à flux laminaire stérile.
4. Extraction de l’ARN total du cerveau des larves de drosophile et des cellules S2
- Cerveau larvaire : Éliminer le PBS par brève centrifugation (8 s d’essorage court à 5 000 × g).
- Ajouter 600 μL de tampon de lyse de l’ARN et homogénéiser 10x avec un pilon en plastique. Inspectez visuellement le tube au stéréomicroscope pour vous assurer d’une lyse complète.
- Centrifuger à 1 000 × g pendant 5 min à 4 °C pour éliminer les débris tissulaires. Transvasez le surnageant éliminé dans un tube de microcentrifugation sans nucléase.
- Isolez l’ARN à l’aide d’un kit de micropréparation d’ARN selon les instructions du fabricant.
REMARQUE : L’utilisation d’un kit de micropréparation d’ARN est essentielle pour les échantillons de cerveau larvaire en raison de la faible quantité d’ARN présente dans les échantillons. - Cellules S2 : Retirer le PBS et isoler l’ARN selon les instructions du fabricant.
- Mesurez le rendement et la qualité de l’ARN par spectrophotométrie et électrophorèse sur gel d’agarose.
- Déterminez la pureté et la quantité d’ARN extrait en mesurant la densité optique de l’ARN extrait à A260 nm et A280 nm, respectivement. Assurez-vous que le rapport A260 nm/A280 nm est de ≥2,0 et que la concentration d’ARN est de >350 ng/μL pour les applications en aval.
REMARQUE : Un rendement typique en ARN de 10 cerveaux de larves de drosophile est de ~500-800 ng/μL ou 2,5-4 μg dans 5 μL. Pour les cellules S2, le rendement est de ~2-3 μg/μL (15-30 μg dans 15 μL). L’ARN isolé peut être stocké à -80 °C pour un stockage à long terme.
- Déterminez la pureté et la quantité d’ARN extrait en mesurant la densité optique de l’ARN extrait à A260 nm et A280 nm, respectivement. Assurez-vous que le rapport A260 nm/A280 nm est de ≥2,0 et que la concentration d’ARN est de >350 ng/μL pour les applications en aval.
5. Préparation du gel d’ARN et de l’électrophorèse
- Gel d’ARN dénaturant à 1,5 % (100 ml)
REMARQUE : Le formaldéhyde est toxique par contact avec la peau et par inhalation de vapeurs ; manipulez-le dans une hotte chimique.- Dissoudre trois comprimés d’agarose (1,5 g) dans 82 mL de tampon MOPS (fichier supplémentaire 1) jusqu’à ce que les comprimés se désagrègent complètement pour former de fines particules.
- Chauffer la bouillie d’agarose au micro-ondes jusqu’à ce que la solution soit claire et que toutes les particules soient complètement dissoutes.
- Refroidir la solution à ~60 °C.
- Ajouter 18 ml de formaldéhyde à 37 %, puis mélanger en remuant doucement. Versez la solution dans le plateau de coulée et laissez-la se solidifier dans une hotte.
- Préparation d’échantillons d’ARN et électrophorèse
- Diluer l’échantillon d’ARN à 200 ng (dans 5 μL) et ajouter 5 μL de colorant de charge 2x ARN.
- Chauffer les échantillons à 70 °C pendant 5 min dans un bain sec.
- Chargez 2 μL d’échelle d’ARN dans la première voie et 10 μL d’échantillons dans les voies adjacentes.
- Effectuer une électrophorèse dans un tampon MOPS à 100 V pendant 60 min pour un gel de 5 x 6 cm.
REMARQUE : Ajustez les conditions d’électrophorèse en fonction de la taille des amplicons. - Visualisez le gel sur un transilluminateur UV.
REMARQUE : La présence d’une seule bande de ~600 nucléotides indique une préparation d’ARN intacte (voir Figure 2A).
6. Mesure de la longueur de la queue Poly(A)
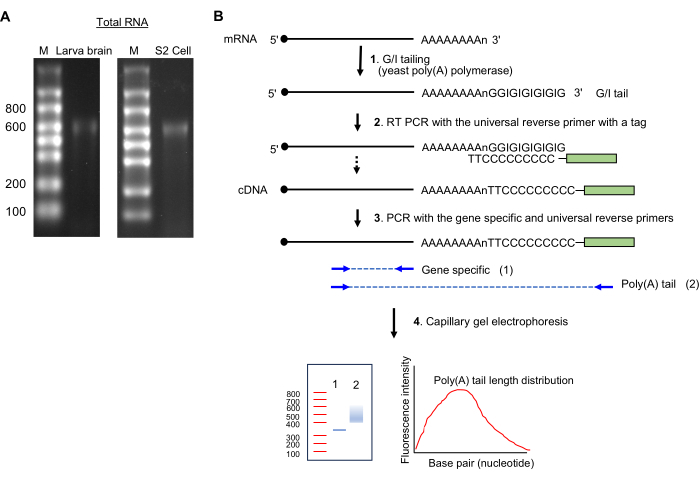
Figure 2 : Préparation de l’échantillon d’ARN et test de la queue poly(A). (A) Les images sur gel d’ARN montrent l’ARN total du cerveau de la larve de drosophile (à gauche) et des cellules S2 (à droite) sur un gel d’agarose au formaldéhyde à 1,5 %. Les tailles d’échelle d’ARN simple brin sont indiquées en nucléotides sur la voie M. Notez une bande d’ARN majeure à ~600 nt, qui provient de l’ARNr. (B) Schémas de l’essai de queue poly(A). Abréviation : G/I = guanosine/inosine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Résidus gastro-intestinaux (figure 2B)
- En gardant les réactifs sur de la glace, préparez le mélange suivant (20 μL) : jusqu’à 14 μL d’échantillon d’ARN total (1 μg), 4 μL de 5 mélanges tampons Tail et 2 μL de mélange d’enzymes 10 x Tail.
- Incuber à 37 °C pendant 60 min dans un thermocycleur.
- Ajouter 1,5 μL de solution de butée arrière ; Garder sur glace pendant 2 min.
REMARQUE : Procédez à la transcription inverse ou conservez les échantillons d’ARN à queue gastro-intestinale à -80 °C jusqu’à ce qu’ils soient prêts à procéder à la transcription inverse.
- Transcription inverse et amplification par PCR
- Synthétiser l’ADNc en préparant le mélange et en l’incubant dans les conditions décrites dans le fichier supplémentaire 1.
- Diluer les échantillons d’ADNc et effectuer une PCR pour amplifier l’ADN comme indiqué dans le fichier supplémentaire 1.
7. Analyse du produit PCR par électrophorèse sur gel d’agarose
- Analyser une petite portion (2-5 μL) des produits de PCR de l’étape 6.2.2 sur un gel d’agarose à 2,5 % par électrophorèse à 100 V pendant 45 min pour le contrôle de la qualité.
- Vérifiez la spécificité de la PCR pour les réactions spécifiques aux gènes et à la queue en séquençant les bandes de PCR extraites sur gel.
8. Électrophorèse capillaire
- Effectuez une électrophorèse sur gel à haute résolution sur 1 μL de produits PCR (0,5-5 ng/μL) à partir de PCR spécifiques au gène et à la poly(A) à l’aide du bioanalyseur avec un kit ADN haute sensibilité. Recherchez des pics bien résolus indiquant une exécution réussie.
9. Analyse des données : mesure de la longueur de la queue en poly(A) (figure 3)
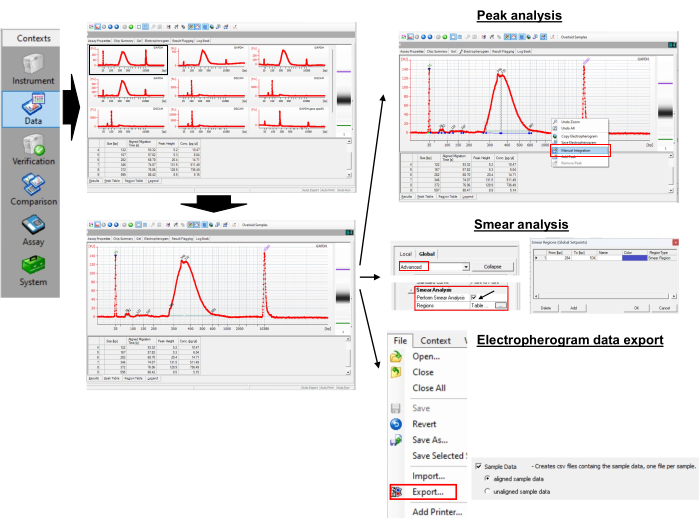
Figure 3 : Mesure de la longueur de la queue et de la valeur de crête du Poly(A). Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Accès aux données
- Pour accéder aux données, ouvrez le fichier xad dans le logiciel.
- Sélectionnez le nom de l’exemple ou l’échelle dans l’arborescence.
REMARQUE : Cela affichera le résultat sous forme d’électrophérogrammes ou d’images de type gel pour les échantillons sélectionnés. La paire de bases inférieure de 35 points de référence (pb) et la paire de points de repère supérieure de 10 380 pb sont les étalons internes utilisés pour aligner les données de l’échelle (50 à 7 000 pb) avec les données des puits d’échantillonnage. - Zoomez et dézoomez sur les électrophéromes et les images de type gel pour afficher les détails.
- Réalisation d’analyses de pics et de frottis
- Pour obtenir les tailles de pic, ouvrez l’électrophérogramme d’un échantillon sélectionné.
- Cliquez avec le bouton droit de la souris sur l’électrophérogramme et sélectionnez l’intégration manuelle pour sélectionner manuellement les pics en faisant glisser la ligne horizontale.
- Observez les valeurs de crête dans le tableau Peak. Identifiez le sommet avec la plus grande hauteur de pic. Il s’agit d’un pic de longueur de queue poly(A) pour un échantillon individuel. Dans l’exemple présenté, il est de 346 pb.
- Activez l’icône Afficher/Masquer les points de consigne dans le menu supérieur et attendez qu’un nouveau panneau apparaisse sur le côté droit.
- Sélectionnez Avancé, faites défiler vers le bas pour trouver Effectuer une analyse de frottis, puis cochez la case. Cela ajoutera la table des régions à l’onglet électrophérogramme.
- Sélectionnez un électrophérogramme dans un échantillon et accédez à la table Région, qui affiche les menus De [bp] et À [bp]. Pour définir le début et la fin [bp], cliquez avec le bouton droit de la souris sur l’électrophérogramme et sélectionnez Région pour ajouter Ajouter une région.
- Cliquez avec le bouton droit de la souris sur n’importe quelle cellule du tableau des régions et sélectionnez Modifier les régions pour faire apparaître une nouvelle petite fenêtre dans laquelle des régions personnalisées peuvent être définies.
REMARQUE : Par exemple, nous avons utilisé une région de 300 pb à 550 pb pour le GAPDH. La PCR GAPDH spécifique au gène a donné un pic à 265 pb. L’amorce universelle (tableau 1) prolonge la longueur de la poly(A) PCR de 35 pb par recuit aux ARN à queue G/I. Ainsi, le premier nucléotide d’adénine sur l’ARN GAPDH commence à 300 pb (265 + 35). Nous avons arbitrairement limité la longueur maximale de la queue poly(A) à 250 (300 + 250 = 550). À partir de la table de région, le programme renvoie la taille moyenne dans la région comme 387 pb. - Utilisez l’équation (1) pour calculer la longueur de la queue poly(A) sur l’ARNm d’intérêt :
Longueur de la queue Poly(A) = (A - B - 35) (1)
Où A est la pb moyenne du produit de PCR poly(A) spécifique de l’électrophérogramme (c.-à-d. 387 pb pour le GAPDH), B est la pb maximale du produit de PCR spécifique au gène de l’électrophérogramme (c.-à-d. 265 pb pour le GAPDH) et « 35 » est la longueur de l’étiquette d’amorce inverse universelle.
NOTE : D’après le calcul ci-dessus, la longueur moyenne de la queue poly(A) du GAPDH est de 387 - 265 - 35 = 87 pb.
10. Visualisation de la distribution de la longueur de la queue poly(A)
- Exporter les données sous forme de fichier csv sous Fichier | Exporter | Exemples de données pour récupérer des exemples de données.
REMARQUE : Le fichier csv exporté affiche le temps d’exécution plutôt que bp sur l’axe X. - Accédez à un électrophérogramme d’un échantillon et cochez Afficher les tailles dans l’onglet Électrophérogramme pour convertir automatiquement la table des régions de la table des régions de bp en temps d’exécution. Dans l’exemple, 70,39 s à 86,28 s correspondent à 300 pb à 550 pb.
- Ouvrez le fichier csv et sélectionnez les valeurs comprises entre 70,39 s et 86,28 s de temps d’exécution pour générer un graphique. Pour visualiser les tailles de bp sur l’axe X sur le graphique, exportez l’électrophérogramme avec les tailles de bp sous forme de fichier image et superposez-le sur le graphique généré dans la feuille de calcul. Cela correspondra de manière appropriée aux tailles de pb sur la distribution de queue poly(A).
Access restricted. Please log in or start a trial to view this content.
Résultats
Ici, nous avons analysé la longueur de la queue poly(A) de Dscam1 et GAPDH à partir de cerveaux larvaires de drosophile (Figure 4). Des ARN isolés ont été visualisés sur un gel d’agarose pour le contrôle de la qualité. Une seule bande d’ARN d’une taille d’environ 600 nucléotides indique une préparation d’ARN intacte (Figure 2A). Les ARN ont été soumis à la queue G/I et à l’électrophorèse capillaire à haute ...
Access restricted. Please log in or start a trial to view this content.
Discussion
Dans ce protocole, nous décrivons la technique de dissection du cerveau larvaire de la drosophile au stade du 3e stade errant ainsi que la préparation de l’échantillon à partir de cellules S2 de la drosophile. En raison de la nature labile des ARNm, le prélèvement d’échantillons nécessite une prudence accrue. Pour la dissection cérébrale larvaire, les cerveaux ne doivent pas être endommagés pendant l’isolement et ne doivent pas être maintenus en solution pendant une durée ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Cette étude a été financée par la subvention de l’Institut national des troubles neurologiques et des accidents vasculaires cérébraux R01NS116463 à J.K., et par la plateforme d’imagerie cellulaire et moléculaire de l’Université du Nevada, à Reno, qui a été soutenue par la subvention des National Institutes of Health P20GM103650 et utilisée pour la recherche rapportée dans cette étude.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
Références
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435(2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622(2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57(2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294(2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon