需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
果 蝇 幼虫脑和细胞系的Poly A尾长测量
摘要
该协议描述了一种有效且可靠的方法,用于量化 果蝇 神经系统中目的基因的poly(A)长度,该方法可以很容易地适应来自其他物种的组织或细胞类型。
摘要
聚腺苷酸化是一种重要的转录后修饰,可将 poly(A) 尾部添加到 mRNA 分子的 3' 末端。poly(A)尾巴的长度受细胞过程的严格调节。mRNA 多聚腺苷酸化的失调与异常基因表达和各种疾病有关,包括癌症、神经系统疾病和发育异常。因此,了解多聚腺苷酸化的动力学对于揭示 mRNA 加工和转录后基因调控的复杂性至关重要。
本文介绍了一种测量从 果蝇 幼虫脑和 果蝇 施耐德 S2 细胞中分离的 RNA 样品中 poly(A) 尾长的方法。我们采用鸟苷/肌苷 (G/I) 拖尾方法,该方法涉及使用酵母聚 (A) 聚合酶在 mRNA 的 3' 末端酶促添加 G/I 残基。这种修饰可保护 RNA 的 3' 末端免受酶降解。然后使用通用反义引物对受保护的全长poly(A)尾部进行反转录。随后,使用靶向目标基因的基因特异性寡核苷酸以及用于逆转录的通用序列寡核苷酸进行PCR扩增。
这会产生包含目的基因的poly(A)尾部的PCR产物。由于聚腺苷酸化不是一种均匀的修饰,并且会导致不同长度的尾部,因此PCR产物显示出一系列尺寸,导致琼脂糖凝胶上的涂抹图案。最后,对PCR产物进行高分辨率毛细管凝胶电泳,然后使用poly(A)PCR产物和基因特异性PCR产物的大小进行定量。该技术为分析poly(A)尾长提供了一种简单可靠的工具,使我们能够更深入地了解控制mRNA调控的复杂机制。
引言
大多数真核 mRNA 通过经典 poly(A) 聚合酶添加非模板化腺苷,在细胞核的 3' 末端进行转录后多聚腺苷化。完整的 poly(A) 尾在 mRNA 的整个生命周期中至关重要,因为它对 mRNA 核输出至关重要1,促进与 poly(A) 结合蛋白的相互作用以提高翻译效率2,并赋予抗降解性3。在某些情况下,poly(A) 尾部也可以在细胞质中延伸,这是由非经典 poly(A) 聚合酶4 促进的。在细胞质中,poly (A) 尾长动态变化并影响 mRNA 分子的寿命。已知许多聚合酶和脱烯酰化酶可调节尾长 5,6,7。例如,poly(A)尾巴的缩短与翻译抑制相关,而poly(A)尾巴的延长增强了翻译8,9。
不断积累的基因组研究已经证明了poly(A)尾长在真核生物学各个方面的基本意义。这包括在生殖细胞发育、早期胚胎发育、学习和记忆的神经元突触可塑性以及炎症反应中的作用10。已经开发了许多用于测量poly(A)尾部长度的方法和测定方法。例如,RNase H/oligo(dT)测定利用RNase H在存在或不存在oligo(dT)的情况下研究poly(A)尾长11,12。研究poly(A)尾部的其他方法包括3'末端的PCR扩增,例如cDNA末端poly(A)测试(RACE-PAT)的快速扩增(RACE-PAT)12,13和连接酶介导的poly(A)测试(LM-PAT)14。PAT测定的进一步修改包括ePAT15和sPAT16。酶促 G 尾17,18 或 3' 端的 G/I 尾是 PAT 测定的其他变体。这些技术的进一步修改包括使用荧光标记引物以及毛细管凝胶电泳进行高分辨率分析,称为高分辨率poly(A)测试(Hire-PAT)19。这些 PCR 驱动的检测可实现快速、高灵敏度的 poly(A) 长度定量。
随着下一代测序的发展,PAL-seq20 和 TAIL-seq21 等高通量测序方法允许在转录组范围内进行多聚腺苷酸化分析。然而,这些方法仅提供 36-51 个核苷酸的短测序读数。因此,FLAM-Seq22 是为全长 mRNA 的全局尾长分析而开发的,并提供长读长。纳米孔技术23 提供 PCR 非依赖性、直接 RNA 或直接 cDNA 测序,用于 poly(A) 尾长估计。然而,这些高通量方法并非没有局限性。它们需要大量的起始材料,价格昂贵且耗时。此外,使用高通量方法分析稀有转录本可能极具挑战性,而当需要分析少量转录本、用于试点实验和其他方法的验证时,基于低通量PCR的方法仍然具有优势。
我们最近证明 Dscam1 mRNA 在果蝇中含有短的 poly(A) 尾巴,这需要使用 G/I 拖尾方法24 将细胞质 poly(A) 结合蛋白与 Dscam1 3'UTR 进行非经典结合。在这里,我们提供了一种简化的程序,用于组织制备和定量来自果蝇神经系统和果蝇 S2 细胞的 poly(A) 长度的 mRNA。
研究方案
1. 饲养和选择果蝇幼虫
- 在加湿培养箱中,在25°C的标准苍蝇食品培养基上维持/培养苍蝇菌株(w1118,野生型)。
- 选10只游荡的3龄幼虫产卵后72小时。
- 将幼虫放入35毫米的空培养皿中,并使用镊子将幼虫转移到装有自来水的新培养皿中,轻轻清洗它们。执行此操作 2 次以去除任何剩余的食物。
2.从果蝇幼虫的大脑分离(图1)
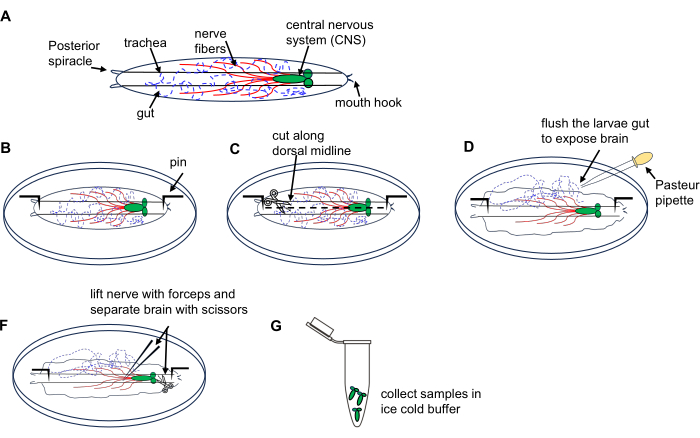
图1:3龄游荡期果蝇幼虫脑的解剖。 (A)果蝇幼虫示意图。(B-G)幼虫解剖。请点击这里查看此图的较大版本.
- 将10只幼虫放在含有冰冷PBS的解剖盘上。
- 将幼虫背侧朝上放置(通过沿其长度延伸的气管插管识别),并将每一端固定在培养皿的底部,然后在后端的体壁上做一个小切口。
- 使用显微解剖剪刀沿背中线向前端切割体壁。
- 使用巴斯德移液管在培养皿中用PBS进行3次短暂冲洗幼虫内部,以暴露大脑。
- 使用镊子定位并抬起大脑,并使用显微解剖剪刀小心地将其隔离。
- 将解剖的大脑转移到1.5mL微量离心管中,该离心管在冰上装满了冰冷的PBS;收集所有幼虫的大脑。如第 4 节所述,使用 RNA 微制备试剂盒进行 RNA 提取。
注意:在15分钟内解剖10只幼虫,以防止组织损伤和RNA降解。
3. 果蝇 S2 施耐德细胞
- 在25°C下,在25°C下在补充有10%胎牛血清(FBS)的果蝇Schneider培养基中培养果蝇S2细胞,密度为8×106至10 ×10 6个细胞/ mL,活力至少为90%。
- 在 50 mL 无菌锥形管中,用补充有 10% FBS 的施耐德果蝇培养基将细胞稀释至 2.5 ×10 6 个细胞/mL,该培养基已预热至 25 °C。
- 将 8 mL 细胞悬液(20 × 106 个细胞)转移到 100 mm 培养板中,并加入 4 mL 培养基使其达到 12 mL(第 1 天)。
- 将培养的细胞在25°C下在湿润培养箱中孵育。
注意:细胞在12-16小时(第2天)后松散地粘附在板上。 - 用适当的 DNA 质粒转染细胞24.
- 在加湿培养箱中孵育48小时。
- 孵育后,通过轻轻移液(第 4 天)加入 5 mL 冰冷的 PBS 来收集细胞。
- 将细胞转移到 15 mL 试管中。
- 通过在4°C下以1,000× g 离心5分钟来沉淀细胞。
- 通过轻轻移液用冰冷的PBS冲洗细胞2次,并通过在4°C下以1,000× g 离心5分钟来收集细胞。
- 使用 RNA 小量制备试剂盒进行 RNA 提取。
注意: 在无菌层流罩内执行以下步骤。
4. 从果蝇幼虫脑和 S2 细胞中提取总 RNA
- 幼虫脑: 通过短暂离心(在5,000× g下短旋8秒)除去PBS。
- 加入 600 μL RNA 裂解缓冲液,并用塑料研杵匀浆 10 倍。在体视显微镜下目视检查试管以确保完全裂解。
- 在4°C下以1,000× g 离心5分钟以除去组织碎片。将清除的上清液转移到无核酸酶的微量离心管中。
- 根据制造商的说明,使用 RNA 微量制备试剂盒分离 RNA。
注意:由于样品中存在少量 RNA,因此使用 RNA 微制备试剂盒对于幼虫脑样品至关重要。 - S2 细胞: 根据制造商的说明去除 PBS 并分离 RNA。
- 通过分光光度法和琼脂糖凝胶电泳测量RNA产量和质量。
- 通过分别在A260nm 和A280nm处测量提取的RNA的光密度来确定提取的RNA的纯度和数量。确保 A260 nm/A 280 nm 比值为 ≥2.0,下游应用的 RNA 浓度为 >350 ng/μL。
注:10 个 果蝇 幼虫大脑的典型 RNA 产量为 ~500-800 ng/μL 或 5 μL 中的 2.5-4 μg。对于 S2 细胞,产率为 ~2-3 μg/μL(15 μL 中为 15-30 μg)。分离的RNA可以储存在-80°C下进行长期储存。
- 通过分别在A260nm 和A280nm处测量提取的RNA的光密度来确定提取的RNA的纯度和数量。确保 A260 nm/A 280 nm 比值为 ≥2.0,下游应用的 RNA 浓度为 >350 ng/μL。
5. RNA凝胶的制备和电泳
- 1.5% 变性 RNA 凝胶 (100 mL)
注意:甲醛通过皮肤接触和吸入蒸气有毒;在化学通风橱中处理它。- 将三片琼脂糖片剂(1.5g)溶解在82mL MOPS缓冲液(补充文件1)中,直至片剂完全分解形成细颗粒。
- 在微波炉中加热琼脂糖浆料,直到溶液澄清并且所有颗粒完全溶解。
- 将溶液冷却至~60°C。
- 加入 18 mL 37% 甲醛,然后轻轻旋转混合。将溶液倒入铸造托盘中,使其在通风橱中凝固。
- RNA样品制备和电泳
- 将 RNA 样品稀释至 200 ng(5 μL 中),并加入 5 μL 2x RNA 上样染料。
- 在干浴中将样品在70°C下加热5分钟。
- 在第一泳道中加载 2 μL RNA 分子量标准,在相邻泳道中加载 10 μL 样品。
- 在100V的MOPS缓冲液中进行电泳60分钟,以获得5×6cm凝胶。
注意:根据扩增子大小调整电泳条件。 - 在紫外透射仪上可视化凝胶。
注意:单条带~600核苷酸大小的存在表明RNA制备完整(见 图2A)。
6. Poly(A)尾长测量
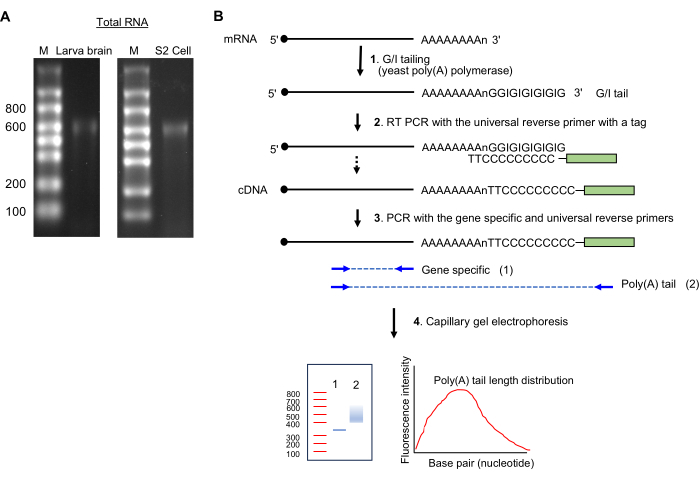
图 2:RNA 样品制备和 poly(A)-tail 测定。 (A) RNA 凝胶图像显示 1.5% 甲醛琼脂糖凝胶上来自 果蝇 幼虫脑(左)和 S2 细胞(右)的总 RNA。单链RNA分子量标准大小显示在泳道M上的核苷酸中,注意~600 nt处的主要RNA条带,它来自rRNA。(B) poly(A)-tail测定示意图。 缩写:G/I = 鸟苷/肌苷。 请点击这里查看此图的较大版本.
- GI拖尾(图2B)
- 将试剂保持在冰上,制备以下混合物 (20 μL):最多 14 μL 总 RNA 样品 (1 μg)、4 μL 5x Tail 缓冲液混合物和 2 μL 10x Tail 酶混合物。
- 在37°C下在热循环仪中孵育60分钟。
- 加入 1.5 μL 尾部止动液;在冰上保持2分钟。
注意:继续逆转录或将GI尾RNA样品储存在-80°C,直到准备好进行逆转录。
- 逆转录和PCR扩增
- 通过制备混合物并在 补充文件 1 中描述的条件下孵育来合成 cDNA。
- 稀释 cDNA 样品并进行 PCR 以扩增 DNA,如 补充文件 1 所示。
7. 琼脂糖凝胶电泳法PCR产物分析
- 通过在100V下电泳45分钟,在2.5%琼脂糖凝胶上分析步骤6.2.2中的一小部分(2-5μL)PCR产物,以进行质量控制。
- 通过对凝胶提取的PCR条带进行测序,验证PCR对基因特异性和尾部特异性反应的特异性。
8. 毛细管电泳
- 使用带有高灵敏度 DNA 试剂盒的生物分析仪对来自基因特异性和多 (A) 特异性 PCR 的 1 μL PCR 产物 (0.5-5 ng/μL) 进行高分辨率凝胶电泳。寻找分辨率高的峰值,表明运行成功。
9. 数据分析:poly(A)尾长测量(图3)
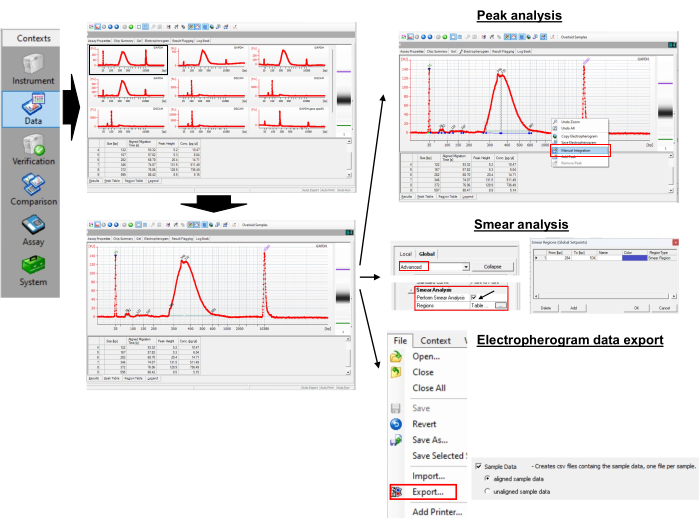
图 3:Poly(A) 尾部长度和峰值测量。 请点击这里查看此图的较大版本.
- 访问数据
- 要访问数据,请在软件中打开 xad 文件。
- 在树视图面板中选择示例名称或梯形图。
注意:这将以所选样品的电泳图或凝胶状图像的形式显示结果。下标记 35 碱基对 (bp) 和上标记 10,380 bp 是用于将标准数据 (50-7,000 bp) 与样品孔数据对齐的内标。 - 放大和缩小电泳图和凝胶状图像以显示细节。
- 执行峰和涂片分析
- 要获得峰尺寸,请打开所选样品的电泳图。
- 右键单击电泳图并选择 手动积分 ,通过拖动水平线手动选择峰。
- 观察 "峰值"表中的峰值。识别具有最大峰高的峰。这是单个样品的poly(A)尾长峰。在所示示例中,它是 346 bp。
- 启用顶部菜单上的 "显示/隐藏设定点 "图标,然后等待右侧弹出一个新面板。
- 选择 "高级",向下滚动以找到 "执行拖尾分析",然后选中该复选框。这会将 Region 表 添加到电泳图选项卡中。
- 从样品中选择一个电泳图,然后转到 "区域"(Region) 表,该表显示 "从 [bp] "和 "到"[bp]" 菜单。要设置开始和结束 [bp],请右键单击 电泳图 ,然后选择 "区域 "以添加 "添加区域"。
- 右键单击 "区域"(Region) 表格 中的任何单元格,然后选择 "修改区域"(Modify Regions ),弹出一个小的新窗口,可在其中设置自定义区域。
注:例如,我们对 GAPDH 使用了 300 bp 至 550 bp 的区域。基因特异性GAPDH PCR在265 bp处产生峰值。通用引物(表1)通过退火到G/I-tailed RNA将poly(A)PCR的长度延长35 bp。因此,GAPDH RNA 上的第一个腺嘌呤核苷酸从 300 bp (265 + 35) 开始。我们任意将最大poly(A)尾部长度限制为250(300 + 250 = 550)。从区域表中,程序返回区域内的平均大小为 387 bp。 - 使用公式 (1) 计算目标 mRNA 上的 poly(A) 尾长:
Poly(A) 尾长 = (A - B - 35) (1)
其中 A 是电泳图中 poly(A) 特异性 PCR 产物的平均 bp(即 GAPDH 为 387 bp),B 是电泳图中基因特异性 PCR 产物的峰值 bp(即 GAPDH 为 265 bp),"35"是通用反向引物标签的长度。
注:根据上述计算,GAPDH 的平均 poly(A) 尾长为 387 - 265 - 35= 87 bp。
10. 可视化poly(A)尾长分布
- 将数据导出为 csv 文件,位于 "文件"|"导出 |用于检索示例数据的示例数据 。
注意:导出的 csv 文件在 X 轴上显示 运行时间 而不是 bp 。 - 转到样品的电泳图,然后选中"电泳图"选项卡上的"显示尺寸",以自动将"区域表"上的区域表从 bp 转换为运行时。在本例中,70.39 秒至 86.28 秒对应于 300 bp 至 550 bp。
- 打开 csv 文件,选择运行时间 的 70.39 秒 到 86.28 秒 之间的值以生成图形。要在图表上将 bp 大小可视化到 X 轴,请将具有 bp 大小的电泳图导出为图像文件,并将其叠加在电子表格中生成的图表上。这将适当匹配 poly(A) 尾部分布的 bp 大小。
结果
在这里,我们分析了果蝇幼虫大脑中 Dscam1 和 GAPDH 的 poly(A) 尾长(图 4)。分离的RNA在琼脂糖凝胶上可视化,用于质量控制。大约 600 个核苷酸大小的单个 RNA 条带表示完整的 RNA 制备(图 2A)。使用 Agilent 2100 生物分析仪对 RNA 进行 G/I 拖尾和高分辨率毛细管电泳。使用 Agilent 2100 Expert 软件导出凝胶图像并进行相应组装。来自GAP...
讨论
在该方案中,我们描述了从徘徊的3龄阶段解剖果蝇幼虫大脑的技术以及果蝇S2细胞的样品制备。由于 mRNA 的不稳定性,样本采集需要格外小心。对于幼虫脑夹层,大脑在隔离期间不应受损,也不应长时间保存在溶液中。将解剖时间保持在 8-10 分钟进行一轮解剖是必不可少的。用RNA酶抑制剂补充解剖液也可能是有益的。至于 S2 细胞培养和转染,请在层流罩中执行所有步骤。
...披露声明
作者没有需要披露的利益冲突。
致谢
这项研究得到了美国国家神经疾病和中风研究所R01NS116463 JK 的支持,以及内华达大学里诺分校的细胞和分子成像核心设施,该设施得到了美国国立卫生研究院资助P20GM103650的支持,并用于本研究中报告的研究。
材料
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
参考文献
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435 (2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622 (2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57 (2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294 (2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。