È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazione della lunghezza della coda di Poly A dal cervello e dalla linea cellulare della larva di Drosophila
In questo articolo
Riepilogo
Il protocollo descrive un metodo efficiente e affidabile per quantificare la lunghezza del poli(A) del gene di interesse dal sistema nervoso della Drosophila , che può essere facilmente adattato a tessuti o tipi cellulari di altre specie.
Abstract
La poliadenilazione è una modificazione post-trascrizionale cruciale che aggiunge code di poli(A) all'estremità 3' delle molecole di mRNA. La lunghezza della coda poli(A) è strettamente regolata dai processi cellulari. La disregolazione della poliadenilazione dell'mRNA è stata associata a un'espressione genica anomala e a varie malattie, tra cui cancro, disturbi neurologici e anomalie dello sviluppo. Pertanto, comprendere le dinamiche della poliadenilazione è fondamentale per svelare le complessità dell'elaborazione dell'mRNA e della regolazione genica post-trascrizionale.
Questo articolo presenta un metodo per misurare la lunghezza della coda di poli(A) in campioni di RNA isolati da cervelli larvali di Drosophila e cellule S2 di Drosophila Schneider. Abbiamo utilizzato l'approccio di coda della guanosina/inosina (G/I), che prevede l'aggiunta enzimatica di residui di G/I all'estremità 3' dell'mRNA utilizzando la poli(A) polimerasi del lievito. Questa modifica protegge l'estremità 3' dell'RNA dalla degradazione enzimatica. Le code protette in poli(A) a tutta lunghezza vengono quindi trascritte inversamente utilizzando un primer antisenso universale. Successivamente, l'amplificazione PCR viene eseguita utilizzando un oligo gene-specifico che ha come bersaglio il gene di interesse, insieme a un oligo di sequenza universale utilizzato per la trascrizione inversa.
Questo genera prodotti di PCR che comprendono le code di poli(A) del gene di interesse. Poiché la poliadenilazione non è una modifica uniforme e si traduce in code di lunghezza variabile, i prodotti PCR mostrano una gamma di dimensioni, portando a un modello di striscio sul gel di agarosio. Infine, i prodotti PCR vengono sottoposti a elettroforesi su gel capillare ad alta risoluzione, seguita da quantificazione utilizzando le dimensioni dei prodotti poly(A) PCR e del prodotto PCR gene-specifico. Questa tecnica offre uno strumento semplice e affidabile per l'analisi delle lunghezze della coda di poli(A), consentendoci di ottenere informazioni più approfondite sugli intricati meccanismi che regolano la regolazione dell'mRNA.
Introduzione
La maggior parte degli mRNA eucariotici sono post-trascrizionalmente poliadenilati al loro terminale 3′ nel nucleo mediante l'aggiunta di adenosine non modellate da poli(A) polimerasi canoniche. Una coda di poli(A) intatta è fondamentale per tutto il ciclo di vita dell'mRNA, in quanto è essenziale per l'esportazione nucleare dell'mRNA1, facilita l'interazione con le proteine che legano il poli(A) per migliorare l'efficienza traduzionale2 e conferisce resistenza alla degradazione3. In alcuni casi, la coda di poli(A) può anche subire un'estensione nel citoplasma, facilitata dalle poli(A) polimerasi non canoniche4. Nel citoplasma, la lunghezza della coda poli (A) cambia dinamicamente e influenza la durata della vita della molecola di mRNA. Numerose polimerasi e deadenilasi sono note per modulare la lunghezza della coda 5,6,7. Ad esempio, l'accorciamento delle code di poli(A) è correlato alla repressione traslazionale, mentre l'allungamento delle code di poli(A) migliora la traduzione 8,9.
Numerosi studi genomici hanno dimostrato l'importanza fondamentale della lunghezza della coda poli(A) in vari aspetti della biologia eucariotica. Ciò include i ruoli nello sviluppo delle cellule germinali, nello sviluppo embrionale precoce, nella plasticità sinaptica neuronale per l'apprendimento e la memoria e nella risposta infiammatoria10. Sono stati sviluppati numerosi metodi e saggi per misurare le lunghezze della coda di poli(A). Ad esempio, il saggio della RNasi H/oligo(dT) sfrutta la RNasi H in presenza o assenza di oligo(dT) per studiare la lunghezza della coda della poli(A)11,12. Altri metodi per studiare la coda di poli(A) includono l'amplificazione PCR delle estremità 3', come l'amplificazione rapida delle estremità del cDNA, il test di poli(A) (RACE-PAT)12,13 e il test di poli(A) MEDIATO DA LIGASI (LM-PAT)14. Ulteriori modifiche del test PAT includono ePAT15 e sPAT16. La coda enzimatica G17,18 o la coda G/I dell'estremità 3' sono altre varianti del test PAT. Un'ulteriore modifica di queste tecniche include l'uso di primer marcati con fluorescenza insieme all'elettroforesi su gel capillare per l'analisi ad alta risoluzione, denominata test poli(A) ad alta risoluzione (Hire-PAT)19. Questi saggi basati sulla PCR consentono una quantificazione rapida e ad alta sensibilità della lunghezza del poli(A).
Con lo sviluppo del sequenziamento di nuova generazione, un metodo di sequenziamento ad alto rendimento, come PAL-seq20 e TAIL-seq21, consente analisi di poliadenilazione su scala a livello di trascrittoma. Tuttavia, questi metodi forniscono solo brevi letture di sequenziamento di 36-51 nucleotidi. Pertanto, FLAM-Seq22 è stato sviluppato per la profilazione globale della lunghezza della coda dell'mRNA a lunghezza intera e fornisce letture lunghe. La tecnologia Nanopore23 fornisce un sequenziamento diretto dell'RNA o del cDNA diretto indipendente dalla PCR per le stime della lunghezza della coda poli(A). Tuttavia, questi metodi ad alta produttività non sono privi di limitazioni. Richiedono grandi quantità di materiali di partenza, sono costosi e richiedono tempo. Inoltre, l'analisi di trascritti rari può essere estremamente impegnativa con metodi ad alto rendimento e i metodi basati su PCR a basso rendimento forniscono ancora un vantaggio quando è necessario analizzare un piccolo numero di trascritti, per esperimenti pilota e convalida di altri metodi.
Abbiamo recentemente dimostrato che gli mRNA di Dscam1 contengono brevi code di poli(A) in Drosophila, il che richiede un legame non canonico della proteina citoplasmatica che lega il poli(A) su Dscam1 3'UTR utilizzando il metodo di coda G/I24. Qui forniamo una procedura semplificata per la preparazione dei tessuti e la quantificazione della lunghezza dei poli(A) di mRNA dal sistema nervoso di Drosophila e dalle cellule di Drosophila S2.
Protocollo
1. Allevamento e selezione delle larve di Drosophila
- Mantenere/coltivare il ceppo di mosca (w1118, wildtype) su terreno di coltura standard a 25 °C in un'incubatrice umidificata.
- Selezionare 10 larve vaganti di 3° stadio 72 h dopo la deposizione delle uova.
- Mettere le larve in una capsula di Petri vuota da 35 mm e lavarle delicatamente trasferendole nella nuova capsula contenente l'acqua del rubinetto usando una pinza. Fallo 2 volte per rimuovere eventuali residui di cibo.
2. Isolamento cerebrale dalle larve di Drosophila (Figura 1)
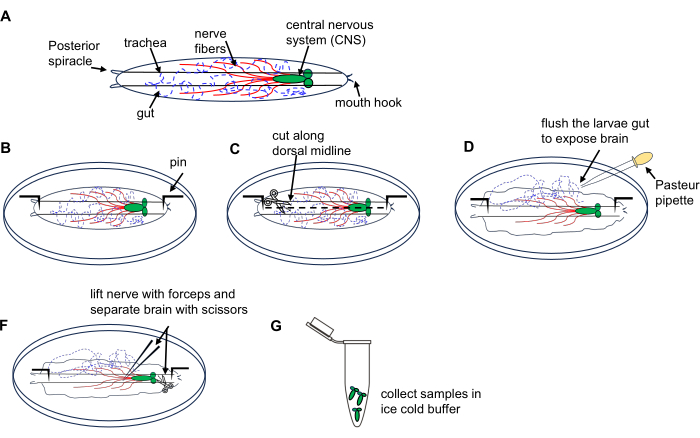
Figura 1: Dissezione del cervello larvale di Drosophila dal 3° stadio di vagabondaggio di stadio. (A) Disegni schematici della larva di Drosophila. (B-G) Dissezione delle larve. Fare clic qui per visualizzare una versione più grande di questa figura.
- Metti 10 larve su un piatto di dissezione contenente PBS ghiacciato.
- Posizionare la larva con il lato dorsale rivolto verso l'alto (identificato dai tubi tracheali che corrono lungo la sua lunghezza) e fissare ciascuna estremità sul fondo del piatto, quindi praticare una piccola incisione sulla parete del corpo all'estremità posteriore.
- Tagliare la parete del corpo lungo la linea mediana dorsale verso l'estremità anteriore usando le forbici per microdissezione.
- Sciacquare brevemente l'interno della larva usando una pipetta Pasteur 3x con PBS nel piatto per esporre il cervello.
- Individua e solleva il cervello usando le pinze e isolalo con cura usando le forbici per microdissezione.
- Trasferire i cervelli sezionati in una provetta per microcentrifuga da 1,5 mL riempita con PBS ghiacciato su ghiaccio; Raccogli tutti i cervelli delle larve. Procedere con l'estrazione dell'RNA utilizzando un kit di micropreparazione dell'RNA come descritto nel paragrafo 4.
NOTA: Eseguire una dissezione di 10 larve entro 15 minuti per prevenire danni ai tessuti e la degradazione dell'RNA.
3. Cellule di Schneider di Drosophila S2
- Coltivare cellule di Drosophila S2 nel terreno di Drosophila Schneider integrato con il 10% di siero fetale bovino (FBS) a 25 °C in un'incubatrice umidificata fino a una densità di 8 × 10,da 6 a 10 ×10, 6 cellule/mL con una vitalità minima del 90%.
- In una provetta conica sterile da 50 mL, diluire le cellule a 2,5 × 106 cellule/mL con terreno di Drosophila di Schneider integrato con FBS al 10% preriscaldato a 25 °C.
- Trasferire 8 mL della sospensione cellulare (20 × 106 cellule) in una piastra di coltura da 100 mm e aggiungere 4 mL di terreno per arrivare a 12 mL (giorno 1).
- Incubare le cellule coltivate a 25 °C in un incubatore umidificato.
NOTA: Le cellule aderiscono liberamente alla piastra dopo 12-16 ore (giorno 2). - Trasfettare le cellule con plasmidi di DNA appropriati24.
- Incubare per 48 ore in un'incubatrice umidificata.
- Dopo l'incubazione, raccogliere le cellule aggiungendo 5 mL di PBS ghiacciato mediante pipettaggio delicato (giorno 4).
- Trasferire le cellule in una provetta da 15 ml.
- Pellettare le cellule centrifugando a 1.000 × g per 5 minuti a 4 °C.
- Sciacquare le cellule 2 volte con PBS ghiacciato mediante pipettaggio delicato e raccogliere le cellule centrifugandole a 1.000 × g per 5 minuti a 4 °C.
- Eseguire l'estrazione dell'RNA utilizzando un kit di minipreparazione dell'RNA.
NOTA: Eseguire i seguenti passaggi all'interno di una cappa sterile a flusso laminare.
4. Estrazione totale dell'RNA dal cervello delle larve di Drosophila e dalle cellule S2
- Cervello larvale: Rimuovere il PBS mediante centrifugazione breve (centrifuga breve di 8 s a 5.000 × g).
- Aggiungere 600 μL di tampone di lisi dell'RNA e omogeneizzare 10x con un pestello di plastica. Ispezionare visivamente il tubo con uno stereomicroscopio per garantire una lisi completa.
- Centrifugare a 1.000 × g per 5 minuti a 4 °C per rimuovere i detriti di tessuto. Trasferire il surnatante eliminato in una provetta per microcentrifuga priva di nucleasi.
- Isolare l'RNA utilizzando un kit di micropreparazione dell'RNA secondo le istruzioni del produttore.
NOTA: L'uso di un kit di micropreparazione dell'RNA è essenziale per i campioni di cervello larvale a causa della piccola quantità di RNA presente nei campioni. - Cellule S2: Rimuovere il PBS e isolare l'RNA secondo le istruzioni del produttore.
- Misurare la resa e la qualità dell'RNA mediante spettrofotometria ed elettroforesi su gel di agarosio.
- Determinare la purezza e la quantità di RNA estratto misurando la densità ottica dell'RNA estratto rispettivamente aA 260 nm e A280 nm. Assicurarsi che il rapporto A260 nm/A280 nm sia ≥2,0 e che la concentrazione di RNA sia >350 ng/μL per le applicazioni a valle.
NOTA: Una resa tipica di RNA da 10 cervelli di larve di Drosophila è ~500-800 ng/μL o 2,5-4 μg in 5 μL. Per le cellule S2, la resa è di ~2-3 μg/μL (15-30 μg in 15 μL). L'RNA isolato può essere conservato a -80 °C per la conservazione a lungo termine.
- Determinare la purezza e la quantità di RNA estratto misurando la densità ottica dell'RNA estratto rispettivamente aA 260 nm e A280 nm. Assicurarsi che il rapporto A260 nm/A280 nm sia ≥2,0 e che la concentrazione di RNA sia >350 ng/μL per le applicazioni a valle.
5. Preparazione del gel di RNA ed elettroforesi
- 1,5% gel di RNA denaturante (100 mL)
NOTA: La formaldeide è tossica attraverso il contatto con la pelle e l'inalazione di vapori; maneggiarlo in una cappa chimica.- Sciogliere tre compresse di agarosio (1,5 g) in 82 mL di tampone MOPS (File supplementare 1) fino a quando le compresse non si rompono completamente per formare particelle fini.
- Scaldare l'impasto di agarosio in un forno a microonde fino a quando la soluzione è limpida e tutte le particelle sono completamente sciolte.
- Raffreddare la soluzione a ~60 °C.
- Aggiungere 18 ml di formaldeide al 37%, quindi mescolare agitando delicatamente. Versare la soluzione nella vaschetta di colata e lasciarla solidificare in una cappa aspirante.
- Preparazione del campione di RNA ed elettroforesi
- Diluire il campione di RNA a 200 ng (in 5 μL) e aggiungere 5 μL di colorante di caricamento RNA 2x.
- Riscaldare i campioni a 70 °C per 5 minuti in un bagno asciutto.
- Caricare 2 μL di scala dell'RNA nella prima corsia e 10 μL di campioni nelle corsie adiacenti.
- Eseguire l'elettroforesi in tampone MOPS a 100 V per 60 minuti per un gel di 5 x 6 cm.
NOTA: Regolare le condizioni di elettroforesi in base alle dimensioni dell'amplificatore. - Visualizzare il gel su un transilluminatore UV.
NOTA: La presenza di una singola banda di ~600 nucleotidi indica una preparazione intatta dell'RNA (vedere la Figura 2A).
6. Misurazione della lunghezza della coda in polietilene (A)
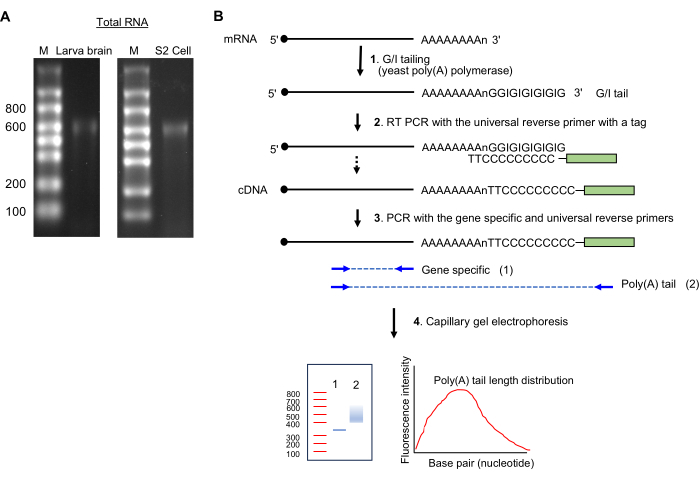
Figura 2: Preparazione del campione di RNA e saggio della poli(A)-coda. (A) Le immagini del gel di RNA mostrano l'RNA totale del cervello della larva di Drosophila (a sinistra) e delle cellule S2 (a destra) su un gel di agarosio all'1,5% di formaldeide. Le dimensioni della scala dell'RNA a singolo filamento sono mostrate nei nucleotidi sulla corsia M. Si noti un importante banding di RNA a ~600 nt, che proviene dall'rRNA. (B) Schemi del saggio della poli(A)-coda. Abbreviazione: G/I = guanosina/inosina. Fare clic qui per visualizzare una versione più grande di questa figura.
- Coda GI (Figura 2B)
- Mantenendo i reagenti in ghiaccio, preparare la seguente miscela (20 μL): fino a 14 μL di campione di RNA totale (1 μg), 4 μL di miscela tampone 5x Tail e 2 μL di miscela enzimatica 10x Tail.
- Incubare a 37 °C per 60 minuti in termociclatore.
- Aggiungere 1,5 μL della soluzione di arresto della coda; Tenere sul ghiaccio per 2 min.
NOTA: Procedere alla trascrizione inversa o conservare i campioni di RNA GI a -80 °C fino al momento di procedere alla trascrizione inversa.
- Trascrizione inversa e amplificazione PCR
- Sintetizzare il cDNA preparando la miscela e incubandola nelle condizioni descritte nel file supplementare 1.
- Diluire i campioni di cDNA ed eseguire la PCR per amplificare il DNA come indicato nel file supplementare 1.
7. Analisi del prodotto PCR mediante elettroforesi su gel di agarosio
- Analizzare una piccola porzione (2-5 μL) dei prodotti della PCR della fase 6.2.2 su un gel di agarosio al 2,5% mediante elettroforesi a 100 V per 45 minuti per il controllo di qualità.
- Verificare la specificità della PCR per le reazioni gene-specifiche e coda-specifiche sequenziando le bande PCR estratte con gel.
8. Elettroforesi capillare
- Eseguire l'elettroforesi su gel ad alta risoluzione su 1 μL di prodotti PCR (0,5-5 ng/μL) da PCR gene-specifica e poli(A) specifica utilizzando il bioanalizzatore con un kit di DNA ad alta sensibilità. Cercare picchi ben risolti che indichino un'esecuzione riuscita.
9. Analisi dei dati: misurazione della lunghezza della coda in poli(A) (Figura 3)
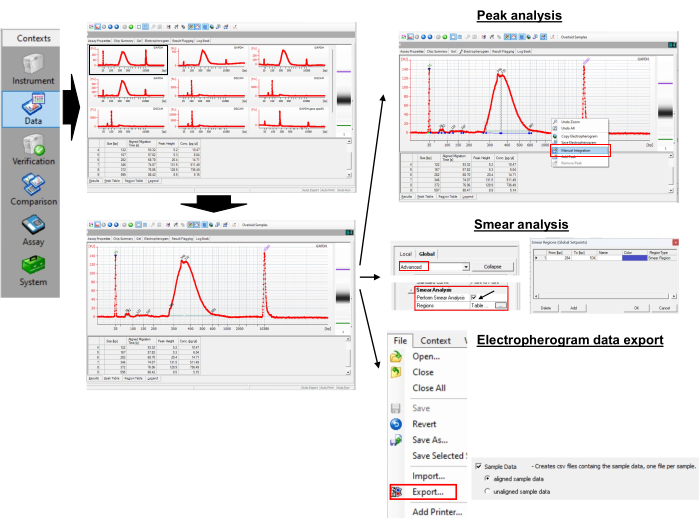
Figura 3: Misurazione della lunghezza della coda e del valore di picco di Poly(A). Fare clic qui per visualizzare una versione più grande di questa figura.
- Accesso ai dati
- Per accedere ai dati, aprire il file xad nel software.
- Selezionare il nome del campione o la scala nel pannello della vista ad albero.
NOTA: Questo mostrerà il risultato come elettroferogrammi o immagini simili a gel per i campioni selezionati. Il marcatore inferiore 35 coppia di basi (bp) e il marcatore superiore 10.380 bp sono gli standard interni utilizzati per allineare i dati ladder (50-7.000 bp) con i dati dei pozzetti campione. - Ingrandisci e rimpicciolisci gli elettroferogrammi e le immagini gelatinose per visualizzare i dettagli.
- Esecuzione dell'analisi dei picchi e delle striscie
- Per ottenere le dimensioni dei picchi, aprire l'elettroferogramma di un campione selezionato.
- Fare clic con il pulsante destro del mouse sull'elettroferogramma e selezionare l'integrazione manuale per selezionare manualmente i picchi trascinando la linea orizzontale.
- Osservare i valori di picco nella tabella Picco. Identificare il picco con l'altezza del picco maggiore. Questo è un picco di lunghezza della coda di poli(A) per un singolo campione. Nell'esempio mostrato, è di 346 bp.
- Abilita l'icona Mostra/Nascondi setpoint nel menu in alto e attendi che venga visualizzato un nuovo pannello sul lato destro.
- Selezionare Avanzate, scorrere verso il basso fino a trovare Esegui analisi strisame e selezionare la casella di controllo. In questo modo la tabella Regione verrà aggiunta alla scheda dell'elettroferogramma.
- Selezionare un elettroferogramma da un campione e passare alla tabella Regione, che mostra il menu Da [bp] e A [bp]. Per impostare l'inizio e la fine [bp], fare clic con il pulsante destro del mouse sull'elettroferogramma e selezionare Regione per aggiungere Aggiungi regione.
- Fare clic con il pulsante destro del mouse su qualsiasi cella nella tabella Regione e selezionare Modifica regioni per visualizzare una nuova piccola finestra in cui è possibile impostare le regioni personalizzate.
NOTA: Ad esempio, abbiamo utilizzato una regione compresa tra 300 bp e 550 bp per GAPDH. La PCR GAPDH gene-specifica ha prodotto un picco a 265 bp. Il primer universale (Tabella 1) estende la lunghezza della poli(A) PCR di 35 bp tramite ricottura a RNA con coda G/I. Pertanto, il primo nucleotide di adenina sull'RNA GAPDH inizia a 300 bp (265 + 35). Abbiamo arbitrariamente limitato la lunghezza massima della coda poly(A) a 250 (300 + 250 = 550). Dalla tabella delle regioni, il programma restituisce la dimensione media all'interno della regione come 387 bp. - Utilizzare l'equazione (1) per calcolare la lunghezza della coda di poli(A) sull'mRNA di interesse:
Lunghezza della coda Poly(A) = (A - B - 35) (1)
Dove A è il bp medio del prodotto PCR poli(A) specifico dall'elettroferogramma (cioè 387 bp per GAPDH), B è il picco bp del prodotto PCR gene-specifico dall'elettroferogramma (cioè 265 bp per GAPDH) e "35" è la lunghezza del tag primer inverso universale.
NOTA: Dal calcolo precedente, la lunghezza media della coda poly(A) di GAPDH è 387 - 265 - 35 = 87 bp.
10. Visualizzazione della distribuzione della lunghezza della coda in poli(A)
- Esportare i dati come file csv in File | Esporta | Dati di esempio per recuperare i dati di esempio.
NOTA: il file csv esportato visualizza il tempo di esecuzione anziché bp sull'asse X. - Passare a un elettroferogramma di un campione e selezionare Mostra dimensioni nella scheda Elettroferogramma per convertire automaticamente la tabella delle regioni nella tabella delle regioni da bp a tempo di esecuzione. Nell'esempio, da 70,39 s a 86,28 s corrispondono a 300 bp a 550 bp.
- Aprire il file csv e selezionare i valori compresi tra 70,39 s e 86,28 s di runtime per generare un grafico. Per visualizzare le dimensioni bp sull'asse X sul grafico, esportare l'elettroferogramma con le dimensioni bp come file immagine e sovrapporlo al grafico generato nel foglio di calcolo. Questo corrisponderà in modo appropriato alle dimensioni bp sulla distribuzione della coda poli (A).
Risultati
Qui, abbiamo analizzato la lunghezza della coda poli(A) di Dscam1 e GAPDH dai cervelli larvali di Drosophila (Figura 4). Gli RNA isolati sono stati visualizzati su un gel di agarosio per il controllo di qualità. Una singola banda di RNA di circa 600 nucleotidi indica una preparazione di RNA intatta (Figura 2A). Gli RNA sono stati sottoposti al tailing G/I e all'elettroforesi capillare ad alta risoluzione utilizzando un bioanalizzatore...
Discussione
In questo protocollo, descriviamo la tecnica per sezionare il cervello larvale di Drosophila dal 3° stadio di stadio errante e la preparazione del campione dalle cellule di Drosophila S2. A causa della natura labile degli mRNA, la raccolta dei campioni richiede particolare cautela. Per la dissezione cerebrale larvale, il cervello non deve essere danneggiato durante l'isolamento e non deve essere tenuto in soluzione per una durata prolungata. È essenziale mantenere il tempo di dissezione a 8...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo studio è stato supportato dal National Institute of Neurological Disorders and Stroke Grant R01NS116463 a J.K. e dalla struttura Cellular and Molecular Imaging Core presso l'Università del Nevada, Reno, che è stata supportata dal National Institutes of Health Grant P20GM103650 e utilizzata per la ricerca riportata in questo studio.
Materiali
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
Riferimenti
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435 (2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622 (2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57 (2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294 (2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon