Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Messung der Poly-A-Schwanzlänge aus Drosophila-Larvengehirn und Zelllinien
In diesem Artikel
Zusammenfassung
Das Protokoll beschreibt eine effiziente und zuverlässige Methode zur Quantifizierung der Poly(A)-Länge des interessierenden Gens aus dem Drosophila-Nervensystem , die leicht an Gewebe oder Zelltypen anderer Spezies angepasst werden kann.
Zusammenfassung
Die Polyadenylierung ist eine entscheidende posttranskriptionelle Modifikation, bei der Poly(A)-Schwänze an das 3'-Ende von mRNA-Molekülen angehängt werden. Die Länge des Poly(A)-Schwanzes wird durch zelluläre Prozesse streng reguliert. Eine Dysregulation der mRNA-Polyadenylierung wurde mit abnormaler Genexpression und verschiedenen Krankheiten in Verbindung gebracht, darunter Krebs, neurologische Störungen und Entwicklungsstörungen. Daher ist das Verständnis der Dynamik der Polyadenylierung von entscheidender Bedeutung, um die Komplexität der mRNA-Prozessierung und der posttranskriptionellen Genregulation zu entschlüsseln.
In dieser Arbeit wird eine Methode zur Messung der Poly(A)-Schwanzlängen in RNA-Proben vorgestellt, die aus Drosophila-Larvengehirnen und Drosophila-Schneider-S2-Zellen isoliert wurden. Wir verwendeten den Guanosin/Inosin (G/I)-Tailing-Ansatz, bei dem G/I-Reste enzymatisch am 3'-Ende der mRNA unter Verwendung von Hefe-Poly(A)-Polymerase hinzugefügt werden. Diese Modifikation schützt das 3'-Ende der RNA vor enzymatischem Abbau. Die geschützten Poly(A)-Schwänze in voller Länge werden dann mit einem universellen Antisense-Primer rückwärts transkribiert. Anschließend wird die PCR-Amplifikation mit einem genspezifischen Oligo durchgeführt, das auf das interessierende Gen abzielt, zusammen mit einem universellen Sequenzoligo, das für die reverse Transkription verwendet wird.
Dadurch werden PCR-Produkte erzeugt, die die Poly(A)-Schwänze des interessierenden Gens umfassen. Da die Polyadenylierung keine einheitliche Modifikation ist und zu unterschiedlich langen Schwänzen führt, weisen die PCR-Produkte eine Reihe von Größen auf, was zu einem Abstrichmuster auf Agarosegel führt. Abschließend werden die PCR-Produkte einer hochauflösenden Kapillargelelektrophorese unterzogen, gefolgt von einer Quantifizierung anhand der Größen der Poly(A)-PCR-Produkte und des genspezifischen PCR-Produkts. Diese Technik bietet ein einfaches und zuverlässiges Werkzeug zur Analyse von Poly(A)-Schwanzlängen, das es uns ermöglicht, tiefere Einblicke in die komplizierten Mechanismen der mRNA-Regulation zu gewinnen.
Einleitung
Die meisten eukaryotischen mRNAs werden an ihrem 3′-Terminus im Zellkern durch Zugabe von nicht-template-Adenosinen durch kanonische Poly(A)-Polymerasen posttranskriptionell polyadenyliert. Ein intakter Poly(A)-Schwanz ist während des gesamten Lebenszyklus von mRNA von entscheidender Bedeutung, da er für den mRNA-Kernexportunerlässlich ist 1, die Interaktion mit Poly(A)-bindenden Proteinen erleichtert, um die Translationseffizienz zu verbessern2, und Resistenz gegen Abbauverleiht 3. In bestimmten Fällen kann der Poly(A)-Schwanz auch im Zytoplasma gedehnt werden, was durch nicht-kanonische Poly(A)-Polymerasen erleichtert wird4. Im Zytoplasma ändert sich die Poly(A)-Schwanzlänge dynamisch und beeinflusst die Lebensdauer des mRNA-Moleküls. Zahlreiche Polymerasen und Deadenylasen sind dafür bekannt, die Schwanzlänge zu modulieren 5,6,7. Zum Beispiel korreliert die Verkürzung der Poly(A)-Schwänze mit der translationalen Repression, während die Verlängerung der Poly(A)-Schwänze die Translation verstärkt 8,9.
Zunehmende genomische Studien haben die grundlegende Bedeutung der Poly(A)-Schwanzlänge in verschiedenen Facetten der eukaryotischen Biologie gezeigt. Dazu gehören Rollen bei der Keimzellentwicklung, der frühen Embryonalentwicklung, der neuronalen synaptischen Plastizität für Lernen und Gedächtnis und der Entzündungsreaktion10. Es wurden zahlreiche Methoden und Assays zur Messung von Poly(A)-Schwanzlängen entwickelt. Beispielsweise nutzt der RNase H/Oligo(dT)-Assay die Vorteile von RNase H in Gegenwart oder Abwesenheit von Oligo(dT), um die Poly(A)-Schwanzlänge zu untersuchen11,12. Andere Methoden zur Untersuchung des Poly(A)-Schwanzes umfassen die PCR-Amplifikation von 3'-Enden, wie z. B. die schnelle Amplifikation von cDNA-Enden, den Poly(A)-Test (RACE-PAT)12,13 und den Ligase-vermittelten Poly(A)-Test (LM-PAT)14. Weitere Modifikationen des PAT-Assays umfassen ePAT15 und sPAT16. Enzymatisches G-Tailing 17,18 oder G/I-Tailing des 3'-Endes sind weitere Variationen des PAT-Assays. Eine weitere Modifikation dieser Techniken umfasst die Verwendung von fluoreszenzmarkierten Primern zusammen mit einer Kapillargelelektrophorese für hochauflösende Analysen, die als hochauflösender Poly(A)-Test (Hire-PAT)19 bezeichnet wird. Diese PCR-gesteuerten Assays ermöglichen eine schnelle und hochempfindliche Quantifizierung der Poly(A)-Länge.
Mit der Entwicklung der Next-Generation-Sequenzierung ermöglicht eine Hochdurchsatz-Sequenzierungsmethode wie PAL-seq20 und TAIL-seq21 Polyadenylierungsanalysen im transkriptomweiten Maßstab. Diese Methoden liefern jedoch nur kurze Sequenzierungs-Reads von 36-51 Nukleotiden. Daher wurde FLAM-Seq22 für die globale Schwanzlängenprofilierung von mRNA in voller Länge entwickelt und bietet lange Lesevorgänge. Die Nanoporentechnologie23 bietet PCR-unabhängige, direkte RNA- oder direkte cDNA-Sequenzierung für die Schätzung der Poly(A)-Schwanzlänge. Diese Hochdurchsatzmethoden sind jedoch nicht ohne Einschränkungen. Sie benötigen große Mengen an Ausgangsmaterialien, sind teuer und zeitaufwändig. Darüber hinaus kann die Analyse seltener Transkripte mit Hochdurchsatzmethoden äußerst anspruchsvoll sein, und PCR-basierte Methoden mit niedrigem Durchsatz bieten immer noch einen Vorteil, wenn eine kleine Anzahl von Transkripten analysiert werden muss, für Pilotexperimente und die Validierung anderer Methoden.
Wir haben kürzlich gezeigt, dass Dscam1-mRNAs kurze Poly(A)-Schwänze in Drosophila enthalten, was eine nicht-kanonische Bindung des zytoplasmatischen Poly(A)-bindenden Proteins an Dscam1 3'UTR unter Verwendung der G/I-Tailing-Methode24 erfordert. Hier bieten wir ein optimiertes Verfahren zur Gewebepräparation und Quantifizierung der Poly(A)-Länge von mRNAs aus dem Drosophila-Nervensystem und Drosophila-S2-Zellen .
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Aufzucht und Selektion von Drosophila-Larven
- Den Fliegenstamm (w1118, Wildtyp) auf Standard-Fliegenfuttermedium bei 25 °C in einem befeuchteten Inkubator pflegen/kultivieren.
- Wählen Sie 10 wandernde Larven des 3. Stadiums 72 h nach der Eiablage aus.
- Legen Sie die Larven in eine leere 35-mm-Petrischale und waschen Sie sie vorsichtig, indem Sie die Larven mit einer Pinzette in die neue Schale mit Leitungswasser geben. Tun Sie dies 2x, um restliche Lebensmittel zu entfernen.
2. Isolierung des Gehirns von Drosophila-Larven (Abbildung 1)
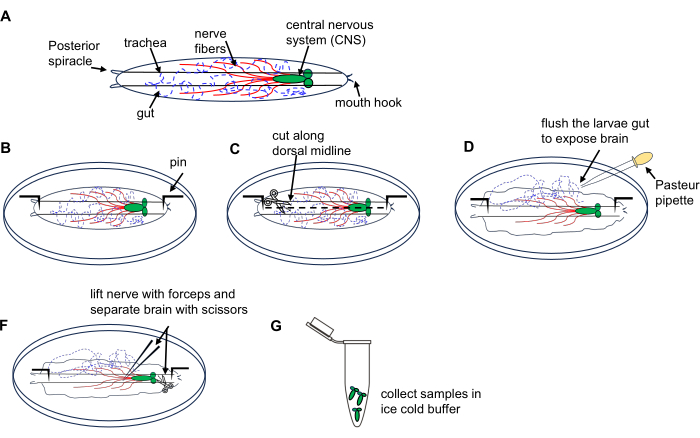
Abbildung 1: Sektion des Gehirns der Drosophila-Larve aus dem 3. Wanderstadium des Stadiums. (A) Schematische Zeichnungen der Drosophila-Larve. (B-G) Larven-Sezierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Legen Sie 10 Larven auf eine Sezierschale mit eiskaltem PBS.
- Legen Sie die Rückenseite der Larve nach oben (gekennzeichnet durch Trachealtuben, die entlang ihrer Länge verlaufen) und stecken Sie jedes Ende an den Boden der Schale, gefolgt von einem kleinen Schnitt an der Körperwand am hinteren Ende.
- Schneiden Sie die Körperwand entlang der dorsalen Mittellinie in Richtung des vorderen Endes mit einer Mikrodissektionsschere.
- Spülen Sie das Innere der Larve kurz mit einer Pasteurpipette 3x mit PBS in der Schale, um das Gehirn freizulegen.
- Lokalisieren und heben Sie das Gehirn mit der Pinzette an und isolieren Sie es vorsichtig mit einer Mikrodissektionsschere.
- Übertragen Sie die präparierten Gehirne in ein 1,5-ml-Mikrozentrifugenröhrchen, das mit eiskaltem PBS auf Eis gefüllt ist. sammle alle Larvengehirne. Fahren Sie mit der RNA-Extraktion mit einem RNA-Microprep-Kit fort, wie in Abschnitt 4 beschrieben.
HINWEIS: Führen Sie innerhalb von 15 Minuten eine Sektion von 10 Larven durch, um Gewebeschäden und RNA-Abbau zu vermeiden.
3. Drosophila S2 Schneider-Zellen
- Züchten Sie Drosophila S2-Zellen in Drosophila Schneiders Medium, ergänzt mit 10 % fötalem Rinderserum (FBS) bei 25 °C in einem befeuchteten Inkubator auf eine Dichte von 8 × 106 bis 10 × 106 Zellen/ml mit einer Lebensfähigkeit von mindestens 90 %.
- In einem sterilen konischen 50-ml-Röhrchen werden die Zellen auf 2,5 × 106 Zellen/ml mit Schneider's Drosophila-Medium verdünnt, das mit 10 % FBS ergänzt wird, das auf 25 °C vorgewärmt wurde.
- 8 ml der Zellsuspension (20 × 106 Zellen) in eine 100-mm-Kulturplatte geben und 4 ml Medium hinzufügen, um 12 ml (Tag 1) zu erhalten.
- Inkubieren Sie die kultivierten Zellen bei 25 °C in einem befeuchteten Inkubator.
HINWEIS: Die Zellen haften nach 12-16 h (Tag 2) locker an der Platte. - Transfizieren Sie die Zellen mit geeigneten DNA-Plasmiden24.
- 48 Stunden in einem befeuchteten Inkubator inkubieren.
- Sammeln Sie nach der Inkubation Zellen, indem Sie 5 ml eiskaltes PBS durch sanftes Pipettieren (Tag 4) hinzufügen.
- Übertragen Sie die Zellen in ein 15-ml-Röhrchen.
- Pelletieren Sie die Zellen durch Zentrifugieren bei 1.000 × g für 5 min bei 4 °C.
- Spülen Sie die Zellen 2x mit eiskaltem PBS durch schonendes Pipettieren und sammeln Sie die Zellen durch Zentrifugieren bei 1.000 × g für 5 min bei 4 °C.
- Führen Sie die RNA-Extraktion mit einem RNA-Miniprep-Kit durch.
HINWEIS: Führen Sie die folgenden Schritte in einer sterilen Laminar-Flow-Haube aus.
4. Gesamt-RNA-Extraktion aus Drosophila-Larven, Gehirn und S2-Zellen
- Larvengehirn: Entfernen Sie PBS durch kurzes Zentrifugieren (8 s kurzes Schleudern bei 5.000 × g).
- Fügen Sie 600 μl RNA-Lysepuffer hinzu und homogenisieren Sie 10x mit einem Kunststoffstößel. Überprüfen Sie das Röhrchen visuell unter einem Stereomikroskop, um eine vollständige Lyse sicherzustellen.
- Bei 1.000 × g 5 min bei 4 °C zentrifugieren, um Gewebereste zu entfernen. Den geklärten Überstand in ein nukleasefreies Mikrozentrifugenröhrchen überführen.
- Isolieren Sie RNA mit einem RNA-Microprep-Kit gemäß den Anweisungen des Herstellers.
HINWEIS: Die Verwendung eines RNA-Microprep-Kits ist für die Larvenhirnproben aufgrund der geringen Menge an RNA in den Proben unerlässlich. - S2-Zellen: Entfernen Sie PBS und isolieren Sie RNA gemäß den Anweisungen des Herstellers.
- Messen Sie die RNA-Ausbeute und -Qualität durch Spektrophotometrie und Agarose-Gelelektrophorese.
- Bestimmen Sie die Reinheit und Menge der extrahierten RNA durch Messung der optischen Dichte der extrahierten RNA bei A260 nm bzw. A280 nm. Stellen Sie sicher, dass das Verhältnis von A260 nm/Azu 280 nm ≥2,0 und die RNA-Konzentration >350 ng/μl für Downstream-Anwendungen beträgt.
HINWEIS: Eine typische RNA-Ausbeute von 10 Drosophila-Larvengehirnen beträgt ~500-800 ng/μl oder 2,5-4 μg in 5 μl. Für S2-Zellen beträgt die Ausbeute ~2-3 μg/μl (15-30 μg in 15 μl). Isolierte RNA kann bei -80 °C für die Langzeitlagerung gelagert werden.
- Bestimmen Sie die Reinheit und Menge der extrahierten RNA durch Messung der optischen Dichte der extrahierten RNA bei A260 nm bzw. A280 nm. Stellen Sie sicher, dass das Verhältnis von A260 nm/Azu 280 nm ≥2,0 und die RNA-Konzentration >350 ng/μl für Downstream-Anwendungen beträgt.
5. Herstellung von RNA-Gel und Elektrophorese
- 1,5 % denaturierendes RNA-Gel (100 ml)
HINWEIS: Formaldehyd ist giftig durch Hautkontakt und Einatmen von Dämpfen; Behandeln Sie es in einem chemischen Abzug.- Lösen Sie drei Agarosetabletten (1,5 g) in 82 ml MOPS-Puffer (Supplemental File 1), bis die Tabletten vollständig zerfallen und feine Partikel bilden.
- Die Agarose-Aufschlämmung in der Mikrowelle erhitzen, bis die Lösung klar ist und sich alle Partikel vollständig aufgelöst haben.
- Die Lösung auf ~60 °C abkühlen lassen.
- Fügen Sie 18 ml 37% Formaldehyd hinzu und mischen Sie es durch sanftes Schwenken. Gießen Sie die Lösung in die Gießschale und lassen Sie sie in einem Abzug erstarren.
- RNA-Probenvorbereitung und Elektrophorese
- Verdünnen Sie die RNA-Probe auf 200 ng (in 5 μl) und fügen Sie 5 μl 2x RNA-Ladefarbstoff hinzu.
- Die Proben werden 5 Minuten lang in einem Trockenbad auf 70 °C erhitzt.
- Laden Sie 2 μl RNA-Leiter in die erste Spur und 10 μl Proben in die benachbarten Spuren.
- Führen Sie die Elektrophorese in MOPS-Puffer bei 100 V für 60 min für ein 5 x 6 cm großes Gel durch.
HINWEIS: Passen Sie die Elektrophoresebedingungen in Abhängigkeit von den Amplikongrößen an. - Visualisieren Sie das Gel auf einem UV-Transilluminator.
HINWEIS: Das Vorhandensein einer einzelnen Bande ~600 Nukleotidgröße weist auf eine intakte RNA-Präparation hin (siehe Abbildung 2A).
6. Messung der Poly(A)-Schwanzlänge
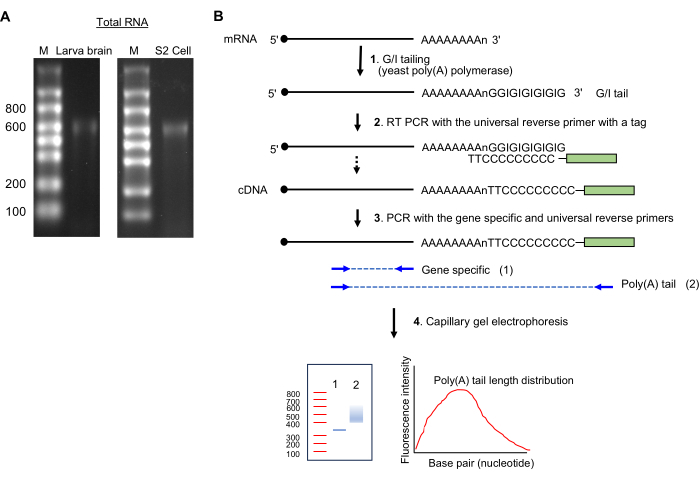
Abbildung 2: RNA-Probenvorbereitung und der Poly(A)-Schwanz-Assay. (A) Die RNA-Gel-Bilder zeigen die Gesamt-RNA aus dem Drosophila-Larvengehirn (links) und den S2-Zellen (rechts) auf einem 1,5%igen Formaldehyd-Agarose-Gel. Einzelsträngige RNA-Leitergrößen sind in Nukleotiden auf Spur M dargestellt. Beachten Sie eine Haupt-RNA-Bandierung bei ~600 nt, die von rRNA stammt. (B) Schematische Darstellung des Poly(A)-Schwanz-Assays. Abkürzung: G/I = Guanosin/Inosin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- GI-Tailing (Abbildung 2B)
- Lassen Sie die Reagenzien auf Eis und bereiten Sie die folgende Mischung (20 μl) vor: bis zu 14 μl Gesamt-RNA-Probe (1 μg), 4 μl 5x Tail-Puffermischung und 2 μl 10x Tail-Enzymmischung.
- Bei 37 °C 60 min in einem Thermocycler inkubieren.
- Fügen Sie 1,5 μl der Endanschlaglösung hinzu; 2 Minuten auf Eis halten.
HINWEIS: Fahren Sie mit der reversen Transkription fort oder lagern Sie die GI-tailed RNA-Proben bei -80 °C, bis Sie mit der reversen Transkription fortfahren können.
- Reverse Transkription und PCR-Amplifikation
- Synthetisieren Sie cDNA, indem Sie die Mischung vorbereiten und unter den in der Zusatzdatei 1 beschriebenen Bedingungen inkubieren.
- Verdünnen Sie die cDNA-Proben und führen Sie eine PCR durch, um die DNA zu amplifizieren, wie in der Zusatzdatei 1 angegeben.
7. PCR-Produktanalyse mittels Agarose-Gelelektrophorese
- Analysieren Sie eine kleine Portion (2-5 μl) der PCR-Produkte aus Schritt 6.2.2 auf einem 2,5%igen Agarosegel durch Elektrophorese bei 100 V für 45 min zur Qualitätskontrolle.
- Überprüfen Sie die Spezifität der PCR für gen- und schwanzspezifische Reaktionen durch Sequenzierung der gelextrahierten PCR-Banden.
8. Kapillarelektrophorese
- Führen Sie eine hochauflösende Gelelektrophorese an 1 μl PCR-Produkten (0,5-5 ng/μl) aus genspezifischer und poly(A)-spezifischer PCR mit dem Bioanalysator mit einem hochempfindlichen DNA-Kit durch. Suchen Sie nach gut aufgelösten Spitzen, die auf einen erfolgreichen Lauf hinweisen.
9. Datenanalyse: Poly(A)-Schwanzlängenmessung (Abbildung 3)
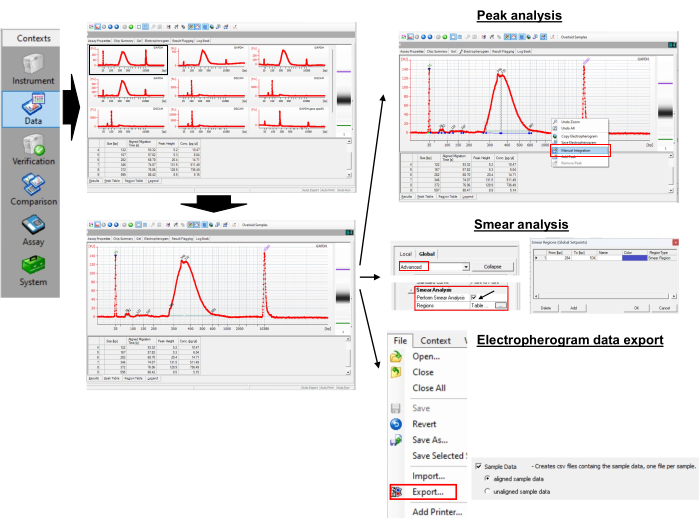
Abbildung 3: Messung der Poly(A)-Schwanzlänge und des Spitzenwerts. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Zugriff auf Daten
- Um auf die Daten zuzugreifen, öffnen Sie die xad-Datei in der Software.
- Wählen Sie den Beispielnamen oder den Kontaktplan im Strukturansichtsfenster aus.
HINWEIS: Dies zeigt das Ergebnis als Elektropherogramme oder gelartige Bilder für die ausgewählten Proben. Der untere Marker 35 Basenpaar (bp) und der obere Marker 10.380 bp sind die internen Standards, die verwendet werden, um die Kontaktplandaten (50-7.000 bp) mit den Daten aus den Probenvertiefungen abzugleichen. - Vergrößern und verkleinern Sie die Elektropherogramme und gelartigen Bilder, um die Details anzuzeigen.
- Durchführung von Peak- und Smear-Analysen
- Um die Peakgrößen zu erhalten, öffnen Sie das Elektropherogramm einer ausgewählten Probe.
- Klicken Sie mit der rechten Maustaste auf das Elektropherogramm und wählen Sie Manuelle Integration , um Peaks manuell auszuwählen, indem Sie die horizontale Linie ziehen.
- Beobachten Sie die Spitzenwerte in der Tabelle Peak. Identifizieren Sie den Peak mit der größten Peakhöhe. Dies ist ein Peak der Poly(A)-Schwanzlänge für eine einzelne Probe. Im gezeigten Beispiel sind es 346 bp.
- Aktivieren Sie das Symbol Sollwerte ein-/ausblenden im oberen Menü und warten Sie, bis auf der rechten Seite ein neues Fenster angezeigt wird.
- Wählen Sie Erweitert aus, scrollen Sie nach unten, um Abstrichanalyse durchführen zu finden, und aktivieren Sie das Kontrollkästchen. Dadurch wird die Tabelle Region zur Registerkarte Elektropherogramm hinzugefügt.
- Wählen Sie ein Elektropherogramm aus einer Probe aus und wechseln Sie zur Tabelle Region, in der die Menüs Von [bp] und Bis [bp] angezeigt werden. Um den Start und das Ende [bp] festzulegen, klicken Sie mit der rechten Maustaste auf das Elektropherogramm und wählen Sie Region , um Region hinzufügen hinzuzufügen.
- Klicken Sie mit der rechten Maustaste auf eine beliebige Zelle in der Tabelle Region und wählen Sie Regionen ändern, um ein kleines neues Fenster zu öffnen, in dem benutzerdefinierte Regionen festgelegt werden können.
HINWEIS: Zum Beispiel haben wir einen Bereich von 300 bp bis 550 bp für GAPDH verwendet. Die genspezifische GAPDH-PCR ergab einen Peak von 265 bp. Der universelle Primer (Tabelle 1) verlängert die Länge der Poly(A)-PCR um 35 bp durch Annealing auf G/I-tailed RNAs. So beginnt das erste Adenin-Nukleotid auf GAPDH-RNA bei 300 bp (265 + 35). Wir haben die maximale Poly(A)-Schwanzlänge willkürlich auf 250 begrenzt (300 + 250 = 550). Aus der Regionstabelle gibt das Programm die durchschnittliche Größe innerhalb der Region mit 387 bp zurück. - Verwenden Sie Gleichung (1), um die Poly(A)-Schwanzlänge auf der interessierenden mRNA zu berechnen:
Poly(A) Schwanzlänge = (A - B - 35) (1)
Dabei ist A der durchschnittliche bp des poly(A)-spezifischen PCR-Produkts aus dem Elektropherogramm (d. h. 387 bp für GAPDH), B ist der maximale bp des genspezifischen PCR-Produkts aus dem Elektropherogramm (d. h. 265 bp für GAPDH) und "35" ist die Länge des universellen umgekehrten Primer-Tags.
HINWEIS: Aus der obigen Berechnung ergibt sich die durchschnittliche Poly(A)-Schwanzlänge von GAPDH von 387 - 265 - 35 = 87 bp.
10. Visualisierung der Poly(A)-Schwanzlängenverteilung
- Exportieren Sie Daten als CSV-Datei unter Datei | Exportieren | Beispieldaten , um Beispieldaten abzurufen.
HINWEIS: Die exportierte CSV-Datei zeigt auf der X-Achse die Laufzeit anstelle von bp an. - Gehen Sie zu einem Elektropherogramm einer Probe und aktivieren Sie Größen anzeigen auf der Registerkarte Elektropherogramm , um die Bereichstabelle in der Regionstabelle automatisch von bp in die Laufzeit zu konvertieren. Im Beispiel entsprechen 70,39 s bis 86,28 s 300 bp bis 550 bp.
- Öffnen Sie die CSV-Datei, und wählen Sie die Werte zwischen 70,39 s und 86,28 s Laufzeit aus, um ein Diagramm zu erstellen. Um die bp-Größen auf der X-Achse im Diagramm zu visualisieren, exportieren Sie das Elektropherogramm mit bp-Größen als Bilddatei und überlagern Sie es mit dem in der Tabelle generierten Diagramm. Dies entspricht den bp-Größen bei der Poly(A)-Schwanzverteilung.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Hier analysierten wir die Poly(A)-Schwanzlänge von Dscam1 und GAPDH aus Drosophila-Larvengehirnen (Abbildung 4). Isolierte RNAs wurden zur Qualitätskontrolle auf einem Agarosegel visualisiert. Eine einzelne RNA-Bande bei einer Nukleotidgröße von etwa 600 weist auf eine intakte RNA-Präparation hin (Abbildung 2A). Die RNAs wurden mit einem Agilent 2100 Bioanalysator dem G/I-Tailing und der hochauflösenden Kapillarelektrophorese unt...
Access restricted. Please log in or start a trial to view this content.
Diskussion
In diesem Protokoll beschreiben wir die Technik zur Sezierung des Drosophila-Larvengehirns aus dem wandernden 3. Stadium sowie die Probenvorbereitung aus Drosophila S2-Zellen. Aufgrund der labilen Natur von mRNAs erfordert die Probenentnahme besondere Vorsicht. Bei der Larvendissektion des Gehirns sollte das Gehirn während der Isolation nicht beschädigt und nicht über einen längeren Zeitraum in Lösung gehalten werden. Es ist wichtig, die Sezierzeit für eine Sezierrunde auf 8-10 Minuten ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Studie wurde vom National Institute of Neurological Disorders and Stroke Grant R01NS116463 an J.K. und der Cellular and Molecular Imaging Core Facility an der University of Nevada, Reno, unterstützt, die von den National Institutes of Health Grant P20GM103650 unterstützt und für die in dieser Studie berichtete Forschung verwendet wurde.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
Referenzen
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435(2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622(2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57(2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294(2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten