A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מדידת אורך זנב Poly A מזחלי דרוזופילה מוח וקו תאים
In This Article
Summary
הפרוטוקול מתאר שיטה יעילה ואמינה לכימות אורך הפולי(A) של הגן המעניין ממערכת העצבים דרוזופילה , שניתן להתאים בקלות לרקמות או סוגי תאים ממינים אחרים.
Abstract
פוליאדנילציה היא שינוי חיוני לאחר שעתוק המוסיף זנבות פולי(A) לקצה 3' של מולקולות mRNA. אורך זנב הפולי(A) מווסת היטב על ידי תהליכים תאיים. דיסרגולציה של פוליאדנילציה mRNA נקשרה לביטוי גנים חריג ולמחלות שונות, כולל סרטן, הפרעות נוירולוגיות והפרעות התפתחותיות. לכן, הבנת הדינמיקה של פוליאדנילציה חיונית לפענוח המורכבות של עיבוד mRNA ובקרת גנים לאחר שעתוק.
מאמר זה מציג שיטה למדידת אורכי זנב פולי(A) בדגימות RNA שבודדו ממוחות זחלי דרוזופילה ומתאי דרוזופילה שניידר S2. השתמשנו בגישת הזנב גואנוזין/אינוזין (G/I), הכוללת הוספה אנזימטית של שאריות G/I בקצה ה-3' של mRNA באמצעות פולימראז שמרים (A). שינוי זה מגן על קצה ה-3' של הרנ"א מפני התפרקות אנזימטית. זנבות הפולי(A) המוגנים באורך מלא מתועתקים לאחור באמצעות פריימר אנטיסנס אוניברסלי. לאחר מכן, הגברה PCR מבוצעת באמצעות אוליגו ספציפי לגן המכוון לגן המעניין, יחד עם אוליגו רצף אוניברסלי המשמש לשעתוק לאחור.
זה מייצר תוצרי PCR המקיפים את זנבות הפולי(A) של הגן המעניין. מכיוון שפוליאדנילציה אינה שינוי אחיד וגורמת לזנבות באורכים משתנים, מוצרי ה-PCR מציגים מגוון גדלים, מה שמוביל לדפוס מריחה על ג'ל אגרוז. לבסוף, מוצרי ה- PCR נתונים לאלקטרופורזה של ג'ל נימי ברזולוציה גבוהה, ואחריה כימות באמצעות הגדלים של מוצרי ה- PCR poly(A) ומוצר ה- PCR הספציפי לגן. טכניקה זו מציעה כלי פשוט ואמין לניתוח אורכי זנב פולי(A), ומאפשרת לנו לקבל תובנות עמוקות יותר על המנגנונים המורכבים השולטים בוויסות mRNA.
Introduction
רוב ה-mRNA האיקריוטי עובר פוליאדנילציה לאחר שעתוק בנקודת הסיום של 3′ בגרעין על ידי תוספת של אדנוזין ללא תבנית על ידי פולימראזות פולי(A) קנוניות. זנב פולי(A) שלם הוא חיוני לאורך מחזור החיים של mRNA, מכיוון שהוא חיוני לייצוא גרעיני של mRNA1, מאפשר אינטראקציה עם חלבונים קושרי פולי(A) כדי לשפר את יעילות התרגום2, ומקנה עמידות בפני פירוק3. במקרים מסוימים, זנב פולי(A) יכול גם לעבור הארכה בציטופלסמה, בסיוע פולימראז (A) לא קנוני4. בציטופלסמה, אורך הזנב של פולי (A) משתנה באופן דינמי ומשפיע על תוחלת החיים של מולקולת ה-mRNA. פולימרזות ודדנילזות רבות ידועות באפנון אורך הזנב 5,6,7. לדוגמה, קיצור זנבות פולי(A) מתואם עם דיכוי תרגומי, ואילו התארכות זנבות פולי(A) משפרת את התרגום 8,9.
מחקרים גנומיים מצטברים הוכיחו את המשמעות הבסיסית של אורך הזנב הפולי(A) על פני היבטים שונים של הביולוגיה האיקריוטית. זה כולל תפקידים בהתפתחות תאי נבט, התפתחות עוברית מוקדמת, פלסטיות סינפטית עצבית ללמידה וזיכרון, והתגובה הדלקתית10. פותחו שיטות ומבדקים רבים למדידת אורכי זנב פולי(A). לדוגמה, בדיקת RNase H/oligo(dT) מנצלת את RNase H בנוכחות או היעדר oligo(dT) כדי לחקור את אורך הזנב של poly(A)11,12. שיטות אחרות לחקר זנב פולי(A) כוללות הגברה PCR של קצוות 3' כגון הגברה מהירה של cDNA קצוות בדיקת poly(A) (RACE-PAT)12,13 ובדיקת poly(A) בתיווך ליגאז (LM-PAT)14. שינויים נוספים בבדיקת PAT כוללים ePAT15 ו- sPAT16. זנב G אנזימטי17,18 או זנב G/I של קצה 3' הן וריאציות אחרות של מבחן PAT. שינוי נוסף של טכניקות אלה כולל שימוש בפריימרים המסומנים באופן פלואורסצנטי יחד עם אלקטרופורזה של ג'ל נימי לאנליזה ברזולוציה גבוהה, המכונה מבחן פולי(A) ברזולוציה גבוהה (Hire-PAT)19. בדיקות מונחות PCR אלה מאפשרות כימות אורך פולי(A) מהיר ובעל רגישות גבוהה.
עם הפיתוח של ריצוף הדור הבא, שיטת ריצוף בתפוקה גבוהה, כגון PAL-seq20 ו- TAIL-seq21, מאפשרת ניתוח פוליאדנילציה בקנה מידה רחב של תעתוק. עם זאת, שיטות אלה מספקות רק קריאות רצף קצרות של 36-51 נוקלאוטידים. לכן, FLAM-Seq22 פותח עבור פרופיל אורך זנב גלובלי של mRNA באורך מלא ומספק קריאות ארוכות. טכנולוגיית ננופור23 מספקת ריצוף RNA ישיר או cDNA ישיר שאינו תלוי ב-PCR עבור הערכות אורך זנב פולי(A). עם זאת, שיטות אלה בעלות תפוקה גבוהה אינן נטולות מגבלות. הם דורשים כמויות גדולות של חומרי התחלה, הם יקרים וגוזלים זמן. יתר על כן, ניתוח תמלילים נדירים יכול להיות מאתגר ביותר עם שיטות תפוקה גבוהה, ושיטות מבוססות PCR בתפוקה נמוכה עדיין מספקות יתרון כאשר יש צורך לנתח מספר קטן של תמלילים, לניסויי פיילוט ותיקוף של שיטות אחרות.
לאחרונה הוכחנו כי Dscam1 mRNA מכיל זנבות פולי(A) קצרים בדרוזופילה, מה שמחייב קשירה לא קנונית של החלבון הקושר פולי(A) ציטופלזמי על Dscam1 3'UTR באמצעות שיטת זנב G/I24. כאן אנו מספקים הליך יעיל להכנת רקמות וכימות אורך פולי(A) של mRNA ממערכת העצבים דרוזופילה ותאי דרוזופילה S2.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. גידול ובחירת זחלי דרוזופילה
- תחזוק/תרבית את זן הזבוב (W1118, wildtype) על מזון זבובים סטנדרטי בטמפרטורה של 25°C באינקובטור לח.
- בחר 10 זחלי כוכב נודדים 3rd 72 שעות לאחר הטלת ביצים.
- מניחים את הזחלים בכלי פטרי ריק בקוטר 35 מ"מ ושוטפים אותם בעדינות על ידי העברת הזחלים לצלחת החדשה המכילה מי ברז באמצעות מלקחיים. בצעו פעולה זו פעמיים כדי להסיר את שאריות המזון.
2. בידוד מוחי מזחלי דרוזופילה (איור 1)
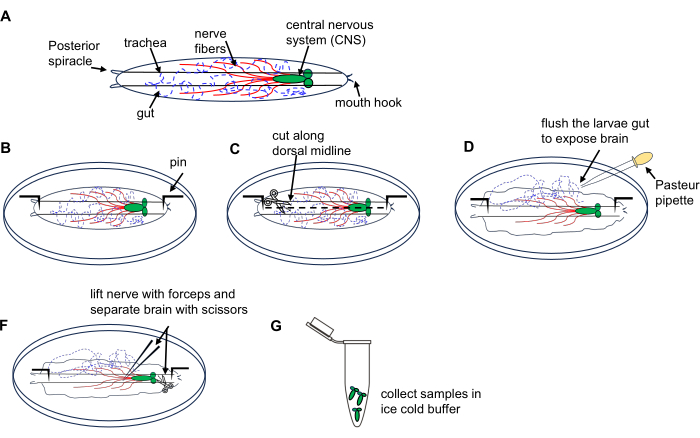
איור 1: דיסקציה של מוח זחל דרוזופילה משלב נדידת כוכב 3. (A) רישומים סכמטיים של זחלי דרוזופילה. (ב-ג) דיסקציה של הזחל. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
- מניחים 10 זחלים על צלחת דיסקציה המכילה PBS קר כקרח.
- מניחים את הצד הגבי של הזחל כלפי מעלה (מזוהה על ידי צינורות קנה הנשימה העוברים לאורכו) ומצמידים כל קצה לתחתית הצלחת ולאחר מכן מבצעים חתך קטן בדופן הגוף בקצה האחורי.
- חותכים את דופן הגוף לאורך קו האמצע הגבי לכיוון הקצה הקדמי באמצעות מספריים microdissection.
- לשטוף בקצרה את הפנים של הזחל באמצעות פיפטה פסטר 3x עם PBS בצלחת כדי לחשוף את המוח.
- אתרו והרימו את המוח באמצעות המלקחיים ובודדו אותו בזהירות באמצעות מספריים מיקרודיסקציה.
- להעביר את המוחות המנותחים לצינור מיקרוצנטריפוגה של 1.5 מ"ל מלא PBS קר כקרח על קרח; אספו את כל מוחות הזחלים. המשך במיצוי RNA באמצעות ערכת מיקרו-הכנה של RNA כמתואר בסעיף 4.
הערה: בצע דיסקציה של 10 זחלים תוך 15 דקות כדי למנוע נזק לרקמות והתפרקות RNA.
3. דרוזופילה S2 תאי שניידר
- לגדל תאי Drosophila S2 בתווך של Drosophila Schneider בתוספת 10% סרום בקר עוברי (FBS) ב 25 ° C באינקובטור לח לצפיפות של 8 × 106 עד 10 × 106 תאים / מ"ל עם מינימום 90% כדאיות.
- בצינור חרוטי סטרילי של 50 מ"ל, יש לדלל תאים ל-2.5 × 106 תאים/מ"ל עם מדיום דרוזופילה של שניידר בתוספת 10% FBS שחומם מראש ל-25°C.
- מעבירים 8 מ"ל של תרחיף התא (20 × 10,6 תאים) לתוך צלחת תרבית 100 מ"מ ומוסיפים 4 מ"ל של מדיום כדי להגיע ל 12 מ"ל (יום 1).
- לדגור על התאים בתרבית ב 25 מעלות צלזיוס באינקובטור לח.
הערה: התאים נצמדים באופן רופף לצלחת לאחר 12-16 שעות (יום 2). - להדביק את התאים עם פלסמידים DNA מתאים24.
- דגירה במשך 48 שעות באינקובטור לח.
- לאחר הדגירה, לאסוף תאים על ידי הוספת 5 מ"ל של PBS קר כקרח על ידי pipetting עדין (יום 4).
- מעבירים את התאים לצינור של 15 מ"ל.
- גלולה את התאים על ידי צנטריפוגה ב 1,000 × גרם במשך 5 דקות ב 4 ° C.
- שטפו את התאים 2x עם PBS קר כקרח על ידי פיפטינג עדין ואספו את התאים על ידי צנטריפוגה שלהם ב 1,000 × גרם במשך 5 דקות ב 4 ° C.
- בצע מיצוי RNA באמצעות ערכת RNA miniprep.
הערה: בצע את השלבים הבאים בתוך מכסה מנוע סטרילי של זרימה למינרית.
4. מיצוי RNA כולל מזחלי דרוזופילה, מוח ותאי S2
- מוח הזחל: הסר PBS על ידי צנטריפוגה קצרה (8 שניות סיבוב קצר ב 5,000 × גרם).
- הוסף 600 μL של חיץ RNA lysis והומוגניזציה 10x עם מזיק פלסטיק. בדוק חזותית את הצינור תחת מיקרוסקופ סטריאו כדי להבטיח ליזה מלאה.
- צנטריפוגה ב-1,000 × גרם למשך 5 דקות ב-4°C לסילוק פסולת רקמות. מעבירים את הסופרנאטנט המפונה לתוך צינור מיקרוצנטריפוגה נטול נוקלאז.
- בודדו RNA באמצעות ערכת RNA microprep בהתאם להוראות היצרן.
הערה: השימוש בערכת מיקרופרפ RNA חיוני לדגימות מוח הזחל בגלל כמות הרנ"א הקטנה הקיימת בדגימות. - תאי S2: הסר PBS ובודד RNA בהתאם להוראות היצרן.
- מדידת תפוקת RNA ואיכותו על ידי ספקטרופוטומטריה ואלקטרופורזה של ג'ל אגרוז.
- לקבוע את הטוהר והכמות של RNA מופק על ידי מדידת הצפיפות האופטית של RNA מופק ב A260 ננומטר ו A280 ננומטר, בהתאמה. ודא שיחס Aשל 260 ננומטר/A280 ננומטר הוא ≥2.0 וריכוז הרנ"א הוא >350 ננוגרם/מיקרוליטר עבור יישומים במורד הזרם.
הערה: תפוקת RNA טיפוסית ממוחות של 10 זחלי דרוזופילה היא ~500-800 ננוגרם/מיקרוליטר או 2.5-4 מיקרוגרם ב-5 מיקרוליטר. עבור תאי S2, התפוקה היא ~ 2-3 מיקרוגרם / μL (15-30 מיקרוגרם ב 15 μL). RNA מבודד יכול להיות מאוחסן ב -80 °C לאחסון לטווח ארוך.
- לקבוע את הטוהר והכמות של RNA מופק על ידי מדידת הצפיפות האופטית של RNA מופק ב A260 ננומטר ו A280 ננומטר, בהתאמה. ודא שיחס Aשל 260 ננומטר/A280 ננומטר הוא ≥2.0 וריכוז הרנ"א הוא >350 ננוגרם/מיקרוליטר עבור יישומים במורד הזרם.
5. הכנת ג'ל RNA ואלקטרופורזה
- 1.5% ג'ל RNA דנטורינג (100 מ"ל)
הערה: פורמלדהיד רעיל באמצעות מגע עור ושאיפת אדים; טפל בו במכסה אדים כימי.- ממיסים שלוש טבליות אגרוז (1.5 גרם) ב-82 מ"ל של מאגר MOPS (קובץ משלים 1) עד שהטבליות מתפרקות לחלוטין ויוצרות חלקיקים עדינים.
- מחממים את תרחיף האגרוז במיקרוגל עד שהתמיסה ברורה וכל החלקיקים מומסים לחלוטין.
- מצננים את התמיסה ל~60 מעלות צלזיוס.
- מוסיפים 18 מ"ל של 37% פורמלדהיד, ומערבבים בערבול עדין. יוצקים את התמיסה למגש היציקה ונותנים לה להתמצק במכסה אדים.
- הכנת דגימת RNA ואלקטרופורזה
- לדלל את דגימת RNA ל 200 ng (ב 5 μL) ולהוסיף 5 μL של 2x RNA טעינת צבע.
- חממו את הדגימות בטמפרטורה של 70°C למשך 5 דקות באמבט יבש.
- עומס 2 μL של סולם RNA בנתיב הראשון ו 10 μL של דגימות בנתיבים הסמוכים.
- בצע אלקטרופורזה במאגר MOPS במתח של 100 וולט למשך 60 דקות עבור ג'ל בגודל 5 x 6 ס"מ.
הערה: התאם את תנאי האלקטרופורזה בהתאם לגדלי האמפליקון. - דמיינו את הג'ל על טרנסילטור UV.
הערה: נוכחות של פס יחיד ~ 600 גודל נוקלאוטיד מצביע על הכנת RNA שלם (ראה איור 2A).
6. מדידת אורך זנב Poly(A)
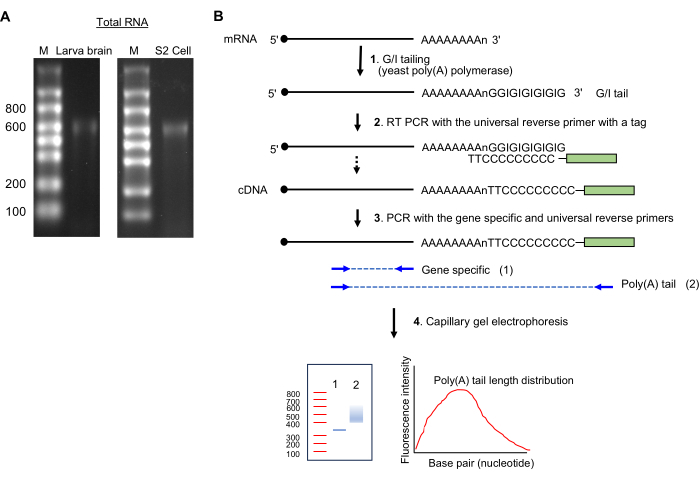
איור 2: הכנת דגימת רנ"א ובדיקת פולי(A)-זנב. (A) תמונות ג'ל הרנ"א מראות רנ"א כולל ממוח זחל דרוזופילה (משמאל) ותאי S2 (מימין) על ג'ל אגרוז פורמלדהיד 1.5%. גדלים של סולם RNA חד-גדילי מוצגים בנוקלאוטידים בנתיב M. שימו לב לפס RNA עיקרי ב~600 nt, שהוא מ-rRNA. (B) סכמות של בדיקת זנב פולי(A). קיצור: G/I = גואנוזין/אינוזין. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
- זנב במערכת העיכול (איור 2B)
- שמירה על ריאגנטים על קרח, להכין את התערובת הבאה (20 μL): עד 14 μL של דגימת RNA הכוללת (1 מיקרוגרם), 4 μL של 5x תערובת חיץ זנב, ו 2 μL של 10x תערובת אנזימי זנב.
- דגירה ב 37 ° C במשך 60 דקות thermocycler.
- הוסף 1.5 μL של תמיסת עצירת הזנב; שומרים על קרח למשך 2 דקות.
הערה: המשך לשעתוק לאחור או אחסן את דגימות ה- RNA עם זנב GI ב- -80 °C עד שיהיה מוכן להמשיך לשעתוק הפוך.
- תמלול לאחור והגברת PCR
- סנתז cDNA על ידי הכנת התערובת ודגירתה בתנאים המתוארים בקובץ משלים 1.
- לדלל את דגימות cDNA ולבצע PCR כדי להגביר את ה- DNA כפי שמצוין בקובץ משלים 1.
7. ניתוח מוצר PCR על ידי אלקטרופורזה ג'ל אגרוז
- נתח חלק קטן (2-5 μL) של מוצרי PCR משלב 6.2.2 על ג'ל אגרוז 2.5% על ידי אלקטרופורזה ב 100 V במשך 45 דקות לבקרת איכות.
- אמת את הספציפיות של PCR עבור תגובות ספציפיות לגן וזנב ספציפיות על ידי ריצוף רצועות PCR שחולצו בג'ל.
8. אלקטרופורזה נימית
- בצע אלקטרופורזה בג'ל ברזולוציה גבוהה על 1 μL של מוצרי PCR (0.5-5 ng/μL) מ- PCR ספציפי לגן ופולי(A) ספציפי באמצעות הביואנלייזר עם ערכת DNA בעלת רגישות גבוהה. חפשו פסגות פתורות היטב המעידות על ריצה מוצלחת.
9. ניתוח נתונים: מדידת אורך זנב poly(A) (איור 3)
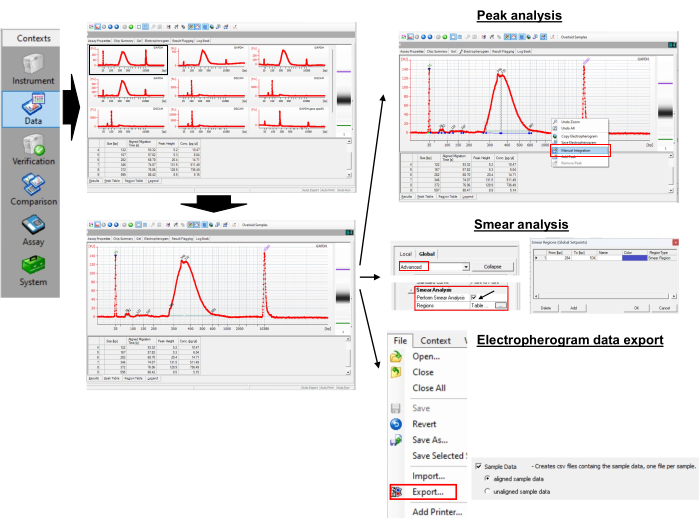
איור 3: אורך זנב Poly(A) ומדידת ערך שיא. לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
- גישה לנתונים
- כדי לגשת לנתונים, פתח את קובץ xad בתוכנה.
- בחרו בשם הדגימה או בסולם בחלונית תצוגת העץ.
הערה: פעולה זו תציג את התוצאה כאלקטרופרוגרמות או תמונות דמויות ג'ל עבור הדגימות שנבחרו. הסמן התחתון 35 זוג הבסיס (bp) והסמן העליון 10,380 bp הם התקנים הפנימיים המשמשים ליישור נתוני הסולם (50-7,000 bp) עם נתונים מבארות הדגימה. - הגדל והקטן את התצוגה של אלקטרופרוגרמות ותמונות דמויות ג'ל כדי להציג את הפרטים.
- ביצוע ניתוח שיא והכפשה
- כדי להשיג את גדלי השיא, פתח את האלקטרופרוגרמה של מדגם שנבחר.
- לחץ לחיצה ימנית על אלקטרופרוגרמה ובחר שילוב ידני כדי לבחור פסגות באופן ידני על ידי גרירת הקו האופקי.
- התבונן בערכי השיא בטבלה 'מרב'. זהה את הפסגה עם גובה השיא הגדול ביותר. זהו שיא של אורך זנב פולי(A) עבור מדגם בודד. בדוגמה המוצגת, הוא 346 bp.
- הפעל את סמל הצג/הסתר נקודות בתפריט העליון והמתן להופעת חלונית חדשה בצד ימין.
- בחר מתקדם, גלול מטה כדי למצוא את Perform Smear Analysis ובחר בתיבת הסימון. פעולה זו תוסיף את טבלת האזור לכרטיסייה אלקטרופרוגרמה.
- בחר אלקטרופרוגרמה מדגימה ועבור אל הטבלה אזור, המציגה את התפריט מאת [bp] ותפריט אל [bp]. כדי להגדיר את ההתחלה והסיום [bp], לחץ לחיצה ימנית על האלקטרופרוגרמה ובחר אזור כדי להוסיף אזור.
- לחץ באמצעות לחצן העכבר הימני על תאים כלשהם בטבלת Region ובחר Modify Regions כדי ליצור חלון חדש קטן שבו ניתן להגדיר אזורים מותאמים אישית.
הערה: לדוגמה, השתמשנו באזור של 300 bp עד 550 bp עבור GAPDH. ה-GAPDH PCR הספציפי לגן הניב שיא של 265 bp. הפריימר האוניברסלי (טבלה 1) מאריך את אורך ה-PCR של פולי(A) ב-35 bp באמצעות חישול ל-RNA זנב-G/I. לפיכך, נוקלאוטיד אדנין הראשון על RNA GAPDH מתחיל ב 300 bp (265 + 35). הגבלנו באופן שרירותי את אורך הזנב המרבי של פולי(A) ל-250 (300 + 250 = 550). מטבלת האזורים, התוכנית מחזירה את הגודל הממוצע בתוך האזור כ- 387 bp. - השתמש במשוואה (1) כדי לחשב את אורך הזנב של poly(A) על mRNA של עניין:
אורך זנב Poly(A) = (A - B - 35) (1)
כאשר A הוא ממוצע bp של מוצר PCR ספציפי לפולי(A) מאלקטרופרוגרמה (כלומר, 387 bp עבור GAPDH), B הוא שיא bp של מוצר PCR ספציפי לגן מאלקטרופרוגרמה (כלומר, 265 bp עבור GAPDH), ו- "35" הוא האורך של תג פריימר הפוך אוניברסלי.
הערה: מהחישוב לעיל, אורך הזנב הממוצע של poly(A) של GAPDH הוא 387 - 265 - 35 = 87 bp.
10. הדמיה של התפלגות אורך זנב poly(A)
- ייצוא נתונים כקובץ csv תחת קובץ | ייצוא | נתונים לדוגמה כדי לאחזר נתונים לדוגמה.
הערה: קובץ ה- csv המיוצא מציג זמן ריצה במקום bp בציר X. - עבור אל אלקטרופרוגרמה של דגימה ובדוק את הצג גדלים בכרטיסיה אלקטרופרוגרמה כדי להמיר באופן אוטומטי את טבלת האזורים בטבלת האזורים מ- bp לזמן ריצה. בדוגמה, 70.39 שניות עד 86.28 שניות מתאימות ל- 300 bp עד 550 bp.
- פתח את קובץ ה- csv ובחר את הערכים בין 70.39 שניות ל- 86.28 שניות של זמן ריצה כדי ליצור גרף. כדי להמחיש גדלי bp לציר X בתרשים, יצא את האלקטרופרוגרמה עם גדלי bp כקובץ תמונה ושכב אותה על הגרף שנוצר בגיליון האלקטרוני. זה יתאים כראוי לגדלי bp בהתפלגות זנב poly(A).
Access restricted. Please log in or start a trial to view this content.
תוצאות
כאן ניתחנו את אורך הזנב של פולי(A) של Dscam1 ו-GAPDH ממוחות של זחלי דרוזופילה (איור 4). RNA מבודד הודגם על ג'ל אגרוז לבקרת איכות. רצועת רנ"א בודדת בגודל של כ-600 נוקלאוטידים מצביעה על הכנת רנ"א שלמה (איור 2A). RNAs היו נתונים לזנב G/I ולאלקטרופורזה נימית ברזולוצי...
Access restricted. Please log in or start a trial to view this content.
Discussion
בפרוטוקול זה, אנו מתארים את הטכניקה לנתח את מוח הזחל Drosophila משלב 3rdinstar נודד, כמו גם את הכנת הדגימה מתאי Drosophila S2. בשל אופיים העלוב של mRNAs, איסוף הדגימות דורש זהירות יתרה. במקרה של דיסקציה מוחית של הזחל, המוח לא אמור להיפגע במהלך הבידוד ואין לשמור אותו בתמיסה למשך זמן ממושך. שמירה ...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין ניגודי עניינים לחשוף.
Acknowledgements
מחקר זה נתמך על ידי המכון הלאומי להפרעות נוירולוגיות ושבץ מוחי R01NS116463 לג'יי.קיי., ומתקן הליבה של הדמיה תאית ומולקולרית באוניברסיטת נבאדה, רינו, אשר נתמך על ידי המכונים הלאומיים לבריאות P20GM103650 ושימש למחקר שדווח במחקר זה.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
References
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435(2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622(2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57(2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294(2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved