需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
选择依赖和独立生成CRISPR / Cas9介导的哺乳动物细胞基因敲除
摘要
遗传操纵体细胞系的能力的最新进展对于基础和应用研究具有巨大的潜力。在这里,我们提出了CRISPR / Cas9在哺乳动物细胞系中产生敲除生产和筛选的两种方法,使用和不使用选择标记。
摘要
CRISPR / Cas9基因组工程系统通过允许精确的基因组编辑,很少的努力,彻底改变了生物学。在引导特异性的单指导RNA(sgRNA)的指导下,Cas9蛋白在靶位点切割两条DNA链。 DNA断裂可以触发非同源末端连接(NHEJ)或同源性定向修复(HDR)。 NHEJ可以引入导致帧转移突变的小缺失或插入,而HDR允许更大和更精确的扰动。在这里,我们提出了通过将建立的CRISPR / Cas9方法与下游选择/筛选的两个选项相结合来产生敲除细胞系的方案。 NHEJ方法使用单个sgRNA切割位点和选择无关筛选,其中以高通量方式通过点免疫印迹评价蛋白质产生。 HDR方法使用跨越感兴趣基因的两个sgRNA切割位点。与提供的HDR模板一起,该方法可以实现删除几十kb,由插入的可选电阻标记辅助。讨论每种方法的适用应用和优点。
引言
稳定的遗传改变比细胞扰动的瞬时方法提供了一个优点,这可以在其效率和持续时间上变化。由于目标特异性核酸酶的发展,如锌指核酸酶1,2,3,4,5,转录激活物样效应核酸酶(TALEN) 6,7,8,9 ,近年来,基因组编辑越来越普及和衍生自聚集的,定期交织的短回文重复(CRISPR)系统的RNA引导的核酸酶10 。
CRISPR / Cas9编辑机器改编自细菌和古菌用于防御病毒感染的免疫系统ass ="xref"> 11,12,13。在这个过程中,入侵病毒序列的短的20-30nt片段被引入到基因组基因座中,作为侧重于重复单元14,15的 "间隔物"。随后的转录和RNA加工产生小的CRISPR相关RNA 16 (crRNA),其与反式激活的crRNA17(tracrRNA)一起与效应物Cas9核酸内切酶组装。因此,crRNA提供对Cas9靶向的特异性,指导复合物切割互补的病毒DNA序列并防止进一步的感染18,19 。目标DNA中的任何"原始孢子"序列都可以作为crRNA的来源,只要它直接与短原型相邻基序(PAM)5'相连,在化脓性链球菌 Cas9 20。宿主CRISPR基因座中间隔物附近的PAM序列的缺失区分自身和非自身,防止宿主的靶向。由于其普遍性和灵活性,该生物系统已经被强力地适应于基因组编辑,使得几乎任何PAM相邻的DNA位点都可以被靶向。在该版本中,进一步的修改将crRNA和tracrRNA融合到装载到Cas9蛋白21中的单个引导RNA(sgRNA)组分中。
在真核细胞中Cas9和sgRNA表达后,Cas9蛋白在目标基因座处切割两条DNA链。在没有合适的同源序列区域的情况下,细胞通过非同源末端连接(NHEJ) 22,23,24修复了该断裂,其通常引入小的缺失或很少插入。定位开放时阅读框,修复可能导致产生非功能蛋白质产物的翻译移码。相比之下,当提供具有大的同源区域的外源模板时,细胞可以通过同源性定向修复25,26固定双链断裂。该途径允许在基因组中进行更大的精确的缺失,置换或插入,以及引入可切除的选择标记27 。
在这里,我们提出了通过这两种CRISPR / Cas9方法产生敲除细胞系的方案( 图1A )。 NHEJ方法使用单个sgRNA切割位点和选择无关筛选,因此需要很少的前期准备。当使用这种方法时,必须设计指导与最有可能产生敲除的转录物5'末端附近的外显子互补的RNA。自从修改在这种情况下,到基因组的离子量很小,筛选克隆克隆基于斑点印迹,其中以高通量方式评估蛋白质产物。我们使用ELAV样1蛋白(ELAVL1)敲除生产线的生成作为例子。第二种方法依赖于同源性定向修复(HDR),并使用跨越感兴趣的基因或区域的两个sgRNA切割位点,允许几十kb的缺失。在切割位点侧面具有两个具有同源性区域的质粒提供替代模板( 图1B ),引入可选择的抗性标记,其提高敲除产生效率。该方法也可适用于引入具有适当设计的同源臂的基因修饰。在这种情况下,新的DNA片段的整合允许基于PCR的筛选( 图1C )。在这里,我们使用Pumilio RNA结合家族成员2(PUM2)敲除生产线的生成作为例子。
研究方案
1.找出需要删除的同源区域
注意:仅在使用基于选择的编辑时才需要。
- 在所需的缺失位点的任一侧选择两个最初为1.5-2 kb的区域,这将作为HDR模板中的同源臂( 图1A )。确定缺乏任何一条(GGTCTC)上的BsaI识别位点以促进克隆的区域。如果BsaI位点是不可避免的,则使用替代型IIs限制酶(BsmBI,SapI,BbsI)并修饰受体质粒(pUC19-BsaI),抗性标记供体质粒(pGolden-Neo或pGolden-Hygro)中的相应位点,同源臂PCR产物突出。
2.产生Cas9-sgRNA表达质粒
- 定义诱变的靶向位点。对于单切,无选择方法,目标5'近端编码外显子增加非功能的概率功能突变对于双切法,选择两个跨越基因必需的位点。
注意:根据我们的经验,有效地创建高达53 kb的删除。 - 输入200到300 bp的目标区域序列到crispr.mit.edu设计工具。对于基于选择的克隆,使用近似于缺失位点的200-300bp的鉴定的同源区( 图1A )。选择每个目标区域最高排名的sgRNA的2-3个,以解释其活动的差异,并分离不具有相同潜在的脱靶效应的独立克隆。
- 为了设计具有适当突出端的用于插入表达质粒的双链体寡核苷酸,省略末端PAM序列(NGG),并将5-CACC突出端附着,然后附加G至顶链寡核苷酸。对于底部链寡核苷酸,将5-AAAC突出端附加到反向互补的靶序列,随后是3'-C,如下面介绍:
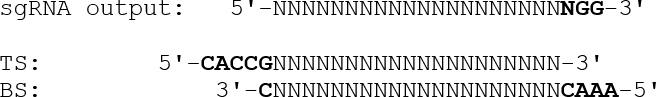
- 使用Zhang实验室方案29所述的金门克隆28 ,将对应于sgRNA的合成寡核苷酸插入pSpCas9(BB)质粒。
同源性修复模板质粒的生成
注意:仅在使用基于选择的编辑时才需要。同源性修复模板质粒由两个与两个sgRNA靶位点之外的基因组互补的两个区域的耐药盒组成( 图1B )。
- 从步骤1.1中鉴定的更广泛的同源区域,选择距设计的Cas9切割位点不超过5-10bp的800-1,000bp 26 ,用作同源臂( 图1A )。确保不包括sgRNA目标位点及其PAM在同源臂中,这将导致CRISPR / Cas9切割模板质粒本身。如果同源性武器含有内部BsaI位点,则使用其他sgRNA位点。
- PCR使用高保真DNA聚合酶根据制造商的说明书使用具有引入BsaI位点(GGTCTC)的另外的5'序列的正向引物和反向引物以及同源区两端的独特突出端扩增基因组DNA的同源性臂。突出部分必须包含适当的序列以产生正确的装配顺序和取向,如下所述( 图1B ):
左同源臂FP突出:5'GGGTCTCA GGCC
左同源臂RP突出:5'GGGTCTCT CACG
右侧同源臂FP突出:5'GGGTCTCA GTCC
右侧同源臂RP突出:5'GGGTCTCT CCAC - 检查同源性臂适当的扩增在进行前进行凝胶电泳。
- 通过金门克隆28将具有电阻盒的右侧和左侧同源臂区域组装成HDR模板质粒。使用pGolden-Neo或pGolden-Hygro质粒30作为loxP-侧翼抗性盒(loxP-PGK-Neo-pA-loxP或loxP-PGK-Hyg-pA-loxP)的来源。使用pUC19-BsaI,在多克隆区域中具有BsaI位点的修饰的pUC19,并将其他位置的BsaI位点作为受体载体(可根据要求提供)消除。对于三个插入和矢量,使用1:1:1:1的比例。
- 准备以下反应混合物30 :
右侧同源臂PCR产物 0.06 pmol(30-40ng) 左同源臂PCR产物 0.06 pmol(30-40ng) pGolden-Neo质粒(100ng /μL) 1µ l pUC19-BsaI质粒(100ng /μL) 1μL 2x T7 DNA连接酶缓冲液 5μL BsaI(10 U /μL) 0.75μL T7 DNA连接酶(3000 U /μL) 0.25μL 水 最多10μL - 使用以下热循环仪参数:37℃5分钟,20℃5分钟。重复30个周期。
- 用制造商的说明书用外切核酸酶处理克隆产物,消化任何剩余的线性化DNA。
- 根据提供给细胞的方案将反应混合物转化为感受态大肠杆菌菌株,并在含有氨苄青霉素的培养皿上培养。
- 准备以下反应混合物30 :
- 要使用正确的模板组合识别克隆,请执行细菌扩增组装的插入物的同源臂亚区域(因为整个插入物可能太大而不能可靠地扩增)。使用一个引物退火到侧翼质粒序列,另一个引物与抗性盒的边缘互补( 图1B ,序列对Neo和Hygro插入物都是共同的),产生大约200bp的装配依赖性产物包含的同源臂:
pUC19-Bsal-Left:5'GGCTCGTATGTTGTGTGGAATTGTGAG
电阻左:5'AAAAGCGCCTCCCCTACCC
pUC19-BsaI-Right:5'GCTATTACGCCAGCTGGCGAAA
抵抗权:5'AAGACAATAGCAGGCATGCTGGG- 用无菌牙签挑取细菌菌落,并将细菌悬浮在20μL水中。在95℃下加热15分钟,并在桌面离心机中以最大速度旋转5分钟。立即放在冰上。
- 准备以下PCR混合物31/ SUP>:
稀释菌落模板 1.25μL 10x Taq反应缓冲液 1.25μL 20mM dNTPs 0.25μL 10μM正向引物 0.25μL 10μM反向引物 0.25μL Taq聚合酶 0.25μL 水 9μL - 使用以下热循环仪参数:95℃30秒,(95℃30秒,61℃30秒,72℃1分钟/ kb),重复25个循环,72℃2分钟。
- 通过琼脂糖凝胶电泳检查PCR产物的正确扩增子大小。
- 任选地,来自具有正确插入片段大小和顺序的单个菌落的微量制备质粒DNA通过Sanger测序与上述扩增引物的同源臂区。
4.将CRISPR成分转染到培养细胞中
- 转染前:在37℃和5%CO 2下补充有10%FBS的DMEM培养基中培养T-REx293细胞。
- 将细胞板放置在6孔板上并生长至约70%汇合。包括一个未经转染的控件的井。
- 使用商业转染方案用2.5μg总质粒转染细胞。同时保持未转染的控制。
注意:转染方法和效率因细胞类型而异。在实验前确定系统的适当转染方法。- 对于用选择的细胞进行转染,使用0.75μgCas9-sgRNA1(左切),0.75μgCas9-sgRNA2(右切)和1.0μg同源重组模板。
- 用于转染在没有选择的细胞上,使用2.5μgCas9-sgRNA质粒。
5.药物选择
- 转染后48 h,用合适的药物(新霉素/潮霉素)处理细胞。进行选择,直到未转染对照的所有细胞死亡(通常为Neo为3-5天,Hygro为7-14天)。
注意:对于新霉素和潮霉素,分别使用浓度为500μg/ mL和10μg/ mL的T-REx293细胞。对于其他细胞类型,可能有用的是在未转染的细胞上进行药物的先前滴定以确定有效浓度。
6.克隆种群的分离
- 选择后,在原始细胞中生长细胞至100%融合。将种子细胞以0.33个细胞/孔的密度进入96孔平板。
注意:播种三个96孔板是一个很好的起点,以确保分离多于一个正确的克隆,但是或多或少可能需要dep结束于sgRNA和HDR效率。 - 在2-4周的时间内观察菌落,直到菌落对眼睛是可见的。用无菌移液器吸头挑取可见的菌落,并在新的孔中重新种植以促进单层生长。当使用悬浮细胞时,甚至可以使用更低的接种密度来确保更大比例的单菌落孔。
7.候选人筛选
- 筛选候选人不使用选择:斑点印迹
- 将个体克隆培养至50-100%融合。通过在孔内移液来移走单层细胞。
- 将100μL总体积的90μL等分成干净的微量离心管。以6000rpm离心5分钟,除去培养基,并在10μL1x Lysis Buffer或1x SDS加载缓冲液(5x:250mM Tris-Cl pH 6.8,8%SDS,0.1%溴酚蓝,40%甘油,100mM DTT)。在其余的细胞中加入90μL新的培养基我继续传播。
- 将1μL细胞裂解液移至干燥的硝酸纤维素膜上,形成一个点。在两个单独的膜上将每个样品两次打印,产生两个相同的样品样式。
- 在室温下将TBST(Tris缓冲盐水+ 0.01%吐温20:8g NaCl,0.2g KCl,3g Tris碱,至多1L蒸馏水,pH 7.4) 31中的5%乳块中的膜封闭1小时摇摆,这里和整个程序)。
- 在一个膜上使用针对靶蛋白的一抗进行印迹,另一个使用对照蛋白(微管蛋白,GAPDH或任何其他预期不会改变的蛋白质)的一抗。在TBST + 5%牛奶中使用推荐的一级抗体稀释度(对于ELAVL1为0.2μg/ mL,在这种情况下为Pum2为0.1μg/ mL)。在室温下孵育1小时。
- 用TBST洗涤3次5分钟。
- 在T中用合适的HRP缀合的二抗孵育每个膜BST + 5%牛奶在室温下1小时。
- 用TBST洗涤3次5分钟。
- 根据制造商的说明书应用化学发光底物溶液(参见材料表),并将数字化学发光成像仪上的印迹膜成像。
- 使用适当的软件量化靶和对照蛋白的点强度。
- 消除具有低控制蛋白信号的候选者,并计算目标的背景扣除比例以控制剩余候选物的蛋白质强度。选择最低通过率的候选物,并通过蛋白质印迹进一步验证。
- 筛选候选人使用选择:菌落PCR
- 使用DNA准备试剂盒收集细胞裂解物(参见材料表 )。
- 在新的96孔中重复个体菌落,生长至100%融合。
- 从一组克隆中删除介质,a并从试剂盒中将细胞重悬于30μL提取缓冲液中。转移到干净的1.5 mL微量离心管中。
- 将溶液加热至96°C 15 min,冷却至室温。
- 从试剂盒中加入30μL稳定缓冲液。混合好
- 通过菌落PCR鉴定野生型和单等位基因/双等位基因/双等位突变体系。
- 设计两组单独的PCR引物(针对目标基因座的左侧和右侧),以基于电阻盒的成功或不成功的整合扩增同源臂周围的区域( 图2C )。在每一侧,使用在同源臂外退火的通用底漆(红色)。将其与与内源序列互补的相应配对引物(跨越同源区域(蓝色))进行测试,以测试野生型等位基因。使用与插入的电阻盒互补的另一配对引物(参见部分引物)2.3),以测试所需的突变。
- (推荐)使用原始选择的大块细胞群体的细胞裂解物验证引物组,因为它将含有WT和突变体等位基因的混合物( 图2D )。
- 准备以下反应混合物:
细胞裂解物 0.5μL 10倍KOD缓冲液 1.25μL 25mM MgSO 4 0.75μL 2 mM dNTPs 1.25μL 10μM正向引物 0.375μL 10μM反向引物 0.375μL KOD聚合酶 0.25μL 水 7.75μL - 使用以下热循环仪条件:95℃2分钟,(95℃20s,Primer Tm 10s,70℃20s / kb)重复25个循环。
- 通过琼脂糖凝胶电泳可视化PCR产物。
- 对预测整合的每一侧重复菌落PCR检测。选择并扩展显示存在整合的候选者,而不删除被删除区域的内源序列产物。
- 通过蛋白质印迹和/或RT-qPCR验证阳性候选物。
- 使用DNA准备试剂盒收集细胞裂解物(参见材料表 )。
8.通过测序验证基因组突变
- 通过苯酚氯仿提取分离突变体细胞系的基因组DNA 31 。
- PCR扩增sgRNA靶区,使用5'末端的引物,每端加入BsaI限制性位点。
注意:对于使用HDR编辑的克隆,其中两个等位基因可能相同,可以直接对PCR产物进行测序。 - 通过金门克隆将扩增区克隆到pUC19-BsaI中。
- 将反应混合物转化为感受态大肠杆菌菌株。
- 来自6-10个单独菌落的Miniprep质粒DNA,并通过Sanger测序序列克隆的区域。
结果
为了产生ELAVL1敲除生产线,可以使用强大的抗体,因此使用单个sgRNA进行编辑( 图1A ,左侧),然后进行点免疫印迹。独立地转染三种sgRNA以比较效率并排除所得克隆中的靶效应。在将克隆群体的细胞裂解物收集并印迹到两个硝酸纤维素膜上之后,对ELAVL1和PUM2(作为归一化对照)检测印迹( 图2A )。 ELAVL1与PUM2信号的量化比例?...
讨论
CRISPR / Cas9系统允许有效地产生稳定的基因组修饰,这提供了与其他瞬时操作方法更一致的替代方案。在这里,我们提出了两种在哺乳动物细胞系中快速鉴定CRISPR / Cas9基因敲除的方法。这两种方法都需要很少的细胞材料,因此可以在克隆培养的早期阶段进行测试,节省时间和试剂。为了提高两种方法的效率,我们建议测试多种sgRNA,因为效率因序列和基因组位置而异。此外,使用多个sgRNA可用于通...
披露声明
作者宣称他们没有竞争的经济利益。
致谢
作者要感谢Gissell Sanchez,Megan Lee和Jason Estep的实验帮助,Weifeng Gu和Xuemei Chen分享试剂。
材料
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
参考文献
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。