Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Memeli Hücrelerinde CRISPR / Cas9 aracılı Gen Knockout'larının Seçime Bağlı ve Bağımsız Üretimi
Bu Makalede
Özet
Somatik hücre dizilerini genetik olarak manipüle etme becerisindeki son gelişmeler, temel ve uygulamalı araştırmalar için büyük potansiyele sahiptir. Burada, CRISPR / Cas9'un ürettiği nakavt üretimi ve seçilebilir işaretlerin kullanılıp kullanılmadığı memeli hücre hatlarında tarama için iki yaklaşım sunuyoruz.
Özet
CRISPR / Cas9 genom mühendisliği sistemi, az çaba sarf ederek hassas genom düzenlemesine izin vererek biyolojiye devrim yarattı. Spesifikliği kazandıran tek bir rehber RNA (sgRNA) yardımıyla Cas9 proteini, her iki DNA zincirini hedeflenen lokusta parçalamaktadır. DNA kopması homolog olmayan bitiş katılma (NHEJ) veya homoloji yönelimli onarım (HDR) tetikleyebilir. HDR, daha büyük ve daha hassas pertürbasyonlara izin verirken, NHEJ, çerçeve kayması mutasyonlarına yol açan küçük delesyonlar veya eklemeler başlatabilir. Burada, çökertilmiş hücre hatlarının üretilmesi için protokolleri, kurulu CRISPR / Cas9 yöntemlerini downstream seçim / tarama için iki seçenek ile birleştirerek sunuyoruz. NHEJ yaklaşımı, tek bir sgRNA kesim bölgesini ve seleksiyondan bağımsız taramayı kullanır; burada, protein üretiminin nokta imünoblotuyla yüksek verimli bir şekilde değerlendirildiği yerdir. HDR yaklaşımı ilgi geni boyunca uzanan iki sgRNA kesim bölgesini kullanır. Sağlanan HDR şablonuyla birlikte bu yöntem silme gerçekleştirebilirEklenebilen seçilebilir direnç işaretleyicisinin yardımıyla, onlarca kb arasında değişir. Her bir yöntemin uygun uygulamaları ve avantajları tartışılmıştır.
Giriş
Kararlı genetik değişiklikler, verimlilik ve süre bakımından değişken olabilen geçici hücresel pertürbasyon yöntemlerine göre bir avantaj sağlar. Genomik düzenleme, çinko parmak nükleazları 1 , 2 , 3 , 4 , 5 , transkripsiyon aktivatörü benzeri efektör nükleazları (TALEN'ler) 6 , 7 , 8 , 9 gibi hedefe spesifik nükleazların gelişimi nedeniyle son yıllarda giderek yaygınlaşmaktadır. Ve kümelenmiş, düzenli olarak aralıklı kısa palindromik tekrarlardan (CRISPR) türemiş RNA yönlendirmeli nükleazlar 10 .
CRISPR / Cas9 düzenleme makineleri bakterilerin ve arkeaların viral enfeksiyonlara karşı savunmak için kullandıkları bağışıklık sisteminden adapte edilmiştirAss = "xref"> 11 , 12 , 13 . Bu süreçte, işgalci viral dizinin kısa, 20-30 nt fragmanları, tekrar eden birimler 14 , 15 tarafından çevrelenen "aralayıcılar" olarak genomik bir lokasyona dahil edilir. Ardından transkripsiyon ve RNA işleme, bir aktive edici crRNA 17 (tracrRNA) ile efektör Cas9 endonükleazı birleştiren küçük CRISPR ilişkili RNA'lar16 (crRNA'lar) üretir. Dolayısıyla, crRNA'lar Cas9 hedeflemesine özgüllük sağlar, kompleksi tamamlayıcı viral DNA dizilerini bölmeye yönlendirir ve diğer enfeksiyonları önler 18,19. Hedeflenen DNA'daki herhangi bir "protospacer" sekans, kısa bir protospazere komşu motife (PAM) doğrudan 5 ', S. pyogenes Cas9 durumunda ise NGG olduğu sürece, crRNA'nın kaynağı olarak görev yapabilir 20. Ev sahibinin CRISPR lokusu içindeki boşluk yakınında PAM sekansının olmaması, benliğin kendisi ile ben olmayan arasındaki ayrımı yaparak, ev sahibinin hedeflenmesini engeller. Evrenselliği ve esnekliği nedeniyle, bu biyolojik sistem genomik düzenleme için güçlü bir şekilde adapte edilmiştir, böylelikle hemen hemen her PAM bitişik DNA sitesi hedeflenebilir. Bu sürümde, başka bir değişiklik, crRNA ve tracRNA'yı Cas9 protein 21'e yüklenen tek bir rehber RNA (sgRNA) bileşenine kaynaştırdı.
Ökaryotik hücrelerde Cas9 ve bir sgRNA ekspresyonu üzerine Cas9 proteini, her iki DNA kolunu hedeflenen lokusta parçalamaktadır. Uygun homolog bir dizinin yokluğunda hücre bu kopuşu tipik olarak küçük delesyonlar veya nadiren eklemeler başlatan homolog olmayan uç birleştirme (NHEJ) 22 , 23 , 24 aracılığıyla tespit eder. Bir açık hedefleme yaparkenOkuma çerçevesi, onarım muhtemelen işlevsel olmayan bir protein ürünü üreten bir translasyonel çerçeve kaymasına yol açar. Aksine, geniş homoloji bölgelerine sahip bir dışsal şablon ile sağlandığında, hücre homolog yöneltilmiş onarım 25 , 26 ile çift iplikçik kırılmasını sabitleyebilir. Bu yol, genomda daha büyük hassas silme, değiştirme veya eklemeye izin verir ve ölebilir seçim işaretleri 27'nin eklenmesi ile birlikte.
Burada, bu iki CRISPR / Cas9 yöntemlerinden biri ile nakavt hücre hatları üretmek için protokoller sunuyoruz ( Şekil 1A ). NHEJ yaklaşımı, tek bir sgRNA kesim bölgesini ve seçiciden bağımsız taramayı kullanır ve bu nedenle çok fazla ön hazırlık gerektirir. Bu yöntemi kullanırken, transkriptin 5 'ucundaki eksonları tamamlayan ve en iyi nakavt üreten kılavuz RNA'lar tasarlanmalıdır. Modifikattan beriBu durumda iyonların genoma küçük olması, nakavt klonlarının taranması, protein ürününün yüksek verimli bir şekilde değerlendirildiği nokta lekelerine dayanıyor. Bir örnek olarak ELAV benzeri 1 protein (ELAVL1) nakavt hatları neslini kullanırız. İkinci yaklaşım, homoloji yönelimli onarım (HDR) üzerine kuruludur ve onlarca kb silinmesine izin vererek ilgi geni veya bölgeyi kapsayan iki sgRNA kesim bölgesini kullanır. Yarılma bölgelerinin yanındaki iki homoloji bölgesine sahip bir plazmid, nakavt üretiminin verimliliğini arttıran seçilebilir bir direnç markeri tanıtan bir değiştirme şablonu ( Şekil 1B ) sağlar. Bu yöntem, uygun şekilde tasarlanmış homoloji kollarıyla gen modifikasyonlarını başlatmak için uyarlanabilir. Bu durumda, yeni bir DNA parçasının entegrasyonu PCR'ye dayalı taramaya izin verir ( Şekil 1C ). Burada, örnek olarak Pumilio RNA bağlayıcı aile üyesi 2 (PUM2) nakavt hatları neslini kullanıyoruz.
Protokol
1. İstenilen Silinme Çevresindeki Homoloji Bölgelerinin Belirlenmesi
NOT: Yalnızca seçim temelli düzenleme kullanılıyorsa gereklidir.
- HDR şablonunda homoloji kolları olarak görev yapacak istenen silme yerinin her iki tarafında başlangıçta 1.5-2 kb olan iki bölge seçin ( Şekil 1A ). Klonlamayı kolaylaştırmak için her iki zincirde (GGTCTC) BsaI tanıma sitelerinden yoksun bölgeler belirleyin. BsaI siteleri kaçınılmazsa alternatif bir IIs kısıtlama enzimi (BsmBI, SapI, BbsI) kullanın ve alıcı plazmitteki karşılık gelen siteleri (pUC19-BsaI), direnç markörü verici plasmidini (pGolden-Neo veya pGolden-Hygro) değiştirin ve Homoloji kolu PCR ürünü çıkıntıları.
2. Cas9-sgRNA Ekspresyon Plazmidlerinin Üretimi
- Mutajenez için hedef alan (lar) ı tanımlayın. Tek kesimli, seçime bağlı olmayan yöntem için, bir non-fu olasılığını artırmak için 5 'yakınlık kodlama eksonlarını hedefleyinNasyonal mutasyon. İki kesimli yöntem için, gerektiği kadar geni kapsayan iki site seçin.
NOT: Deneyimlerimize göre 53 kb'ye kadar olan silinmeler verimli bir şekilde oluşturulmuştur. - Hedeflenen bölge diziliminin 200 ila 300 bp'lik değerini crispr.mit.edu tasarım aracına girin. Seçim temelli klonlama için, silme lokusuna yakın olan 200-300 bp'lik tanımlanmış homoloji bölgelerini kullanın ( Şekil 1A ). Faaliyet bölgelerindeki farklılıkları açıklamak ve aynı potansiyel dışı etkileri paylaşmayan bağımsız klonları izole etmek için hedef bölgeye göre en üst sıralarda yer alan sgRNA'lardan 2-3'ünü seçin.
- İfade plazmidine yerleştirilmek üzere uygun çıkıntılar ile duplekslenmiş oligonükleotitleri tasarlamak için, biten PAM dizisini (NGG) atlayın ve bir 5'-CACC çıkıntısı takiben bir üst üste oligonoza bitişik ekleyin. Alt iplikçiği oligo için, ters tamamlanmış hedef dizisine bir 5'-AAAC çıkıntı ekleyin, bunu takiben 3'-C, ilAşağıda lustrated:
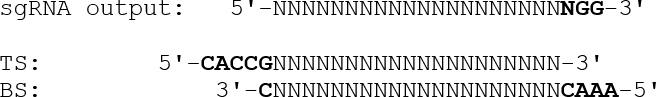
- SgRNA'lara karşılık gelen sentetik oligonükleotitleri, Zhang laboratuvar protokolü 29'da tarif edildiği gibi Golden Gate klonlama 28'i kullanarak pSpCas9 (BB) plasmidine yerleştirin.
Homolojiye Yönelik Tamir Şablonu Plazmidlerinin Üretilmesi
NOT: Yalnızca seçim temelli düzenleme kullanılıyorsa gereklidir. Homoloji yönelimli onarım şablonu plazmid, iki sgRNA hedef alanının hemen dışındaki genomu tamamlayan iki bölge tarafından çevrelenmiş bir ilaç direnci kasetinden oluşur ( Şekil 1B ).
- Adım 1.1'de tanımlanan daha geniş homoloji bölgelerinden, homoloji kolları olarak görev yapmak üzere tasarlanmış Cas9 kesim bölgelerinden 800-1000 bp 26 en fazla 5-10 bp uzaklıkta olanı seçin ( Şekil 1A ). SgRNA'yı içermediğinden emin olunHedef bölge ve PAM'sini homoloji kollarında kullanır, çünkü şablon plazmitinin CRISPR / Cas9 bölünmesine neden olur. Homoloji kolları dahili BsaI siteleri içeriyorsa, alternatif sgRNA siteleri kullanın.
- PCR, homolog bölgenin herhangi bir ucunda BsaI bölgelerini (GGTCTC) ve benzersiz çıkıntıları tanıtan ilave 5 'sekansı olan ileri ve geri primerleri kullanarak üreticinin talimatlarına göre yüksek doğrulukta bir DNA polimerazı kullanarak genomik DNA'dan homoloji kollarını yükseltmektedir. Çıkıntılar, aşağıda açıklandığı gibi ( Şekil 1B ) doğru montaj sırasını ve yönünü üretmek için doğru dizileri içermelidir:
Sol homoloji kolu FP çıkıntısı: 5 'GGGTCTCA GGCC
Sol homoloji kolu RP çıkıntısı: 5 'GGGTCTCT CACG
Doğru homoloji kolu FP çıkıntısı: 5 'GGGTCTCA GTCC
Doğru homoloji kolu RP çıkıntısı: 5 'GGGTCTCT CCAC - Uygun olan amplifık için homoloji kollarını kontrol edinİleriye ilerlemeden önce jel elektroforezi yoluyla verin.
- Sağ ve sol homoloji kol bölgelerini direnç kaseti ile Golden Gate klonlama 28 ile bir HDR şablon plasmidine birleştirin. LoxP ile çevrili direnç kasetleri (loxP-PGK-Neo-pA-loxP veya loxP-PGK-Hyg-pA-loxP) kaynağı olarak pGolden-Neo veya pGolden-Hygro plazmidlerini 30 kullanın. Çoklu klonlama bölgesinde BsaI siteleri bulunan bir pUC19-BsaI, alıcı vektör olarak BSAI bölgelerini başka yerlerde elimine eden değiştirilmiş bir pUC19 kullanın (istek üzerine temin edilebilir). Üç ekleme ve vektör için 1: 1: 1: 1 oranını kullanın.
- Aşağıdaki reaksiyon karışımını 30 hazırlayın:
Doğru homoloji kolu PCR ürünü 0.06 pmol (30-40ng) Sol homoloji kolu PCR ürünü 0.06 pmol (30-40ng) PGolden-Neo plasmid (100 ng / ml) 1 &181. L PUC19-BsaI plasmid (100 ng / ml) 1 uL 2x T7 DNA ligaz tamponu 5 uL BsaI (10 U / uL) 0,75 μL T7 DNA ligazı (3000 U / uL) 0.25 uL Su 10 μL'ye kadar - Aşağıdaki termal çevrimci parametrelerini kullanın: 5 dakika boyunca 37 ° C, 5 dakika için 20 ° C. 30 döngü için tekrarlayın.
- Geriye kalan doğrusallaştırılmış DNA'yı sindirmek için üreticinin talimatlarına göre klonlama ürününe bir eksonükleaz verin.
- Reaksiyon karışımını hücrelerle verilen protokole göre yetkili bir E. coli suşuna, ampisilin içeren kaplarda plaka haline getirin.
- Aşağıdaki reaksiyon karışımını 30 hazırlayın:
- Doğru şablon düzeneğiyle klonları tanımlamak için, bakteri uygulamak(Tamamı eklemenin güvenilir şekilde büyütülmesi için çok büyük olabilmesi nedeniyle) birleştirilen ekin homoloji kolu alt bölümlerini büyütmek için al koloni PCR. Kenar altındaki plazmid dizisine bir primer tavlama ve direnç kasetinin kenarına tamamlayıcı ikinci bir primer kullanın ( Şekil 1B , sıra hem Neo hem de Hygro eklerinde ortaktır), yaklaşık 200 bp daha uzun bir montaja bağlı ürünü üretir Içerilen homoloji kolu:
PUC19-Bsal-Left: 5 'GGCTCGTATGTTGTGTGGAATTGTGAG
Direnç-Sol: 5 'AAAAGCGCCTCCCCTACCC
PUC19-BsaI-Sağ: 5 'GCTATTACGCCAGCTGGCGAAA
Direnç-Sağ: 5 'AAGACAATAGCAGGCATGCTGGG- Steril kürdan ile bakteri kolonileri seçin ve 20 μL su bakterileri askıya al. 95 ° C'de 15 dakika ısıtın ve bir masa santrifüjünde 5 dakika süreyle maksimum hızda aşağı doğru döndürün. Hemen buzda bekletin.
- Aşağıdaki PCR karışımını hazırlayın 31 </ Sup>:
Seyreltik koloni şablonu 1.25 μL 10x Taq reaksiyon tamponu 1.25 μL 20 mM dNTP'ler 0.25 uL 10 uM İleri primer 0.25 uL 10 uM Geri astar 0.25 uL Taq Polimeraz 0.25 uL Su 9 uL - Aşağıdaki termal siklör parametrelerini kullanın: 30 saniye süreyle 95 ° C, (95 ° C 30 saniye, 30 saniye için 61 ° C, 1 dakika / kb için 72 ° C) 25 devir için tekrarlayın, 72 ° C'de 2 dakika boyunca tekrarlayın.
- Agaroz jel elektroforezi ile doğru amplikon boyutu için PCR ürününü kontrol edin.
- İsteğe bağlı olarak, doğru ekleme boyutu ve sırası ile bireysel kolonilerden miniprep plazmid DNAE homoloji kol bölgeleri yukarıda adı geçen amplifikasyon primerleri ile Sanger dizilişiyle.
4. CRISPR bileşenlerinin kültürlenmiş hücrelere transferi
- Transfeksiyondan önce: 37 ° C'de ve% 5 C02'de% 10 FBS ile takviye edilmiş DMEM ortamında T-REx293 hücrelerini kültürleyin.
- 6-kuyucuklu plakalara hücreleri plaklayın ve yaklaşık% 70 konfluansa kadar büyür. Transfekte edilmemiş bir kontrol için bir kuyu ekleyin.
- Ticari bir transfeksiyon protokolü kullanarak 2.5 ug toplam plazmid ile transfekte hücreler. Buna paralel olarak, transfekte edilmemiş kontrolü muhafaza edin.
NOT: Transfeksiyon yöntemi ve verimliliği hücre tipine göre değişir. Deneyden önce sistem için uygun transfeksiyon yöntemini belirleyin.- Hücrelerin seçimle transfekte edilmesi için 0.75 μg Cas9-sgRNA 1 (sol kesim), 0.75 μg Cas9-sgRNA 2 (sağ kesim) ve 1.0 μg homolog rekombinasyon şablonu kullanın.
- Transfecti içinSeçim yapılmadan hücrelerin açık olmasını istiyorsanız, 2.5 μg Cas9-sgRNA plazmid kullanın.
5. İlaç Seçimi
- Transfeksiyondan 48 saat sonra hücreleri uygun ilaçla tedavi edin (Neomisin / Hygromycin). Transfekte edilmemiş kontrol hücresindeki tüm hücreler ölünceye kadar seçim yapın (genellikle Neo için 3-5 gün, Hygro için 7-14 gün).
NOT: T-REx293 hücreleri için Neomisin ve Hygromycin için sırasıyla 500 μg / mL ve 10 μg / mL konsantrasyonları kullanın. Diğer hücre türleri için etkin konsantrasyonu belirlemek için transfekte edilmemiş hücreler üzerinde ilacın önceden titrasyonunun yapılması yararlı olabilir.
6. Klon Popülasyonlarının İzolasyonu
- Seçildikten sonra orijinal kuyudaki hücreleri% 100 konfluansa kadar büyütün. Kuyu başına 0.33 hücre yoğunluğunda 96 oyuklu plakalara hücreler tohumlandı.
NOT: Üç 96 yuvalı plakanın tohumlanması, birden fazla doğru klonun izolasyonunu sağlamak için iyi bir başlangıç noktasıdır, ancak daha fazla ya da daha azına ihtiyaç duyulabilir.SgRNA ve HDR verimliliklerine son verir. - Koloniler gözle görünene kadar, 2-4 hafta süreyle kolonilere dikkat edin. Steril bir pipet ucu ile görünür koloniler seçin ve tek katmanlı büyümeyi teşvik etmek için yeni kuyularda yeniden sıkıştırın. Süspansiyon hücrelerini kullanırken, tek koloni kuyularının daha büyük bir kısmını sağlamak için daha düşük ekim yoğunlukları kullanılabilir.
7. Tarama Adayları
- Seçim kullanmadan adayları tarama: nokta leke
- Bireysel klonları% 50-100 konfluansa yükseltin. Kuyu içinde pipetleme yaparak hücrelerin tek katmanını çıkarın.
- Temiz bir mikrosantrifüj tüpüne 100 uL'lik toplam hacmden 90 μL'ye bölün. 5 dakika boyunca 6000 rpm'de aşağı döndürün, ortamı alın ve 1 x Lysis Tamponu veya 1x SDS yükleme tamponu (5x: 250 mM Tris-Cl pH 6.8,% 8 SDS,% 0.1 bromofenol mavisi,% 40'lık 10 uL'lik hücre pelletini lize edin. Gliserol, 100 mM DTT). Wel'de hücrelerin geri kalanına 90 uL yeni medya ekleyinYayılmaya devam edeceğim.
- Bir nokta oluşturmak için kuru nitroselüloz zar üzerine 1 μL hücre lizatı pipetleyin. Her örneği iki ayrı zar üzerinde iki kez lekeleyerek iki aynı örnek örneği oluşturun.
- TBST (Tris Tamponlu Tuz +% 0.01 Tween 20: 8 g NaCl, 0.2 g KCI, 3 g Tris baz, 1 L damıtılmış suya kadar, pH 7.4) içinde% 5 sütteki membranları oda sıcaklığında 1 saat bloke edin Sallama, burada ve prosedür boyunca).
- Bir membranda hedef proteine karşı birincil antikor ve bir kontrol proteini için birincil antikor (tubulin, GAPDH veya değişmesi beklenmeyen diğer proteinler) kullanılarak leke. TBST +% 5 sütte önerilen primer antikor seyreltmesini (ELAVL1 için 0.2 μg / mL ve bu durumda Pum2 için 0.1 μg / mL) kullanın. 1 saat oda sıcaklığında inkübe edin.
- TBST ile 3 kez 15 dakika yıkayın.
- Her membranı T'de uygun HRP ile konjüge edilmiş sekonder antikorla inkübe edinBST + oda sıcaklığında 1 saat boyunca% 5 süt.
- TBST ile 3 kez 15 dakika yıkayın.
- Üreticinin talimatlarını izleyerek kemilüminesan substrat çözeltisi uygulayın (Malzeme Tablosuna bakın) ve blotlanmış membranı bir dijital kemilüminesans görüntüleyicide görüntüleyin.
- Uygun yazılım kullanarak hedef ve kontrol proteinleri için nokta yoğunluklarını nicelleştirin.
- Düşük kontrol protein sinyali ile adayları yok edin ve geri kalan adaylar için protein yoğunluklarını kontrol etmek için hedefin arka plandan çıkarılmış oranlarını hesaplayın. Geçiş için en düşük oranlara sahip adayları seçin ve batı leke ile daha fazla doğrulama yapın.
- Seçim kullanılarak tarama adayları: koloni PCR
- Bir DNA hazırlama kiti kullanarak hücre lizatı toplayın (Bkz. Malzeme Tablosu ).
- Tek tek kolonileri yeni bir 96 kuyucukta çoğaltın ve% 100 konfluene kadar büyür.
- Bir grup klondan ortam çıkarın,Kitten 30 μL ekstraksiyon tamponu içindeki hücreleri tekrar süspansiyon haline getirin. Temiz bir 1.5 mL'lik mikrosantrifüj tüpüne aktarın.
- Çözeltiyi 96 ° C'ye 15 dakika ısıtın ve oda sıcaklığına soğumasını bekleyin.
- Kitten 30 μL Stabilizasyon tamponu ekleyin. İyice karıştırın.
- Yabani tip ve monoelale / bialelik mutant hatlarını koloni PCR ile tanımlayın.
- Direnç kasetinin başarılı veya başarısız entegrasyonuna dayalı olarak homoloji kollarının etrafındaki bölgeleri büyütmek için iki ayrı PCR primeri seti (hedeflenen lokusun sol ve sağ tarafı için) tasarlayın ( Şekil 2C ). Her iki tarafta, homoloji kolunun dışında tavlanmış ortak bir primer (kırmızı) kullanın. Vahşi tipli alleli test etmek için homoloji bölgesini (mavi) kapsayan endojen diziyi tamamlayan karşılık gelen eşleştirilmiş bir primerle kullanın. Takılan direnç kasetini tamamlayan başka bir çift astar kullanın (bkz. Bölümlerdeki primerler2.3), arzu edilen mutasyonu test etmek için.
- (Önerilir) Başlangıçtaki seçilmiş toplu hücre popülasyonundan hücre lisatı kullanılarak primer setlerini doğrulayın, çünkü hem WT hem de mutant allellerin bir karışımını içerecektir ( Şekil 2D ).
- Aşağıdaki reaksiyon karışımını hazırlayın:
Hücre lisatı 0.5 uL 10x KOD tampon 1.25 μL 25 mM MgS04 0,75 μL 2 mM dNTP'ler 1.25 μL 10 uM İleri primer 0.375 uL 10 uM Geri astar 0.375 uL KOD polimeraz 0.25 uL Su 7,75 μL - Aşağıdaki termal çevrimci koşullarını kullanın: 95 ° C'de 2 dakika, (95 ° C'de 20 saniye, Primer Tm'de 10 saniye, 70 ° C'de 20 s / kb) tekrarlama.
- PCR ürününü agaroz jel elektroforezi ile görselleştirin.
- Tahmini entegrasyonun her iki tarafı için koloni PCR testini tekrarlayın. Silinmiş bölge için entegrasyonun varlığını ve içsel sıra ürününü göstermeyen adayları seçin ve genişletin.
- Pozitif adayları western blot ve / veya RT-qPCR ile doğrulayın.
- Bir DNA hazırlama kiti kullanarak hücre lizatı toplayın (Bkz. Malzeme Tablosu ).
8. Sırayla Sıralama Genomik Mutasyonunu Doğrulayın
- Genomik DNA'yı mutant hücre çizgilerinden fenol kloroform ekstraksiyonu ile izole edin 31 .
- PCR, her ucuna BsaI kısıtlama siteleri ekleyen 5 'çıkıntılı primerler kullanılarak sgRNA hedef bölgesini çoğaltılır.
NOT: HDR ile düzenlenmiş klonlar için, her iki allel de aynı olabilir, PCR ürünü doğrudan dizilenebilir. - Golden Gate klonlama ile güçlendirilmiş bölgeyi pUC19-BsaI'ye klonlayın.
- Reaksiyon karışımı, yetkili bir E. coli suşuna dönüştürülür.
- 6-10 bireysel koloniden plazmid DNA'yı miniiprep edin ve Sanger dizilemesi ile klonlanmış bölgeyi dizinleyin.
Sonuçlar
ELAVL1 nakavt hatlarının üretilmesi için sağlam bir antikor mevcuttu, bu nedenle tek sgRNA'lar ( Şekil 1A , solda) kullanılarak düzenleme, ardından nokta immünoblot uygulandı. Elde edilen klonlarda verimlilikleri karşılaştırmak ve hedef etkileri dışarıda bırakmak için bağımsız olarak üç sgRNA transfekte edildi. Hücre lizatlarını, iki nitroselüloz zar üzerine klonal popülasyondan toplayıp blotladıktan sonra lekeler hem ELA...
Tartışmalar
CRISPR / Cas9 sistemi, diğer geçici manipülasyon yöntemlerine daha tutarlı bir alternatif sağlayan stabil genomik modifikasyonların verimli bir şekilde oluşturulmasına izin vermiştir. Burada, memeli hücre hatlarında CRISPR / Cas9 gen nakavtın hızlı tanımlanması için iki yöntem sunduk. Her iki yöntem de çok az hücresel malzeme gerektirir, bu nedenle test, klonal kültüre ait erken safhalarda yapılabilir, zamandan ve reaktiflerden tasarruf edilebilir. Her iki yöntemin verimliliğini artırmak iç...
Açıklamalar
Yazarlar, rekabet eden mali çıkarlarının olmadığını beyan ettiler.
Teşekkürler
Yazarlar, deneysel yardım için Gissell Sanchez, Megan Lee ve Jason Estep'i ve reaktifleri paylaşmak için Weifeng Gu ve Xuemei Chen'i onaylamak istiyorlar.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
Referanslar
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır