JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
포유류 세포에서 CRISPR / Cas9 매개 유전자 knockouts의 선택 의존 및 독립적 생성
요약
체세포를 유 전적으로 조작하는 능력의 최근 진보는 기본 및 응용 연구에 큰 가능성을 안겨줍니다. 여기, 우리는 선택 마커의 사용 여부에 관계없이 포유류 세포주에서 CRISP / Cas9 생성 녹아웃 생산 및 스크리닝을위한 두 가지 접근법을 제시한다.
초록
CRISPR / Cas9 게놈 공학 시스템은 적은 노력으로 정확한 게놈 편집을 가능하게함으로써 생물학에 혁명을 일으켰습니다. 특이성을 부여하는 단일 가이드 RNA (sgRNA)에 의해 유도 된 Cas9 단백질은 표적 유전자좌에서 DNA 가닥 둘 모두를 절단합니다. DNA가 끊어지면 NHEJ (homologous end joining) 또는 homology directed repair (HDR)가 유발 될 수 있습니다. NHEJ는 프레임 변이 돌연변이를 유도하는 작은 결실 또는 삽입을 도입 할 수있는 반면, HDR은 더 크고 더 정확한 섭동을 허용한다. 여기, 우리는 다운 스트림 선택 / 스크리닝을위한 두 가지 옵션으로 설립 CRISPR / Cas9 방법을 결합하여 녹아웃 세포 라인을 생성하기위한 프로토콜을 제시합니다. NHEJ 접근법은 하나의 sgRNA 절단 사이트와 선택에 의존하지 않는 스크리닝을 사용하며, 단백질 생산은 도트 면역 블롯 (dot immunoblot)으로 높은 처리량 방식으로 평가됩니다. HDR 접근법은 관심있는 유전자를 스팬하는 2 개의 sgRNA 절단 부위를 사용합니다. 제공된 HDR 템플릿과 함께이 메소드는 삭제를 수행 할 수 있습니다.삽입 된 선택 저항 마커의 도움을 받아 수십 kb의 적절한 방법과 각 방법의 장점에 대해 설명합니다.
서문
안정한 유전 적 변형은 일시적인 세포 섭동 (perturbation)의 방법보다 이점을 제공하는데, 이는 효율성과 지속 기간이 가변적 일 수 있습니다. 게놈 편집은 징크 핑거 핵산 분해 효소 1 , 2 , 3 , 4 , 5 , 전사 활성제 유사 이펙터 핵산 효소 (TALENs) 6 , 7 , 8 , 9 와 같은 표적 특이 적 뉴 클레아 제의 개발로 인해 최근에 점점 더 보편화되고있다. (CRISPR) 시스템 10 에서 유래 된 RNA 유도 뉴 클레아 제를 포함한다.
CRISPR / Cas9 편집 기계는 박테리아와 고세균이 바이러스 감염을 막기 위해 사용하는 면역계로부터 변형되었습니다.ass = "xref"> 11 , 12 , 13 . 이 과정에서, 침입 바이러스 서열의 짧은 20-30nt 단편이 반복 단위 14 , 15 에 의해 둘러싸인 "스페이서"로서 게놈 궤적에 편입된다. 후속 전사 및 RNA 프로세싱은 trans-activating crRNA 17 (tracrRNA)과 함께 작동 자 Cas9 엔도 뉴 클레아 제와 함께 조립되는 작은 CRISPR- 관련 RNAs 16 (crRNA)을 생성한다. 따라서 crRNA는 Cas9 표적화에 대한 특이성을 제공하고, 상보적인 바이러스 DNA 서열을 절단하고 추가 감염을 예방하기 위해 복합체를 유도한다 18 , 19 . 표적 DNA에있는 어떤 "protospacer"서열이라도 짧은 protospacer 인접 모티프 (PAM), S. pyogenes Casg의 경우 NGG에 직접 5 '있는 한, crRNA의 원천이 될 수있다 20. 숙주의 CRISPR 궤적의 스페이서 근처에 PAM 서열이 없다는 것은 자기와 비 - 자기를 구별하여 숙주의 표적화를 방지한다. 이 생물학적 시스템은 보편성과 유연성으로 인해 거의 모든 PAM 인접 DNA 사이트를 타겟팅 할 수 있도록 게놈 편집에 강력하게 적응했습니다. 이 버전에서는 추가 변경으로 crRNA와 tracrRNA를 Cas9 단백질 21 에로드되는 단일 가이드 RNA (sgRNA) 성분으로 융합시켰다.
진핵 세포에서 Cas9와 sgRNA의 발현시, Cas9 단백질은 표적 유전자좌에서 두 DNA 가닥을 절단한다. 상 동성 서열의 적당한 영역이없는 경우, 세포는 전형적으로 작은 결실 또는 드물게 삽입을 유도하는 비 - 상 동성 말단 결합 (NHEJ) 22 , 23 , 24 를 통해 이러한 단절을 고정시킨다. 공개를 타겟팅 할 때판독 프레임에서, 수리는 비 기능성 단백질 생성물을 생성하는 번역적인 골격 이동을 유발할 수있다. 대조적으로, 큰 상 동성 영역을 갖는 외인성 템플레이트가 제공 될 때, 세포는 상 동성 지시에 의한 보수에 의해 이중 가닥 절단을 고칠 수있다 25 , 26 . 이 경로는 excisable 선택 마커 27 도입과 함께 게놈에서 더 큰 정확한 삭제, 교체 또는 삽입을 허용합니다.
여기, 우리는 두 가지 CRISPR / Cas9 방법 ( 그림 1A ) 중 하나에 의해 녹아웃 세포 라인을 생성하기위한 프로토콜을 제시합니다. NHEJ 접근법은 단일 sgRNA 절단 사이트 및 선발 독립 스크리닝을 사용하므로 초기 준비가 거의 필요하지 않습니다. 이 방법을 사용할 때 녹아웃을 생성 할 가능성이 가장 높은 전사 인자의 5 '말단 근처에있는 엑손과 상보적인 RNA를 고안해야합니다. 수정 이후이 경우 게놈에 대한 이온은 작으며, 녹아웃 클론을 스크리닝하는 것은 도트 블롯을 기반으로하며, 여기서 단백질 생산물은 고효율 방식으로 평가됩니다. 우리는 ELAV와 유사한 1 단백질 (ELAVL1) 녹아웃 라인을 예로 들어 사용합니다. 두 번째 접근법은 homology directed repair (HDR)에 의존하고 관심있는 유전자 또는 관심 영역에 걸친 2 개의 sgRNA 절단 부위를 사용하여 수십 kb의 결실을 허용합니다. 절단 부위의 측면에 위치하는 두 개의 상 동성 영역을 갖는 플라스미드는 녹아웃 생성의 효율을 증가시키는 선택 가능한 저항 마커를 도입하는 대체 주형 ( 도 1b )을 제공한다. 이 방법은 적절하게 설계된 상 동성 암으로 유전자 변형을 도입하는 데에도 적용될 수있다. 이 경우 새로운 DNA 단편을 통합하면 PCR 기반의 스크리닝이 가능합니다 ( 그림 1C ). 여기에서는 Pumilio RNA binding family member 2 (PUM2) knockout line의 생성을 예로 사용합니다.
프로토콜
1. 바람직한 제거 주위의 상 동성 영역의 동정
참고 : 선택 기반 편집을 사용하는 경우에만 필요합니다.
- HDR 템플릿 ( 그림 1A )에서 상 동성 암 역할을 원하는 원하는 deletecus의 양쪽에 두 개의 영역, 처음 1.5-2 킬로바이트를 선택합니다. 복제를 용이하게하기 위해 두 가닥 (BsGI)에서 BsaI 인식 부위가 부족한 부위를 확인하십시오 (GGTCTC). BsaI 사이트가 불가피한 경우, 다른 유형 IIs 제한 효소 (BsmBI, SapI, BbsI)를 사용하여 수용자 플라스미드 (pUC19-BsaI), 저항 마커 기증자 플라스미드 (pGolden-Neo 또는 pGolden-Hygro) 상 동성 암 PCR 제품 오버행.
2. Cas9-sgRNA 발현 플라스미드의 생성
- 돌연변이 유발을위한 표적 사이트를 정의하십시오. 단일 절단, 선택없는 방법의 경우, 비 - 후 (non-fu)의 확률을 증가시키기 위해 5 '근위 코딩 엑손을 표적으로한다nctional 돌연변이. 두 컷 방법의 경우, 필요에 따라 많은 유전자를 포함하는 두 개의 사이트를 선택하십시오.
참고 : 우리의 경험에 따르면 최대 53kb의 삭제가 효율적으로 만들어집니다. - crispr.mit.edu 디자인 툴에 타겟 지역 시퀀스 200 ~ 300 bp를 입력합니다. 선택 기반 복제의 경우, deletion locus ( 그림 1A )의 근위에있는 확인 된 상동 성 영역의 200-300 bp를 사용하십시오. 타겟 지역별로 가장 높은 순위의 sgRNA를 2-3 가지 선택하여 활동 차이를 고려하고 동일한 잠재적 타겟 외 효과를 공유하지 않는 독립 클론을 격리하십시오.
- 발현 플라스미드에 삽입하기 위해 적절한 오버행을 갖는 이중 가닥 올리고 뉴클레오티드를 디자인하기 위해, 말단 PAM 서열 (NGG)을 생략하고 5'-CACC 오버행을 첨부 한 다음 G를 맨 위 가닥 oligo에 첨부한다. 하단 strand oligo의 경우, reverse-complemented 표적 서열에 5'-AAAC 돌출부를 추가하고,이어서 3'-C를 첨부한다.아래 lustrated :
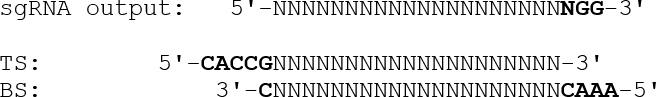
- Zhang 랩 프로토콜 29 에 설명 된대로 골든 게이트 클로닝을 사용하여 pSpCas9 (BB) 플라스미드에 sgRNA에 해당하는 합성 올리고 뉴클레오티드를 삽입합니다.
3. Homology-directed 수리 템플릿 Plasmids의 생성
참고 : 선택 기반 편집을 사용하는 경우에만 필요합니다. 상 동성 지시 한 수선 템플렛 플라스미드는 2 개의 sgRNA 표적 위치의 바로 저쪽에 게놈에 상보적인 2 개의 지역에 의해 측면에 마약 저항 카세트로 이루어져있다 ( 그림 1B ).
- 1.1 단계에서 확인한 더 넓은 상 동성 영역에서 상 동성 암 ( 그림 1A )으로 사용하기 위해 설계된 Cas9 절단 부위로부터 800-1,000 bp 26을 5 ~ 10 bp 이상 멀리 선택하십시오. sgRNA가 포함되지 않도록하십시오.표적 부위와 그 PAM을 상 동성 암에 삽입하면 이것이 플라스미드 자체의 CRISPR / Cas9 분열을 일으킬 것이다. 상 동성 암이 내부 BsaI 부위를 포함한다면 대체 sgRNA 부위를 사용하십시오.
- PCR은 BsaI 사이트 (GGTCTC)와 상 동체 영역의 양쪽 끝 부분에 독특한 오버행을 도입하는 추가 5 '서열을 갖는 순방향 및 역방향 프라이머를 사용하여 제조사의 지침에 따라 고 충실도 DNA 중합 효소를 사용하여 게놈 DNA로부터 상 동성 암을 증폭시킨다. 오버행은 올바른 조립 순서와 방향을 생성하기 위해 아래에 설명 된대로 적절한 순서를 포함해야합니다 ( 그림 1B ).
좌측 상 동성 암 FP 돌출부 : 5 'GGGTCTCA GGCC
왼쪽 상 동성 암 RP 돌출부 : 5 'GGGTCTCT CACG
오른쪽 상 동성 암 FP 돌출부 : 5 'GGGTCTCA GTCC
오른쪽 상 동성 암 RP 돌출부 : 5 'GGGTCTCT CCAC - 적절한 amplif에 대한 상 동성 암 검사앞으로 진행하기 전에 겔 전기 영동을 통한 배양.
- 골든 게이트 클로닝 28 에 의해 저항 카세트와 오른쪽 및 왼쪽 상 동성 암 지역을 HDR 템플릿 플라스미드로 조립. pGolden - Neo 또는 pGolden - Hygro plasmids 30 을 loxP- 측면 저항 카세트 (loxP-PGK-Neo-pA-loxP 또는 loxP-PGK-Hyg-pA-loxP)의 공급원으로 사용하십시오. 다중 클로닝 영역에서 BsaI 사이트를 갖는 변형 된 pUC19 인 pUC19-BsaI를 사용하고, 다른 곳의 BsaI 사이트를 수령 벡터 (요청시 이용 가능)로서 제거 하였다. 3 개의 인서트 및 벡터에 대해 1 : 1 : 1 : 1의 비율을 사용하십시오.
- 다음 반응 혼합물 30을 조제한다 :
오른쪽 상 동성 암 PCR 제품 0.06 pmol (30-40ng) 왼쪽 상 동성 암 PCR 제품 0.06 pmol (30-40ng) pGolden-Neo 플라스미드 (100 ng / μL) 1 &# 181; L pUC19-BsaI 플라스미드 (100ng / ㎕) 1 μL 2x T7 DNA 리가 아제 완충액 5 μL BsaI (10U / μL) 0.75 μL T7 DNA 리가 제 (3000 U / μL) 0.25 μL 물 최대 10 μL - 다음 thermocycler 매개 변수를 사용하십시오 : 37 ° C 5 분, 20 ° C 5 분. 30 사이클을 반복합니다.
- 나머지 선형화 된 DNA를 소화하기 위해 제조업체의 지침에 따라 exonuclease로 클로닝 제품을 처리하십시오.
- 세포와 함께 제공된 프로토콜에 따라 반응 혼합물을 유능한 E. coli 균주로 변형시키고 암피실린 함유 접시에 플레이트를 만듭니다.
- 다음 반응 혼합물 30을 조제한다 :
- 올바른 템플릿 어셈블리로 클론을 식별하려면 박테리아를 수행하십시오(전체 인서트가 너무 커서 증폭을 증폭시킬 수 없기 때문에) 조립 된 인서트의 상 동성 암 부분 영역을 증폭시키기위한 al colony PCR. 인접한 플라스미드 서열에 하나의 프라이머를 사용하고 저항 카세트의 가장자리에 상보적인 두 번째 프라이머 ( 그림 1B , 서열은 Neo와 Hygro inserts 모두 공통이다)를 사용하여 약 200 bp보다 긴 어셈블리 의존성 생성물을 생성한다 포함 된 상 동성 암 :
pUC19-Bsal- 좌측 : 5 'GGCTCGTATGTTGTGTGGAATTGTGAG
저항 - 왼쪽 : 5 'AAAAGCGCCTCCCCTACCC
pUC19-BsaI- 오른쪽 : 5' GCTATTACGCCAGCTGGCGAAA
저항 - 오른쪽 : 5 'AAGACAATAGCAGGCATGCTGGG- 멸균 된 이쑤시개로 세균성 콜로니를 선택하고 물 20 μL로 박테리아를 정지시킵니다. 95 ° C에서 15 분간 가열하고 탁상 원심 분리기에서 최대 속도로 5 분간 스핀 다운합니다. 즉시 얼음 위에 놓으십시오.
- 다음 PCR 혼합물을 준비하십시오. 31 </ sup> :
희석 식민지 템플릿 1.25 μL 10x Taq 반응 완충액 1.25 μL 20mM dNTPs 0.25 μL 10 μM 포워드 프라이머 0.25 μL 10 μM Reverse primer 0.25 μL Taq 폴리 메라 이제 0.25 μL 물 9 μL - 다음 thermocycler 매개 변수를 사용하십시오 : 95 ° C 30 초, (95 ° C 30 초, 61 ° C 30 초, 72 ° C 1 분 / kb) 25 ℃, 2 분간 72 ℃에서 반복하십시오.
- 아가 로스 겔 전기 영동으로 정확한 amplicon 크기에 대한 PCR 제품을 확인하십시오.
- 선택적으로, 정확한 삽입 크기 및 순서를 갖는 개별 콜로니로부터의 miniprep 플라스미드 DNAe 상 동체 암 부위를 증폭시켰다.
4. 배양 세포로의 CRISPR 성분의 트랜 스펙 션
- transfection하기 전에 : 37 ° C와 5 % CO 2 에서 10 % FBS로 보충 된 DMEM 배지에서 배양 T-REx293 세포.
- 6 잘 접시에 플레이트 세포와 70 % confluency로 성장. 트랜 스펙 션되지 않은 컨트롤을위한 웰을 포함하십시오.
- 상업 transfection 프로토콜을 사용하여 2.5 μg 총 플라스미드와 세포를 Transfect. 동시에, 트랜스 팩트되지 않은 컨트롤을 유지하십시오.
참고 : 형질 전환 방법 및 효율은 세포 유형에 따라 다릅니다. 실험 전에 시스템에 대한 적절한 형질 전환 방법을 결정하십시오.- 선택으로 세포 transfection, 0.75 μg Cas9 - sgRNA 1 (왼쪽 컷), 0.75 μg Cas9 - sgRNA 2 (오른쪽 컷), 1.0 μg 상동 재조합 템플릿을 사용합니다.
- transfecti의 경우선택하지 않고 세포에 2.5 μg Cas9-sgRNA 플라스미드를 사용한다.
5. 마약 선택
- transfection 후 48 시간, 적절한 약물 (Neomycin / Hygromycin)으로 세포를 치료하십시오. untransfected 컨트롤 (일반적으로 3-5 일 Neo, Hygro에 대한 7-14 일)에 죽을 때까지 선택을 수행하십시오.
참고 : T-REx293 세포의 경우 Neomycin과 Hygromycin에는 각각 500 μg / mL와 10 μg / mL의 농도를 사용하십시오. 다른 세포 유형의 경우, 효과적인 농도를 결정하기 위해 감염되지 않은 세포에서 약물의 사전 적정을 수행하는 것이 유용 할 수 있습니다.
6. 클론 개체의 분리
- 선택 후 원래의 우물에서 세포를 100 % confluency로 성장시킵니다. 세포를 96 웰 플레이트에 0.33 세포 / 웰의 밀도로 접종한다.
참고 : 세 개의 96 웰 플레이트에 시드를하면 올바른 클론을 하나 이상 분리 할 수있는 좋은 출발점이 될 수 있지만 더 많거나 적게 필요할 수 있습니다. depsgRNA 및 HDR 효율성이 끝납니다. - 식민지가 눈에 보일 때까지 2-4 주에 걸쳐 식민지를 관찰하십시오. 멸균 피펫 팁으로 보이는 식민지를 선택하고 monolayer 성장을 장려하기 위해 새로운 우물에 reseed. 서스펜션 세포를 사용할 때 더 낮은 배양 밀도를 사용하여 단일 콜로니 우물의보다 많은 부분을 확보 할 수 있습니다.
7. 상영 후보자
- 선택을 사용하지 않고 선별 후보 : 도트 블롯
- 개별 클론을 50-100 %의 confluency로 성장시킵니다. 웰 내에서 피펫 팅하여 세포의 단일 층을 제거하십시오.
- 깨끗한 마이크로 원심 분리 튜브에 총 부피 100 μL 중 분취 량 90 μL. 5 분 동안 6,000 rpm으로 스핀 다운하고, 배지를 제거하고, 1X Lysis Buffer 또는 1x SDS loading buffer (5x : 250 mM Tris-Cl pH 6.8, 8 % SDS, 0.1 % bromophenol blue, 40 % 글리세롤, 100 mM DTT). 웰에있는 세포의 나머지에 새로운 미디어 90 μL를 추가하십시오계속 전파하십시오.
- 점을 형성하는 건조 nitrocellulose 막 위에 세포 lysate 1 μL를 피펫. 각 샘플을 두 개의 별도 멤브레인에서 두 번 블로 팅하여 두 개의 동일한 샘플 패턴을 만듭니다.
- 실온에서 TBST (트리스 완충 식염수 + 0.01 % 트윈 20 : 8g NaCl, 0.2g KCl, 3g 트리스 염기, 최대 1L 증류수, pH 7.4) 31 중 5 % 여기에서 그리고 절차 전반에 걸쳐서 흔들림).
- 하나의 막에있는 표적 단백질에 대한 1 차 항체와 대조 단백질 (튜브린, GAPDH 또는 다른 것으로 예상되는 다른 단백질)에 대한 1 차 항체를 사용하여 블로 팅합니다. TBST + 5 % 우유에서 1 차 항체 희석액 (ELAVL1은 0.2 μg / mL, Pum2는 0.1 μg / mL)을 사용하십시오. 실온에서 1 시간 동안 품어 낸다.
- 5 분 동안 TBST로 3 번 씻는다.
- T에서 각 막을 적절한 HRP- 접합 이차 항체와 함께 배양한다.BST + 실온에서 1 시간 동안 5 % 우유.
- 5 분 동안 TBST로 3 번 씻는다.
- 제조업체 지침에 따라 화학 발광 기질 용액 (재료 표 참조)을 적용하고 디지털 화학 발광 이미 저에서 블 ted된 막을 묘사하십시오.
- 적절한 소프트웨어를 사용하여 표적 단백질과 대조 단백질의 도트 농도를 정량화하십시오.
- 낮은 제어 단백질 신호로 후보 물질을 제거하고, 나머지 후보 물질에 대한 단백질 강도를 제어하기 위해 표적의 배경 - 뺀 비율을 계산하십시오. Western blot으로 통과 및 추가 확인을 위해 가장 낮은 비율의 후보를 선택하십시오.
- 선택의 사용을 가진 후보자를 검열하십시오 : 식민지 PCR
- DNA prep 키트를 사용하여 세포 용 해물을 수집하십시오 ( 재료 표 참조).
- 새로운 96 well에서 개별 콜로니를 복제하고 100 % confluency까지 성장시킵니다.
- 한 세트의 복제본에서 미디어를 제거합니다.키트에서 추출 버퍼 30 μL에 세포를 resuspend. 깨끗한 1.5 ML microcentrifuge 튜브로 전송하십시오.
- 용액을 96 ℃에서 15 분간 가열하고 실온으로 냉각시킨다.
- 키트에서 안정화 버퍼 30 μL를 추가합니다. 잘 섞다.
- 식민지 PCR에 의해 wildtype 및 monallelic / biallelic 돌연변이 라인을 식별합니다.
- 저항 카세트의 성공적인 또는 성공적이지 않은 통합 ( 그림 2C )에 기반한 상 동성 암 주위의 영역을 증폭시키기 위해 두 개의 개별 PCR 프라이머 세트 (표적 유전자좌의 왼쪽과 오른쪽에 대해)를 설계하십시오. 양쪽에는 상 동성 암 외부에 어닐링하는 공통 프라이머 (적색)를 사용하십시오. 야생형 대립 유전자를 테스트하기 위해 상 동성 영역 (파란색)에 걸친 내인성 서열에 상보적인 상응하는 쌍을 이루는 프라이머와 함께 사용하십시오. 삽입 된 저항 카세트와 상보적인 또 다른 한 쌍의 프라이머를 사용하십시오 (섹션의 프라이머 참조).2.3), 원하는 돌연변이를 시험한다.
- (권장) WT와 돌연변이 대립 유전자가 모두 포함되어 있기 때문에 원래 선택된 벌크 세포군의 세포 용 해물을 사용하여 프라이머 세트를 확인합니다 ( 그림 2D ).
- 다음 반응 혼합물을 준비하십시오 :
세포 용 해물 0.5 μL 10x KOD 버퍼 1.25 μL 25mM MgSO4 0.75 μL 2 mM dNTPs 1.25 μL 10 μM 포워드 프라이머 0.375 μL 10 μM Reverse primer 0.375 μL KOD 중합 효소 0.25 μL 물 7.75 μL - 다음 thermocycler 조건을 사용하십시오 : 95 ℃에서 2 분, (95 ℃에서 20 초, Primer Tm에서 10 초, 70 ℃에서 20 초 / kb) 25 사이클을 반복하십시오.
- 아가 로스 겔 전기 영동으로 PCR 산물을 시각화하십시오.
- 예측 통합의 각 측면에 대한 식민지 PCR 테스트를 반복합니다. 통합의 존재와 삭제 된 지역에 대한 내인성 서열 생성물이없는 후보 물질을 선택하고 확장하십시오.
- 웨스턴 블랏 및 / 또는 RT-qPCR로 양성 반응자를 확인합니다.
- DNA prep 키트를 사용하여 세포 용 해물을 수집하십시오 ( 재료 표 참조).
8. 시퀀싱으로 게놈 돌연변이 확인
- 페놀 클로로포름 추출 31 에 의해 돌연변이 세포주에서 게놈 DNA를 분리합니다.
- PCR은 각 말단에 BsaI 제한 부위를 부가하는 5 '오버행을 갖는 프라이머를 사용하여 sgRNA 표적 영역을 증폭시킨다.
참고 : 두 대립 유전자가 동일 할 수있는 HDR로 편집 한 클론의 경우 PCR 산물을 직접 시퀀싱 할 수 있습니다. - 골든 게이트 복제에 의해 증폭 된 영역을 pUC19-BsaI로 클로닝한다.
- 반응 혼합물을 유능한 대장균 균주로 변형시킨다.
- 6 ~ 10 개의 개별 콜로니에서 Miniprep 플라스미드 DNA를 추출하고 Sanger 시퀀싱으로 클로닝 된 영역을 시퀀싱합니다.
결과
ELAVL1 knockout line을 생성하기 위해 강력한 항체를 사용할 수 있었으므로 단일 sgRNA ( 그림 1A , 왼쪽)를 사용하여 편집 한 다음 도트 면역 블롯을 수행했습니다. 3 개의 sgRNA를 독립적으로 트랜 스펙 션하여 효율을 비교하고 결과 클론에서 표적 효과를 배제했습니다. clonal population으로부터 2 개의 nitrocellulose membrane으로 세포 용 해물을 채취하고 blotting 한 ?...
토론
CRISPR / Cas9 시스템은 다른 일시적인 조작 방법에 대한보다 일관된 대안을 제공하는 안정한 게놈 변형의 효율적인 생성을 가능하게했다. 여기, 우리는 포유 동물 세포 라인에서 CRISPR / Cas9 유전자 녹아웃의 신속한 식별을위한 두 가지 방법을 제시했습니다. 두 가지 방법 모두 세포질이 거의 필요하지 않으므로 클론 배양의 초기 단계에서 시험을 수행하여 시간과 시약을 절약 할 수 있습니다. 두 가?...
공개
저자는 경쟁적인 금전적 이해 관계가 없다고 선언합니다.
감사의 말
저자는 Gissell Sanchez, Megan Lee 및 Jason Estep을 실험 지원을 위해, Weifeng Gu와 Xuemei Chen은 시약 공유에 대해 인정하고 싶습니다.
자료
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
참고문헌
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유