Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Génération indépendante et dépendante de la sélection de CRISPR / Cas9-Meded Knockouts dans les cellules de mammifères
Dans cet article
Résumé
Les progrès récents dans la capacité de manipuler génétiquement les lignées cellulaires somatiques offrent un grand potentiel pour la recherche fondamentale et appliquée. Ici, nous présentons deux approches pour la production et le dépistage de knock-out créés par CRISPR / Cas9 dans des lignées cellulaires de mammifères, avec et sans utilisation de marqueurs sélectionnables.
Résumé
Le système d'ingénierie du génome CRISPR / Cas9 a révolutionné la biologie en permettant un montage précis du génome avec peu d'effort. Guidé par un seul ARN guide (sgRNA) qui confère une spécificité, la protéine Cas9 clive les deux brins d'ADN au locus ciblé. La rupture de l'ADN peut déclencher une jointure terminale non homologue (NHEJ) ou une réparation dirigée par homologie (HDR). NHEJ peut introduire de petites suppressions ou des insertions qui conduisent à des mutations par déplacement de trame, tandis que HDR permet des perturbations plus grandes et plus précises. Ici, nous présentons des protocoles pour générer des lignées cellulaires knock-out en couplant les méthodes CRISPR / Cas9 établies avec deux options pour la sélection / sélection en aval. L'approche NHEJ utilise un seul site de coupe de sgRNA et un dépistage indépendant de la sélection, où la production de protéines est évaluée par immunotransfert par points de manière à haut débit. L'approche HDR utilise deux sites de sgRNA coupés qui couvrent le gène d'intérêt. Ensemble avec un modèle HDR fourni, cette méthode peut éliminerDe dizaines de kb, aidé par le marqueur de résistance sélectionnable inséré. Les applications et avantages appropriés de chaque méthode sont discutés.
Introduction
Les modifications génétiques stables offrent un avantage sur les méthodes transitoires de perturbation cellulaire, qui peuvent être variables dans leur efficacité et leur durée. L'édition génomique est devenue de plus en plus courante ces dernières années en raison du développement de nucléases spécifiques cibles, telles que les nucléases 1 , 2 , 3 , 4 , 5 , les nucléases effectrices de l'activateur de la transcription (TALEN) 6 , 7 , 8 , 9 Et des nucléases guidées par l'ARN dérivées du système de répétitions palindromiques courtes (CRISPR) regroupées, régulièrement intercalées 10 .
La machine d'édition CRISPR / Cas9 est adaptée d'un système immunitaire que les bactéries et les archées utilisent pour se défendre contre les infections viralesAss = "xref"> 11 , 12 , 13 . Dans ce processus, des fragments courts de 20-30 nt de séquence virale envahissante sont incorporés dans un locus génomique comme des "entretoises" flanqués par des unités répétitives 14 , 15 . La transcription subséquente et le traitement de l'ARN génèrent de petits ARN 16 associés à CRISPR (crRNAs) qui, conjointement avec un CRRNA 17 trans-activant (tracrRNA), assemblent avec l'endonucléase Eff9 Cas9. Les crRNAs fournissent donc une spécificité au ciblage Cas9, guidant le complexe pour cliver des séquences d'ADN virales complémentaires et prévenir d'autres infections 18 , 19 . Toute séquence "protospacer" dans l'ADN ciblé peut servir de source de l'ARNrm, pourvu qu'elle soit directement à 5 'd'un motif adjacent de protospacer court (PAM), NGG dans le cas de S. pyogenes Cas9 20. L'absence de la séquence PAM près de l'espaceur dans le locus CRISPR de l'hôte se distingue entre soi-même et non-soi, empêchant le ciblage de l'hôte. En raison de son universalité et de sa flexibilité, ce système biologique a été puissamment adapté pour l'édition génomique, de telle sorte que presque n'importe quel site d'ADN adjacente au PAM peut être ciblé. Dans cette version, une autre modification a fusionné le crRNA et l'ARNt de trac en un seul composant guide RNA (sgRNA) qui est chargé dans la protéine Cas9 21 .
Lors de l'expression de Cas9 et d'un sgRNA dans des cellules eucaryotes, la protéine Cas9 clive les deux brins d'ADN au locus ciblé. En l'absence d'une région appropriée de séquence homologue, la cellule corrige cette rupture par l'intermédiaire d'une jointure finale non homologue (NHEJ) 22 , 23 , 24 , qui introduit typiquement de petites deletions ou, rarement, des insertions. Lorsque vous ciblez unCadre de lecture, la réparation entraîne probablement un changement de cadre translationnel qui produit un produit protéique non fonctionnel. En revanche, lorsqu'elle est munie d'un modèle exogène avec de grandes régions d'homologie, la cellule peut réparer la rupture à double brin par une réparation dirigée par homologie 25 , 26 . Cette voie permet des deletions, des remplacements ou des insertions plus importantes dans le génome, associées à l'introduction de marqueurs de sélection excisables 27 .
Ici, nous présentons des protocoles pour générer des lignées cellulaires knock-out par l'une ou l'autre de ces deux méthodes CRISPR / Cas9 ( Figure 1A ). L'approche NHEJ utilise un seul site de coupe de sgRNA et un dépistage indépendant de la sélection, et nécessite donc une préparation précieuse. Lors de l'utilisation de cette méthode, il convient de concevoir des ARN de guide complémentaires aux exons près de la fin de la transcription 5 ', qui sont les plus susceptibles de produire un knock-out. Depuis le modificatLes ions du génome dans ce cas sont petits, le dépistage des clones knock-out est basé sur des transferts de points, où le produit protéique est évalué à haut débit. Nous utilisons par exemple la génération de liaisons knock-out ELAV-like 1 (ELAVL1). La deuxième approche repose sur la réparation dirigée par homologie (HDR) et utilise deux sites de sgRNA coupés qui couvrent le gène ou la région d'intérêt, ce qui permet de supprimer des dizaines de kb. Un plasmide avec deux régions d'homologie qui flanquent les sites de clivage fournit un modèle de remplacement ( Figure 1B ), en introduisant un marqueur de résistance sélectionnable qui augmente l'efficacité de la génération de knock-out. Cette méthode peut également être adaptée pour introduire des modifications de gènes avec des bras d'homologie correctement conçus. Dans ce cas, l'intégration d'un nouveau fragment d'ADN permet un dépistage par PCR ( Figure 1C ). Ici, nous utilisons la génération des lignes knock-out 2 de la famille de liaison Pumilio RNA (PUM2) à titre d'exemple.
Protocole
1. Identification des régions d'homologie autour de la suppression désirée
REMARQUE: nécessaire uniquement si vous utilisez l'édition basée sur la sélection.
- Sélectionnez deux régions, initialement 1,5-2 kb, de chaque côté du locus de suppression désiré, qui serviront d'armes d'homologie dans le modèle HDR ( Figure 1A ). Identifiez les régions qui manquent de sites de reconnaissance BsaI sur l'un ou l'autre des deux (GGTCTC) pour faciliter le clonage. Si les sites BsaI sont inévitables, utilisez une enzyme de restriction de type II (BsmBI, SapI, BbsI) et modifiez les sites correspondants dans le plasmide récepteur (pUC19-BsaI), le plasmide donneur de marqueur de résistance (pGolden-Neo ou pGolden-Hygro) et Le bras de homologie produit des montants supérieurs.
2. Génération de plasmides d'expression Cas9-sgRNA
- Définissez le (s) site (s) de ciblage pour la mutagenèse. Pour la méthode à coupe unique, sans sélection, cibler les exons de codage proximaux 5 'pour augmenter la probabilité d'un non-fuMutation nationale. Pour la méthode à deux coups, sélectionnez deux sites qui couvrent la majeure partie du gène si nécessaire.
REMARQUE: dans notre expérience, les suppressions allant jusqu'à 53 kb sont créées efficacement. - Entre 200 à 300 pb de la séquence de région ciblée dans l'outil de conception crispr.mit.edu. Pour le clonage basé sur la sélection, utilisez 200-300 pb des régions d'homologie identifiées qui sont proximales au locus de suppression ( Figure 1A ). Sélectionnez 2-3 des sgRNAs les mieux classés par région ciblée pour tenir compte des différences dans leur activité et pour isoler des clones indépendants qui ne partagent pas les mêmes effets hors cible potentiels.
- Pour concevoir des oligonucléotides duplexés avec des surplombs appropriés pour insertion dans le plasmide d'expression, omettre la séquence de PAM de fin (NGG) et ajouter un surplomb de 5'-CACC suivi d'un G sur l'oligo de brin supérieur. Pour l'oligo de brin inférieur, ajoutez un surplomb de 5 '-AAAC à la séquence cible complémentaire inverse, suivi d'un 3'-C, car ilIllustré ci-dessous:
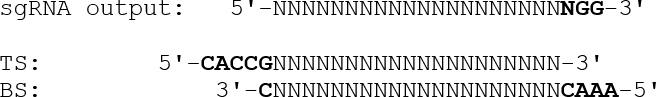
- Insérez des oligonucleotides synthétiques correspondant aux sgRNA dans le plasmide pSpCas9 (BB) en utilisant le clonage Golden Gate 28 comme décrit dans le protocole de laboratoire Zhang 29 .
3. Génération de plasmides de modèles de réparation dirigés par l'homologie
REMARQUE: nécessaire uniquement si vous utilisez l'édition basée sur la sélection. Le plasmide de modèle de réparation dirigé par homologie est constitué d'une cassette de résistance aux médicaments flanquée de deux régions qui sont complémentaires du génome juste à l'extérieur des deux sites cibles de sgRNA ( figure 1B ).
- À partir des régions d'homologie plus larges identifiées à l'étape 1.1, sélectionnez 800-1,000 bp 26 à plus de 5-10 pb des sites de coupe Cas9 conçus pour servir d'armes d'homologie ( figure 1A ). Assurez-vous de ne pas inclure le sgRNASite cible et son PAM dans les bras d'homologie, car cela provoquera le clivage CRISPR / Cas9 du plasmide modèle lui-même. Si les bras d'homologie contiennent des sites BsaI internes, utilisez d'autres sites d'sgRNA.
- La PCR amplifie les armes d'homologie à partir de l'ADN génomique à l'aide d'une ADN polymerase haute fidélité, selon les instructions du fabricant, en utilisant des amorces avant et arrière avec une séquence 5 'supplémentaire qui introduit des sites BsaI (GGTCTC) et des surplombs uniques sur l'une ou l'autre extrémité de la région homologique. Les surplombs doivent contenir des séquences appropriées pour générer un bon ordre et une bonne orientation de l'assemblage, comme décrit ci-dessous ( Figure 1B ):
Bras de homologie gauche FP overhang: 5 'GGGTCTCA GGCC
Arme d'homologie gauche bras de surtension: 5 'GGGTCTCT CACG
Bras d'homologie droit FP overhang: 5 'GGGTCTCA GTCC
Le bras droit de l'homologie est en surtension: 5 'GGGTCTCTC CCAC - Vérifiez les bras d'homologie pour un amplif appropriéPar électrophorèse sur gel avant de continuer.
- Assembler les régions de bras d'homologie droite et gauche avec la cassette de résistance dans un plasmide modèle HDR par le clonage Golden Gate 28 . Utilisez les plasmides pGolden-Neo ou pGolden-Hygro 30 comme source de cassettes de résistance flanées LoxP (loxP-PGK-Neo-pA-loxP ou loxP-PGK-Hyg-pA-loxP). Utilisez pUC19-BsaI, un pUC19 modifié avec des sites BsaI dans la région de clonage multiple et éliminé les sites BsaI ailleurs, en tant que vecteur récepteur (disponible sur demande). Utilisez un rapport de 1: 1: 1: 1 pour les trois inserts et le vecteur.
- Préparer le mélange réactionnel 30 suivant:
Produit de PCR droit de l'homologie 0,06 pmol (30-40ng) Produit de PCR d'armes d'homologie gauche 0,06 pmol (30-40ng) Plasmide pGolden-Neo (100 ng / μL) 1 &# 181; L PUC19-BsaI plasmide (100 ng / μL) 1 μL 2x T7 ADN ligase tampon 5 μL BsaI (10 U / μL) 0,75 μL T7 ADN ligase (3000 U / μL) 0,25 μL Eau Jusqu'à 10 μL - Utilisez les paramètres suivants du thermocycleur: 37 ° C pendant 5 min, 20 ° C pendant 5 min. Répétez pendant 30 cycles.
- Traiter le produit de clonage avec une exonucléase selon les instructions du fabricant pour digérer tout ADN linéarisé restant.
- Transforme le mélange réactionnel en une souche E. coli compétente selon le protocole fourni avec les cellules, et plaque sur des plats contenant de l'ampicilline.
- Préparer le mélange réactionnel 30 suivant:
- Pour identifier les clones avec l'assemblage de gabarit correct, effectuer des bactériesPCR de colonisation pour amplifier les sous-régions du bras d'homologie de l'insert assemblé (car l'insert entier peut être trop grand pour amplifier de manière fiable). Utiliser un recuit d'amorce à la séquence du plasmide flanquant et une seconde amorce complémentaire au bord de la cassette de résistance ( Figure 1B , la séquence est commune aux inserts Neo et Hygro), générant un produit dépendant de l'assemblage qui est d'environ 200 pb plus long que Le bras d'homologie contenu:
PUC19-Bsal-Left: 5 'GGCTCGTATGTTGTGTGGAATTGTGAG
Résistance à gauche: 5 'AAAAGCGCCTCCCCTACCC
PUC19-BsaI-Right: 5 'GCTATTACGCCAGCTGGCGAAA
Résistance-Droite: 5 'AAGACAATAGCAGGCATGCTGGG- Choisissez des colonies bactériennes avec des cure-dents stériles et suspendez les bactéries dans 20 μl d'eau. Faire chauffer à 95 ° C pendant 15 minutes et centrifuger dans une centrifugeuse de table à vitesse maximale pendant 5 min. Placez immédiatement sur la glace.
- Préparer le mélange PCR suivant 31 </ Sup>:
Modèle de colonie diluée 1,25 μL 10 x tampon de réaction Taq 1,25 μL DNTP de 20mM 0,25 μL Amorce directe de 10 μM 0,25 μL 10 μM Amorce inversée 0,25 μL Taq Polymerase 0,25 μL Eau 9 μL - Utilisez les paramètres suivants du thermocycleur: 95 ° C pendant 30 s, (95 ° C 30 s, 61 ° C pendant 30 s, 72 ° C pendant 1 minute / kb), répétez pendant 25 cycles, 72 ° C pendant 2 min.
- Vérifier le produit de PCR pour une taille d'amplicon correcte par électrophorèse sur gel d'agarose.
- Facultativement, l'ADN de plasmide miniprep provenant de colonies individuelles avec une taille d'insert correcte et une séquenceE les régions de bras d'homologie par séquençage de Sanger avec les amorces d'amplification mentionnées ci-dessus.
4. Transfection des composants CRISPR dans les cellules cultivées
- Avant la transfection: culturez les cellules T-REx293 dans du milieu DMEM complété par 10% de FBS à 37 ° C et 5% de CO 2 .
- Placer les cellules sur des plaques à 6 puits et atteindre jusqu'à environ 70% de confluence. Inclure un puits pour un contrôle non transfecté.
- Cellules transfectées avec 2,5 pg de plasmide total en utilisant un protocole de transfection commercial. En parallèle, maintenez le contrôle non transfecté.
REMARQUE: la méthode de transfection et l'efficacité varient en fonction du type de cellule. Déterminer la méthode de transfection appropriée pour le système avant l'expérience.- Pour la transfection de cellules avec sélection, utiliser 0,75 μg Cas9-sgRNA 1 (coupe gauche), 0,75 μg Cas9-sgRNA 2 (coupe droite) et 1,0 μg de modèle de recombinaison homologue.
- Pour transfectiSur les cellules sans sélection, utiliser un plasmide Cas9-sgRNA de 2,5 μg.
5. Sélection des médicaments
- 48 h après la transfection, traiter les cellules avec le médicament approprié (Neomycine / Hygromycine). Effectuez la sélection jusqu'à ce que toutes les cellules du contrôle non transfecté meurent (généralement 3-5 jours pour Neo, 7-14 jours pour Hygro).
REMARQUE: Utilisez des concentrations de 500 μg / mL et 10 μg / mL pour Neomycine et Hygromycine, respectivement pour les cellules T-REx293. Pour d'autres types de cellules, il peut être utile d'effectuer un titrage préalable de médicament sur des cellules non transfectées pour déterminer la concentration effective.
6. Isolation des populations clonales
- Augmenter les cellules à 100% de confluence dans le puits d'origine après sélection. Cellules de graines dans des plaques à 96 puits à une densité de 0,33 cellules par puits.
REMARQUE: L'ensemencement de trois plaques à 96 puits est un bon point de départ pour assurer l'isolement de plus d'un clone correct, mais plus ou moins peut-être nécessaire depSe terminant par l'efficacité du sgRNA et du HDR. - Observez les colonies pendant une période de 2-4 semaines, jusqu'à ce que les colonies soient visibles aux yeux. Choisir des colonies visibles avec une pointe de pipette stérile et réensemencer de nouveaux puits afin d'encourager la croissance monocouche. Lors de l'utilisation de cellules en suspension, des densités de semis encore plus faibles peuvent être utilisées pour assurer une plus grande proportion de puits de colonie unique.
7. Candidats au dépistage
- Dépistage des candidats sans sélection: dot blot
- Augmenter les clones individuels à 50-100% de confluence. Désinstaller la monocouche de cellules en pipettant dans le puits.
- Aliquoter 90 μL du volume total de 100 μL à un tube de microcentrifugeuse propre. Verser à 6000 tr / min pendant 5 min, enlever les milieux et lier le culot cellulaire dans 10 μL de 1x tampon de lyse ou 1x tampon de chargement SDS (5 fois: Tris-Cl 250 mM pH 6,8, SDS à 8%, bleu de bromophénol 0,1%, 40% Glycerol, 100 mM de DTT). Ajouter 90 μL de nouveaux médias au reste des cellules dans le bienvenueL pour continuer à se propager.
- Pipettez 1 μL de lysat cellulaire sur une membrane de nitrocellulose sèche pour former un point. Nettoyer chaque échantillon deux fois sur deux membranes séparées, en créant deux modèles d'échantillons identiques.
- Bloquer les membranes dans du lait à 5% dans TBST (Tris Buffered Saline + 0,01% Tween 20: 8 g de NaCl, 0,2 g de KCl, 3 g de base de Tris, jusqu'à 1 L d'eau distillée, pH 7,4) 31 pendant 1 h à température ambiante ( Avec le basculement, ici et tout au long de la procédure).
- Blot en utilisant l'anticorps primaire contre la protéine cible sur une membrane et l'anticorps primaire pour une protéine de contrôle (tubuline, GAPDH ou toute autre protéine qui ne devrait pas changer) d'autre part. Utilisez la dilution d'anticorps primaire recommandée (0,2 μg / mL pour ELAVL1 et 0,1 μg / mL pour Pum2 dans ce cas) dans TBST + 5% de lait. Incuber 1 h à température ambiante.
- Laver 3 fois avec TBST pendant 5 min.
- Incuber chaque membrane avec un anticorps secondaire conjugué HRP approprié dans TBST + 5% de lait pendant 1 h à température ambiante.
- Laver 3 fois avec TBST pendant 5 min.
- Appliquer une solution de substrat de chimioluminescence (voir la Table des matériaux) en suivant les instructions du fabricant et image de la membrane blotée sur un imageur de chimioluminescence numérique.
- Quantifier les intensités de points pour la cible et contrôler les protéines en utilisant le logiciel approprié.
- Éliminer les candidats avec un faible signal de protéines de contrôle, et calculer les ratios de cible de base de l'arrière-plan pour contrôler les intensités de protéines pour les candidats restants. Sélectionnez les candidats avec les taux les plus bas pour le passage et une validation ultérieure par western blot.
- Dépistage des candidats à l'aide de la sélection: PCR de la colonie
- Recueillir le lysat cellulaire à l' aide d'un kit de préparation d' ADN (voir la table des matériaux ).
- Dupliquer les colonies individuelles dans un nouveau 96 puits et augmenter jusqu'à 100% de confluence.
- Supprimez les médias d'un ensemble de clones, unEt résister à des cellules dans 30 μL de tampon d'extraction du kit. Transférer à un tube de microcentrifugeuse propre de 1,5 mL.
- Chauffer la solution à 96 ° C pendant 15 minutes et laisser refroidir à température ambiante.
- Ajouter 30 μL de tampon de stabilisation du kit. Bien mélanger.
- Identifiez les lignes mutantes de type sauvage et monoalonique / bialélique par PCR par colonie.
- Concevez deux ensembles distincts d'amorces de PCR (pour le côté gauche et le côté droit du locus ciblé) pour amplifier les régions autour des bras d'homologie en fonction de l'intégration réussie ou non réussie de la cassette de résistance ( Figure 2C ). De chaque côté, utilisez une amorce commune (rouge) qui recule à l'extérieur du bras d'homologie. Utilisez-le avec une amorce appariée correspondante qui est complémentaire de la séquence endogène, couvrant la région d'homologie (bleu), pour tester l'allèle de type sauvage. Utilisez une autre amorce appariée, complémentaire à la cassette de résistance insérée (voir les amorces dans la section2.3), pour tester la mutation souhaitée.
- (Recommandé) Validez les ensembles d'amorces en utilisant le lysat cellulaire à partir de la population de cellules en vrac sélectionnée d'origine, puisqu'il contiendra un mélange d'allèles WT et mutants ( Figure 2D ).
- Préparer le mélange réactionnel suivant:
Lysat cellulaire 0,5 μL 10x tampon KOD 1,25 μL 25 mM MgSO4 0,75 μL DNTP 2 mM 1,25 μL Amorce directe de 10 μM 0,375 μL 10 μM Amorce inversée 0,375 μL KOD polymerase 0,25 μL Eau 7,75 μL - Utilisez les conditions suivantes du thermocycleur: 95 ° C pendant 2 min (95 ° C pendant 20 s, Primer Tm pendant 10 s, 70 ° C pendant 20 s / kb) pendant 25 cycles.
- Visualiser le produit PCR par électrophorèse sur gel d'agarose.
- Répétez le test de PCR de colonie pour chaque côté de l'intégration prévue. Sélectionnez et développez les candidats qui présentent une présence d'intégration et aucun produit de séquence endogène pour la région supprimée.
- Valider les candidats positifs par western blot et / ou RT-qPCR.
- Recueillir le lysat cellulaire à l' aide d'un kit de préparation d' ADN (voir la table des matériaux ).
8. Vérifier la mutation génomique par séquençage
- Isoler l'ADN génomique des lignées cellulaires mutantes par extraction au phénol chloroforme 31 .
- La PCR amplifie la région cible de sgRNA, en utilisant des amorces avec des surplombs de 5 'qui ajoutent des sites de restriction BsaI à chaque extrémité.
NOTE: Pour les clones édités avec HDR, où les deux allèles peuvent être identiques, le produit de PCR peut être séquencé directement. - Cloner la région amplifiée dans pUC19-BsaI par le clonage Golden Gate.
- Transformer le mélange réactionnel en une souche E. coli compétente.
- ADN du plasmide Miniprep de 6 à 10 colonies individuelles et séquence de la région clonée par séquençage de Sanger.
Résultats
Pour la génération de lignes knock-out ELAVL1, un anticorps robuste était disponible, de sorte que l'édition utilisant des sgRNAs simples ( Figure 1A , à gauche) a été effectuée, suivie d'un immunoblot point. Trois sgRNAs ont été transfectés de manière indépendante pour comparer l'efficacité et éliminer les effets cibles dans les clones résultants. Après avoir recueilli et bloqué les lysats cellulaires à partir de populations c...
Discussion
Le système CRISPR / Cas9 a permis une génération efficace de modifications génomiques stables, qui fournissent une alternative plus cohérente aux autres méthodes de manipulation transitoires. Ici, nous avons présenté deux méthodes pour l'identification rapide des knockouts du gène CRISPR / Cas9 dans les lignées cellulaires de mammifères. Les deux méthodes nécessitent peu de matériel cellulaire, de sorte que les tests peuvent être effectués dans les premiers stades de la culture clonale, en économis...
Déclarations de divulgation
Les auteurs déclarent qu'ils n'ont pas d'intérêts financiers concurrents.
Remerciements
Les auteurs souhaitent remercier Gissell Sanchez, Megan Lee et Jason Estep pour l'aide expérimentale, et Weifeng Gu et Xuemei Chen pour le partage des réactifs.
matériels
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
Références
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon