このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
哺乳類細胞におけるCRISPR / Cas9媒介遺伝子ノックアウトの選択依存性および独立した世代
要約
体細胞株を遺伝子操作する能力の最近の進歩は、基礎研究および応用研究の大きな可能性を秘めている。ここでは、選択マーカーを使用した場合と使用しない場合の、哺乳類細胞株におけるCRISP / Cas9生成ノックアウト産生およびスクリーニングのための2つのアプローチを提示する。
要約
CRISPR / Cas9ゲノムエンジニアリングシステムは、わずかな労力で正確なゲノム編集を可能にすることによって生物学に革命をもたらしました。特異性を与える単一のガイドRNA(sgRNA)によって導かれ、Cas9タンパク質は、標的遺伝子座で両方のDNA鎖を切断する。 DNA破壊は、非相同末端結合(NHEJ)または相同性指向修復(HDR)のいずれかを引き起こす可能性がある。 NHEJは、フレームシフト突然変異をもたらす小さな欠失または挿入を導入することができ、一方、HDRはより大きくより正確な摂動を可能にする。ここでは、確立されたCRISPR / Cas9法を下流の選択/スクリーニングの2つの選択肢と結びつけることによってノックアウト細胞株を生成するためのプロトコールを提示する。 NHEJアプローチでは、単一のsgRNA切断部位および選択非依存性スクリーニングを使用し、ここでタンパク質産生は、ハイスループット様式でドットイムノブロットによって評価される。 HDRアプローチは、目的の遺伝子にまたがる2つのsgRNA切断部位を使用する。提供されたHDRテンプレートと一緒に、この方法は削除を達成することができます挿入された選択可能な抵抗マーカーによって助けられた数十kbの大きさであった。それぞれの方法の適切なアプリケーションと利点について説明します。
概要
安定した遺伝子改変は、細胞摂動の一時的な方法よりも利点を提供し、その効率および持続時間は可変であり得る。ゲノム編集は、亜鉛フィンガーヌクレアーゼ1,2,3,4,5、転写アクチベーター様エフェクターヌクレアーゼ(TALEN)6,7,8,9などの標的特異的ヌクレアーゼの開発のために近年ますます一般的になってきているクラスター化され、規則的に間隔を置いて配置された短いパリンドローム反復(CRISPR)システム10に由来するRNA誘導ヌクレアーゼを含む。
CRISPR / Cas9編集機は、細菌や古細菌がウイルス感染を防御するために使用する免疫システムに適合していますass = "xref"> 11,12,13。このプロセスでは、侵入するウイルス配列の短い20〜30ntフラグメントが、繰り返しユニット14,15に隣接する「スペーサー」としてゲノム座に組み込まれる。その後の転写およびRNAプロセシングにより、トランス活性化crRNA 17 (tracrRNA)とともに、エフェクターCas9エンドヌクレアーゼと組み立てられる小さなCRISPR関連RNA16(crRNA)が生成される。このようにして、crRNAは、相補性ウイルスDNA配列を切断し、さらなる感染を予防するために、複合体を誘導するCas9標的化への特異性を提供する18,19。標的DNA中のいずれかの「プロトスペーサー」配列は、短いprotospacer隣接モチーフ(PAM)、ストレプトコッカス・ピオゲネス Cas9の場合のNGG 20。宿主のCRISPR遺伝子座のスペーサーの近くにPAM配列がないことは、自己と非自己とを区別し、宿主の標的化を妨げる。その普遍性と柔軟性のために、この生物学的システムは、ほぼ全てのPAMに隣接するDNA部位を標的とすることができるように、ゲノム編集に強力に適合している。このバージョンでは、さらなる改変により、crRNAおよびtracrRNAが、Cas9タンパク質21にロードされる単一のガイドRNA(sgRNA)成分に融合した。
Cas9およびsgRNAを真核細胞で発現させると、Cas9タンパク質は、標的遺伝子座で両方のDNA鎖を切断する。相同配列の適切な領域が存在しない場合、細胞は、典型的には小さな欠失またはまれに挿入を導入する非相同末端結合(NHEJ) 22,23,24を介してこのブレークを固定する。オープンをターゲットとする場合修復は、おそらく非機能的なタンパク質産物を産生する翻訳フレームシフトにつながる。対照的に、大きな相同性領域を有する外因性鋳型を提供する場合、細胞は相同性指向修復により二本鎖切断を固定することができる25,26 。この経路は、切除可能な選択マーカー27の導入と相まって、ゲノムにおけるより大きな正確な欠失、置換または挿入を可能にする。
ここでは、これらの2つのCRISPR / Cas9法( 図1A )のいずれかによってノックアウト細胞株を生成するためのプロトコールを提示する。 NHEJアプローチは、単一のsgRNA切断部位および選択非依存性スクリーニングを使用するため、ほとんど準備を必要としない。この方法を使用する場合、ノックアウトを生成する可能性が最も高い、転写物の5 '末端近くのエクソンに相補的な誘導RNAを設計しなければならない。変更後この場合、ゲノムへのイオンの量は少なく、ノックアウトクローンのスクリーニングはドットブロットに基づいており、タンパク質産物はハイスループットの様式で評価される。我々はELAV様1タンパク質(ELAVL1)ノックアウト系の生成を例として使用する。第2のアプローチは相同性指向修復(HDR)に依拠し、数十kbの欠失を可能にする目的の遺伝子または関心領域にまたがる2つのsgRNA切断部位を使用する。切断部位に隣接する2つの相同性領域を有するプラスミドは、ノックアウト生成の効率を増加させる選択可能な耐性マーカーを導入する置換テンプレート( 図1B )を提供する。この方法は、適切に設計された相同性アームを用いて遺伝子改変を導入するために適合させることもできる。この場合、新しいDNA断片の組み込みにより、PCRベースのスクリーニングが可能になる( 図1C )。ここでは、Pumilio RNA binding family member 2(PUM2)ノックアウト系統の生成を例として使用します。
プロトコル
1.所望の欠失周辺の相同性領域の同定
注:選択ベース編集を使用している場合のみ必要です。
- HDR鋳型( 図1A )の相同性アームとして機能する所望の欠失座の両側に、最初は1.5-2kbの2つの領域を選択する。クローニングを容易にするために、いずれかの鎖(GGTCTC)上のBsaI認識部位を欠く領域を同定する。 BsaI部位が避けられない場合は、別のタイプIIs制限酵素(BsmBI、SapI、BbsI)を使用し、レシピエントプラスミド(pUC19-BsaI)、耐性マーカードナープラスミド(pGolden-NeoまたはpGolden-Hygro)相同性アームPCR産物のオーバーハング。
Cas9-sgRNA発現プラスミドの作製
- 突然変異誘発のための標的化部位を定義する。シングルカット、選択のない方法では、非フーの確率を高めるために5 '近位コーディングエクソンを標的とするnctional突然変異。 2カット法では、必要に応じて多くの遺伝子にまたがる2つのサイトを選択します。
注:私たちの経験では、53 kbまでの削除が効率的に作成されています。 - ターゲット地域配列の200から300 bpをcrispr.mit.eduデザインツールに入力します。選択ベースのクローニングのために、欠失座位の近位にある同定された相同性領域の200〜300bpを使用する( 図1A )。標的領域ごとに最高位にランク付けされたsgRNAのうち2-3個を選択して、それらの活性の相違を説明し、同じ潜在的なオフターゲット効果を共有しない独立したクローンを単離する。
- 発現プラスミドに挿入するための適切なオーバーハングを有する二本鎖オリゴヌクレオチドを設計するためには、終結PAM配列(NGG)を省略し、5'-CACCオーバーハングに続いてGを先頭鎖オリゴに付加する。下鎖オリゴについては、5'-AAACオーバーハングを逆相補的な標的配列に付加し、続いて3'-Cをiil以下を欲しがる:
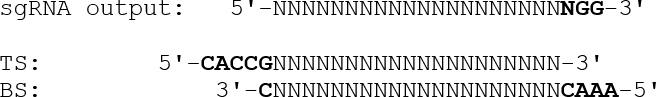
- Zhang labプロトコール29に記載されるように、ゴールデンゲートクローニング28を使用して、sgRNAに対応する合成オリゴヌクレオチドをpSpCas9(BB)プラスミドに挿入する。
相同性修復テンプレートプラスミドの作製
注:選択ベース編集を使用している場合のみ必要です。相同性指向修復テンプレートプラスミドは、2つのsgRNA標的部位のすぐ外側のゲノムに相補的な2つの領域に隣接する薬剤耐性カセットからなる( 図1B )。
- ステップ1.1で同定されたより広い相同性領域から、設計されたCas9切断部位から800〜1,000bpを超えて5〜10bpを選択し、相同性アーム( 図1A )として機能させる。 sgRNAを含まないようにしてくださいこれは、鋳型プラスミド自体のCRISPR / Cas9切断を引き起こすであろうからである。相同性アームに内部BsaI部位が含まれている場合は、別のsgRNA部位を使用する。
- ホモロジー領域のいずれかの末端にBsaI部位(GGTCTC)およびユニークなオーバーハングを導入する追加の5 '配列を有する順方向プライマーおよび逆方向プライマーを使用して、製造者の指示に従って、高忠実度DNAポリメラーゼを用いてゲノムDNAから相同性アームをPCR増幅する。オーバーハングには、正しいアセンブリ順序と方向を生成するための適切なシーケンスが含まれている必要があります( 図1B )。
左相同アームFP突出部:5 'GGGTCTCA GGCC
左ホモロジーアームRPオーバーハング:5 'GGGTCTCT CACG
右相同アームFPオーバーハング:5 'GGGTCTCA GTCC
右相同アームRPオーバーハング:5 'GGGTCTCT CCAC - 適切なamplifについて相同性アームをチェックする前進する前にゲル電気泳動による増幅。
- ゴールデンゲートクローニング28によって、抵抗カセットを有する左右の相同性アーム領域をHDRテンプレートプラスミドに組み込む。 loxPに隣接する耐性カセット(loxP-PGK-Neo-pA-loxPまたはloxP-PGK-Hyg-pA-loxP)の供給源として、pGolden-NeoまたはpGolden-Hygroプラスミド30を使用する。複数のクローニング領域にBsaI部位を有する修飾pUC19であるpUC19-BsaIを使用し、他の場所でBsaI部位を除去したものをレシピエントベクター(要請に応じて入手可能)として使用する。 3つのインサートとベクターに1:1:1:1の比率を使用します。
- 以下の反応混合物30を調製する:
右相同アームPCR産物 0.06 pmol(30-40ng) 左ホモロジーアームPCR産物 0.06 pmol(30-40ng) pGolden-Neoプラスミド(100ng /μL) 1µ L pUC19-BsaIプラスミド(100ng /μL) 1μL 2×T7 DNAリガーゼ緩衝液 5μL BsaI(10U /μL) 0.75μL T7 DNAリガーゼ(3000U /μL) 0.25μL 水 最大10μL - 次のサーモサイクラーパラメーターを使用してください:37℃で5分間、20℃で5分間。 30サイクル繰り返します。
- 残りの線状化DNAを消化するために、製造者の指示に従ってエキソヌクレアーゼを用いてクローニング製品を処理する。
- 細胞とともに供給されたプロトコールに従って反応混合物をコンピテント大腸菌株に形質転換し、アンピシリン含有皿にプレートする。
- 以下の反応混合物30を調製する:
- 正しいテンプレートアセンブリを持つクローンを同定するには、バクテリア(全インサートが大きすぎて確実に増幅することができないため)組み立てられたインサートの相同性アーム小領域を増幅するためのαコロニーPCRを行う。隣接するプラスミド配列に1つのプライマーをアニーリングし、抵抗カセットの端に相補的な第2のプライマーを使用して( 図1B 、配列はNeoとHygroの両方のインサートに共通)、約200 bp含まれる相同性アーム:
pUC19-Bsal-左:5 'GGCTCGTATGTTGTGTGGAATTGTGAG
抵抗性 - 左:5 'AAAAGCGCCTCCCCTACCC
pUC19-BsaI-右:5 'GCTATTACGCCAGCTGGCGAAA
抵抗 - 右:5 'AAGACAATAGCAGGCATGCTGGG- 滅菌爪楊枝で細菌のコロニーを選び、細菌を20μLの水に懸濁する。 95℃で15分間加熱し、卓上遠心分離機で最大速度で5分間スピンダウンする。直ちに氷上に置く。
- 以下のPCR混合物を調製する</ sup>:
コロニーテンプレートを希釈する 1.25μL 10×Taq反応緩衝液 1.25μL 20mMのdNTP 0.25μL 10μMフォワードプライマー 0.25μL 10μMリバースプライマー 0.25μL Taqポリメラーゼ 0.25μL 水 9μL - 以下のサーマルサイクラーパラメーターを使用してください:95℃30秒間(95℃30秒間、61℃30秒間、72℃1分/ kb)、25サイクル、72℃で2分間繰り返す。
- アガロースゲル電気泳動で正確なアンプリコンサイズについてPCR産物をチェックする。
- 任意選択的に、正確なインサートサイズおよび配列thを有する個々のコロニーからのminiprepプラスミドDNA上記の増幅プライマーによるサンガー配列決定による相同性アーム領域を含む。
4.培養細胞へのCRISPR成分のトランスフェクション
- トランスフェクションの前に:37℃、5%CO 2で 10%FBSを補充したDMEM培地中のT-REx293細胞を培養する。
- 6ウェルプレートに細胞をプレートし、約70%のコンフルエントまで増殖させる。トランスフェクションされていない対照用のウェルを含める。
- 市販のトランスフェクションプロトコールを用いて2.5μgの全プラスミドを細胞にトランスフェクトする。並行して、トランスフェクトされていないコントロールを維持する。
注:トランスフェクションの方法と効率は細胞の種類によって異なります。実験前にシステムに適したトランスフェクション方法を決定する。- 選択した細胞をトランスフェクションするために、0.75μgCas9-sgRNA 1(左カット)、0.75μgCas9-sgRNA 2(右カット)および1.0μgの相同組換えテンプレートを使用する。
- トランスフェクションの場合2.5μgのCas9-sgRNAプラスミドを使用する。
5.薬物選択
- トランスフェクションの48時間後、適切な薬物(ネオマイシン/ハイグロマイシン)で細胞を処理する。トランスフェクトされていないコントロールのすべての細胞が死ぬまで選択を行う(通常、Neoでは3〜5日、Hygroでは7〜14日)。
注:T-REx293細胞については、それぞれネオマイシンおよびハイグロマイシンに対して500μg/ mLおよび10μg/ mLの濃度を使用してください。他の細胞型については、有効濃度を決定するために、トランスフェクションされていない細胞に薬物を予め滴定することが有用であり得る。
クローン集団の単離
- 選択後、元のウェルに100%コンフルエンシーまで細胞を増殖させる。細胞を96ウェルプレートに0.33細胞/ウェルの密度で播種する。
注:3つの96ウェルプレートを播種することは、複数の正しいクローンの分離を確実にするための良い出発点ですが、多かれ少なかれ必要な場合があります。depsgRNAおよびHDR効率が終わる。 - コロニーが目に見えるまで、2〜4週間にわたってコロニーを観察する。滅菌ピペットチップで目に見えるコロニーを拾い、単層の増殖を促すために新しいウェルに再播種する。懸濁細胞を使用する場合、播種密度をさらに低くして、単一コロニーウェルのより大きな割合を確保することができる。
7.審査の候補者
- 選択を使用しない候補のスクリーニング:ドットブロット
- 個々のクローンを50-100%のコンフルエンシーまで増殖させる。ウェル内でピペッティングすることによって細胞の単層を除去する。
- 清潔なマイクロ遠心チューブに100μL全量の90μLを分注する。 6000rpmで5分間スピンダウンし、培地を除去し、1×溶解バッファーまたは1×SDSローディングバッファー(5×250mM Tris-Cl pH6.8,8%SDS、0.1%ブロモフェノールブルー、40%グリセロール、100mMのDTT)を含む。ウエル内の細胞の残りに90μLの新しい培地を加える。lは伝搬を続ける。
- 1μLの細胞溶解物を乾燥ニトロセルロースメンブレン上にピペットしてドットを形成する。 2つの別々の膜で各サンプルを2回ブロットし、2つの同一パターンのサンプルを作成する。
- TBST(トリス緩衝食塩水+ 0.01%Tween20:8g NaCl、0.2g KCl、3gトリス塩基、最大1L蒸留水、pH7.4)中の5%ミルク中の膜を室温で1時間ブロックする(ロッキング、ここでは、手順全体を通して)。
- 1つの膜上の標的タンパク質に対する一次抗体および対照タンパク質(チューブリン、GAPDH、または他のいずれのタンパク質も変化しない)に対する一次抗体を用いてブロットする。 TBST + 5%ミルクで推奨される一次抗体希釈液(ELAVL1では0.2μg/ mL、Pum2では0.1μg/ mL)を使用してください。室温で1時間インキュベートする。
- TBSTで5分間3回洗浄する。
- 各メンブレンをT中の適切なHRPコンジュゲート二次抗体とインキュベートする。BST + 5%ミルクを室温で1時間インキュベートする。
- TBSTで5分間3回洗浄する。
- 製造元の指示に従って化学発光基質溶液(材料表を参照)を適用し、ブロットした膜をデジタル化学イメージャで画像化する。
- 適切なソフトウェアを使用して、標的タンパク質および対照タンパク質のドット強度を定量化する。
- 低いコントロールタンパク質シグナルを有する候補物質を排除し、残りの候補について、タンパク質強度を制御するための標的のバックグラウンドを差し引いた比を計算する。継代およびウェスタンブロットによるさらなる検証のために最も低い比を有する候補を選択する。
- 選択を使用して候補をスクリーニングする:コロニーPCR
- DNAプレップキット( 材料表を参照)を使用して細胞ライセートを収集する。
- 新しい96ウェルに個々のコロニーを複製し、100%コンフルエンシーまで増殖させる。
- クローンの1つのセットからメディアを削除します。キットからの抽出バッファー30μL中の細胞を再懸濁する。清潔な1.5 mLのマイクロ遠心チューブに移す。
- 溶液を96℃で15分間加熱し、室温まで冷却する。
- 安定化バッファー30μLをキットから加えます。よく混ぜます。
- コロニーPCRにより、野生型および単対立遺伝子/二対立遺伝子変異体を同定する。
- 耐性カセット( 図2C )の成功または不成功のいずれかに基づいて相同性アーム周辺の領域を増幅するための2つの別個のPCRプライマーセット(標的遺伝子座の左側および右側)を設計する。それぞれの側で、相同性アームの外側にアニーリングする共通のプライマー(赤色)を使用します。野生型対立遺伝子を試験するために、相同性領域(青色)にわたる内因性配列に相補的な対応する対のプライマーと共に使用する。挿入された耐性カセットに相補的な別のペアのプライマーを使用する(セクション2.3)、所望の突然変異を試験する。
- (推奨)元の選択されたバルク細胞集団からの細胞ライセートを使用してプライマーセットを検証する.WTおよび変異対立遺伝子の両方の混合物を含むからである( 図2D )。
- 以下の反応混合物を調製する:
細胞ライセート 0.5μL 10×KOD緩衝液 1.25μL 25mM MgSO4 0.75μL 2mMのdNTP 1.25μL 10μMフォワードプライマー 0.375μL 10μMリバースプライマー 0.375μL KODポリメラーゼ 0.25μL 水 7.75μL - 次のサーマルサイクラー条件を使用してください:95℃で2分間(95℃で20秒間、プライマーTmで10秒間、70℃で20秒間/ kb)を25サイクル繰り返します。
- アガロースゲル電気泳動でPCR産物を視覚化する。
- 予想される組込みの両側についてコロニーPCR試験を繰り返す。インテグレーションの存在を示し、削除された領域の内因性配列産物を示さない候補を選択して広げる。
- ウエスタンブロットおよび/またはRT-qPCRによって陽性候補を検証する。
- DNAプレップキット( 材料表を参照)を使用して細胞ライセートを収集する。
8.シーケンシングによるゲノム突然変異の確認
- フェノールクロロホルム抽出による変異細胞株からのゲノムDNAの単離31 。
- 各末端にBsaI制限部位を付加する5 'オーバーハングを有するプライマーを用いて、sgRNA標的領域をPCR増幅する。
注:両方の対立遺伝子が同一である可能性があるHDRで編集されたクローンの場合、PCR産物を直接配列決定することができます。 - 増幅した領域をゴールデンゲートクローニングによってpUC19-BsaIにクローニングする。
- 反応混合物をコンピテント大腸菌株に形質転換する 。
- 6〜10個の個々のコロニーからのMiniprepプラスミドDNAを回収し、Sanger配列決定によりクローン化領域を配列決定する。
結果
ELAVL1ノックアウト系統の作製には、頑強な抗体が利用できたので、単一のsgRNA( 図1A 、左)を用いた編集を行い、次にドットイムノブロットを行った。 3つのsgRNAを独立してトランスフェクトして、効率を比較し、得られたクローンの標的効果を排除した。クローン集団からの細胞溶解物を2つのニトロセルロースメンブレン上に集め、ブロット?...
ディスカッション
CRISPR / Cas9システムは、安定したゲノム改変の効率的な生成を可能にし、他の一時的な操作方法のより一貫した代替物を提供する。ここでは、哺乳動物細胞株におけるCRISPR / Cas9遺伝子ノックアウトの迅速な同定のための2つの方法を提示した。どちらの方法も細胞材料をほとんど必要としないため、クローン培養の初期段階で試験を行い、時間と試薬を節約できます。両方の方法の効率を上げ...
開示事項
著者らは、競合する金銭的利益がないと宣言している。
謝辞
著者は、実験支援のためにGissell Sanchez、Megan Lee、Jason Estep、試薬を共有するWeifeng GuとXuemei Chenを認めたいと思っています。
資料
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
参考文献
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved