A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הבחירה תלויה עצמאית הדור של CRISPR / Cas9 בתיווך גין Knockouts בתאי יונקים
In This Article
Summary
ההתקדמות האחרונות ביכולת לתמרן גנטית קווי תאים סומטיים להחזיק פוטנציאל גדול עבור מחקר בסיסי ויישומי. כאן, אנו מציגים שתי גישות עבור CRISPR / Cas9 שנוצר נוקאאוט הייצור והקרנה שורות תאים יונקים, עם וללא שימוש סמנים לבחירה.
Abstract
מערכת ההנדסה הגנטית של CRISPR / Cas9 חוללה מהפכה ביולוגית בכך שהיא מאפשרת עריכה מדויקת של הגנום עם מעט מאמץ. מודרך על ידי מדריך יחיד RNA (sgRNA) המעניק סגוליות, חלבון Cas9 cleaves שני גדילי DNA ב מוקד ממוקדת. ההפצה DNA יכול להפעיל או שאינם הומולוגיים סוף שהצטרף (NHEJ) או הומולוגיה מכוונת לתקן (HDR). NHEJ יכול להציג מחיקות קטנות או הוספות אשר להוביל מוטציות מסגרת משמרת, בעוד HDR מאפשר הפרעות גדול יותר מדויק. כאן, אנו מציגים פרוטוקולים להפקת קווי תא נוקאאוט על ידי צימוד הוקמה CRISPR / Cas9 שיטות עם שתי אפשרויות לבחירה במורד / הקרנה. הגישה NHEJ משתמשת באתר חיתוך יחיד sgRNA ובחירה עצמאית הבחירה, שבו ייצור החלבון מוערכת על ידי אימונובלוט נקודה בצורה תפוקה גבוהה. הגישה HDR משתמש בשני sgRNA לחתוך אתרים כי טווח הגן של עניין. יחד עם תבנית HDR בתנאי, שיטה זו יכולה להשיג מחיקהשל עשרות kb, בסיוע סמן התנגדות לבחירה לבחירה. היישומים והיתרונות המתאימים של כל שיטה נדונים.
Introduction
שינויים גנטיים יציבים מספקים יתרון על פני שיטות חולפות של הפרעות סלולר, אשר יכול להיות משתנה ביעילות שלהם משך. עריכה גנומית הפכה נפוצה יותר ויותר בשנים האחרונות עקב התפתחות של נוקלאזות ספציפיות למטרה, כגון נוקלאזות של אבץ אצבע , 1 , 2 , 3 , 4 , 5 , ו RNA מונחה נוקלאזות הנגזרות, מקובצים בקביעות interinded קצר palindromic חוזר (CRISPR) מערכת 10 .
CRISPR / Cas9 מכונות העריכה מותאמת מתוך המערכת החיסונית כי חיידקים ארכאי להשתמש כדי להגן מפני זיהומים ויראלייםהתחת = "xref"> 11 , 12 , 13 . בתהליך זה, קצרים, 20-30 nt שברי רצף ויראלי פולש משולבים לתוך לוקוס גנומי כמו "מרווחים" מוקף יחידות חוזרות 14 , 15 . תעתיק לאחר עיבוד RNA מייצר קטן CRISPR הקשורים RNAs 16 (crRNAs) כי, יחד עם הפעלת crRNA 17 (tracrRNA) מפעיל, להרכיב עם endonuclease Cas9 effector. CRRNAs ובכך לספק ספציפיות למיקוד Cas9, המנחה את המתחם לדבק רצפים דנ"א ויראלי משלימה ומניעת זיהומים נוספים 18 , 19 . כל רצף "protospacer" בדנ"א ממוקד יכול לשמש כמקור של ה- CRRNA, כל עוד הוא ישירות 5 'כדי מוטיב קצר סמוך protospacer (PAM), NGG במקרה של S. pyogenes Cas9 20. העדר רצף ה- PAM ליד המרווח באזור של ה- CRISPR המארח מבדיל בין העצמי לבין הלא-עצמי, ומנע את המיקוד של המארח. בגלל האוניברסליות והגמישות שלה, מערכת ביולוגית זו הותאמה בעוצמה לעריכה גנומית, כך שכמעט כל אתר דנ"א הצמוד ל- PAM יכול להיות ממוקד. בגרסה זו, שינוי נוסף התמזגו crRNA ו tracrRNA לתוך RNA מדריך יחידה (sgRNA) רכיב נטען לתוך חלבון Cas9 21 .
עם הביטוי של Cas9 ו sgRNA בתאים אוקריוטים, חלבון Cas9 cleaves שני גדילי דנ"א על מוקד ממוקדת. בהיעדר אזור מתאים של רצף הומולוגי, התא מתקן את ההפסקה הזאת באמצעות חיבור לא הומולוגי (NHEJ) 22 , 23 , 24 , אשר בדרך כלל מציג מחיקות קטנות או, לעתים נדירות, כניסות. בעת מיקוד פתוחמסגרת הקריאה, התיקון עלול להוביל frameeshift translational שמייצר מוצר חלבון שאינו פונקציונלי. לעומת זאת, כאשר מסופק עם תבנית אקסוגני עם אזורים גדולים של הומולוגיה, התא יכול לתקן את הפער פעמיים גדיל על ידי הומולוגיה מכוונת לתקן 25 , 26 . נתיב זה מאפשר מחיקות מדויקות יותר, תחליפים או הוספות בגנום, יחד עם הכנסת סמני הבחירה הנבחרים 27 .
כאן, אנו מציגים פרוטוקולים ליצירת שורות תאים נוקאאוט על ידי אחד משני אלה CRISPR / Cas9 שיטות ( איור 1 א ). הגישה NHEJ משתמשת באתר חיתוך יחיד sgRNA ובחירה עצמאית ההקרנה, ולכן דורש הכנה מראש upfront. בעת שימוש בשיטה זו, מדריך RNAs משלימים אקסונים ליד הקצה 5 'של התמליל, אשר סביר להניח לייצר נוקאאוט, חייב להיות מתוכנן. מאז modificatיונים לגנום במקרה זה הם קטנים, ההקרנה שיבוטים נוקאאוט מבוסס על כתמים נקודה, שבו המוצר חלבון מוערך בצורה תפוקה גבוהה. אנו משתמשים בדוגמה של ELAVL 1 כמו חלבון 1 (ELAVL1). הגישה השנייה מסתמכת על תיקון הומולוגיה מכוונת (HDR) ומשתמשת בשני אתרי חיתוך sgRNA המשתרעים על פני הגן או אזור העניין, ומאפשרים מחיקות של עשרות קילובייט. פלסמיד עם שני אזורים של הומולוגיה כי האגף אתרי מחשוף מספק תבנית תחליף ( איור 1B ), מציגה סמן התנגדות לבחירה כי מגביר את היעילות של הדור נוקאאוט. שיטה זו יכולה גם להיות מותאם כדי להציג שינויים גנים עם זרועות הומולוגיה מתוכנן כראוי. במקרה זה, שילוב של קטע DNA חדש מאפשר סינון מבוסס PCR ( איור 1C ). כאן, אנו משתמשים הדור של Pumilio RNA מחייב בן משפחה 2 (PUM2) נוקאוט שורות כדוגמה.
Protocol
1. זיהוי של הומולוגיה אזורים סביב המחיקה הרצוי
הערה: יש צורך רק אם אתה משתמש בעריכה המבוססת על בחירה.
- בחר שני אזורים, בהתחלה 1.5-2 kb, משני צדי המקום הרצוי המחיקה, אשר ישמשו זרועות הומולוגיה בתבנית HDR ( איור 1 א ). זהה אזורים ללא אתרי הכרה ב- BSAI בשני החוטים (GGTC) כדי להקל על שיבוט. אם אתרי BSAI הם בלתי נמנעים, להשתמש באנזים הגבלת סוג IIs חלופי (BsmBI, SapI, BbsI) ולשנות את האתרים המתאימים של פלסמיד הנמען (pUC19-BSAI), סמן ההתנגדות התורם פלסמיד (pGolden-Neo או pGolden- היגרו), ו זרוע הומולוגיה PCR - -.
2. הדור של Cas9-sgRNA ביטוי פלסמידים
- הגדר את אתרי המיקוד עבור mutagenesis. עבור יחיד לחתוך, בחירה חופשית שיטה, יעד 5 'אקסונים קידוד הפרוקסימלי כדי להגדיל את ההסתברות של non-fuמוטציה פונקציונלית. עבור שני לחתוך שיטה, בחר שני אתרים כי להקיף את כמות הגן כפי הצורך.
הערה: מניסיוננו, מחיקות של עד 53 kb נוצרות ביעילות. - קלט 200-300 BP של רצף האזור ממוקד לתוך כלי העיצוב של crispr.mit.edu. עבור שיבוט מבוסס הבחירה, השתמש 200-300 BP של אזורים הומולוגיה זיהו כי הם הפרוקסימלי לוקוס המחיקה ( איור 1 א ). בחר 2-3 של הדירוג הגבוה ביותר sgRNAs לכל אזור ממוקד כדי להסביר את ההבדלים בפעילות שלהם, כדי לבודד שיבוטים עצמאיים שאינם חולקים את הפוטנציאל אותו מחוץ היעד הפוטנציאל.
- כדי לעצב oligonucleotides דו-צדדית עם overhangs המתאים להכנסת הביטוי פלסמיד, להשמיט את רצף PAM מסתיים (NGG) ו לצרף 5'-CACC הסככה ואחריו G אל החוט העליון אוליגו. עבור גדיל התחתונה אוליגו, לצרף 5'-AAAC התלוי על רצף היעד המשלים לאחור, ואחריו 3'-C, כמו ilמבוהל מתחת:
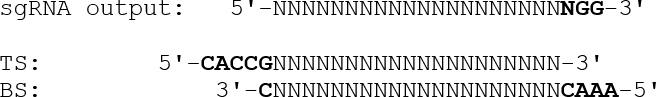
- הכנס oligonucleotides סינתטי המתאים sgRNAs לתוך pSpCas9 (BB) פלסמיד באמצעות שער הזהב שיבוט 28 כמתואר בפרוטוקול מעבדה ג 'אנג 29 .
3. הדור של הומולוגיה מכוונת תיקון תבנית פלסמידים
הערה: יש צורך רק אם אתה משתמש בעריכה המבוססת על בחירה. ההומולוגיה מכוונת תיקון התבנית פלסמיד מורכב קלטת עמידות התרופה מוקף על ידי שני אזורים המשלימים את הגנום רק מחוץ לשני אתרי היעד sgRNA ( איור 1 ב ).
- מתוך אזורים ההומולוגיה רחב יותר מזוהה בשלב 1.1, בחר 800-1,000 bp 26 לא יותר מ 5-10 BP הרחק מעוצב Cas9 אתרים לחתוך, לשמש זרועות הומולוגיה ( איור 1 א ). הקפד לא לכלול את sgRNAאתר היעד שלה PAM בזרועות ההומולוגיה, כמו זה יגרום CRISPR / Cas9 cleavage של התבנית פלסמיד עצמו. אם הזרועות ההומולוגיה מכילות אתרי BSAI פנימיים, השתמש באתרי sgRNA חלופיים.
- PCR להגביר את זרועות ההומולוגיה של דנ"א גנומי באמצעות פולימראז DNA גבוהה נאמנות, לפי הוראות היצרן, באמצעות primers קדימה ויורדים עם רצף 5 'נוספים זה מציג את אתרי BSAI (GGTCTC) ו overhangs ייחודי משני צדי האזור ההומולוגיה. את overhangs חייב להכיל sequences הנכון כדי ליצור בסדר הרכבה נכונה וכיוון, כפי שמתואר להלן ( איור 1 ב ):
שמאל זרוע ההומולוגיה זרוע FP: 5 'GGGTCTCA GGCC
שמאל הידומולוגיה זרוע RP overhang: 5 'GGGTCTCT CACG
זרוע הומולוגיה ימין זרוע FP: 5 'GGGTCTCA GTCC
זרוע ההומולוגיה הנכונה: "5 GGGTCTCT CCAC - בדוק נשק הומולוגיה עבור amplif המתאיםקרח באמצעות אלקטרופורזה ג'ל לפני שתמשיך קדימה.
- להרכיב את ימין ושמאל זרוע ההומולוגיה אזורים עם קלטת התנגדות לתוך HDR תבנית פלסמיד על ידי גולדן שער שיבוט 28 . השתמש pGolden-Neo או pGolden-היגרו פלסמידים 30 כמקור של קלטות התנגדות loxP-flanked (loxP-PGK-Neo-pA-loxP או loxP-PGK-Hyg-pA-loxP). השתמש pUC19-BSAI, PUC19 שונה עם אתרים BSAI באזור שיבוט מרובים וביטל אתרי BSAI במקום אחר, כמו וקטור הנמען (זמין על פי בקשה). השתמש ביחס של 1: 1: 1: 1 עבור שלושת התוספות והקטור.
- הכן את תערובת התגובה הבאה 30 :
נכון זרוע ההומולוגיה PCR המוצר 0.06 pmol (30-40ng) שמאל זרוע ההומולוגיה PCR המוצר 0.06 pmol (30-40ng) PGolden-Neo פלסמיד (100 ng / μL) /# 181; L PUC19-BSAI פלסמיד (100 ng / μL) 1 μL 2x T7 DNA חיץ ligase 5 μL BSAI (10 U / μL) 0.75 μL T7 DNA ligase (3000 U / μL) 0.25 μL מַיִם עד 10 μL - השתמש בפרמטרים thermocycler הבאים: 37 מעלות צלזיוס למשך 5 דקות, 20 מעלות צלזיוס למשך 5 דקות. חזור על 30 מחזורים.
- פנקו את המוצר שיבוט עם exonuclease לכל הוראות היצרן לעכל משם כל DNA ליניארית הנותרים.
- להפוך את תערובת התגובה לתוך זן E. מוסמכת coli בהתאם לפרוטוקול שסופקו עם התאים, ואת צלחת על מאכלים המכילים ampicillin.
- הכן את תערובת התגובה הבאה 30 :
- כדי לזהות שיבוטים עם הרכבה התבנית הנכונה, לבצע חיידקיםאל מושבה PCR כדי להגביר את זרועות ההומולוגיה subregions של הכנס המורכבת (כמו להכניס את כל יכול להיות גדול מדי כדי להגביר באופן מהימן). השתמש חישול אחד תחל על רצף פלסמיד אגף פריימר השני משלימים את הקלטת של התנגדות קלטת ( איור 1 ב , רצף נפוץ הן ניאו והיגרו מוסיף), יצירת מוצר תלויי הרכבה, כי הוא כ 200 BP זמן ארוך יותר מאשר זרוע ההומולוגיה הכלולה:
PUC19-Bsal-Left: 5 'GGCTCGTATGTTGTGTGGAATTGTGAG
התנגדות- Left: 5 'AAAAGCGCCTCCCCTACCC
PUC19-BSAI-Right: 5 'GCTATTACGCCAGCTGGCGAAA
התנגדות-מימין: 5 'AAGACAATAGCAGGCATGCTGGG- בחר מושבות חיידקים עם קיסמים קיסריים להשעות את החיידקים במים 20 μL. מחממים על 95 מעלות צלזיוס למשך 15 דקות, ספין למטה צנטריפוגה השולחן במהירות מקסימלית במשך 5 דקות. מניחים מיד על הקרח.
- הכן את תערובת ה- PCR הבאה/ Sup>
מדולל תבנית המושבה 1.25 μL 10x חיץ התגובה Taq 1.25 μL 20mM dNTPs 0.25 μL 10 מיקרומטר קדימה תחל 0.25 μL 10 מיקרומטר הפוך פריימר 0.25 μL Taq פולימראז 0.25 μL מַיִם 9 μL - השתמש פרמטרים thermocycler הבאה: 95 מעלות צלזיוס למשך 30 שניות, (95 מעלות צלזיוס 30 מעלות, 61 מעלות צלזיוס למשך 30 שניות, 72 מעלות צלזיוס במשך 1 דקות / Kb), לחזור על 25 מחזורים, 72 מעלות צלזיוס למשך 2 דקות.
- בדוק את המוצר PCR עבור גודל amplicon הנכון על ידי אלקטרופורזה agarose ג'ל.
- לחלופין, DNA miniprep פלסמיד מ מושבות בודדים עם גודל הכנס הנכון ורצף הE homology זרוע אזורים על ידי סנגר רצף עם primers הגברה שהוזכרו לעיל.
4. transfection של רכיבי CRISPR לתוך תאים תרבותיים
- לפני transfection: תרבות T-Rex293 תאים בתקשורת DMEM בתוספת 10% FBS ב 37 ° C ו 5% CO 2 .
- תאים צלחת על 6 צלחות היטב לגדול כ confluency 70%. כלול גם עבור שליטה untransfected.
- Transfect תאים עם 2.5 מיקרוגרם סה"כ פלסמיד באמצעות פרוטוקול transfection מסחרי. במקביל, לשמור על שליטה untransfected.
הערה: שיטת Transfection ויעילות להשתנות בהתאם לסוג התא. קביעת השיטה המתאימה transfection עבור המערכת לפני הניסוי.- עבור transfection של תאים עם הבחירה, להשתמש 0.75 מיקרוגרם Cas9-sgRNA 1 (חתך שמאל), 0.75 מיקרוגרם Cas9-sgRNA 2 (לחתוך מימין), ו 1.0 microg הומולוגיים רקומבינציה תבנית.
- עבור transfectiעל תאים ללא בחירה, להשתמש 2.5 מיקרוגרם Cas9-sgRNA פלסמיד.
5. בחירת סמים
- 48 שעות לאחר transfection, לטפל בתאים עם התרופה המתאימה (Neomycin / Hygromycin). לבצע את הבחירה עד שכל התאים בשליטה untransfected למות (בדרך כלל 3-5 ימים עבור ניאו, 7-14 ימים עבור היגרו).
הערה: שימוש בריכוז של 500 מיקרוגרם / מ"ל ו 10 מיקרוגרם / מ"ל עבור Neomycin ו Hygromycin, בהתאמה עבור T-Rex293 תאים. עבור סוגי תאים אחרים, זה עשוי להיות שימושי לבצע טיטרציה מוקדמת של התרופה על תאים untransfected לקבוע את הריכוז האפקטיבי.
6. בידוד של אוכלוסיות Clonal
- לגדל תאים 100% confluency בבאר המקורי לאחר הבחירה. תאים זרע לתוך 96 צלחות היטב בצפיפות של 0.33 תאים לכל טוב.
הערה: זריעת שלוש 96 צלחות טוב היא נקודת התחלה טובה כדי להבטיח בידוד של יותר אחד שיבוט נכונה, אבל פחות או יותר ייתכן שיהיה צורך depמסתיים על יעילות sgRNA ו HDR. - שימו לב המושבות על פני 2-4 שבועות תקופה, עד מושבות גלויים לעין. בחר מושבות גלוי עם קצה פיפטה סטרילית reseed בבארות חדשות לעודד צמיחה monolayer. בעת שימוש בתאים ההשעיה, אפילו צפיפות זריעה נמוכה יותר ניתן להשתמש כדי להבטיח חלק גדול יותר של בארות יחיד המושבה.
7. סקר המועמדים
- הקרנת מועמדים ללא שימוש בבחירה: כתם נקודה
- לגדל שיבוטים בודדים confluency 50-100%. סילוק monolayer של תאים על ידי pipetting בתוך הבאר.
- Aliquot 90 μL של נפח 100 μL הכולל צינור microcentrifuge נקי. ספין למטה ב 6000 סל"ד במשך 5 דקות, להסיר מדיה, ו lyse תא גלולה ב 10 μL של 1x תמוגה חיץ או 1x SDS טוען חיץ (5x: 250 מ"מ טריס- CL pH 6.8, 8% SDS, 0.1% ברומופנול כחול, 40% גליצרול, 100 מ"מ DTT). הוסף 90 μL של מדיה חדשה לשאר התאים ב wel• להמשיך ולהפיץ.
- פיפטה 1 μL של lysate התא על קרום nitrocellulose יבש כדי ליצור נקודה. כתם כל פעמיים על שני קרום נפרד, יצירת שני דפוסים זהים של דגימות.
- לחסום את הממברנות בחלב 5 ב TBST (טריס שנאגרו מלוחים + 0.01% Tween 20: 8 גרם NaCl, 0.2g KCl, 3 גרם בסיס Tris, עד 1 L מים מזוקקים, pH 7.4) 31 עבור 1 שעה בטמפרטורת החדר ( עם נדנדה, כאן לאורך כל התהליך).
- כתם באמצעות נוגדנים ראשוניים נגד חלבון המטרה על קרום אחד, ואת הנוגדן העיקרי עבור חלבון שליטה (טובולין, GAPDH, או כל חלבון אחר שלא צפוי לשנות) מצד שני. השתמש דילול נוגדן ראשוני מומלץ (0.2 מיקרוגרם / מ"ל עבור ELAVL1 ו 0.1 מיקרוגרם / מ"ל עבור Pum2 במקרה זה) ב TBST + 5% חלב. דגירה 1 שעה בטמפרטורת החדר.
- לשטוף 3 פעמים עם TBST במשך 5 דקות.
- דגירה כל קרום עם המתאים HRP מצומדות נוגדנים משני ב TBST + 5% חלב עבור 1 שעה בטמפרטורת החדר.
- לשטוף 3 פעמים עם TBST במשך 5 דקות.
- החל פתרון chemiluminescence המצע (ראה טבלה חומרים) בעקבות הוראות היצרן ואת התמונה קרום מנוקב על imager דיגיטלית chemiluminescence.
- לכמת עוצמות נקודה עבור היעד וחלבונים שליטה באמצעות התוכנה המתאימה.
- לחסל את המועמדים עם האות חלבון שליטה נמוכה, ולחשב יחסי יחס-הרקע של היעד לשלוט בעוצמות חלבון עבור המועמדים הנותרים. בחר את המועמדים עם היחס הנמוך ביותר עבור מעבר אימות נוסף על ידי כתם המערבי.
- הקרנת מועמדים עם הבחירה של בחירה: מושבה PCR
- איסוף lysate התא באמצעות ערכת הכנה DNA (ראה טבלת חומרים ).
- כפולות מושבות בודדות חדש 96 גם לגדול עד 100% confluency.
- הסר מדיה מקבוצה אחת של שיבוטים, אNd resuspend תאים μL 30 של חיץ החילוץ מתוך הערכה. העברה צינור נקי 1.5 מ"ל microcentrifuge.
- מחממים את הפתרון ל 96 מעלות צלזיוס למשך 15 דקות ולתת מגניב לטמפרטורת החדר.
- הוסף 30 μL של חיץ ייצוב מן הערכה. לערבב היטב.
- זיהוי wildtype ו monoallelic / biallelic שורות מוטציה על ידי המושבה PCR.
- עיצוב שתי קבוצות נפרדות של primers PCR (בצד שמאל וימין של מוקד לוקוס) כדי להגביר את האזורים סביב זרועות הומולוגיה מבוסס על שילוב מוצלח או לא מוצלח של קלטת התנגדות ( איור 2 ג ). בכל צד, להשתמש פריימר משותף (אדום) כי anneals מחוץ זרוע ההומולוגיה. השתמש בו עם פריימר משויך המקביל כי הוא משלים את רצף אנדוגני, פורש באזור ההומולוגיה (כחול), כדי לבדוק את אלל wild-type. השתמש בפריימר משויך אחר, המשלים לקלטת ההתנגדות שהוכנסה (ראה פריימרים בסעיף2.3), כדי לבדוק את המוטציה הרצויה.
- (מומלץ) לאמת את ערכות פריימר באמצעות lysate התא מתוך האוכלוסייה שנבחרה בתא המקומי בתפזורת, שכן הוא יכיל תערובת של אללים WT ו מוטנטים ( איור 2 ד ).
- הכינו את תערובת התגובה הבאה:
נייד lysate 0.5 μL 10x חיץ KOD 1.25 μL 25 מ"מ MgSO4 0.75 μL 2 mN dNTPs 1.25 μL 10 מיקרומטר קדימה תחל 0.375 μL 10 מיקרומטר הפוך פריימר 0.375 μL פולימרז KOD 0.25 μL מַיִם 7.75 μL - השתמש בתנאי thermocycler הבאים: 95 מעלות צלזיוס למשך 2 דקות, (95 מעלות צלזיוס למשך 20 שניות, פריימר TM במשך 10 שניות, 70 מעלות צלזיוס למשך 20 s / kb) חזור על 25 מחזורים.
- דמיינו את מוצר ה- PCR על ידי אלקטרופורזה agarose ג'ל.
- חזור על בדיקת המושבה PCR עבור כל צד של האינטגרציה החזוי. בחר והרחב מועמדים המציגים נוכחות של אינטגרציה וללא מוצר אנדוגני רצף עבור האזור שנמחק.
- לאמת מועמדים חיוביים על ידי כתם המערבי ו / או RT-qPCR.
- איסוף lysate התא באמצעות ערכת הכנה DNA (ראה טבלת חומרים ).
8. אמת את המוטציה הגנומית על ידי רצף
- בידוד DNA גנומי מקווי תא מוטציה על ידי הפקת chloroform פנול 31 .
- PCR להגביר את אזור היעד sgRNA, באמצעות primers עם 5 'overhangs כי להוסיף אתרים הגבלת BSAI לכל קצה.
הערה: עבור שיבוטים שנערכו עם HDR, כאשר האללים עשויים להיות זהים, ניתן יהיה לרשום את מוצר ה- PCR ישירות. - לשכפל את האזור מוגבר לתוך pUC19-BSAI על ידי שיבוט שער הזהב.
- להפוך את תערובת התגובה לתוך זן E. המוסמכת coli .
- Miniprep DNA פלסמיד מ 6-10 מושבות בודדות רצף האזור משובטים על ידי רצף סנגר.
תוצאות
עבור הדור של שורות ELOWL1 נוקאאוט, נוגדנים חזקים היה זמין, כך העריכה באמצעות sgRNAs יחיד ( איור 1 א , משמאל) בוצעה, ואחריו אימונובלוט נקודה. שלושה sgRNAs היו transfected באופן עצמאי כדי להשוות את היעילות כדי לשלול את ההשפעות היעד שיבוטים וכתוצאה מכך. ?...
Discussion
מערכת CRISPR / Cas9 אפשרה ייצור יעיל של שינויים גנומיים יציבים, המספקים חלופה עקבית יותר לשיטות מניפולציה אחרות. הנה, הצגנו שתי שיטות לזיהוי מהיר של CRISPR / Cas9 גן knockouts בקווים תא יונקים. שתי השיטות דורשות חומר סלולרי קטן, ולכן הבדיקה יכולה להתבצע בשלבים המוקדמים של התרבות המשו...
Disclosures
המחברים מצהירים כי אין להם אינטרסים פיננסיים מתחרים.
Acknowledgements
המחברים היו רוצים להודות Gissell Sanchez, מייגן לי, וג 'ייסון Estep לסיוע ניסיוני, וייפנג גו ו Xuemei חן על שיתוף ריאגנטים.
Materials
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
References
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved