Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Отборочное и независимое генерирование CRISPR / Cas9-опосредованных генов-нокаутов в клетках млекопитающих
В этой статье
Резюме
Недавние достижения в способности генетически манипулировать соматическими клеточными линиями имеют большой потенциал для фундаментальных и прикладных исследований. Здесь мы представляем два подхода для производства и скрининга генерируемых нокаутом CRISPR / Cas9 в клеточных линиях млекопитающих с использованием и без использования селективных маркеров.
Аннотация
Инженерная система генома CRISPR / Cas9 произвела революцию в области биологии, позволив без особого труда точно редактировать геном. Управляемый одной направляющей РНК (sgRNA), которая придает специфичность, белок Cas9 расщепляет обе линии ДНК в целевом локусе. Разрыв ДНК может вызывать либо не гомологичное концевое соединение (NHEJ), либо гомологичный направленный ремонт (HDR). NHEJ может вводить небольшие делеции или вставки, которые приводят к мутациям смены кадров, тогда как HDR допускает большие и более точные возмущения. Здесь мы представляем протоколы для генерации клеточных линий нокаута путем связывания установленных методов CRISPR / Cas9 с двумя вариантами выбора / скрининга ниже по течению. Подход NHEJ использует единый сайт расщепления sgRNA и независимый от отбора скрининг, где производство белка оценивается точным иммуноблотом с высокой пропускной способностью. В методе HDR используются два сайта, разрезающих sgRNA, которые охватывают интересующий ген. Вместе с предоставленным шаблоном HDR этот метод может обеспечить удалениеИз десятков kb, чему способствует вставленный селективный маркер сопротивления. Обсуждаются соответствующие приложения и преимущества каждого метода.
Введение
Стабильные генетические изменения обеспечивают преимущество перед переходными методами клеточного возмущения, которые могут быть переменными по эффективности и продолжительности. Геномное редактирование становится все более распространенным в последние годы из-за развития целевых специфических нуклеаз, таких как нуклеазы цинка-пальца 1 , 2 , 3 , 4 , 5 , транскрипционные активатор-подобные эффекторные нуклеазы (TALENs) 6 , 7 , 8 , 9 И RNA-направленные нуклеазы, полученные из кластеризованной, регулярно интерполированной системы коротких палиндромных повторов (CRISPR) 10 .
Механизм редактирования CRISPR / Cas9 адаптирован из иммунной системы, которую используют бактерии и археи для защиты от вирусных инфекцийAss = "xref"> 11 , 12 , 13 . В этом процессе короткие, 20-30 н.т. фрагменты инвазионной вирусной последовательности включены в геномный локус в виде «спейсеров», фланкированных повторяющимися звеньями 14 , 15 . Последующая транскрипция и обработка РНК генерируют небольшие CRISPR-ассоциированные РНК 16 (crRNAs), которые вместе с транс-активирующей кРНК 17 (tracrRNA) собираются с эффекторной эндонуклеазой Cas9. Таким образом, кРНК обладают специфичностью к таргетированию каз9, направляя комплекс для расщепления комплементарных последовательностей вирусных ДНК и предотвращения дальнейших инфекций 18 , 19 . Любая последовательность «protospacer» в целевой ДНК может служить источником crRNA, если она непосредственно 5 'к короткому протоспасферу, смежному мотиву (PAM), NGG в случае S. pyogenes Cas9 20. Отсутствие последовательности PAM вблизи спейсера в локусе CRISPR хоста различает «я» и «не-я», предотвращая нацеливание хоста. Из-за своей универсальности и гибкости, эта биологическая система была сильно адаптирована для геномного редактирования, так что почти любой сайт, расположенный рядом с PAM, может быть нацелен. В этой версии дополнительная модификация сливала crRNA и tracrRNA в один направляющий РНК (sgRNA) компонент, который загружается в белок Cas9 21 .
При экспрессии Cas9 и sgRNA в эукариотических клетках белок Cas9 расщепляет обе линии ДНК в целевом локусе. В отсутствие подходящей области гомологичной последовательности клетка фиксирует этот разрыв через не гомологичное концевое соединение (NHEJ) 22 , 23 , 24 , которое обычно вводит небольшие делеции или, реже, вставки. При таргетинге на открытыйРамка считывания, ремонт, вероятно, приводит к поступательному сдвигу кадров, который создает нефункциональный белковый продукт. Напротив, при наличии экзогенной матрицы с большими областями гомологии клетка может фиксировать разрыв двух нитей с помощью гомологичного направленного ремонта 25 , 26 . Этот маршрут допускает более крупные точные удаления, замены или вставки в геноме в сочетании с введением маркеров подакцизного выделения 27 .
Здесь мы представляем протоколы для генерации клеточных линий нокаута одним из этих двух методов CRISPR / Cas9 ( рисунок 1A ). Подход NHEJ использует единый сайт расщепления sgRNA и независимый от отбора скрининг и, следовательно, требует небольшой предварительной подготовки. При использовании этого метода необходимо разработать направляющие РНК, дополняющие экзоны около 5'-конца транскрипта, которые, скорее всего, будут производить нокаут. Поскольку modificatИонов в этом геноме мало, скрининг на клоны для нокаутов основан на дот-блотах, где белковый продукт оценивается с высокой пропускной способностью. В качестве примера мы используем выработку линий выведения ELAV-like 1 (ELAVL1). Второй подход основан на восстановлении гомологии (HDR) и использует два сайта среза sgRNA, которые охватывают ген или область интереса, что позволяет удалять десятки килобайт. Плазмида с двумя регионами гомологии, которые расположены по участкам расщепления, обеспечивает шаблон замены ( фиг. 1B ), вводя селективный маркер сопротивления, который повышает эффективность генерации нокаута. Этот метод также может быть адаптирован для введения модификаций генов с правильно разработанными головками гомологии. В этом случае интеграция нового фрагмента ДНК позволяет проводить скрининг на основе ПЦР ( рис. 1С ). Здесь мы используем в качестве примера создание линий нокаута 2-го члена семейства Pumilio RNA (PUM2).
протокол
1. Идентификация областей гомологии вокруг желаемого удаления
ПРИМЕЧАНИЕ. Необходимо только при использовании редактирования на основе выбора.
- Выберите две области, первоначально 1,5-2 kb, по обе стороны от желаемого локуса для удаления, которые будут служить в качестве гомологического оружия в шаблоне HDR ( рис. 1A ). Определите регионы, в которых отсутствуют сайты распознавания BsaI ни на одной из сторон (GGTCTC), чтобы облегчить клонирование. Если сайты BsaI неизбежны, используйте альтернативный рестрикционный фермент IIs (BsmBI, SapI, BbsI) и модифицируйте соответствующие сайты в реципиентной плазмиде (pUC19-BsaI), резистентную маркерную донорную плазмиду (pGolden-Neo или pGolden-Hygro) и Плечи продукта PCR с гомологией.
2. Генерация плазмид экспрессии каз9-sgРНК
- Определите сайт (ы) для таргетинга для мутагенеза. Для метода однократной, без выбора, целевого 5 'проксимального кодирования экзонов для увеличения вероятности не-fuМутацией. Для метода двух разрезов выберите два сайта, которые по мере необходимости занимают столько же гена.
ПРИМЕЧАНИЕ. По нашему опыту эффективно удаляются до 53 кб. - Введите 200-300 бит целевой последовательности областей в инструмент проектирования crispr.mit.edu. Для клонирования на основе выбора используйте 200-300 пар оснований идентифицированных областей гомологии, которые расположены ближе к локусу для удаления ( рисунок 1A ). Выберите 2-3 из наиболее ранжированных sgRNAs для каждого целевого региона, чтобы учесть различия в их активности, и изолировать независимые клоны, которые не имеют одинаковых потенциальных побочных эффектов.
- Для создания дуплексных олигонуклеотидов с соответствующими выступами для введения в плазмиду экспрессии опустить конечную последовательность PAM (NGG) и добавить 5'-CACC-высадку, а затем G на олигонуклеотид верхней нити. Для олигонуклеотидов нижней нити добавьте 5'-AAAC навес к последовательности с обратной добавленной мишенью, за которой следует 3'-C, так как ilНиже:
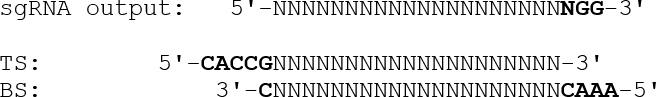
- Вставьте синтетические олигонуклеотиды, соответствующие sgRNAs в плазмиду pSpCas9 (BB) с использованием клонирования Golden Gate 28, как описано в лабораторном протоколе Zhang 29 .
3. Генерация гомологично-ориентированных шаблонов восстановления шаблонов
ПРИМЕЧАНИЕ. Необходимо только при использовании редактирования на основе выбора. Гомологически ориентированная восстанавливающая шаблонная плазмида состоит из кассеты с лекарственной устойчивостью, фланкированной двумя регионами, которые комплементарны геному, расположенному за пределами двух целевых сайтов sgRNA ( фиг. 1В ).
- Из более широких областей гомологий, определенных на шаге 1.1, выберите 800-1000 bp 26 не более чем на 5-10 б.п. от разработанных участков среза Cas9, чтобы служить в качестве оружия гомологии ( рис. 1A ). Не включайте sgRNAЦелевого сайта и его PAM в руках гомологии, поскольку это приведет к расщеплению CRISPR / Cas9 самой плазменной плазмы. Если в слоях гомологии имеются внутренние сайты BsaI, используйте альтернативные сайты sgRNA.
- ПЦР усиливают руки гомологии из геномной ДНК с использованием высоконадежной ДНК-полимеразы в соответствии с инструкциями производителя, используя прямые и обратные праймеры с дополнительной 5'-последовательностью, которая вводит сайты BsaI (GGTCTC) и уникальные выступы на обоих концах области гомологии. Выступы должны содержать правильные последовательности для формирования правильного порядка сборки и ориентации, как описано ниже ( рисунок 1B ):
Левая рука гомологии FP свеса: 5 'GGGTCTCA GGCC
Левый гомологический рычаг RP свес: 5 'GGGTCTCT CACG
Правый гомологический рычаг FP overhang: 5 'GGGTCTCA GTCC
Правый гомологический рычаг RP свес: 5 'GGGTCTCT CCAC - Проверьте оружие гомологии для соответствующего амплифаС помощью гель-электрофореза перед продолжением вперед.
- Соберите правую и левую области горизонтальной гомологии с помощью кассеты сопротивления в плазмиду шаблона HDR с помощью клонирования Golden Gate 28 . Используйте плазмиды pGolden-Neo или pGolden-Hygro 30 в качестве источника кассет сопротивления loxP-фланкированных (loxP-PGK-Neo-pA-loxP или loxP-PGK-Hyg-pA-loxP). Используйте pUC19-BsaI, модифицированный pUC19 с сайтами BsaI в области множественного клонирования и удалил сайты BsaI в другом месте, в качестве вектора-получателя (доступно по запросу). Используйте соотношение 1: 1: 1: 1 для трех вставок и вектора.
- Подготовьте следующую реакционную смесь 30 :
Правый гомологический рука PCR-продукт 0,06 пмоль (30-40 нг) Левый гомологический рука PCR-продукт 0,06 пмоль (30-40 нг) Плазмида pGolden-Neo (100 нг / мкл) 1 &# 181; L Плазмиду pUC19-BsaI (100 нг / мкл) 1 мкл 2x T7 ДНК-лигазный буфер 5 мкл BsaI (10 U / мкл) 0,75 мкл ДНК-лигазу T7 (3000 U / мкл) 0,25 мкл вода До 10 мкл - Используйте следующие параметры термоциклера: 37 ° С в течение 5 мин, 20 ° С в течение 5 мин. Повторите операцию на 30 циклов.
- Обработайте клонирующий продукт экзонуклеазой по инструкциям производителя, чтобы переварить оставшуюся линеаризованную ДНК.
- Превращают реакционную смесь в компетентный штамм E.coli в соответствии с протоколом, поставляемым с клетками, и планшет на ампициллинсодержащих чашках.
- Подготовьте следующую реакционную смесь 30 :
- Чтобы идентифицировать клоны с помощью правильной сборки шаблона, выполнитеЧтобы амплифицировать подошвы подгонки гомологии собранной вставки (поскольку вся вставка может быть слишком большой для надежного усиления). Используйте один отжиг праймера к фланкирующей последовательности плазмиды и второй праймер, дополняющий кромку кассеты с сопротивлением ( фиг. 1В , эта последовательность является общей как для вкладышей Neo, так и Hygro), генерируя зависимый от сборки продукт, который приблизительно на 200 б.п. больше, чем Содержащаяся гомология:
PUC19-Bsal-Left: 5 'GGCTCGTATGTTGTGTGGAATTGTGAG
Сопротивление слева: 5 'AAAAGCGCCTCCCCTACCC
PUC19-BsaI-Right: 5 'GCTATTACGCCAGCTGGCGAAA
Сопротивление справа: 5 'AAGACAATAGCAGGCATGCTGGG- Выбирайте колонии бактерий со стерильными зубочистками и суспендируйте бактерии в 20 мкл воды. Нагреть при 95 ° С в течение 15 мин и открутить в столешнице центрифугу при максимальной скорости в течение 5 мин. Поместите сразу на лед.
- Подготовьте следующую смесь ПЦР 31 </ SUP>:
Развернуть шаблон колонии 1,25 мкл 10x реакционный буфер Taq 1,25 мкл 20 мМ dNTP 0,25 мкл 10 мкМ Прямой праймер 0,25 мкл 10 мкМ Обратный праймер 0,25 мкл Taq-полимераза 0,25 мкл вода 9 мкл - Используйте следующие параметры термоциклера: 95 ° C в течение 30 с (95 ° C 30 с, 61 ° C в течение 30 с, 72 ° C в течение 1 мин / кб), повторите в течение 25 циклов, 72 ° C в течение 2 мин.
- Проверьте продукт ПЦР на правильный размер ампликона с помощью электрофореза в агарозном геле.
- По желанию, минипреп-плазмидная ДНК из отдельных колоний с правильными размерами вставки и последовательностьюE гомологичных областей секреции Sanger с упомянутыми выше усилительными праймерами.
4. Трансфекция компонентов CRISPR в культивируемые клетки
- До трансфекции: клетки культуры T-REx293 в среде DMEM, дополненные 10% FBS при 37 ° C и 5% CO 2 .
- Плиты клеток на 6-луночные планшеты и растут примерно до 70% слияния. Включите колодец для нетрансфекционного контроля.
- Трансфектные клетки с общей плазмидой 2,5 мкг с использованием протокола коммерческой трансфекции. Параллельно поддерживайте нетрансфицированный контроль.
ПРИМЕЧАНИЕ. Метод трансфекции и эффективность варьируются в зависимости от типа ячейки. Определите подходящий метод трансфекции для системы перед экспериментом.- Для трансфекции клеток с селекцией используйте 0,75 мкг Cas9-sgRNA 1 (левый срез), 0,75 мкг Cas9-sgRNA 2 (правый разрез) и образец гомологичной рекомбинации 1,0 мкг.
- Для трансфекцииНа клетках без отбора, используйте 2,5 мкг плазмиды Cas9-sgRNA.
5. Выбор лекарств
- Через 48 ч после трансфекции обрабатывают клетки соответствующим препаратом (неомицин / гигромицин). Выполните выбор до тех пор, пока все клетки в нетрансфицированном контрольном штампе (обычно 3-5 дней для Нео, 7-14 дней для Hygro).
ПРИМЕЧАНИЕ. Используйте концентрации 500 мкг / мл и 10 мкг / мл для неомицина и гигромицина соответственно для клеток T-REx293. Для других типов клеток может быть полезно провести предварительное титрование лекарственного средства на нетрансфицированных клетках для определения эффективной концентрации.
6. Изоляция клональных групп населения
- Выращивайте клетки до 100% слияния в исходном колоде после отбора. Высевают клетки в 96-луночные планшеты с плотностью 0,33 клеток на лунку.
ПРИМЕЧАНИЕ. Поселение трех 96-луночных планшетов является хорошей отправной точкой для обеспечения изоляции более чем одного правильного клона, но может потребоваться более или менееЗаканчивая эффективностью sgRNA и HDR. - Наблюдайте за колониями в течение 2-4 недель, пока колонии не будут видны глазу. Выбирайте видимые колонии со стерильным наконечником пипетки и подавайте в новые лунки, чтобы стимулировать рост монослоев. При использовании суспензионных клеток даже более низкая плотность посева может быть использована для обеспечения большей доли колоний с колониями.
7. Кандидаты на скрининг
- Отбор кандидатов без использования выбора: дот-блот
- Растут отдельные клоны до 50-100% слияния. Выдвиньте монослой клеток путем пипетирования внутри колодца.
- Аликвоту 90 мкл общего объема 100 мкл в чистую микроцентрифужную пробирку. Скручивают при 6000 об / мин в течение 5 мин, удаляют среду и осаждают лизис клеток в 10 мкл 1x буфера для лизиса или 1x буфера для загрузки SDS (5x: 250 мМ Tris-Cl, pH 6,8, 8% SDS, 0,1% бромфенолового синего, 40% Глицерин, 100 мМ DTT). Добавьте 90 мкл новых носителей в оставшуюся часть клеток вL продолжить распространение.
- Внесите 1 мкл клеточного лизата на сухую нитроцеллюлозную мембрану, чтобы сформировать точку. Промойте каждый образец дважды на две отдельные мембраны, создавая две идентичные образцы образцов.
- Блокировать мембраны в 5% молоке в TBST (Трис-буферный солевой раствор + 0,01% Твин 20: 8 г NaCl, 0,2 г KCl, 3 г Трис-основания, до 1 л дистиллированной воды, pH 7,4) 31 в течение 1 часа при комнатной температуре ( С качанием, здесь и на протяжении всей процедуры).
- Блот с использованием первичного антитела против целевого белка на одной мембране и первичное антитело для контрольного белка (тубулин, GAPDH или любой другой белок, который, как ожидается, не изменится), с другой. Используйте рекомендуемое первичное разведение антител (0,2 мкг / мл для ELAVL1 и 0,1 мкг / мл для Pum2 в этом случае) в TBST + 5% молока. Инкубируйте 1 ч при комнатной температуре.
- Промыть 3 раза TBST в течение 5 мин.
- Инкубируйте каждую мембрану с соответствующим HRP-конъюгированным вторичным антителом в TBST + 5% молока в течение 1 часа при комнатной температуре.
- Промыть 3 раза TBST в течение 5 мин.
- Примените раствор хемилюминесцентного субстрата (см. Таблицу материалов) в соответствии с инструкциями производителя и изобразите сфотографированную мембрану на цифровом хемилюминесцентном изображении.
- Определите количественные значения интенсивностей точек для целевого и контрольного белков с использованием соответствующего программного обеспечения.
- Исключить кандидатов с низким сигналом контрольного белка и рассчитать фоновые вычитаемые отношения мишени для контроля интенсивности белка для остальных кандидатов. Выберите кандидатов с самыми низкими коэффициентами для прохода и дальнейшей проверки с помощью вестерн-блоттинга.
- Скрининг кандидатов с использованием селекции: колония PCR
- Собирайте лизат клеток с помощью набора для подготовки ДНК (см. Таблицу материалов ).
- Дублируйте отдельные колонии в новом 96-луночном колоде и произрастайте до 100% слияния.
- Удалите носитель из одного набора клонов,N ресуспендируют клетки в 30 мкл экстракционного буфера из набора. Перенесите в чистую 1,5-миллилитровую микроцентрифужную пробирку.
- Нагреть раствор до 96 ° С в течение 15 мин и дать охладиться до комнатной температуры.
- Добавьте 30 мкл стабилизирующего буфера из набора. Хорошо перемешать.
- Определите дикого типа и моноаллельные / биаллельные мутантные линии методом колонии PCR.
- Разработайте два отдельных набора ПЦР-праймеров (для левой и правой стороны целевого локуса), чтобы усилить области вокруг рук гомологии на основе успешной или неудачной интеграции резистивной кассеты ( рисунок 2C ). С каждой стороны используйте общий праймер (красный), который отжигает вне руки гомологии. Используйте его с соответствующим парным праймером, который дополняет эндогенную последовательность, охватывая область гомологии (синий), для проверки аллеля дикого типа. Используйте другой парный праймер, дополняющий вставленную резистентную кассету (см. Праймеры в разделе2.3), чтобы проверить желаемую мутацию.
- (Рекомендуется) Проверьте набор праймеров с использованием клеточного лизата из исходной выбранной популяции массивных клеток, так как он будет содержать смесь как WT, так и мутантных аллелей ( рисунок 2D ).
- Подготовьте следующую реакционную смесь:
Клеточный лизат 0,5 мкл Буфер 10x KOD 1,25 мкл 25 мМ MgSO4 0,75 мкл 2 мМ dNTP 1,25 мкл 10 мкМ Прямой праймер 0,375 мкл 10 мкМ Обратный праймер 0,375 мкл Полимераза KOD 0,25 мкл вода 7,75 мкл - Используйте следующие условия термоциклов: 95 ° C в течение 2 минут (95 ° C в течение 20 с, Primer Tm в течение 10 с, 70 ° C в течение 20 с / кб) повторите в течение 25 циклов.
- Визуализируйте продукт ПЦР с помощью электрофореза в агарозном геле.
- Повторите тестирование ПЦР колонии для каждой стороны прогнозируемой интеграции. Выбирайте и расширяйте кандидатов, которые показывают наличие интеграции и отсутствие продукта эндогенной последовательности для удаленной области.
- Подтвердите позитивные кандидаты вестерн-блот и / или RT-qPCR.
- Собирайте лизат клеток с помощью набора для подготовки ДНК (см. Таблицу материалов ).
8. Проверьте геномную мутацию путем секвенирования
- Выделение геномной ДНК из мутантных клеточных линий путем экстракции фенольным хлороформом 31 .
- ПЦР амплифицируют целевую область sgRNA, используя праймеры с 5'-выступами, которые добавляют сайты рестрикции BsaI к каждому концу.
ПРИМЕЧАНИЕ. Для клонов, отредактированных с помощью HDR, где оба аллеля могут быть идентичными, продукт ПЦР можно секвенировать напрямую, - Клонировать амплифицированную область в pUC19-BsaI путем клонирования Golden Gate.
- Превратите реакционную смесь в компетентный штамм E.coli .
- ДНК плазмиды Miniprep из 6-10 отдельных колоний и последовательность клонированной области с помощью секвенирования Сангера.
Результаты
Для генерации линий нокаута ELAVL1 было доступно надежное антитело, поэтому было выполнено редактирование с использованием одиночных sgRNAs ( рис. 1A , слева), а затем точечный иммуноблот. Три sgRNAs трансфицировали независимо, чтобы сравнить эффективность и иск...
Обсуждение
Система CRISPR / Cas9 позволила обеспечить эффективную генерацию стабильных геномных модификаций, которые обеспечивают более последовательную альтернативу другим способам временного манипулирования. Здесь мы представили два метода быстрой идентификации нокаутов гена CRISPR / Cas9 в клеточны...
Раскрытие информации
Авторы заявляют, что у них нет конкурирующих финансовых интересов.
Благодарности
Авторы хотели бы выразить признательность Гисселу Санчесу, Меган Ли и Джейсону Эстепу за экспериментальную помощь, а также Вайфэн Гу и Сюэми Чэнь за обмен реагентами.
Материалы
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
Ссылки
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены