Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Generación dependiente de la selección y independiente de CRISPR / Cas9-mediada Gene Knockouts en células de mamíferos
En este artículo
Resumen
Los recientes avances en la capacidad de manipular genéticamente las líneas celulares somáticas tienen un gran potencial para la investigación básica y aplicada. En este sentido, presentamos dos enfoques para CRISPR / Cas9 generado producción de eliminación y el cribado en líneas de células de mamíferos, con y sin el uso de marcadores seleccionables.
Resumen
El sistema de ingeniería del genoma CRISPR / Cas9 ha revolucionado la biología permitiendo la edición precisa del genoma con poco esfuerzo. Guiado por una sola guía de ARN (sgRNA) que confiere especificidad, la proteína Cas9 escinde ambas cadenas de ADN en el lugar de destino. La ruptura del ADN puede desencadenar la unión final no homóloga (NHEJ) o la reparación dirigida por homología (HDR). NHEJ puede introducir pequeñas supresiones o inserciones que conducen a mutaciones de cambio de marco, mientras que el HDR permite perturbaciones más grandes y más precisas. Aquí, presentamos protocolos para la generación de líneas celulares knockout mediante el acoplamiento CRISPR / Cas9 métodos establecidos con dos opciones para la selección / cribado aguas abajo. El enfoque de NHEJ utiliza un único sitio de corte sgRNA y selección independiente de la selección, donde la producción de proteínas se evalúa por punto inmunoblot en un alto rendimiento. El enfoque HDR utiliza dos sitios de corte sgRNA que abarcan el gen de interés. Junto con una plantilla de HDR proporcionada, este método puede lograr supresiónDe decenas de kb, ayudado por el marcador de resistencia seleccionable insertado. Se discuten las aplicaciones y ventajas apropiadas de cada método.
Introducción
Las alteraciones genéticas estables proporcionan una ventaja sobre los métodos transitorios de perturbación celular, que pueden ser variables en su eficacia y duración. La edición genómica se ha vuelto cada vez más común en los últimos años debido al desarrollo de nucleasas específicas para el objetivo, como las nucleasas de zinc-dedo 1 , 2 , 3 , 4 , 5 , nucleasas efectoras semejantes a activadores de transcripción (TALENs) 6 , 7 , 8 , 9 Y ARN-guiado nucleasas derivadas de las agrupadas, regularmente intercaladas palindromic corto repite (CRISPR) sistema [ 10] .
La maquinaria de edición CRISPR / Cas9 está adaptada de un sistema inmunológico que las bacterias y arqueas utilizan para defenderse contra infecciones viralesAsno = "xref"> 11 , 12 , 13 . En este proceso, fragmentos cortos de 20-30 nt de secuencia viral invasora se incorporan en un locus genómico como "espaciadores" flanqueados por unidades repetidas 14 , 15 . La transcripción subsiguiente y el procesamiento de ARN genera ARNs 16 (CRRNAs) asociados a CRISPR pequeños que, junto con un ARNc trans-activador 17 (tracrRNA), se reúnen con la endonucleasa Cas9 efectora. Los CRRNAs, por lo tanto, proporcionar la especificidad de Cas9 orientación, la guía del complejo para escindir secuencias complementarias de ADN viral y la prevención de nuevas infecciones [ 18 , 19] . Cualquier secuencia "protospacer" en el ADN diana puede servir como fuente del CRRNA, siempre y cuando esté directamente 5 'con un motivo adyacente protospacer corto (PAM), NGG en el caso de S. pyogenes Cas9 20. La ausencia de la secuencia de PAM cerca del espaciador en el locus CRISPR del anfitrión distingue entre el yo y el no-yo, evitando el objetivo del huésped. Debido a su universalidad y flexibilidad, este sistema biológico ha sido poderosamente adaptado para la edición genómica, de tal manera que casi cualquier sitio de ADN PAM adyacente puede ser dirigido. En esta versión, una modificación adicional fundido el crRNA y tracrRNA en un único ARN guía (sgRNA) componente que se carga en el Cas9 proteína [ 21] .
Tras la expresión de Cas9 y un sgRNA en células eucariotas, la proteína Cas9 escinde ambas cadenas de ADN en el locus diana. En ausencia de una región apropiada de secuencia homóloga, la célula fija esta ruptura mediante la unión final no homóloga (NHEJ) 22 , 23 , 24 , que típicamente introduce pequeñas deleciones o, en raras ocasiones, inserciones. Al orientar unaLectura, la reparación conduce probablemente a un desplazamiento de traslación que produce un producto proteico no funcional. Por el contrario, cuando se proporciona una plantilla exógena con grandes regiones de homología, la célula puede fijar la ruptura de doble hebra mediante reparación dirigida por homología 25 , 26 . Esta ruta permite mayores deleciones precisas, sustituciones o inserciones en el genoma, junto con la introducción de marcadores de selección excisable [ 27] .
Aquí, presentamos protocolos para generar knockout líneas celulares por cualquiera de estos dos CRISPR / Cas9 métodos ( Figura 1A ]. El enfoque de NHEJ utiliza un solo sitio de corte sgRNA y selección independiente de la selección, y por lo tanto requiere poca preparación inicial. Cuando se utiliza este método, deben diseñarse RNAs guía complementarios a los exones cerca del extremo 5 'de la transcripción, que son más probables para producir un knockout. Desde la modificaciónIones al genoma en este caso son pequeños, el cribado para los clones knockout se basa en transferencias de punto, donde el producto proteico se evalúa de una manera de alto rendimiento. Utilizamos la generación de líneas de knockout de ELAV-like 1 protein (ELAVL1) como ejemplo. El segundo enfoque se basa en la reparación dirigida por homología (HDR) y utiliza dos sitios de corte de sgRNA que abarcan el gen o región de interés, permitiendo deleciones de decenas de kb. Un plásmido con dos regiones de homología que flanquean los sitios de escisión proporciona una plantilla de reemplazo ( Figura 1B ), introduciendo un marcador de resistencia seleccionable que aumenta la eficiencia de la generación de knockout. Este método también puede adaptarse para introducir modificaciones genéticas con brazos de homología diseñados apropiadamente. En este caso, la integración de un nuevo fragmento de ADN permite el cribado basado en PCR ( Figura 1C ). Aquí, utilizamos la generación de Pumilio RNA vinculante familia miembro 2 (PUM2) knockout líneas como un ejemplo.
Protocolo
1. Identificación de regiones de homología alrededor de la deleción deseada
NOTA: Sólo es necesario si se utiliza la edición basada en la selección.
- Seleccione dos regiones, inicialmente de 1,5-2 kb, a cada lado del locus de deleción deseado, que servirá como brazos de homología en la plantilla HDR ( Figura 1A ). Identificar las regiones que carecen de sitios de reconocimiento BsaI en cada capítulo (GGTCTC) para facilitar la clonación. Si los sitios BsaI son inevitables, utilice una enzima de restricción alternativa de tipo IIs (BsmBI, SapI, BbsI) y modifique los sitios correspondientes en el plásmido receptor (pUC19-BsaI), el plásmido donante marcador de resistencia (pGolden-Neo o pGolden-Hygro) y Homología brazo productos PCR sobresale.
2. Generación de plásmidos de expresión de Cas9-sgRNA
- Definir el (los) sitio (s) de orientación para la mutagénesis. Para el método de corte único, sin selección, los exones proximales de codificación 5 'para aumentar la probabilidad de un no fuMutación funcional. Para el método de dos cortes, seleccione dos sitios que abarquen tanto del gen como sea necesario.
NOTA: En nuestra experiencia, las supresiones de hasta 53 kb se crean eficientemente. - Introduzca 200 a 300 pb de la secuencia de la región objetivo en la herramienta de diseño crispr.mit.edu. Para la clonación basada en selección, utilice 200-300 pb de las regiones de homología identificadas que son proximales al locus de deleción ( Figura 1A ). Seleccione 2-3 de los sgRNAs de mayor rango por región objetivo para tener en cuenta las diferencias en su actividad y para aislar clones independientes que no compartan los mismos potenciales efectos fuera de la meta.
- Para diseñar oligonucleótidos dúplex con salientes adecuados para su inserción en el plásmido de expresión, omitir la secuencia de PAM final (NGG) y añadir un saliente de 5'-CACC seguido por un G al oligo de la hebra superior. Para el oligo de la hebra inferior, añada un saliente 5'-AAAC a la secuencia diana complementada con el reverso, seguido por un 3'-C, como ilIlustrado a continuación:
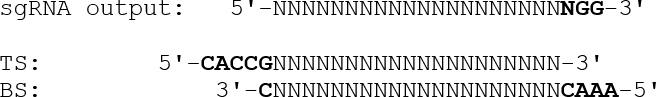
- Insertar oligonucleótidos sintéticos correspondientes a los sgRNAs en el plásmido pSpCas9 (BB) utilizando la clonación Golden Gate 28 como se describe en el protocolo de laboratorio Zhang [ 29] .
3. Generación de Plásmidos Plantilla de Reparación dirigida por Homología
NOTA: Sólo es necesario si se utiliza la edición basada en la selección. El plásmido plantilla de reparación dirigida por homología consiste en un casete de resistencia a fármacos flanqueado por dos regiones que son complementarias al genoma justo fuera de los dos sitios diana sgRNA ( Figura 1B ).
- A partir de las regiones de homología más amplias identificadas en el paso 1.1, seleccionar 800-1.000 pb 26 no más de 5-10 pb de los sitios de corte Cas9 diseñados, para servir como brazos de homología ( Figura 1A ). Asegúrese de no incluir el sgRNAEl sitio diana y su PAM en los brazos de homología, ya que esto causará la escisión CRISPR / Cas9 del propio plásmido molde. Si los brazos de homología contienen sitios BsaI internos, utilice sitios alternativos de sgRNA.
- PCR amplifica los brazos de homología a partir del ADN genómico usando una ADN polimerasa de alta fidelidad, según las instrucciones del fabricante, usando cebadores directos e inversos con secuencia 5 'adicional que introduce sitios BsaI (GGTCTC) y salientes únicos en cada extremo de la región de homología. Los salientes deben contener secuencias apropiadas para generar el orden de montaje y orientación correctos, como se describe a continuación ( Figura 1B ):
Homología izquierda brazo FP overhang: 5 'GGGTCTCA GGCC
Brazo de homología izquierda RP prominente: 5 'GGGTCTCT CACG
Homología derecha brazo FP overhang: 5 'GGGTCTCA GTCC
Brazo de homología derecha brazo de RP: 5 'GGGTCTCT CCAC - Comprobar los brazos de homología para el amplificador apropiadoPor electroforesis en gel antes de continuar adelante.
- Reúna las regiones del brazo de homología derecha e izquierda con el casete de resistencia en un plásmido molde HDR por clonación Golden Gate [ 28] . Utilizar plásmidos pGolden-Neo o pGolden-Hygro 30 como fuente de los casetes de resistencia flanqueados con loxP (loxP-PGK-Neo-pA-loxP o loxP-PGK-Hyg-pA-loxP). Utilizar pUC19-BsaI, un pUC19 modificado con sitios BsaI en la región de clonación múltiple y eliminar sitios BsaI en otra parte, como el vector receptor (disponible a petición). Utilice una proporción de 1: 1: 1: 1 para los tres insertos y el vector.
- Preparar la siguiente mezcla de reacción 30 :
Producto de PCR del brazo de homología derecha 0,06 pmoles (30-40 ng) Producto de PCR del brazo de homología izquierdo 0,06 pmoles (30-40 ng) El plásmido pGolden - Neo (100 ng / μl) 1 &# 181; L PUC19 - BsaI (100 ng / μl) 1 μl 2x tampón de ADN ligasa T7 5 μL BsaI (10 U / μl) 0,75 μL T7 ADN ligasa (3000 U / μl) 0,25 μL Agua Hasta 10 μL - Utilice los siguientes parámetros del termociclador: 37 ° C durante 5 min, 20 ° C durante 5 min. Repita durante 30 ciclos.
- Tratar el producto de clonación con una exonucleasa según las instrucciones del fabricante para digerir cualquier ADN lineal restante.
- Transformar la mezcla de reacción en una cepa E. coli competente de acuerdo con el protocolo suministrado con las células, y la placa sobre platos que contienen ampicilina.
- Preparar la siguiente mezcla de reacción 30 :
- Para identificar clones con el conjunto de plantilla correcto, realizar bacteriasAl colonia PCR para amplificar las subregiones del brazo de homología del inserto montado (ya que el inserto completo puede ser demasiado grande para amplificarse de forma fiable). Utilizar un recocido de cebador a la secuencia de plásmido flanqueante y un segundo cebador complementario al borde del cassette de resistencia ( Figura 1B , la secuencia es común a los insertos Neo e Hygro), generando un producto dependiente del ensamblaje que es aproximadamente 200 pb más largo que El brazo de homología contenida:
PUC19 - Bsal - Izquierda: 5 'GGCTCGTATGTTGTGTGGAATTGTGAG
Resistencia-Izquierda: 5 'AAAAGCGCCTCCCCTACCC
PUC19 - BsaI - Derecha: 5 'GCTATTACGCCAGCTGGCGAAA
Resistencia-Derecha: 5 'AAGACAATAGCAGGCATGCTGGG- Elija colonias bacterianas con mondadientes estériles y suspenda las bacterias en 20 μl de agua. Calentar a 95 ° C durante 15 min, y girar en una centrífuga de mesa a la velocidad máxima durante 5 min. Coloque inmediatamente sobre hielo.
- Preparar la siguiente mezcla de PCR 31 </ Sup>:
Plantilla de colonia diluida 1,25 mu l Tampón de reacción Taq 10x 1,25 mu l 20mM dNTPs 0,25 μL 10 μM Imprimación directa 0,25 μL 10 μM Imprimación inversa 0,25 μL Taq Polimerasa 0,25 μL Agua 9 μL - Utilizar los siguientes parámetros del termociclador: 95 ° C durante 30 s, (95 ° C 30 s, 61 ° C durante 30 s, 72 ° C durante 1 min / kb), repetir durante 25 ciclos, 72 ° C durante 2 min.
- Compruebe el producto de PCR para el tamaño correcto del amplicón mediante electroforesis en gel de agarosa.
- Opcionalmente, el ADN de plásmido de miniprep de colonias individuales con tamaño de inserto correcto y secuenciaE por secuenciación de Sanger con los cebadores de amplificación mencionados anteriormente.
4. Transfección de componentes CRISPR en células cultivadas
- Antes de la transfección: cultivar células T-REx293 en medio DMEM suplementado con FBS al 10% a 37ºC y CO 2 al 5%.
- Placa de células en placas de 6 pozos y crecer hasta aproximadamente el 70% de confluencia. Incluya un pozo para un control no transfectado.
- Transfect células con 2,5 μ g plásmido total utilizando un protocolo de transfección comercial. En paralelo, mantener el control no transfectado.
NOTA: El método de transfección y la eficiencia varían dependiendo del tipo de célula. Determine el método de transfección apropiado para el sistema antes del experimento.- Para la transfección de células con selección, se utilizan 0,75 μg de Cas9-sgRNA 1 (corte a la izquierda), 0,75 μg de Cas9-sgRNA 2 (corte derecho) y 1,0 μg de plantilla de recombinación homóloga.
- Para transfectiDe las células sin selección, se utiliza 2,5 μ g de Cas9-sgRNA plásmido.
5. Selección de fármacos
- 48 h después de la transfección, tratar las células con el fármaco apropiado (Neomicina / Higromicina). Llevar a cabo la selección hasta que todas las células en el control no transfectado mueren (normalmente 3-5 días para Neo, 7-14 días para Hygro).
NOTA: Use concentraciones de 500 μg / mL y 10 μg / mL para Neomicina e Higromicina, respectivamente para las células T-REx293. Para otros tipos de células, puede ser útil llevar a cabo una titulación previa de fármaco en células no transfectadas para determinar la concentración efectiva.
6. Aislamiento de las poblaciones clonales
- Cultivar las células hasta el 100% de confluencia en el pocillo original después de la selección. Seed células en placas de 96 pozos a una densidad de 0,33 células por pocillo.
NOTA: La siembra de tres placas de 96 pocillos es un buen punto de partida para asegurar el aislamiento de más de un clon correcto, pero se puede necesitar más o menos depTerminando en sgRNA y HDR eficiencias. - Observe las colonias durante un período de 2-4 semanas, hasta que las colonias sean visibles al ojo. Elija colonias visibles con una punta de pipeta estéril y vuelva a sembrar en nuevos pozos para estimular el crecimiento de monocapa. Cuando se usan células en suspensión, incluso se pueden utilizar densidades de siembra más bajas para asegurar una mayor proporción de pozos de una sola colonia.
7. Selección de Candidatos
- Selección de candidatos sin el uso de la selección: dot blot
- Crecer clones individuales a 50-100% de confluencia. Desalojar la monocapa de las células pipeteando dentro del pozo.
- Alícuota de 90 μL del volumen total de 100 μL a un tubo de microcentrífuga limpio. Se centrifuga a 6000 rpm durante 5 min, se elimina el medio y se liza el sedimento celular en 10 μl de tampón de lisis 1x o tampón de carga SDS 1x (5x: Tris-Cl 250 mM pH 6,8, SDS al 8%, azul de bromofenol al 0,1% Glicerol, DTT 100 mM). Añadir 90 μl de nuevos medios al resto de las células en elL para continuar propagando.
- Pipetear 1 μl de lisado celular sobre una membrana de nitrocelulosa seca para formar un punto. Blot cada muestra dos veces en dos membranas separadas, creando dos patrones idénticos de muestras.
- Bloquear las membranas en 5% de leche en TBST (solución salina tamponada con Tris + 0,01% de Tween 20: 8 g de NaCl, 0,2 g de KCl, 3 g de base de Tris, hasta 1 l de agua destilada, pH 7,4) 31 durante 1 h a temperatura ambiente Con balanceo, aquí y durante todo el procedimiento).
- Blot usando el anticuerpo primario contra la proteína diana en una membrana, y el anticuerpo primario para una proteína de control (tubulina, GAPDH, o cualquier otra proteína que no se espera que cambie) en el otro. Utilice la dilución recomendada de anticuerpos primarios (0,2 μg / mL para ELAVL1 y 0,1 μg / mL para Pum2 en este caso) en TBST + 5% de leche. Incubar 1 h a temperatura ambiente.
- Lavar 3 veces con TBST durante 5 min.
- Incubar cada membrana con anticuerpo secundario conjugado con HRP apropiado en TBST + 5% de leche durante 1 h a temperatura ambiente.
- Lavar 3 veces con TBST durante 5 min.
- Aplique una solución de sustrato de quimioluminiscencia (véase Tabla de Materiales) siguiendo las instrucciones del fabricante e imprima la membrana manchada en un dispositivo de imagen de quimioluminiscencia digital.
- Cuantificar las intensidades de los puntos para las proteínas objetivo y de control utilizando el software apropiado.
- Eliminar a los candidatos con baja señal de proteína de control, y calcular el fondo substraído ratios de objetivo para controlar la intensidad de proteínas para los candidatos restantes. Seleccione los candidatos con las proporciones más bajas para el paso y la posterior validación por transferencia western.
- Selección de candidatos con el uso de la selección: PCR de colonias
- Recoger el lisado celular usando un kit de preparación de ADN (Ver Tabla de Materiales ).
- Duplicar las colonias individuales en un pozo de 96 nuevos y crecer hasta 100% de confluencia.
- Eliminar los medios de un conjunto de los clones, unY resuspender las células en 30 μl de tampón de extracción del kit. Transferir a un tubo de microcentrífuga limpio de 1,5 ml.
- Calentar la solución a 96 ° C durante 15 min y dejar enfriar a temperatura ambiente.
- Añadir 30 μL de tampón de estabilización del kit. Mezclar bien.
- Identificar líneas mutantes de tipo salvaje y monoalélicas / bialélicas mediante PCR de colonias.
- Diseñar dos conjuntos separados de cebadores de PCR (para el lado izquierdo y derecho del locus objetivo) para amplificar las regiones alrededor de los brazos de homología basado en la integración exitosa o fallida del casete de resistencia ( Figura 2C ). En cada lado, utilice un cebador común (rojo) que se recoje fuera del brazo de homología. Se utiliza con un cebador pareado correspondiente que es complementario a la secuencia endógena, que abarca la región de homología (azul), para ensayar el alelo de tipo salvaje. Utilice otro cebador pareado, complementario al casete de resistencia insertado (ver cebadores en la sección2.3), para probar la mutación deseada.
- (Recomendado) Validar los conjuntos de cebadores usando lisado celular de la población de células a granel original seleccionada, ya que contendrá una mezcla de ambos WT y alelos mutantes ( Figura 2D ).
- Preparar la siguiente mezcla de reacción:
Lisado celular 0,5 μl 10 x tampón KOD 1,25 mu l MgSO _ {4} 25 mM 0,75 μL DNTPs 2 mM 1,25 mu l 10 μM Imprimación directa 0,375 μL 10 μM Imprimación inversa 0,375 μL KOD polimerasa 0,25 μL Agua 7,75 μL - Utilizar las siguientes condiciones del termociclador: 95 ° C durante 2 min, (95 ° C durante 20 s, Primer Tm durante 10 s, 70 ° C durante 20 s / kb) repetir durante 25 ciclos.
- Visualizar el producto de PCR mediante electroforesis en gel de agarosa.
- Repita la prueba de PCR de colonias para cada lado de la integración prevista. Seleccione y expanda candidatos que muestren presencia de integración y ningún producto de secuencia endógena para la región suprimida.
- Validar los candidatos positivos por Western blot y / o RT-qPCR.
- Recoger el lisado celular usando un kit de preparación de ADN (Ver Tabla de Materiales ).
8. Verificar la mutación genómica mediante secuenciación
- Aislar el ADN genómico de líneas celulares mutantes por extracción de fenol cloroformo 31 .
- La PCR amplifica la región diana de sgRNA, utilizando cebadores con salientes 5 'que añaden sitios de restricción BsaI a cada extremo.
NOTA: Para clones editados con HDR, donde ambos alelos pueden ser idénticos, el producto de PCR puede secuenciarse directamente. - Clonar la región amplificada en pUC19-BsaI por clonación Golden Gate.
- Transformar la mezcla de reacción en una cepa E. coli competente.
- Miniprep ADN plasmídico de 6-10 colonias individuales y secuencia de la región clonada por secuenciación de Sanger.
Resultados
Para la generación de ELAVL1 knockout líneas, un anticuerpo robusto estaba disponible, por lo que la edición utilizando sgRNAs único ( Figura 1A , izquierda) se realizó, seguido de inmunotransferencia punto. Se transfectaron tres sgRNAs de forma independiente para comparar las eficiencias y para descartar los efectos deseados en los clones resultantes. Después de recoger y borrar los lisados celulares de las poblaciones clonales en dos membranas ...
Discusión
El sistema CRISPR / Cas9 ha permitido la generación eficiente de modificaciones genómicas estables, que proporcionan una alternativa más consistente a otros métodos de manipulación transitorios. Aquí, hemos presentado dos métodos para la rápida identificación de CRISPR / Cas9 genes knockouts en líneas de células de mamíferos. Ambos métodos requieren poco material celular, por lo que las pruebas se pueden hacer en las primeras etapas del cultivo clonal, ahorrando tiempo y reactivos. Para aumentar la eficienc...
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Los autores desean agradecer a Gissell Sanchez, Megan Lee y Jason Estep por la asistencia experimental, y Weifeng Gu y Xuemei Chen por compartir reactivos.
Materiales
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
Referencias
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados