É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Geração independente dependente da seleção e de genes independentes de CRISPR / Cas9 em células de mamíferos
Neste Artigo
Resumo
Os avanços recentes na capacidade de manipular geneticamente as linhas celulares somáticas possuem grande potencial para pesquisas básicas e aplicadas. Aqui, apresentamos duas abordagens para CRISPR / Cas9 gerado produção knockout e triagem em linhas de células de mamíferos, com e sem o uso de marcadores selecionáveis.
Resumo
O sistema de engenharia do genoma CRISPR / Cas9 revolucionou a biologia ao permitir uma edição precisa do genoma com pouco esforço. Guiado por um único guia de RNA (sgRNA) que confere especificidade, a proteína Cas9 cliva ambas as cadeias de DNA no locus alvo. A ruptura do DNA pode desencadear união final não homóloga (NHEJ) ou reparação direcionada a homologia (HDR). O NHEJ pode introduzir pequenas deleções ou inserções que levam a mutações de mudança de quadro, enquanto o HDR permite perturbações maiores e mais precisas. Aqui, apresentamos protocolos para geração de linhas celulares knockout através do acoplamento de métodos CRISPR / Cas9 estabelecidos com duas opções para seleção / rastreio a jusante. A abordagem NHEJ usa um único local de corte de sgRNA e rastreio independente de seleção, onde a produção de proteína é avaliada por imunotransferência ponto de uma maneira de alto rendimento. A abordagem HDR usa dois sites de corte de sgRNA que abrangem o gene de interesse. Juntamente com um modelo de HDR fornecido, esse método pode ser excluídoDe dezenas de kb, auxiliado pelo marcador de resistência selecionado inserido. São discutidas as aplicações e vantagens apropriadas de cada método.
Introdução
Alterações genéticas estáveis oferecem uma vantagem sobre os métodos transitórios de perturbação celular, que podem ser variáveis em sua eficiência e duração. A edição genômica tornou-se cada vez mais comum nos últimos anos devido ao desenvolvimento de nucleases específicas do alvo, como as nucleases de zinco-dedo 1 , 2 , 3 , 4 , 5 , as nucleases efetoras do tipo ativador da transcrição (TALENs) 6 , 7 , 8 , 9 E nucleases guiadas por RNA derivadas do sistema de repetições palindrômicas curtas agrupadas, regularmente intercaladas (CRISPR) 10 .
A maquinaria de edição CRISPR / Cas9 é adaptada de um sistema imunológico que bactérias e archaea usam para se defender contra infecções viraisAss = "xref"> 11 , 12 , 13 . Neste processo, fragmentos curtos de 20-30 nt de sequência viral invasora são incorporados em um locus genômico como "espaçadores" flanqueados por unidades repetitivas 14 , 15 . A transcrição subsequente e o processamento de RNA geram pequenos RNAs 16 (CRRNAs) associados ao CRISPR que, juntamente com um CRRNA 17 (traRRNA) trans-ativador, se reúnem com a endonuclease efectora Cas9. Os crRNAs proporcionam assim especificidade ao alvo de Cas9, guiando o complexo para separar sequências de DNA viral complementares e prevenir novas infecções 18 , 19 . Qualquer sequência de "protospacer" no DNA alvo pode servir como fonte do crRNA, desde que seja diretamente 5 'para um motivo adjacente prótespacer curto (PAM), NGG no caso de S. pyogenes Cas9 20. A ausência da sequência PAM perto do espaçador no locus CRISPR do host distingue-se de si mesmo e não-auto, evitando a segmentação do hospedeiro. Devido à sua universalidade e flexibilidade, este sistema biológico foi poderosamente adaptado para a edição genômica, de modo que quase qualquer site de DNA adjacente ao PAM pode ser direcionado. Nesta versão, uma modificação adicional fundiu o crRNA e o tracrRNA em um único componente guia RNA (sgRNA) que é carregado na proteína Cas9 21 .
Após a expressão de Cas9 e um sgRNA em células eucarióticas, a proteína Cas9 corta as duas cadeias de DNA no locus alvo. Na ausência de uma região adequada de sequência homóloga, a célula corrige esta ruptura através de união final não homóloga (NHEJ) 22 , 23 , 24 , que tipicamente introduz pequenas deleções ou, raramente, inserções. Ao segmentar umQuadro de leitura, o reparo provavelmente leva a um quadro de translação que produz um produto proteico não funcional. Em contraste, quando fornecido com um modelo exógeno com grandes regiões de homologia, a célula pode corrigir a ruptura de duas vertentes por reparo direcionado por homologia 25 , 26 . Esta rota permite exclusões, substituições ou inserções precisas maiores no genoma, juntamente com a introdução de marcadores de seleção excisáveis 27 .
Aqui, apresentamos protocolos para gerar linhas celulares knockout por qualquer um desses dois métodos CRISPR / Cas9 ( Figura 1A ). A abordagem do NHEJ usa um único site de corte de sgRNA e rastreio independente de seleção e, portanto, requer pouca preparação antecipada. Ao usar este método, os RNAs de guia complementares aos exons próximos ao final 5 'da transcrição, que são mais propensos a produzir um knockout, devem ser projetados. Desde o modificatOs íons para o genoma neste caso são pequenos, a triagem para clones knockout baseia-se em manchas de ponto, onde o produto proteico é avaliado de forma eficiente. Utilizamos a geração de linhas knockout de proteína ELAV-like 1 (ELAVL1) como exemplo. A segunda abordagem baseia-se na reparação direta de homologia (HDR) e usa dois sites de corte de sgRNA que abrangem o gene ou região de interesse, permitindo deleções de dezenas de kb. Um plasmídeo com duas regiões de homologia que flanqueiam os sites de clivagem fornece um modelo de substituição ( Figura 1B ), introduzindo um marcador de resistência selecionável que aumenta a eficiência da geração de nocaute. Este método também pode ser adaptado para introduzir modificações de genes com armas de homologia devidamente projetadas. Neste caso, a integração de um novo fragmento de DNA permite a triagem baseada em PCR ( Figura 1C ). Aqui, usamos a geração de linhas knockout do membro da família Pumilio RNA vinculativo 2 (PUM2) como exemplo.
Protocolo
1. Identificação de regiões de homologia em torno da eliminação desejada
NOTA: necessário somente se estiver usando a edição baseada em seleção.
- Selecione duas regiões, inicialmente 1,5-2 kb, em ambos os lados do locus de exclusão desejado, que servirá como braços de homologia no modelo HDR ( Figura 1A ). Identifique as regiões que carecem de sites de reconhecimento BsaI em ambos os lados (GGTCTC) para facilitar a clonagem. Se os sítios BsaI forem inevitáveis, use uma enzima de restrição tipo II diferente (BsmBI, SapI, BbsI) e modifique os locais correspondentes no plasmídeo receptor (pUC19-BsaI), plasmídeo doador marcador de resistência (pGolden-Neo ou pGolden-Hygro) e Saliência de produtos de PCR do grupo de homologia.
2. Geração de Plasmídeos de Expressão Cas9-SgRNA
- Defina o (s) site (s) de segmentação para mutagênese. Para o método de corte único, sem seleção, exões de codificação proximal de 5 'para aumentar a probabilidade de um não-fuMutação nacional. Para o método de corte duplo, selecione dois sites que ocupam a maior parte do gene conforme necessário.
NOTA: Em nossa experiência, as exclusões de até 53 kb são criadas eficientemente. - Entre 200 a 300 pb da sequência de região alvo na ferramenta de design crispr.mit.edu. Para a clonagem baseada em seleção, use 200-300 pb das regiões de homologia identificadas que são proximais ao locus de exclusão ( Figura 1A ). Selecione 2-3 dos sgRNAs de maior classificação por região segmentada para explicar as diferenças em suas atividades e isolar clones independentes que não compartilham os mesmos efeitos fora do alvo.
- Para projetar oligonucleótidos duplexados com saliências apropriadas para inserção no plasmídeo de expressão, omita a sequência PAM final (NGG) e adicione uma saliência 5 '-CACC seguida de um G no oligo superior. Para o oligo de vertente inferior, adicione uma saliência de 5 a AAAC à sequência alvo complementada inversa, seguida de um 3'-C, como ilIlustrado abaixo:
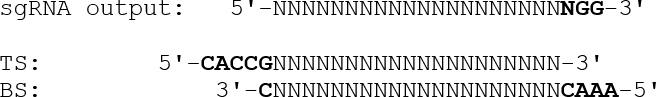
- Insira oligonucleótidos sintéticos correspondentes aos sgRNAs no plasmídeo pSpCas9 (BB) usando a clonagem Golden Gate 28 como descrito no protocolo de laboratório Zhang 29 .
3. Geração de Plasmídeos de Modelo de Reparo Dirigidos por Homologia
NOTA: necessário somente se estiver usando a edição baseada em seleção. O plasmídeo modelo de reparação dirigido por homologia consiste em uma cassete de resistência a fármacos flanqueada por duas regiões que são complementares do genoma, apenas fora dos dois sites alvo de sgRNA ( Figura 1B ).
- Das regiões de homologia mais amplas identificadas no passo 1.1, selecione 800-1,000 bp 26 não mais de 5-10 pb dos locais de corte Cas9 projetados, para servir como os braços de homologia ( Figura 1A ). Certifique-se de não incluir o sgRNASite alvo e seu PAM nos braços de homologia, pois isso causará clivagem CRISPR / Cas9 do próprio plasmídeo modelo. Se os braços de homologia contêm sites BsaI internos, use sites de sgRNA alternativos.
- A amplificação por PCR amplifica os braços de homologia a partir de DNA genômico usando uma polimerase de DNA de alta fidelidade, de acordo com as instruções do fabricante, utilizando iniciadores diretos e reversos com sequência adicional de 5 'que introduz sites BsaI (GGTCTC) e saliências únicas em qualquer extremidade da região de homologia. Os saliências devem conter sequências adequadas para gerar ordem e orientação de montagem corretas, conforme descrito abaixo ( Figura 1B ):
Braço de homologia esquerdo FP overhang: 5 'GGGTCTCA GGCC
Braço de homologia esquerdo RP overhang: 5 'GGGTCTCT CACG
Braço de homologia direito FP overhang: 5 'GGGTCTCA GTCC
Direito braço de homologia RP overhang: 5 'GGGTCTCT CCAC - Verifique os braços de homologia para amplificação apropriadaAtravés de eletroforese em gel antes de prosseguir.
- Monte as regiões do braço de homologia direita e esquerda com a cassete de resistência em um plasmídeo modelo HDR pela clonagem 28 de Golden Gate. Use os plasmídeos pGolden-Neo ou pGolden-Hygro 30 como fonte de cassetes de resistência flangeadas loxP (loxP-PGK-Neo-pA-loxP ou loxP-PGK-Hyg-pA-loxP). Use pUC19-BsaI, uma pUC19 modificada com sites BsaI na região de clonagem múltipla e eliminou sites BsaI em outros lugares, como o vetor destinatário (disponível mediante solicitação). Use uma proporção de 1: 1: 1: 1 para as três inserções e vetor.
- Preparar a seguinte mistura de reacção 30 :
Produto de PCR do direito da homologia 0,06 pmol (30-40ng) Produto de PCR do braço de homologia esquerdo 0,06 pmol (30-40ng) Plasmídeo pGolden-Neo (100 ng / μL) 1 &# 181, L Plasmídeo pUC19-BsaI (100 ng / μL) 1 μL 2x T7 DNA ligase buffer 5 μL BsaI (10 U / μL) 0,75 μL T7 ADN ligase (3000 U / μL) 0,25 μL agua Até 10 μL - Use os seguintes parâmetros do termociclador: 37 ° C durante 5 min, 20 ° C durante 5 min. Repita durante 30 ciclos.
- Trate o produto de clonagem com uma exonuclease por instruções do fabricante para digerir qualquer DNA linear remanescente.
- Transforme a mistura de reação em uma estirpe competente de E. coli de acordo com o protocolo fornecido com as células e placa sobre pratos contendo ampicilina.
- Preparar a seguinte mistura de reacção 30 :
- Para identificar clones com a montagem de modelo correta, execute bactériasPCR de colônia para amplificar as sub-regiões do braço de homologia da inserção montada (como a inserção inteira pode ser muito grande para amplificar de forma confiável). Use um recozimento de iniciador para a sequência do plasmídeo flanqueador e um segundo primário complementar à borda da cassete de resistência ( Figura 1B , a sequência é comum aos insertos Neo e Hygro), gerando um produto dependente da montagem que é aproximadamente 200 pb maior que O braço de homologia contido:
PUC19-Bsal-Left: 5 'GGCTCGTATGTTGTGTGGAATTGTGAG
Resistência à esquerda: 5 'AAAAGCGCCTCCCCTACCC
PUC19-BsaI-Right: 5 'GCTATTACGCCAGCTGGCGAAA
Resistência à direita: 5 'AAGACAATAGCAGGCATGCTGGG- Escolha colônias bacterianas com palitos de palito estéril e suspenda as bactérias em 20 μL de água. Aqueça a 95 ° C durante 15 min e gire para baixo numa centrífuga de mesa à velocidade máxima durante 5 min. Coloque imediatamente sobre gelo.
- Preparar a seguinte mistura de PCR 31 </ Sup>:
Diluir modelo de colônia 1,25 μL 10 x tampão de reação Taq 1,25 μL DNTPs de 20mM 0,25 μL Iniciador direto de 10 μM 0,25 μL Primer iniciador reverso de 10 μM 0,25 μL Taq Polymerase 0,25 μL agua 9 μL - Use os seguintes parâmetros do termociclador: 95 ° C durante 30 s, (95 ° C 30 s, 61 ° C durante 30 s, 72 ° C durante 1 min / kb), repita durante 25 ciclos, 72 ° C durante 2 min.
- Verifique o produto de PCR para o tamanho correto do amplicão por eletroforese em gel de agarose.
- Opcionalmente, o DNA de plasmídeo de miniprep de colônias individuais com tamanho de inserção correto e sequência thE regiões de braços de homologia pela seqüência de Sanger com os iniciadores de amplificação mencionados acima.
4. Transfecção de componentes CRISPR em células cultivadas
- Antes da transfecção: cultivar células T-REx293 em meio DMEM suplementado com 10% de FBS a 37 ° C e 5% de CO 2 .
- Coloque as células em placas de 6 poços e cresça até cerca de 70% de confluência. Inclua um poço para um controle não-transfectado.
- Células transfectadas com 2,5 μg de plasmídeo total utilizando um protocolo de transfecção comercial. Em paralelo, mantenha o controle não transfectado.
NOTA: O método de transfeção e a eficiência variam dependendo do tipo de célula. Determine o método de transfecção apropriado para o sistema antes da experiência.- Para a transfecção de células com seleção, use 0,75 μg de Cas9-sgRNA 1 (corte esquerdo), 0,75 μg de Cas9-sgRNA 2 (corte direito) e 1,0 μg de modelo de recombinação homóloga.
- Para transfectiNas células sem seleção, use 2,5 μg de plasmídeo Cas9-sgRNA.
5. Seleção de medicamentos
- 48 h após a transfecção, tratar as células com o fármaco apropriado (Neomicina / Higromicina). Execute a seleção até que todas as células no controle não transfectado morram (geralmente 3-5 dias para Neo, 7-14 dias para Hygro).
NOTA: Use concentrações de 500 μg / mL e 10 μg / mL para Neomycin e Hygromycin, respectivamente para células T-REx293. Para outros tipos de células, pode ser útil realizar uma titulação prévia de fármaco em células não transfectadas para determinar a concentração efetiva.
6. Isolamento de populações clonais
- Crescer as células para 100% de confluência no poço original após a seleção. Células de semente em placas de 96 poços a uma densidade de 0,33 células por poço.
NOTA: Semear três placas de 96 poços é um bom ponto de partida para garantir o isolamento de mais de um clone correto, mas pode ser necessário mais ou menos depTerminando em sgRNA e HDR eficiências. - Observe as colônias durante um período de 2-4 semanas, até que as colônias estejam visíveis aos olhos. Escolha as colônias visíveis com uma ponta de pipeta esterilizada e volte a colocar em novos poços para incentivar o crescimento de monocamadas. Ao usar células em suspensão, podem ser utilizadas densidades de semeadura mais baixas para garantir uma maior proporção de poços de colônia única.
7. Candidatos de triagem
- Seleção de candidatos sem a seleção: dot blot
- Cresça clones individuais a 50-100% de confluência. Desliga a monocamada de células por pipetagem dentro do poço.
- Alíquota 90 μL do volume total de 100 μL para um tubo de microcentrífuga limpo. Girando a 6000 rpm durante 5 min, retire o meio e lie o sedimento de células em 10 μL de 1x Lysis Buffer ou 1x tampão de carga de SDS (5x: Tris-Cl 250 mM pH 6,8, SDS a 8%, azul de bromofenol a 0,1%, 40% Glicerol, 100 mM de DTT). Adicione 90 μL de novas mídias ao restante das células no bemL para continuar propagando.
- Pipetar 1 μL de lisado celular sobre uma membrana de nitrocelulose seca para formar um ponto. Bloqueie cada amostra duas vezes em duas membranas separadas, criando dois padrões idênticos de amostras.
- Bloquear as membranas em 5% de leite em TBST (Tris Buffered Saline + 0,01% Tween 20: 8 g de NaCl, 0,2 g de KCl, 3 g de base de Tris, até 1 L de água destilada, pH 7,4) 31 durante 1 h à temperatura ambiente ( Com balanço, aqui e durante o procedimento).
- Bloquear usando o anticorpo primário contra a proteína alvo em uma membrana e o anticorpo primário para uma proteína de controle (tubulina, GAPDH ou qualquer outra proteína que não se espera que mude) no outro. Use a diluição recomendada de anticorpos primários (0,2 μg / mL para ELAVL1 e 0,1 μg / mL para Pum2 neste caso) em TBST + 5% de leite. Incubar 1 h à temperatura ambiente.
- Lavar 3 vezes com TBST por 5 min.
- Incubar cada membrana com anticorpo secundário adequado conjugado com HRP em TBST + 5% de leite durante 1 h à temperatura ambiente.
- Lavar 3 vezes com TBST por 5 min.
- Aplicar solução de substrato de quimioluminiscência (ver Tabela de Materiais) seguindo as instruções do fabricante e imagem da membrana manchada em uma imagem digital de quimioluminiscência.
- Quantifique intensidades de ponto para o alvo e controle proteínas usando o software apropriado.
- Elimine os candidatos com sinal de proteína de controle baixo e calcule os índices subtraídos de fundo do alvo para controlar as intensidades de proteína para os restantes candidatos. Selecione os candidatos com os índices mais baixos para a passagem e posterior validação pelo western blot.
- Candidatos de seleção com o uso da seleção: PCR de colônia
- Coletar lisado celular usando um kit de preparação de DNA (ver Tabela de Materiais ).
- Duplique colônias individuais em um novo 96 poços e cresça até 100% de confluência.
- Remova a mídia de um conjunto de clones, umE ressuspender células em 30 μL de tampão de extração do kit. Transfira para um tubo de microcentrífuga limpo de 1,5 mL.
- Aqueça a solução a 96 ° C durante 15 minutos e deixe arrefecer até à temperatura ambiente.
- Adicione 30 μL de tampão de estabilização do kit. Misture bem.
- Identificar linhas mutantes de tipo selvagem e monoalquilo / bialélico por PCR de colônia.
- Desenhe dois conjuntos separados de iniciadores de PCR (para o lado esquerdo e direito do locus alvo) para amplificar as regiões ao redor dos braços de homologia com base na integração bem sucedida ou mal sucedida da cassete de resistência ( Figura 2C ). Em cada lado, use um primário comum (vermelho) que recobre fora do braço de homologia. Use-o com um iniciador emparelhado correspondente que seja complementar à seqüência endógena, abrangendo a região de homologia (azul), para testar o alelo de tipo selvagem. Use outro iniciador emparelhado, complementar a cassete de resistência inserida (veja os iniciadores na seção2.3), para testar a mutação desejada.
- (Recomendado) Valide os conjuntos de iniciadores usando o lisado celular da população celular original selecionada, uma vez que conterá uma mistura de ambos os alelos WT e mutantes ( Figura 2D ).
- Preparar a seguinte mistura reaccional:
Lisado celular 0,5 μL 10x buffer de KOD 1,25 μL MgSO4 25 mM 0,75 μL DNTPs 2 mM 1,25 μL Iniciador direto de 10 μM 0,375 μL Primer iniciador reverso de 10 μM 0,375 μL KOD polimerase 0,25 μL agua 7,75 μL - Use as seguintes condições de termociclador: 95 ° C durante 2 minutos, (95 ° C por 20 s, Primer Tm durante 10 s, 70 ° C durante 20 s / kb), repita durante 25 ciclos.
- Visualize o produto de PCR por eletroforese em gel de agarose.
- Repita o teste de PCR da colônia para cada lado da integração prevista. Selecione e expanda candidatos que mostram presença de integração e nenhum produto de seqüência endógena para a região excluída.
- Valide candidatos positivos por western blot e / ou RT-qPCR.
- Coletar lisado celular usando um kit de preparação de DNA (ver Tabela de Materiais ).
8. Verifique a Mutação Genômica por Seqüenciamento
- Isolar DNA genômico a partir de linhas de células mutantes por extração de fenol clorofórmio 31 .
- A amplificação por PCR da região alvo de sgRNA, usando primers com 5 'overhangs que adicionam os locais de restrição BsaI para cada extremidade.
NOTA: Para clones editados com HDR, onde ambos os alelos podem ser idênticos, o produto de PCR pode ser sequenciado diretamente. - Clonar a região ampliada em pUC19-BsaI pela clonagem Golden Gate.
- Transforme a mistura de reação em uma estirpe competente de E. coli .
- ADN do plasmídeo Miniprep de 6 a 10 colônias individuais e sequencia a região clonada pela seqüência de Sanger.
Resultados
Para a geração de linhas knockout ELAVL1, um anticorpo robusto estava disponível, então a edição usando sgRNAs simples ( Figura 1A , esquerda) foi realizada, seguida de imunotransferência por pontos. Três sgRNAs foram transfectados de forma independente para comparar eficiências e descartar os efeitos alvo nos clones resultantes. Após a coleta e mancha de lisados celulares de populações clonais em duas membranas de nitrocelulose, as manchas...
Discussão
O sistema CRISPR / Cas9 permitiu uma geração eficiente de modificações genômicas estáveis, que proporcionam uma alternativa mais consistente a outros métodos de manipulação transitória. Aqui, apresentamos dois métodos para identificação rápida de knockouts de genes CRISPR / Cas9 em linhas celulares de mamíferos. Ambos os métodos requerem pouco material celular, pelo que o teste pode ser feito em estágios iniciais da cultura clonal, economizando tempo e reagentes. Para aumentar a eficiência de ambos os ...
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Os autores gostariam de reconhecer Gissell Sanchez, Megan Lee e Jason Estep para assistência experimental, e Weifeng Gu e Xuemei Chen por compartilhar reagentes.
Materiais
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
Referências
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados