Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Selektionsabhängige und unabhängige Erzeugung von CRISPR / Cas9-vermittelten Gen-Knockouts in Säugetierzellen
In diesem Artikel
Zusammenfassung
Jüngste Fortschritte in der Fähigkeit, somatische Zelllinien genetisch zu manipulieren, haben großes Potenzial für grundlegende und angewandte Forschung. Hier präsentieren wir zwei Ansätze für CRISPR / Cas9-produzierte Knockout-Produktion und Screening in Säugetierzelllinien, mit und ohne Verwendung von selektierbaren Markern.
Zusammenfassung
Das CRISPR / Cas9 Genom Engineering System hat die Biologie revolutioniert, indem es genaue Genombearbeitung mit wenig Aufwand ermöglicht. Geleitet von einer einzigen Guide-RNA (sgRNA), die Spezifität verleiht, spaltet das Cas9-Protein beide DNA-Stränge am Zielort. Der DNA-Bruch kann entweder nicht-homologe Endverbindungen (NHEJ) oder Homologie gerichtete Reparatur (HDR) auslösen. NHEJ kann kleine Deletionen oder Insertionen einführen, die zu Frame-Shift-Mutationen führen, während HDR größere und präzisere Störungen ermöglicht. Hier präsentieren wir Protokolle zur Erzeugung von Knockout-Zelllinien durch Kopplung von etablierten CRISPR / Cas9-Methoden mit zwei Optionen für die nachgeschaltete Auswahl / Screening. Der NHEJ-Ansatz nutzt eine einzelne sgRNA-Cut-Stelle und selektionsunabhängiges Screening, bei der die Proteinproduktion durch Dot-Immunoblot in einer Hochdurchsatz-Weise beurteilt wird. Der HDR-Ansatz verwendet zwei sgRNA-Cut-Stellen, die das Gen von Interesse überspannen. Zusammen mit einer bereitgestellten HDR-Vorlage kann diese Methode eine Löschung erreichenVon zehn kb, unterstützt durch die eingefügte wählbare Widerstandsmarkierung. Die entsprechenden Anwendungen und Vorteile jeder Methode werden diskutiert.
Einleitung
Stabile genetische Veränderungen bieten einen Vorteil gegenüber transienten Methoden der zellulären Störung, die in ihrer Effizienz und Dauer variabel sein können. Die genomische Bearbeitung ist in den letzten Jahren aufgrund der Entwicklung von zielspezifischen Nukleasen, wie z. B. Zinkfinger-Nukleasen 1 , 2 , 3 , 4 , 5 , Transkriptionsaktivator-ähnliche Effektor-Nukleasen (TALENs) 6 , 7 , 8 , 9 immer häufiger geworden Und RNA-geführte Nukleasen, die aus dem geclusterten, regelmäßig verteilten kurzen palindromischen Wiederholungssystem (CRISPR) 10 gewonnen wurden .
Die CRISPR / Cas9-Bearbeitungsmaschinen werden von einem Immunsystem angepasst, das Bakterien und Archaea zur Verteidigung gegen Virusinfektionen verwendenAss = "xref"> 11 , 12 , 13 . In diesem Prozess werden kurze, 20-30 nt-Fragmente der eindringenden Virussequenz in einen genomischen Locus als "Spacer" eingebaut, die von den Wiederholungseinheiten 14 , 15 flankiert sind. Nachfolgende Transkriptions- und RNA-Verarbeitung erzeugt kleine CRISPR-assoziierte RNAs 16 (crRNAs), die zusammen mit einer transaktivierenden crRNA 17 (tracrRNA) mit der Effektor-Cas9-Endonuklease zusammensetzen. Die crRNAs bieten somit eine Spezifität für das Cas9-Targeting, die den Komplex leitet, um komplementäre virale DNA-Sequenzen zu spalten und weitere Infektionen zu verhindern 18 , 19 . Jede "Protospacer" -Sequenz in der Ziel-DNA kann als Quelle der crRNA dienen, solange sie direkt 5 'zu einem kurzen Protospacer benachbarten Motiv (PAM), NGG im Fall von S. pyogenes Cas9 ist 20 Die Abwesenheit der PAM-Sequenz nahe dem Spacer im CRISPR-Locus des Hosts unterscheidet zwischen Selbst und Nicht-Selbst und verhindert das Targeting des Hosts. Wegen seiner Universalität und Flexibilität ist dieses biologische System mächtig für die genomische Bearbeitung angepasst worden, so dass nahezu jede PAM-benachbarte DNA-Stelle gezielt werden kann. In dieser Version fusionierte eine weitere Modifikation die crRNA und tracrRNA zu einer einzigen RNA (sgRNA) -Komponente, die in das Cas9-Protein 21 geladen wurde.
Bei der Expression von Cas9 und einer sgRNA in eukaryotischen Zellen spaltet das Cas9-Protein beide DNA-Stränge am Zielort. In Abwesenheit einer geeigneten Region der homologen Sequenz fixiert die Zelle diesen Bruch über eine nicht homologe Endverbindung (NHEJ) 22 , 23 , 24 , die typischerweise kleine Deletionen oder selten Insertionen einführt. Bei der Ausrichtung auf eine offeneLeserahmen, führt die Reparatur wahrscheinlich zu einer translationalen Frameshift, die ein nicht-funktionelles Proteinprodukt produziert. Im Gegensatz dazu kann die Zelle, wenn sie mit einer exogenen Schablone mit großen Regionen der Homologie versehen ist, die doppelsträngige Pause durch homologisch gerichtete Reparatur 25 , 26 fixieren. Dieser Weg ermöglicht größere präzise Deletionen, Ersetzungen oder Insertionen im Genom, gepaart mit der Einführung von ausfahrbaren Selektionsmarkern 27 .
Hier stellen wir Protokolle zur Erzeugung von Knockout-Zelllinien durch eine dieser beiden CRISPR / Cas9-Verfahren vor (Abbildung 1A ). Der NHEJ-Ansatz nutzt eine einzelne sgRNA-Cut-Site und selektionsunabhängiges Screening und erfordert daher eine geringe Vorwärtspräparation. Bei der Verwendung dieser Methode müssen RNAs, die komplementär zu Exons in der Nähe des 5'-Endes des Transkripts sind, die am ehesten ein Knockout produzieren, entworfen werden. Seit dem modifikatIonen zum Genom in diesem Fall sind klein, das Screening auf Knockout-Klone basiert auf Punkt-Blots, wo das Protein-Produkt in einer Hochdurchsatz-Weise beurteilt wird. Wir verwenden die Generation von ELAV-ähnlichen 1 Protein (ELAVL1) Knockout Linien als Beispiel. Der zweite Ansatz beruht auf homologisch gerichteter Reparatur (HDR) und verwendet zwei sgRNA-Cut-Stellen, die das Gen oder die Region von Interesse überspannen, was eine Deletionen von zehn kb ermöglicht. Ein Plasmid mit zwei Regionen der Homologie, die die Spaltstellen flankieren, liefert eine Ersatzschablone (Abbildung 1B ), die einen wählbaren Resistenzmarker einführt, der die Effizienz der Knockout-Erzeugung erhöht. Diese Methode kann auch angepasst werden, um Genmodifikationen mit richtig entworfenen Homologiearmen einzuführen. In diesem Fall ermöglicht die Integration eines neuen DNA-Fragments ein PCR-basiertes Screening (Abbildung 1C ). Hier verwenden wir die Generierung von Pumilio RNA Bindung Familienmitglied 2 (PUM2) Knockout Linien als Beispiel.
Protokoll
1. Identifizierung von Homologieregionen um die gewünschte Löschung
HINWEIS: Nur bei selektionsbasierter Bearbeitung erforderlich.
- Wähle zwei Regionen, anfänglich 1,5-2 kb, auf beiden Seiten des gewünschten Löschorts, die als Homologiearme in der HDR-Schablone dienen (Abbildung 1A ). Identifizieren Sie Regionen, die BsaI-Erkennungsstellen auf beiden Strang (GGTCTC) fehlen, um das Klonen zu erleichtern. Wenn BsaI-Stellen unvermeidbar sind, verwenden Sie ein alternatives IIs-Restriktionsenzym (BsmBI, SapI, BbsI) und modifizieren die entsprechenden Stellen im Empfängerplasmid (pUC19-BsaI), Resistenzmarker-Donor-Plasmid (pGolden-Neo oder pGolden-Hygro) und Homologie Arm PCR Produkt Überhänge.
2. Erzeugung von Cas9-sgRNA-Expressionsplasmiden
- Definiere die Zielstelle (n) für die Mutagenese. Für die Single-Cut, Selektions-freie Methode, Ziel 5 'proximale Codierung Exons, um die Wahrscheinlichkeit einer Nicht-FU zu erhöhenNationale Mutation. Für die Zwei-Cut-Methode, wählen Sie zwei Standorte, die so viel von dem Gen wie nötig überspannen.
HINWEIS: Nach unserer Erfahrung werden Löschungen bis zu 53 kb effizient erstellt. - Geben Sie 200 bis 300 bp der Ziel-Sequenz in das crispr.mit.edu-Design-Tool ein. Zur selektionsbasierten Klonierung verwenden Sie 200-300 bp der identifizierten Homologieregionen, die proximal zum Deletionsort sind (Abbildung 1A ). Wählen Sie 2-3 der höchstrangigen sgRNAs pro Zielregion aus, um Unterschiede in ihrer Aktivität zu berücksichtigen und unabhängige Klone zu isolieren, die nicht die gleichen potentiellen Off-Target-Effekte teilen.
- Um duplexierte Oligonukleotide mit geeigneten Überhängen für die Insertion in das Expressionsplasmid zu entwerfen, weglassen sie die endgültige PAM-Sequenz (NGG) und hängen einen 5'-CACC-Überhang an, gefolgt von einem G zum Top-Strang-Oligo. Für den unteren Strang-Oligo, hängen Sie einen 5'-AAAC-Überhang auf die umgekehrt-ergänzte Zielsequenz, gefolgt von einem 3'-C, als ilLustriert unten:
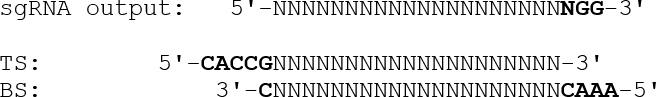
- Insert synthetische Oligonukleotide entsprechend den sgRNAs in das pSpCas9 (BB) Plasmid unter Verwendung der Golden Gate Klonierung 28, wie in dem Zhang Laborprotokoll 29 beschrieben.
3. Erzeugung von Homologie-gerichteten Reparatur-Template-Plasmiden
HINWEIS: Nur bei selektionsbasierter Bearbeitung erforderlich. Das homologiegesteuerte Reparaturschablonenplasmid besteht aus einer Arzneimittelresistenzkassette, die von zwei Regionen flankiert ist, die komplementär zum Genom gerade außerhalb der beiden sgRNA-Zielstellen sind ( 1B ).
- Aus den in Schritt 1.1 identifizierten breiteren Homologie-Regionen wählen Sie 800-1000 bp 26 nicht mehr als 5-10 bp weg von den entworfenen Cas9-Schnittorten, um als Homologie-Arme zu dienen (Abbildung 1A ). Achten Sie darauf, die sgRNA nicht einzuschließenZiel-Site und seine PAM in der Homologie Arme, da dies wird dazu führen, dass CRISPR / Cas9 Spaltung der Vorlage Plasmid selbst. Wenn die Homologiearme interne BsaI-Standorte enthalten, verwenden Sie alternative sgRNA-Stellen.
- PCR amplifiziert Homologiearme aus genomischer DNA unter Verwendung einer hochauflösenden DNA-Polymerase nach den Anweisungen des Herstellers unter Verwendung von Vorwärts- und Rückwärtsprimern mit einer zusätzlichen 5'-Sequenz, die BsaI-Stellen (GGTCTC) und einzigartige Überhänge an beiden Enden der Homologieregion einführt. Die Überhänge müssen korrekte Sequenzen enthalten, um eine korrekte Montageordnung und Orientierung zu erzeugen, wie unten beschrieben (Abbildung 1B ):
Linker Homologiearm FP-Überhang: 5 'GGGTCTCA GGCC
Linker Homologiearm RP-Überhang: 5 'GGGTCTCT CACG
Recht Homologie Arm FP Überhang: 5 'GGGTCTCA GTCC
Recht Homologie Arm RP Überhang: 5 'GGGTCTCT CCAC - Überprüfen Sie die Homologiearme für den entsprechenden VerstärkerÜber die Gelelektrophorese vor dem Fortfahren.
- Montieren Sie die rechte und linke Homologiearmregionen mit der Resistenzkassette in ein HDR Template Plasmid von Golden Gate Cloning 28 . Verwenden Sie pGolden-Neo oder pGolden-Hygro-Plasmide 30 als Quelle für loxP-flankierte Resistenzkassetten (loxP-PGK-Neo-pA-loxP oder loxP-PGK-Hyg-pA-loxP). Verwenden Sie pUC19-BsaI, ein modifiziertes pUC19 mit BsaI-Stellen in der multiplen Klonierungsregion und eliminiert BsaI-Stellen an anderer Stelle als den Empfängervektor (auf Anfrage erhältlich). Verwenden Sie für die drei Einsätze und den Vektor ein Verhältnis von 1: 1: 1: 1.
- Vorbereitung der folgenden Reaktionsmischung 30 :
Richtige Homologie Arm PCR Produkt 0,06 pmol (30-40 ng) Linkes Homologiearm PCR Produkt 0,06 pmol (30-40 ng) PGolden-Neo-Plasmid (100 ng / & mgr; l) 1 &# 181; L PUC19-BsaI-Plasmid (100 ng / & mgr; l) 1 & mgr; l 2x T7 DNA-Ligasepuffer 5 & mgr; l BsaI (10 U / & mgr; l) 0,75 & mgr; l T7-DNA-Ligase (3000 U / & mgr; l) 0,25 & mgr; l Wasser Bis zu 10 μl - Verwenden Sie folgende Thermocycler-Parameter: 37 ° C für 5 min, 20 ° C für 5 min. Wiederholen Sie für 30 Zyklen.
- Behandeln Sie das Klonprodukt mit einer Exonuklease nach den Anweisungen des Herstellers, um jede verbleibende linearisierte DNA zu verdauen.
- Umwandlung der Reaktionsmischung in einen kompetenten E. coli- Stamm gemäß dem mit den Zellen gelieferten Protokoll und Platte auf Ampicillin-haltigen Schalen.
- Vorbereitung der folgenden Reaktionsmischung 30 :
- Um Klone mit der richtigen Schablonenanordnung zu identifizieren, Bakterien ausführenAl Kolonie PCR, um die Homologiearm-Teilbereiche des zusammengebauten Einsatzes zu verstärken (da der gesamte Einsatz zu groß sein kann, um zuverlässig zu verstärken). Verwenden Sie eine Primerglühung für die flankierende Plasmidsequenz und einen zweiten Primer, der komplementär zum Rand der Resistenzkassette ist (Abbildung 1B , die Sequenz ist sowohl für Neo- als auch Hygro-Insertionen üblich), wodurch ein assemblierungsabhängiges Produkt erzeugt wird, das etwa 200 bp länger ist als Der enthaltene Homologiearm:
PUC19-Bsal-Links: 5 'GGCTCGTATGTTGTGTGGAATTGTGAG
Widerstand-Links: 5 'AAAAGCGCCTCCCCTACCC
PUC19-BsaI-rechts: 5 'GCTATTACGCCAGCTGGCGAAA
Widerstands-rechts: 5 'AAGACAATAGCAGGCATGCTGGG- Pick-Bakterien-Kolonien mit sterilen Zahnstochern und suspendieren die Bakterien in 20 μl Wasser. Bei 95 ° C für 15 min erhitzen und in einer Tischzentrifuge bei maximaler Geschwindigkeit für 5 min abreiben. Legen Sie sofort auf Eis.
- Vorbereitung der folgenden PCR-Mischung 31 </ Sup>:
Kolonievorlage verdünnen 1,25 & mgr; l 10x Taq-Reaktionspuffer 1,25 & mgr; l 20mM dNTPs 0,25 & mgr; l 10 μM Vorwärtsprimer 0,25 & mgr; l 10 μM Rückwärtsprimer 0,25 & mgr; l Taq-Polymerase 0,25 & mgr; l Wasser 9 & mgr; l - Verwenden Sie die folgenden Thermocycler-Parameter: 95 ° C für 30 s, (95 ° C 30 s, 61 ° C für 30 s, 72 ° C für 1 min / kb), wiederholen Sie für 25 Zyklen, 72 ° C für 2 min.
- Kontrollieren Sie das PCR-Produkt auf korrekte Amplikongröße durch Agarose-Gelelektrophorese.
- Optional können Miniprep-Plasmid-DNA aus einzelnen Kolonien mit korrekter Insert-Größe und Sequenz thE-Homologie-Armregionen durch Sanger-Sequenzierung mit den oben erwähnten Amplifikations-Primern.
4. Transfektion von CRISPR-Komponenten in kultivierte Zellen
- Vor der Transfektion: Kultur-T-REx293-Zellen in DMEM-Medien, ergänzt mit 10% FBS bei 37 ° C und 5% CO 2 .
- Tafeln Sie Zellen auf 6-Well-Platten und wachsen auf etwa 70% Konfluenz. Füge einen Brunnen für eine untransfizierte Kontrolle ein.
- Transfektion von Zellen mit 2,5 μg Gesamtplasmid unter Verwendung eines kommerziellen Transfektionsprotokolls. Parallel dazu die untransfizierte Kontrolle beibehalten.
HINWEIS: Transfektionsmethode und Effizienz variieren je nach Zelltyp. Bestimmen Sie die geeignete Transfektionsmethode für das System vor dem Experiment.- Für die Transfektion von Zellen mit Selektion verwenden Sie 0,75 μg Cas9-sgRNA 1 (links geschnitten), 0,75 μg Cas9-sgRNA 2 (rechts geschnitten) und 1,0 μg homologe Rekombinationsschablone.
- Für transfectiVon Zellen ohne Selektion, verwenden Sie 2,5 μg Cas9-sgRNA Plasmid.
5. Drug Selection
- 48 h nach der Transfektion behandeln die Zellen mit dem entsprechenden Medikament (Neomycin / Hygromycin). Führen Sie die Selektion durch, bis alle Zellen in der nicht transfizierten Kontrolldüse (typischerweise 3-5 Tage für Neo, 7-14 Tage für Hygro).
HINWEIS: Verwenden Sie Konzentrationen von 500 μg / ml und 10 μg / ml für Neomycin bzw. Hygromycin für T-REx293-Zellen. Für andere Zelltypen kann es nützlich sein, eine vorherige Titration des Arzneimittels auf nicht transfizierten Zellen durchzuführen, um die wirksame Konzentration zu bestimmen.
6. Isolierung von Klonpopulationen
- Wachsen Sie Zellen zu 100% Konfluenz in der ursprünglichen gut nach Auswahl. Seed-Zellen in 96-Well-Platten mit einer Dichte von 0,33 Zellen pro Vertiefung.
HINWEIS: Das Samen von drei Platten mit 96 Vertiefungen ist ein guter Ausgangspunkt, um die Isolierung von mehr als einem korrekten Klon zu gewährleisten, aber mehr oder weniger kann erforderlich seinEnden auf sgRNA und HDR effizienzen. - Beobachten Sie Kolonien über einen Zeitraum von 2-4 Wochen, bis Kolonien für das Auge sichtbar sind. Sehe sichtbare Kolonien mit einer sterilen Pipettenspitze auf und setze in neuen Brunnen ein, um das Monolayer-Wachstum zu fördern. Bei der Verwendung von Suspensionszellen können sogar niedrigere Saatdichten verwendet werden, um einen größeren Anteil an Einzelkolonien-Vertiefungen zu gewährleisten.
7. Screening Kandidaten
- Screening Kandidaten ohne die Verwendung von Auswahl: Dot Blot
- Wachsen Sie einzelne Klone zu 50-100% Konfluenz. Die Monoschicht der Zellen durch Pipettieren innerhalb des Brunnens auslöschen
- Aliquotieren Sie 90 μl des 100 μl Gesamtvolumens zu einem sauberen Mikrozentrifugenröhrchen. 5 Minuten lang bei 6000 U / min abspülen, Medium entfernen und Zellpellet in 10 μl 1x Lysepuffer oder 1x SDS-Beladungspuffer (5x: 250 mM Tris-Cl pH 6,8, 8% SDS, 0,1% Bromphenolblau, 40% Glycerin, 100 mM DTT). Fügen Sie 90 μl neue Medien in den Rest der Zellen in der welIch bleibe weiter fortpflanzen.
- 1 & mgr; l Zelllysat auf eine trockene Nitrozellulosemembran pipettieren, um einen Punkt zu bilden. Blot jede Probe zweimal auf zwei getrennten Membranen, die Schaffung von zwei identischen Muster von Proben.
- Blockieren Sie die Membranen in 5% Milch in TBST (Tris gepufferte Salzlösung + 0,01% Tween 20: 8 g NaCl, 0,2 g KCl, 3 g Tris-Base, bis zu 1 l destilliertem Wasser, pH 7,4) 31 für 1 h bei Raumtemperatur ( Mit Schaukeln, hier und während des ganzen Verfahrens).
- Blot unter Verwendung des primären Antikörpers gegen das Zielprotein auf einer Membran und der primäre Antikörper für ein Kontrollprotein (Tubulin, GAPDH oder irgendein anderes Protein, das sich nicht ändern wird) auf der anderen Seite. Verwenden Sie die empfohlene primäre Antikörperverdünnung (0,2 μg / ml für ELAVL1 und 0,1 μg / ml für Pum2 in diesem Fall) in TBST + 5% Milch. 1 h bei Raumtemperatur inkubieren.
- 3 mal mit TBST für 5 min waschen.
- Inkubieren Sie jede Membran mit einem geeigneten HRP-konjugierten sekundären Antikörper in TBST + 5% Milch für 1 h bei Raumtemperatur.
- 3 mal mit TBST für 5 min waschen.
- Die Chemilumineszenz-Substratlösung (siehe Werkstofftabelle) nach den Anweisungen des Herstellers auftragen und die blotted Membran auf einen digitalen Chemilumineszenz-Imager auftragen.
- Quantifizierung der Punktintensitäten für die Ziel- und Kontrollproteine mit der entsprechenden Software.
- Eliminieren Sie Kandidaten mit niedrigem Kontrollproteinsignal und berechnen Sie Hintergrund-subtrahierte Verhältnisse von Ziel, um Proteinintensitäten für die verbleibenden Kandidaten zu kontrollieren. Wählen Sie die Kandidaten mit den niedrigsten Verhältnissen für Passage und weitere Validierung durch Western Blot.
- Screening Kandidaten mit der Verwendung von Auswahl: Kolonie PCR
- Sammle das Zelllysat unter Verwendung eines DNA- Präparats (siehe Materialtabelle ).
- Duplizieren Sie einzelne Kolonien in einem neuen 96 Brunnen und wachsen bis 100% Konfluenz.
- Medien aus einem Satz der Klone entfernen, aNd die Zellen in 30 μl Extraktionspuffer aus dem Kit resuspendieren. Überführung in ein sauberes 1,5 mL Mikrozentrifugenröhrchen.
- Die Lösung 15 Minuten lang auf 96 ° C erhitzen und auf Raumtemperatur abkühlen lassen.
- Füge 30 μl Stabilisierungspuffer aus dem Kit hinzu. Gut mischen.
- Identifizierung von Wildtyp- und Monoallelic / Biallelic-Mutantenlinien durch Kolonie-PCR.
- Entwerfen Sie zwei getrennte Sätze von PCR-Primern (für die linke und rechte Seite des Zielortes), um Regionen um die Homologiearme zu verstärken, die entweder auf einer erfolgreichen oder erfolglosen Integration der Widerstandskassette basieren (Abbildung 2C ). Auf jeder Seite eine gemeinsame Grundierung (rot) verwenden, die außerhalb des Homologiearms anhebt. Verwenden Sie es mit einem entsprechenden gepaarten Primer, der komplementär zur endogenen Sequenz ist, die den Homologiebereich (blau) überspannt, um das Wildtyp-Allel zu testen. Verwenden Sie einen anderen gepaarten Primer, komplementär zur eingesetzten Resistenzkassette (siehe Primer im Abschnitt2.3), um die gewünschte Mutation zu testen.
- (Empfohlen) Validieren der Primersätze unter Verwendung von Zelllysat aus der ursprünglichen ausgewählten Bulkzellpopulation, da sie eine Mischung aus sowohl WT als auch mutanten Allelen enthält (Abbildung 2D ).
- Vorbereitung der folgenden Reaktionsmischung:
Zelllysat 0,5 & mgr; l 10x KOD Puffer 1,25 & mgr; l 25mM MgSO4 0,75 & mgr; l 2 mM dNTPs 1,25 & mgr; l 10 μM Vorwärtsprimer 0,375 & mgr; l 10 μM Rückwärtsprimer 0,375 & mgr; l KOD-Polymerase 0,25 & mgr; l Wasser 7,75 & mgr; l - Verwenden Sie die folgenden Thermocycler-Bedingungen: 95 ° C für 2 min, (95 ° C für 20 s, Primer Tm für 10 s, 70 ° C für 20 s / kb) Wiederholen für 25 Zyklen.
- Das PCR-Produkt durch Agarose-Gelelektrophorese visualisieren.
- Wiederholen Sie die Kolonie-PCR-Tests für jede Seite der vorhergesagten Integration. Auswählen und Erweitern von Kandidaten, die Vorhandensein von Integration und kein endogenes Sequenzprodukt für die gelöschte Region zeigen.
- Validieren Sie positive Kandidaten durch Western-Blot und / oder RT-qPCR.
- Sammle das Zelllysat unter Verwendung eines DNA- Präparats (siehe Materialtabelle ).
8. Überprüfen Sie die genomische Mutation durch Sequenzierung
- Isolieren Sie genomische DNA aus mutanten Zelllinien durch Phenol-Chloroform-Extraktion 31 .
- PCR amplifizieren die sgRNA-Zielregion unter Verwendung von Primern mit 5'-Überhängen, die BsaI-Restriktionsstellen zu jedem Ende hinzufügen.
HINWEIS: Für Klone, die mit HDR bearbeitet wurden, wo beide Allele identisch sein können, kann das PCR-Produkt direkt sequenziert werden. - Klone die verstärkte Region in pUC19-BsaI durch Golden Gate Klonen.
- Umwandlung der Reaktionsmischung in einen kompetenten E. coli- Stamm.
- Miniprep-Plasmid-DNA aus 6-10 einzelnen Kolonien und Sequenz der klonierten Region durch Sanger-Sequenzierung.
Ergebnisse
Für die Erzeugung von ELAVL1-Knockout-Linien war ein robuster Antikörper verfügbar, so dass die Bearbeitung mit einzelnen sgRNAs (Abbildung 1A , links) durchgeführt wurde, gefolgt von Punkt-Immunoblot. Drei sgRNAs wurden unabhängig transfiziert, um die Effizienz zu vergleichen und die Ziel-Effekte in den resultierenden Klonen auszuschließen. Nach dem Sammeln und Blotting von Zelllysaten aus klonalen Populationen auf zwei Nitrozellulosemembranen wurden ...
Diskussion
Das CRISPR / Cas9-System ermöglicht eine effiziente Erzeugung stabiler genomischer Modifikationen, die eine konsistentere Alternative zu anderen transienten Manipulationsverfahren bieten. Hier haben wir zwei Methoden zur schnellen Identifizierung von CRISPR / Cas9-Gen-Knockouts in Säugetierzelllinien vorgestellt. Beide Methoden erfordern wenig zelluläres Material, so dass Tests in frühen Stadien der Klonalkultur durchgeführt werden können, spart Zeit und Reagenzien. Um die Effizienz beider Methoden zu erhöhen, em...
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Die Autoren möchten Gissell Sanchez, Megan Lee und Jason Estep für experimentelle Hilfe und Weifeng Gu und Xuemei Chen für den Austausch von Reagenzien anerkennen.
Materialien
| Name | Company | Catalog Number | Comments |
| Competent E. coli cells | |||
| plasmid prep kit | |||
| pSpCas9(BB) plasmid | Addgene | 42230 | Ran et al. 2013, cloning sgRNAs |
| BbsI enzyme | ThermoFisher | FD1014 | Ran et al. 2013, cloning sgRNAs |
| T7 DNA ligase | NEB | M0318L | Ran et al. 2013, cloning sgRNAs |
| Tango Buffer | ThermoFisher | BY5 | Ran et al. 2013, cloning sgRNAs |
| PlasmidSafe exonuclease | Epicentre | E3105K | Ran et al. 2013, cloning sgRNAs |
| Q5 hot start high fidelity polymerase | NEB | M0494A | HA amplification |
| pUC19-BsaI | Modified pUC19 plasmid, mutated existing BsaI site and inserted two outward facing BsaI sites after BamHI/EcoRI digestion | ||
| pGolden-Neo | Addgene | 51422 | Resistance cassette |
| pGolden-Hygro | Addgene | 51423 | Resistance cassette |
| BsaI enzyme | NEB | R3535S | Homology arm contruction |
| T7 DNA ligase | NEB | M0318L | |
| PlasmidSafe exonuclease | Epicentre | E3105K | |
| Toothpicks | bacterial PCR | ||
| Taq polymerase | bacterial colony PCR | ||
| HEK293 human cells | |||
| DMEM | Corning | 10-013-CV | |
| FBS | Corning | MT35010CV | |
| Penicillin-Strep (opt.) | Gibco | 15140-122 | |
| 6 well plates | BioLite | 12556004 | |
| TransIT-LTI Transfection Reagent | Mirus | MIR2300 | for lipofection only |
| Opti-MEM | ThermoFisher | 31985062 | for lipofection only |
| G418 (Neomycin) | Sigma Aldrich | A1720-5G | |
| Hygromycin | Sigma Aldrich | H3274-250MG | |
| 96 well plates | ThermoFisher | 12556008 | |
| Passive Lysis Buffer, 5x | Promega | ||
| 1x SDS loading buffer | recipe decribed in protocol | ||
| Nitrocellulose Membrane | Bio-Rad | 162-0115 | |
| TBST | recipe decribed in protocol | ||
| Dehydrated milk | |||
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher | 34075 | for HRP-conjugated secondary antibodies |
| Extracta DNA prep for PCR | Quantabio | 95091-025 | |
| KOD polymerase | Novagen | 71316 |
Referenzen
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- Bibikova, M., et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol. 21 (1), 289-297 (2001).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Mol Cell Biol. 23 (10), 3558-3565 (2003).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New Tools for Genetic Manipulations from Bacterial Immunity Systems. Annu Rev Microbiol. 69, 209-228 (2015).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Karginov, F. V., Hannon, G. J. The CRISPR system: small RNA-guided defense in bacteria and archaea. Mol Cell. 37 (1), 7-19 (2010).
- Wiedenheft, B., Sternberg, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archaea. Nature. 482 (7385), 331-338 (2012).
- Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J., Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol. 60 (2), 174-182 (2005).
- Pourcel, C., Salvignol, G., Vergnaud, G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151 (Pt 3), 653-663 (2005).
- Brouns, S. J., et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 321 (5891), 960-964 (2008).
- Deltcheva, E., et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 471 (7340), 602-607 (2011).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 322 (5909), 1843-1845 (2008).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Guirouilh-Barbat, J., et al. Impact of the KU80 pathway on NHEJ-induced genome rearrangements in mammalian cells. Mol Cell. 14 (5), 611-623 (2004).
- Moore, J. K., Haber, J. E. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 16 (5), 2164-2173 (1996).
- Liang, F., Han, M., Romanienko, P. J., Jasin, M. Homology-directed repair is a major double-strand break repair pathway in mammalian cells. Proc Natl Acad Sci U S A. 95 (9), 5172-5177 (1998).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Luo, Y., Lin, L., Bolund, L., Sorensen, C. B. Efficient construction of rAAV-based gene targeting vectors by Golden Gate cloning. Biotechniques. 56 (5), 263-268 (2014).
- Green, M. R., Sambrook, J., Sambrook, J. . Molecular cloning: a laboratory manual. , (2012).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten