Method Article
用于全基因组 DNA 分析的超长读数测序
摘要
长读序列极大地促进了复杂基因组的组合和结构变异的表征。我们描述了一种利用纳米粒子测序平台生成超长序列的方法。该方法采用优化的 DNA 提取, 然后进行改进的文库准备, 从人体细胞中产生数百条覆盖适度的千列酶读数。
摘要
第三代单分子 DNA 测序技术提供了更长的读取长度, 可以促进复杂基因组的组装和复杂结构变异的分析。纳米粒子平台通过直接测量 DNA 通过毛孔介导的电流变化来执行单分子测序, 并可以以最小的资本成本产生数百千卡卡酶 (kb) 读数。该平台已被许多研究人员用于各种应用。实现更长的测序读取长度是利用纳米粒子测序平台价值的最关键因素。要产生超长读数, 需要特别考虑避免 DNA 断裂并提高效率, 以生成高效测序模板。在这里, 我们提供了超长 DNA 测序的详细协议, 包括从新鲜或冷冻细胞中提取高分子量 (HMW) DNA, 通过机械剪切或转相酶碎片化构建库, 以及在纳米粒子器件上进行测序。从20-25 微克的 HMW DNA 中, 该方法可通过机械剪切达到 N50 读取长度 50-70 kb, 采用转相酶介导的碎片化方法, 使 n50 读长度达到 900-100 kb。该协议可应用于从哺乳动物细胞中提取的 DNA, 用于检测结构变异和基因组组装的全基因组测序。DNA 提取和酶反应的进一步改进将进一步增加读取长度并扩大其效用。
引言
在过去的十年里, 大规模并行和高精度的第二代高通量测序技术推动了生物医学发现和技术创新的爆炸式增长 1、2、3。尽管技术取得了进步, 第二代平台生成的短读数据在解决复杂的基因组区域方面是无效的, 在检测基因组结构变量 (Sv) 方面也是有限的, 而基因组结构变异在人类中发挥着重要作用。进化和疾病4,5。此外, 短读数据无法解决重复变异, 不适合鉴别遗传变异的单倍型阶段6。
最近在单分子测序方面取得的进展提供了更长的读取长度, 这有助于检测sv 7、8、9的全谱, 并提供了精确、完整的复杂组件微生物和哺乳动物基因组6,10。纳米孔平台通过直接测量 dna 通过毛孔11、12、13介导的电流变化来执行单分子测序。与任何现有的 DNA 测序化学不同, 纳米粒子测序可以实时生成长 (数万到数千千比特) 读数, 而无需依赖聚合酶动力学或 DNA 样本的人工扩增。因此, 纳米孔长读测序 (NLR-seq) 为产生远远超过 100 kb 的超长读取长度带来了巨大的希望, 这将极大地推动基因组和生物医学分析 14, 特别是在低复杂性或丰富的基因组的区域 15。
纳米粒子测序的独特之处在于它具有在不受理论长度限制的情况下产生长读数的潜力。因此, 读取长度取决于 DNA 的物理长度, 而 dna 的物理长度直接受到 DNA 完整性和测序模板质量的影响。此外, 根据操作的程度和所涉及的步骤数量, 如移液力和提取条件, DNA 的质量差异很大。因此, 仅仅应用标准的 DNA 提取协议和制造商提供的库构建方法, 就很难产生长读。为此, 我们开发了一种可靠的方法来生成从采集的细胞颗粒开始的超长读取 (数百千维酶) 测序数据。我们在 DNA 提取和库制备过程中采用了多项改进。我们简化了协议, 以排除导致 DNA 降解和损害的不必要程序。该方案由高分子量 DNA 提取、超长 DNA 文库构建和纳米平台测序两部分组成。对于训练有素的分子生物学家来说, 从细胞采集到完成 HMW DNA 提取通常需要 6小时, 根据剪切方法的不同, 图书馆建设需要90分钟或 8小时, DNA 测序的时间最高为48小时。该协议的使用将使基因组学社区能够提高我们对基因组复杂性的认识, 并获得对人类疾病基因组变异的新见解。
研究方案
注: NLR-seq 协议由三个连续步骤组成: 1) 提取高分子量 (HMW) 基因组 DNA;2) 超长 DNA 文库构建, 包括将 HMW DNA 分解成所需的大小和将测序适配器连接到 DNA 端;3) 将自适应连接的 DNA 加载到纳米孔阵列上 (图 1)。
1. HMW DNA 提取
- 试剂设置。通过将100毫升的 PBS (10倍) 添加到900毫升的水和混合, 制成1x 磷酸盐缓冲液 (1, 000 毫升)。在50毫升管中加入 43.5 mL 的水, 制成裂解缓冲液 (50 毫升)。加入500μl 的 Tris (1 M, pH 8.0)、1毫升氯化钠 (氯化钠) (5m)、2.5 毫升乙二胺四乙酸 (EDTA) (0.5 M, pH 8.0) 和 2.5 mL 十二烷基硫酸钠 (SDS) (10%, wt/vol) 到管和混合良好。
注: 此 PBS 缓冲器可在4°c 下存储长达6个月。预先制作的裂解缓冲液可在 RT 中存储长达2个月。 - 检查细胞死亡率并对细胞进行计数。确保活比 > 85%, 总电池数为 30 x 106.
注: 本协议中使用的细胞来自 HG00733 细胞系, 这是一个人类的波多黎各裔淋巴细胞细胞系, 广泛用于1000个基因组联合会的结构变异分析 (见订购资料的材料表), 属于到国际基因组样本资源。 - 在 rt 处以 200 x g离心5分钟收集细胞, 丢弃培养基, 用 1X pbs 缓冲液的5毫升重新悬浮细胞颗粒 (30 x10 6细胞)。在 RT 以 200 x g的速度再次离心 5分钟, 并丢弃上清液。
注: 这种方法可以接受 25-35 x10 6 的单元格。使用的细胞数量的进一步变化需要进一步优化。细胞颗粒可在-80°c 下储存长达6个月。 - 在 1x PBS 缓冲液的200Μl 中重新使用细胞颗粒。如果使用冷冻细胞颗粒, 用 1x PBS 缓冲液的5毫升清洗。在 RT 以 200 x g离心溶液 5分钟, 丢弃上清液, 并在1x-l 的 1X pbs 缓冲液中重新悬浮电池。
- 在50毫升管中制备10毫升的裂解缓冲液。将200Μl 细胞悬浮液加入裂解缓冲液中, 以最高速度旋转 3秒, 在37°c 下将溶液培养1小时。
- 在裂解液中加入2μl 的 RNase a (100 Mg/ml)。轻轻旋转50毫升管混合样品。在37°c 下将溶液生也就是1小时。
- 在裂解液中加入50μl 蛋白酶 K (20mg/ml)。轻轻旋转50毫升管混合样品。在50°c 下将溶液生也就是2小时。在孵育过程中, 每30分钟轻轻混合一次样品。
- 将50毫升管从50°c 中取出, 让支架在 RT 站5分钟。
- 加入10毫升的苯酚层: 氯仿: 异戊醇 (25:24/1, vol/vol2 vol) 到裂解液中, 并在 RPM 的旋转搅拌机上旋转管 (见材料表) 在转速 20 rpm 的烟罩中, 用副管包包 10分钟, 以防止泄漏在旋转过程中。
- 准备两个50毫升凝胶管 (见材料表), 在 1, 500 x 克离心2分钟。
注: 凝胶在含核酸的水相和有机溶剂之间形成稳定的屏障。 - 将样品酚溶液倒进步骤1.10 中制备的50毫升凝胶管中的一个。在 RT 以 3, 000 x g的速度离心溶液10分钟。
- 将上清液倒进新的50毫升管中。加入10毫升的苯酚层: 氯仿: 异戊醇 (25:24:1, vol/vol/vol)), 并在 RPM 的旋转搅拌机上旋转管, 在转速 20 rpm 的通风柜10分钟。
- 用第二制备的凝胶管重复步骤1.11 一次。
- 将上清液倒进新的50毫升管中。加入25毫升的冰凉100% 乙醇, 用手轻轻旋转管, 直到 DNA 沉淀 (图 2)。
注: 降水方法有助于稳定 HMW DNA。 - 弯曲20Μl 尖端, 制成钩子。小心地取出 HMW dna 与钩子, 让液体下降。
- 将 HMW DNA 放入含有40% 乙醇的50毫升管中。用轻轻倒置管3次来清洗 DNA。
- 重复步骤1.15 一次, 从70% 乙醇管中收集 DNA。
- 将 HMW DNA 放入含有 1.8 mL 乙醇的2毫升管中。
- 在 RT 以 10, 000 x克的速度离心清洗后的 HMW dna, 为 3秒. 通过移液中尽可能多地去除剩余乙醇。
注: 在对残留乙醇进行移液处理时, 不要干扰 DNA 颗粒。 - 在37°c 下将2毫升管培养 10分钟, 盖子打开以干燥样品。
- 如果继续执行步骤 2.1 (机械剪切和1D 结扎测序套件), 在 2 mL 管中添加 1 mL 的 TE (10 mM Tris 和 1 mM EDTA, pH 8.0)。
- 如果继续执行步骤 2.2 (使用基于转座机的碎片和快速测序工具), 请添加200Μl 的 10 mm Tris (pH 8.0) 和 0.02% triton X-100。
注: 请勿打扰 DNA 颗粒。让管处于4°c 的黑暗48小时将有助于样品完全重新悬浮。HMW DNA 可在4°c 下保存长达2周。较长的存储时间或其他存储条件可能会引入更多较短的碎片。
2. 超长 DNA 文库建设
注: 有两种方法来构建超长 DNA 库基于两种不同的剪切方法与纳米粒子测序试剂盒相结合。基于机械剪切的库生成的数据为 50-70 kb 的 N50, 用于图书馆建设大约需要8小时。基于转位数据库片段的库生成的 N50 为 90-100 kb 数据, 仅需90分钟进行库构建。机械剪切协议使用相同版本的测序适配器和纳米孔流动细胞的质量, 从相同的 DNA 输入中获得更高的产量。
- 基于机械剪力的图书馆建设
- 解冻并混合结扎试剂盒中的试剂 (见材料表)。解冻 FFPE DNA 修复缓冲液和结束修复--尾矿缓冲液在冰上, 然后涡旋和旋转下混合。解冻适配器混合 (AMX) 和适配器珠子绑定缓冲器 (ABB) 在冰上, 然后移液器和旋转下来混合。在 RT 中用燃料混合 (RBF) 和洗脱缓冲液 (ELUTION) 解冻运行缓冲器, 然后涡旋和旋转下混合。解冻库在 RT 装载珠子 (LLB) 和移液器, 在使用前混合。
- 解冻后, 将所有套件组件放在冰上。只有在需要的时候才会取出酶。将磁珠带到 RT 使用。
注: 有关要使用的磁珠的建议, 请参见材料表。
- 解冻后, 将所有套件组件放在冰上。只有在需要的时候才会取出酶。将磁珠带到 RT 使用。
- 从步骤1.21.1 检查 HMW DNA 的质量和数量。从 Hmw DNA 管中的三个不同位置使用 P200 宽孔提示将20Μl 的 DNA 输送到新的 1.5 mL 管中。取三个等价物中的 1Μl, 使用荧光计检测浓度, 用 uv 读数检测质量。多次检查以确认结果。
注: 预期结果如图 3 a所示。OD2600280 值大约为 1.9, od26/230 值约为2.3。 - 将剩余的 940μl HMW DNA 转移到具有 P1000 宽孔尖端的50毫升管盖中。
- 将所有 DNA 吸入1毫升注射器, 无需针头。
- 将 27 G 针放入注射器上, 轻轻地将所有 DNA 缓慢 (~ 10秒) 喷射到瓶盖中。从注射器上取下 27 G 针。
- 重复步骤2.1.4 和 2.1.5 29次, 共30次通过针头。
注: 剪切的 HMW DNA 可在4°C 的黑暗中储存长达 24小时. 脉冲场凝胶电泳强烈建议进行质量控制 (QC), 但成本高、耗时长。如果在自动脉冲场凝胶电泳机上执行 QC, 使用5-150kb 协议跑20小时。预期的结果如图 4所示。 - 在 0.2 mL 管中加入100Μl 的剪切 HMW DNA (20μg)、15μl 的 FFPE DNA 修复缓冲液、12μl 的 FFPE dna 修复混合物和16μl 的无核水, 准备 DNA 修复反应。将反应混合, 轻轻闪烁 6次, 然后向下旋转以去除气泡。
- 在20°c 下将反应加氢 60分钟, 将样品转移到带有 P200 宽孔尖端的新 1.5 mL 管中。
- 通过移液或涡旋重新扫描磁珠。在 DNA 修复反应中加入143μl 珠 (1x), 轻轻搅拌6次。在 RPM 晚上20转旋转旋转搅拌机上旋转管30分钟。
- 在 RT 以 1, 000 x x 克的速度将样品向下旋转, 在磁性机架上放置 10分钟. 将管子放在磁性机架上, 并丢弃上清液。
- 将管子放在磁性机架上, 在不干扰颗粒的情况下, 加入400Μl 的新鲜制备的70% 乙醇。30秒后去除70% 的乙醇。
- 重复步骤2.1.11 一次。
- 在 RT 以 1, 000 x克的速度将样品向下旋转 2秒, 将管子放回磁性机架上。去除任何残留的乙醇和空气干燥30秒。不要把颗粒擦干。
- 从磁性机架上取下管, 加入103Μl 的 TE (10 mM Tris 和 1 mM EDTA, pH 值 8.0)。轻轻轻触试管, 确保珠子被覆盖在缓冲液中, 并在 RT 的旋转搅拌机上孵育 30分钟. 每5分钟轻轻轻拍一次管, 以帮助颗粒重新悬浮。
- 将磁架上的珠子颗粒至少 10分钟. 用 P200 宽孔尖端将100μl 的洗脱液转移到 0.2 mL 管中。
- 在 0.2 mL 管中加入100Μl 的修复 HMW DNA、14μl 的端部修补-尾矿缓冲液和7μl 的端部修补尾矿混合, 在 0.2 ml 管中制备端部修复和 Da-tha玲反应。通过轻轻闪烁6次混合反应, 然后向下旋转以去除气泡。
- 在20°c 下将反应培养 60分钟, 然后再在65°c 中培养 20分钟, 然后保持在22°C。使用 P200 宽孔尖端将样品转移到新的 1.5 mL 管中。
- 通过移液或涡旋重新扫描磁珠。在末端修复-尾部反应中加入48μl 的珠子 (0.4倍), 然后轻轻搅拌, 将试管轻弹6次。在 RPM 晚上20转旋转旋转搅拌机上旋转管30分钟。
- 重复步骤 2.1.10-2.1.13 一次。
- 从磁性机架上取下管, 加入33Μl 的 TE (10 mM Tris 和 1 mM EDTA, pH 值 8.0)。轻轻轻触试管, 确保珠子被覆盖在缓冲液中, 并在 RT 的旋转搅拌机上孵育 30分钟. 每5分钟轻轻轻拍一次管, 以帮助颗粒重新悬浮。
- 将磁架上的珠子颗粒至少 10分钟. 用 P200 宽孔尖端将30μl 的洗脱液输送到新的 1.5 mL 管中。取额外的1-2 μL, 使用荧光计检测浓度。
注: 预计在此步骤中恢复5-6 微克。 - 在 1.5 mL 样品管中加入30Μl 的端修复 HMW DNA, 20μl 的适配器混合 (AMX 1D), 并加入50μl 的福音结扎主混合物, 准备结扎反应。混合反应, 在每个连续加法之间轻轻闪烁 6次, 并向下旋转以去除气泡。
- 在 RT 中培养反应60分钟。
- 通过移液或涡旋重新扫描磁珠。在结扎反应中加入40Μl 珠 (0.4 x), 轻轻搅拌6次。在 RPM 晚上20转旋转旋转搅拌机上旋转管30分钟。
- 重复步骤2.1.10 一次。
- 在管中加入400Μl 的适配器珠结合 (ABB) 缓冲器。轻轻轻扫管 6次, 重新悬挂珠子。将管子放回磁性机架上, 将珠子与缓冲器分开, 并丢弃上清液。
- 重复步骤2.1.26 一次。
- 在 RT 以 1, 000 x克的速度将样品向下旋转 2秒, 将管子放回磁性机架上。取出任何残留的缓冲液, 并将空气干燥30秒。不要把颗粒擦干。
- 从磁性机架上取下管子, 然后在43μl 洗脱缓冲液中重新悬浮颗粒。轻轻轻触试管, 确保珠子被覆盖在缓冲液中, 并在 RT 旋转搅拌机上孵育 30分钟. 每5分钟轻轻轻触管, 以帮助颗粒重新悬浮。
- 将磁架上的珠子颗粒至少 10分钟. 用 P200 宽孔尖端将40μl 的洗脱液转移到新的 1.5 mL 管中。取额外的1-2 μL, 使用荧光计检测浓度。
注: 预计在此步骤中恢复1-2 微克。基于机械剪切的库已准备好加载。该库可以在冰上存储长达 2小时, 直到加载进行排序 (如果需要)。
- 解冻并混合结扎试剂盒中的试剂 (见材料表)。解冻 FFPE DNA 修复缓冲液和结束修复--尾矿缓冲液在冰上, 然后涡旋和旋转下混合。解冻适配器混合 (AMX) 和适配器珠子绑定缓冲器 (ABB) 在冰上, 然后移液器和旋转下来混合。在 RT 中用燃料混合 (RBF) 和洗脱缓冲液 (ELUTION) 解冻运行缓冲器, 然后涡旋和旋转下混合。解冻库在 RT 装载珠子 (LLB) 和移液器, 在使用前混合。
- 基于转移式碎片的图书馆建设
- 从转座酶试剂盒中解冻试剂 (见材料表)。解冻破碎混合 (FRA) 和快速适配器 (RAP) 在冰上和移液器混合。解冻测序缓冲液 (SQB), 加载珠子 (LB), 冲洗缓冲液 (FLB) 和冲洗系绳 (FLT) 在 RT 和移液器混合。在 RT 上装载珠子 (LB), 在使用前混合。解冻后, 将所有套件组件放在冰上。只有在需要的时候才会取出酶。
- 从步骤1.21.2 检查 HMW DNA 的质量和数量。从 Hmw DNA 管中的三个不同位置使用 P200 宽孔提示将20Μl 的 DNA 输送到新的 1.5 mL 管中。取三个等价物中的 1Μl, 使用荧光计检测浓度, 用 uv 读数检测质量。多次检查以确认结果。
注: 预期结果如图 3 b所示。OD2600280 值大约为 1.9, od26/230 值约为2.3。 - 在 0.2 mL 管中加入22Μl 的 HMW DNA, 10mm Tris (pH 8.0) 的 1Μl, 并加入0.02% 的 Triton X-100 和1μl 的碎裂混合物 (FRA), 在 0.2 ml 管中制备 DNA 标记反应。通过与 P200 宽孔尖的移液混合尽可能缓慢 6次, 注意不要引入气泡。
- 在30°c 下将反应培养 1分钟, 然后在80°c 下培养 1分钟, 然后在4°c 下保持。将混合物转移到一个新的 1.5 mL 管与 P200 宽孔提示, 并立即进入下一步。
- 在 1.5 mL 样品管中加入1μl 快速适配器 (RAP)。通过与 P200 宽孔尖的移液混合尽可能缓慢 6次, 注意不要引入气泡。
- 在 RT 中培养反应60分钟。
注: 基于转储碎片的库已准备好加载。该库可以在冰上存储长达 2小时, 直到加载进行排序 (如果需要)。
3. 纳米孔装置上的测序
- 检查纳米粒子测序装置 (参见材料表)。确保软件和硬件都正常工作, 并且有足够的存储空间。
- 检查流动单元。打开一个新的流动单元, 并将流动单元插入纳米孔设备。选中流单元插入到的位置 (X1-x5) 的框。选择正确的流单元格类型。单击 "检查流单元格" 工作流.单击 "开始测试"按钮, 开始流单元 qc 分析。
注: 如果报告的总活动孔数小于 800, 请使用不同的新流动单元进行测序。 - 准备启动缓冲区。对于基于机械剪切的文库, 在 1.5 mL 管中加入576μl 的运行缓冲液 (RBF) 和624μl 的无核酸酶水。涡流和旋转向下混合启动缓冲液。对于基于转座片碎片的库, 在冲洗缓冲液 (FLB) 管中加入30Μl 的冲洗系 (FLT)。涡流和旋转向下混合启动缓冲液。
- 在流动单元上, 顺时针移动启动端口盖, 以暴露启动端口。
- 将 P1000 移液器设置为 100μl, 并将尖端插入启动口。减少少量的缓冲液 (小于 30μl), 从流动单元中取出任何气泡。一旦少量黄色液体进入尖端, 就停止移液。
- 使用 P1000 移液器通过启动口将启动混合物的800μl 加载到流动单元中。为避免引入气泡, 请先添加30Μl 的启动混合物以覆盖启动口的顶部, 然后将尖端插入启动端口, 然后慢慢添加其余的启动混合物。当剩下大约50Μl 时, 取出尖端。在启动口的顶部添加其余的启动混合物。液体会自行进入体内。
- 让设置孵育5分钟。同时, 在包含库的 1.5 mL 管中准备库组合。
注: 对于基于机械剪切的库, 在 dna 库的40Μl 中添加35Μl 的带有燃料混合 (RBF) 的运行缓冲液。对于基于转座酶片段的文库, 在 dna 文库中添加34Μl 测序缓冲液 (SQB) 和16μl 无核水。 - 轻轻打开流单元样品端口盖, 露出样品端口。如步骤3.5 中所述, 使用 P1000 移液器通过启动口将启动混合物的200Μl 添加到流动单元中。确保启动混合物不会通过样品端口加载到流动单元中。
- 将 P200 移液器设置为80μl。只需在加载前向上和向下移液 6次, 将库与宽孔尖端轻轻混合。
- 通过采样端口将库混合向下加载到流单元中。只有在上一个放置完全加载到端口后, 才添加每个放置。
- 轻轻地将样品口盖放回原处, 确保样品口完全覆盖。逆时针移动启动端口盖, 以覆盖启动端口。合上设备盖。
- 单击"新实验" 工作流。键入库名称, 根据使用的过程选择正确的套件, 并检查设置是否正确 (48小时运行, 实时基本调用 ON)。
- 单击 "开始运行"。10分钟后, 从运行信息中记录流动单元 ID 和活动纳米孔数 (总数和每四个组的数字)。
- 数据分析。在排序的任何时候或运行完成时, 将数据复制到本地计算机或群集。使用 Minimap2 16 (https://github.com/lh3/minimap2) 将序列数据与参考基因组对齐。用 NanoPlot 17 (Https://github.com/wdecoster/NanoPlot) 从原始序列数据和对齐中总结了测序性能。
结果
超长 DNA 测序协议将 HMW DNA 应用于图书馆建设。因此, 在细胞收获步骤中选择活比 & gt;85 的培养良好的细胞至关重要。用于 DNA 提取的细胞数量将影响 HMW DNA 的质量和数量。如果从太多的细胞开始, 细胞裂解效果不好。使用太少的细胞不能产生足够的 DNA 用于文库构建, 因为 HMW DNA 沉淀是通过手轻轻旋转而不是高速离心进行的。图 2中添加冰凉100% 乙醇并旋转后的 HMW dna 示例显示为白色棉花状沉淀物。
在开始库建设之前, 检查输入 DNA 的质量是很重要的。降解、不正确的定量、污染 (例如蛋白质、Rna、洗涤剂、表面活性剂、残留的苯酚或乙醇) 和低分子量 DNA 可对后续程序和最终读取长度产生重大影响。我们建议使用含有 HMW DNA 的管道中三个不同位置的 DNA 进行 QC 分析。从 HMW DNA 的紫外线读数结果中, OD260/od280值约为 1.9, od260 /230 值约为 2.3 (图 3a, b)。在三个测试中, 这些比率值是一致的, 以获得良好的 HMW DNA 样本。不同的剪切方法需要不同体积的输入 DNA。HMW DNA 的浓度需要 gt;200 ngμμl 用于机械剪切, 而它需要是和 gt;1 μμμl 用于转座酶的碎裂。荧光计检测到的浓度比紫外线读数略低。然而, 相同的 HMW DNA 样品的浓度的变异系数要求小于15% 的荧光计和紫外线读数检测。机械剪切应用带有针头的注射器来破坏 HMW DNA, 以便通过针头的次数将影响剪切 DNA 的大小和最终读取长度。建议在剪针后执行尺寸 QC, 以确保大多数 HMW DNA 大于 50kb, 如图 4所示。在机械剪切法中, 考虑到长度和输出, 30次传递产生了最佳的测序结果。
基于机械剪切的库的 N50 为 50-70 kb, 而基于移位的移位块库的 n50 为 90-100 kb。使用 HG00733 细胞系的四次运行结果见表 1。所有四次跑动的读数都超过 2, 300次, 长度超过 100 kb。与基于机械剪切的库 (348 kb 和 387kb) 相比, 基于转载碎片的库的最大长度更长, 而后者的总读数更多, 表明产量较高。基于转储数据库片段的库构造具有更少的步骤和更短的准备时间, 因此它将引入较少的短片段。使用转相酶的两个运行具有较长的平均长度 (& gt;30 kb) 和中位长度 (& gt;10 kb)。此外, 数据显示所有运行都具有一致的高质量 (平均质量得分约为 10.0, ~ 90% 的基本精度)。超过97% 的总碱基使用具有默认设置的 Minimap216与人类参考基因组 (hg19) 对齐。原始读取的预期大小分布如图 5所示。所有运行都有很大比例的数据超过 50 kb, 而移位碎片库具有较高的超长读取率 (例如 > 100 kb)。该协议已成功应用于多个人体细胞系 (补充表 1)。
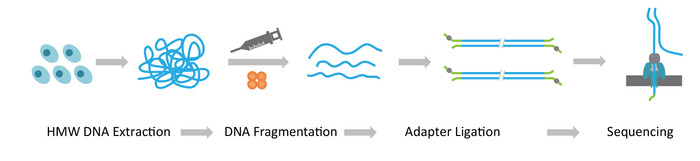
图 1: 纳米孔长读测序 (nlr-seq) 工作流的示意图概述.橙色, 转座酶复合物。黄绿色, 纳米粒子适配器。请点击这里查看此图的较大版本.

图 2: 从酚-氯仿提取法中提取的代表性 DNA 沉淀.白色箭头表示 HMW DNA。请点击这里查看此图的较大版本.

图 3: uv 读数的 HMW DNA 的质量控制结果示例.(a) HMW dna 1.21.1 准备好进行机械剪力库建设。(b) 基于移位碎片的图书馆建设的步骤 1.21.2 HMW dna。请点击这里查看此图的较大版本.

图 4: 通过脉冲场凝胶电泳剪伤 HMW DNA 的质量控制结果.L1: 快速加载 1 kb DNA 阶梯;L2: 快速加载 1 kb 延长 DNA 阶梯。1-8: DNA 具有不同的通过时间通过针剪切。1-3, 无剪切;4、10次;5, 20次;6, 30次;7, 40次;8, 50 次。此 QC 步骤是可选的。请点击这里查看此图的较大版本.

图 5: 纳米粒子超长 dna 文库的预期粒径分布.基于 m s、机械剪力的库。TF, 基于片段的转换库。请点击这里查看此图的较大版本.
| 机械 shearing_rep1 | 机械 shearing_rep2 | 转移酶 fragmentation_rep1 | 转移酶 fragmentation_rep2 | |
| 细胞系 | HG00733 | HG00733 | HG00733 | HG00733 |
| N50 的读数 | 55 180 | 63, 007 | 98, 237 | 95, 629 |
| 超过 100 Kb 的读取次数 | 2, 500 | 3, 082 | 2, 386 | 2, 355 |
| 总读取数 | 97 859 | 80, 465 | 24, 166 | 2, 032 |
| 最大长度 (bp) | 348, 482 | 387 113 | 445 660 | 489 426 |
| 平均长度 (bp) | 17 861 | 20, 395 | 33, 528 | 38 175 |
| 中位长度 (bp) | 5, 335 | 5, 894 | 10, 249 | 15 656 |
| 读取的平均质量 | 10。0 | 10。1 | 9。8 | 10。0 |
| 原始读取的总基础 | 747 889 822 | 1, 411. 932 | 810 229 733 | 80.28, 88 |
| 对齐读取的总基数 | 1, 693 300 832 | 1, 67, 97 5, 925 | 79, 442, 077 | 778 417 627 |
| 总碱基的映射比率 (hg19, Minimap2) | 99.9% | 98.0% | 97.7% | 9.0% |
| 活动毛孔的数量 | 1225:480, 402, 254, 89 | 1058:480, 356, 176, 46 | 958:452, 328, 148, 30 | 1092:487, 367, 195, 43 |
表 1: 具有不同剪切协议的运行的性能指标摘要。
| 图书馆1 | 图书馆2 | |
| 细胞系 | K562 | GM19240 |
| 细胞订购信息 | ATCC, 猫。大声 笑CCL-243 | 科瑞尔研究所, 猫。大声 笑GM19240 |
| 协议 | 机械剪切 | 机械剪切 |
| N50 的读数 | 60, 063 | 55 295 |
| 总读取数 | 193, 783 | 120 807 |
| 中位长度 (bp) | 1, 843 | 4, 688 |
| 平均长度 (bp) | 9 825 | 17 408 |
| 最大长度 (bp) | 588 780 | 212 338 |
| 原始读取的总基础 | 1, 903, 989, 686 | 2, 103, 15, 331 |
| 对齐读取的总基数 | 1, 837, 350, 047 | 1, 997, 419 761 |
| 总碱基的映射比率 (hg19, Minimap2) | 966% | 950% |
| 活动毛孔的数量 | 1111:482, 371, 203, 55 | 1032:447, 333, 196, 56 |
补充表 1: 使用具有机械剪切协议的其他细胞系运行的两个 NLR-seq 摘要。
讨论
原则上, 纳米粒子测序能够产生100kb 到超位的读数, 长度为 11,12,13。影响测序运行性能和数据质量的主要因素有四个: 1) 活动孔数和毛孔活性;2) 运动蛋白, 控制 DNA 通过纳米孔的速度;3) DNA 模板 (长度、纯度、质量、质量);4) 测序适配器结扎效率, 这决定了输入样本中可用的 DNA。前两个因素取决于流单元的版本和制造商提供的测序试剂盒。第二个因素是该协议中的关键步骤 (HMW DNA 提取、剪切和结扎)。
这个协议需要耐心和实践。HMW DNA 的质量对超长 DNA 文库具有重要意义.该协议从收集的细胞开始, 细胞的活性很高 (& gt;85% 的活细胞首选), 限制了死亡细胞的降解 DNA。任何可能对 DNA 造成损害的苛刻过程 (例如, 强烈的干扰、晃动、涡旋、多次移液、反复冻结和解冻) 都应避免。在协议的设计中, 我们在 DNA 提取的整个过程中忽略了移液。在库的结构和测序过程中, 机械剪切后需要移液时, 需要使用宽孔前置。由于纳米孔对腔内缓冲液 12中的化学物质敏感, 因此 dna 中的残留污染物 (例如洗涤剂、表面活性剂、苯酚、乙醇、蛋白质 rna 等) 应尽可能少。考虑到苯酚的长度和收率, 与目前测试的多种不同提取方法相比, 苯酚提取方法显示出最佳和最可重复的结果。
尽管此协议能够生成长读取序列, 但仍存在一些限制。首先, 根据发布时可用的纳米粒子测序装置对该协议进行了优化;因此, 它仅限于选择性纳米粒子测序化学, 在其他类型的长读测序器件中进行测序时可能是次优的。其次, 结果在很大程度上取决于从起始材料 (组织或细胞) 中提取的 DNA 的质量。如果起始 DNA 已经退化或损坏, 读取长度将受到影响。第三, 虽然在检查 DNA 质量的协议中纳入了多个 QC 步骤, 但读数的最终产量和长度可能会受到流动细胞和孔隙活动的影响, 而流动细胞和孔隙活动可能会在纳米粒子测序平台的这一早期阶段发生变化发展。
这里描述的协议使用人类悬浮细胞系样本进行 DNA 提取。优化了剪针过程、HMW DNA 转相酶的比例和结扎时间, 得出了所述的结果。该协议可以通过四种方式扩展。首先, 使用者可以从其他培养的哺乳动物细胞开始, 并与不同数量的细胞、组织、临床样本或其他生物结合使用。需要进一步优化裂解孵育时间、反应量和离心。其次, 很难预测超长读取测序的目标大小。如果读取长度比预期的要短, 用户可以调整基于机械剪切方法的传递时间, 或在基于移位的方法中更改 HMW DNA 与移位酶的比率。在清理步骤中更长的结合和洗脱时间是有帮助的, 因为 HMW DNA 具有很高的粘性。第三, 利用不同的纳米粒子测序装置, 可以调整 DNA 的数量和体积, 以满足测序器的标准。第四, 只有那些 DNA 连接到测序适配器将被测序。为了进一步提高结扎效率, 可以尝试滴定适配器和连接酶浓度。改性结扎时间和聚乙二醇18等分子拥挤剂可用于未来。超长 dna 测序协议与 crispr19,20相结合, 可为目标浓缩测序提供有效的工具。
披露声明
提交人声明, 他们没有相互竞争的经济利益。
致谢
作者感谢朱先生对手稿的评论。该出版物中报告的研究得到了国家卫生研究院国家癌症研究所 P30CA034196一奖的部分支持。内容完全由作者负责, 不一定代表国家卫生研究院的官方观点。
材料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Absolute ethanol | Sigma-Aldrich | E7023 | |
| Agencourt AMPure XPbeads | Beckman | A63881 | magnetic beads for cleanup |
| BD conventional needles | Becton Dickinson | 305136 | 27G, for mechanical shearing |
| BD Luer-Lok syringe | Becton Dickinson | 309628 | for mechanical shearing |
| Blunt/TA Ligase Master Mix | NEB | M0367S | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | for cell counting |
| EDTA | Invitrogen | AM9261 | pH 8.0, 0.5 M, 500 mL |
| Flow Cell | Oxford Nanopore Technologies | FLO-MIN106 | R9.4.1 |
| HG00773 cells | Coriell Institute | HG00733 | cells used in this protocol |
| Ligation Sequencing Kit 1D | Oxford Nanopore Technologies | SQK-LSK108 | nanopore ligation kit |
| MaXtract High Density tubes | Qiagen | 129073 | gel tubes |
| NEBNext FFPE DNA Repair Mix | NEB | M6630S | |
| NEBNext Ultra II End Repair/dA-Tailing Module | NEB | M7546S | |
| Nuclease-free water | Invitrogen | AM9937 | |
| Phosphate-Buffered Saline, PBS | Gibco | 70011044 | 10X, pH 7.4 |
| Phenol:chloroform:IAA | Invitrogen | AM9730 | |
| Proteinase K | Qiagen | 19131 | 20 mg/mL |
| Qubit dsDNA BR Assay Kit | Invitrogen | Q32850 | fluorometer assays for DNA quantification |
| Rapid Sequencing Kit | Oxford Nanopore Technologies | SQK-RAD004 | nanopore transposase kit |
| RNase A | Qiagen | 19101 | 100 mg/mL |
| SDS | Invitrogen | AM9822 | 10% (wt/vol) |
| Sodium chloride solution | Invitrogen | AM9759 | 5.0 M |
| TE buffer | Invitrogen | AM9849 | pH 8.0 |
| Tris | Invitrogen | AM9856 | pH 8.0, 1 M |
| Triton X-100 solution | Sigma-Aldrich | 93443 | ~10% |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Bio-Rad C1000 Thermal Cycler | Bio-Rad | 1851196EDU | |
| Centrifuge 5810R | Eppendorf | 22628180 | |
| Countess II FL Automated Cell Counter | Life Technologies | AMQAF1000 | for cell counting |
| DynaMag-2 Magnet | Life Technologies | 12321D | magnetic rack |
| Eppendorf ThermoMixer | Eppendorf | 5382000023 | for incubation |
| Freezer | LabRepCo | LHP-5-UFMB | |
| GridION | Oxford Nanopore Technologies | GridION X5 | nanopore device used in this protocol |
| HulaMixer Sample Mixer | Thermo Fisher Scientific | 15920D | rotator mixer |
| MicroCentrifuge | Benchmark Scientific | C1012 | |
| NanoDrop ND-1000 Spectrophotometer | Thermo Fisher Scientific | ND-1000 | for UV reading |
| Pippin Pulse | Sage Science | PPI0200 | pulsed-field gel electrophoresis instrument |
| Qubit 3.0 Fluorometer | Invitrogen | Q33216 | fluorometer |
| Refrigerator | LabRepCo | LABHP-5-URBSS | |
| Vortex-Genie 2 | Scientific Industries | SI-A236 | |
| Water bath | VWR | 89501-464 |
参考文献
- Mardis, E. R. Next-generation sequencing platforms. Annual Review of Analytical Chemistry. 6, 287-303 (2013).
- Goodwin, S., McPherson, J. D., McCombie, W. R. Coming of age: ten years of next-generation sequencing technologies. Nature Reviews Genetics. 17 (6), 333-351 (2016).
- Shendure, J., et al. DNA sequencing at 40: past, present and future. Nature. 550 (7676), 345-353 (2017).
- Alkan, C., Coe, B. P., Eichler, E. E. Genome structural variation discovery and genotyping. Nature Reviews Genetics. 12 (5), 363-376 (2011).
- Weischenfeldt, J., Symmons, O., Spitz, F., Korbel, J. O. Phenotypic impact of genomic structural variation: insights from and for human disease. Nature Reviews Genetics. 14 (2), 125-138 (2013).
- Pollard, M. O., Gurdasani, D., Mentzer, A. J., Porter, T., Sandhu, M. S. Long reads: their purpose and place. Human Molecular Genetics. 27 (R2), R234-R241 (2018).
- Cretu Stancu, M., et al. Mapping and phasing of structural variation in patient genomes using nanopore sequencing. Nature Communications. 8 (1), 1326(2017).
- Gong, L., et al. Picky comprehensively detects high-resolution structural variants in nanopore long reads. Nature Methods. 15 (6), 455-460 (2018).
- Sedlazeck, F. J., et al. Accurate detection of complex structural variations using single-molecule sequencing. Nature Methods. 15 (6), 461-468 (2018).
- Jain, M., et al. Nanopore sequencing and assembly of a human genome with ultra-long reads. Nature Biotechnology. 36 (4), 338-345 (2018).
- Jain, M., et al. Improved data analysis for the MinION nanopore sequencer. Nature Methods. 12 (4), 351-356 (2015).
- Deamer, D., Akeson, M., Branton, D. Three decades of nanopore sequencing. Nature Biotechnology. 34 (5), 518-524 (2016).
- Jain, M., Olsen, H. E., Paten, B., Akeson, M. The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community. Genome Biology. 17 (1), 239(2016).
- Editorial, The long view on sequencing. Nature Biotechnology. 36 (4), 287(2018).
- Jain, M., et al. Linear assembly of a human centromere on the Y chromosome. Nature Biotechnology. 36 (4), 321-323 (2018).
- Li, H. Minimap2: pairwise alignment for nucleotide sequences. Bioinformatics. 34, 3094-3100 (2018).
- De Coster, W., D'Hert, S., Schultz, D. T., Cruts, M., Van Broeckhoven, C. NanoPack: visualizing and processing long-read sequencing data. Bioinformatics. 34, 2666-2669 (2018).
- Akabayov, B., Akabayov, S. R., Lee, S. J., Wagner, G., Richardson, C. C. Impact of macromolecular crowding on DNA replication. Nature Communications. 4, 1615(2013).
- Gabrieli, T., Sharim, H., Michaeli, Y., Ebenstein, Y. Cas9-Assisted Targeting of CHromosome segments (CATCH) for targeted nanopore sequencing and optical genome mapping. bioRxiv. , (2017).
- Gabrieli, T., et al. Selective nanopore sequencing of human BRCA1 by Cas9-assisted targeting of chromosome segments (CATCH). Nucleic Acids Research. , (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。