Method Article
全 Genomic DNA 分析のための超長いリード シーケンス
要約
長い読み取りシーケンス大きく複雑なゲノム構造の変化の特徴付けとアセンブリを容易にします。ナノポア ベースのシーケンスのプラットフォームで超長いシーケンスを生成する方法について述べる。アプローチは、人間の細胞から中程度の範囲でプラスミド読み取りの数百人を生成する変更したライブラリの準備に続いて最適化された DNA の抽出を採用しています。
要約
第三世代の 1 分子 DNA シーケンシング技術提供大幅に長く複雑なゲノムと複雑な構造の変形解析のアセンブリを容易にすることができます長さをお読みください。ナノポア プラットフォームは、直接 DNA の通過孔を介した現在の変更の測定によって単一分子シーケンス処理の実行し、最小限の資本コストでプラスミド (kb) の読み取りの数百を生成することができます。このプラットフォームは、さまざまなアプリケーションの多くの研究者によって採用されています。ナノ細孔シーケンシング プラットフォームの価値を活用する最も重要な因子は、長いシーケンスの読み取り距離を達成するため。超長い読み取りを生成するには、DNA の破損を避けるために、生産的な配列のテンプレートを生成する効率を得るために特別な配慮が必要です。ここでは、超長い DNA の塩基配列など新鮮なまたは冷凍細胞、機械的剪断またはトランスポザーゼ断片化によってライブラリーの構築から高分子量分画法 DNA 抽出とナノポア デバイスをシーケンス処理の詳細なプロトコルを提供します。HMW DNA の 20-25 μ g から 90 100 kb の読み取りトランスポザーゼと長さの N50 媒介断片化、およびメソッドは N50 機械的剪断で 50-70 kb の長さの読み出しを実現できます。プロトコルは哺乳類セルから抽出した DNA の構造変形とゲノムのアセンブリの検出の全ゲノム配列を実行するに適用できます。DNA の抽出および酵素反応の追加改善さらにリードの長さを増やすのユーティリティを展開します。
概要
過去 10 年間超並列でかつ精度の高い第二世代高スループット シーケンス テクノロジーは生物医学的発見と技術革新1,2,3の爆発を駆動しています。技術の進歩にもかかわらず第 2 世代のプラットフォームで生成された短鎖リード データは複雑なゲノム領域の解決に効果がないと人間に重要な役割を担うゲノムの構造変形 (SVs) の検出に限られています。進化と病気の4,5。さらに、データの短い読み取りは繰り返し変動を解決することができないおよびハプロタイプが遺伝的変異6段階目の肥えたに適しています。
単一分子の配列を提供しています大幅に長くの最近の進歩を読む長さは、SVs7,8,9の完全なスペクトルおよび複合体の提供正確かつ完全なアセンブリの検出を容易にすることができます。微生物および哺乳類ゲノム6,10。ナノポア プラットフォームは、直接 DNA の通過孔11,12,13を介した現在の変更の測定によって単一分子シーケンスを実行します。ポリメラーゼの反応に依存することがなく、リアルタイムまたは人工に、ナノ細孔シーケンシングが長い (kilobases の何千もの数十) 読み取りを生成とは異なり、任意の既存の DNA シーケンスの化学、DNA サンプルの増幅。したがって、ナノ細孔長い読み低複雑さや繰り返しリッチで特に大きく前進するでしょうゲノム ・ バイオメディカル分析14、100 kb を越えて超長い読み取り距離を生成する (NLR seq) 保持している偉大な約束をシーケンス処理ゲノム15の地域。
ナノ細孔シーケンシングのユニークな特徴は、長いを生成するその可能性を理論上の長さ制限なし読み取りです。したがって、読み取りの長さは DNA の整合性とシーケンス テンプレートの品質によって直接影響を受ける DNA の物理的な長さによって異なります。また、操作やピペッティング力と抽出条件など、関連するステップの数の範囲によって DNA の品質は非常に可変。したがって、ちょうど標準の DNA の抽出のプロトコルや製造元の提供されるライブラリ工法を適用することによって長時間の読み取りを生成する 1 つの挑戦です。これに向けて、堅牢な開発した超長を生成する方法を読む (何百もの kilobases の) 収穫した細胞ペレットから始まるシーケンス データ。DNA の抽出とライブラリの準備の手順で複数の改善点を採用しました。我々 は DNA の劣化や損害賠償を引き起こす不必要な手順を除外するプロトコルを簡素化。このプロトコルは高分子量分画法で構成される DNA の抽出、超長い DNA ライブラリ構築およびナノポア プラットフォームの配列。よく訓練された分子生物学者の通常かかります 6 h セル HMW DNA 抽出、90 分またはライブラリ構築せん断方法によって、DNA シークエンシングのためさらに 48 h までの 8 h の完了するために収穫から。プロトコルの使用はゲノムの複雑性の私達の理解を改善し、人間の病気のゲノムの変化に新しい洞察力を得るためにゲノムのコミュニティに権限を与えます。
プロトコル
注: NLR seq プロトコル 3 連続ステップで構成されています: 1) 高分子の抽出重量分画法ゲノム DNA;任意の大きさに見られる DNA の断片化と dna シーケンス アダプターの ligation が含まれています 2) 超長い DNA ライブラリ構築終了します。・ 3) アダプター結紮 DNA ナノ細孔 (図 1) のアレイへの読み込み。
1. 高分子 DNA の抽出
- 試薬のセットアップ。100 mL の PBS を追加することによってリン酸バッファー生理食塩水 (PBS) バッファー (1,000 mL) × 1 を作る (10 倍) を 900 mL の水とよく混ぜる。換散バッファー (50 mL) を 50 mL チューブに 43.5 mL の水を追加してください。エチレンジアミン四酢酸 (EDTA) (0.5 M、pH 8.0) の 2.5 mL トリス (1 M, pH 8.0)、塩化ナトリウム (NaCl) (5 M) の 1 つの mL の 500 μ L を追加し、ナトリウム ドデシル 2.5 mL チューブ (SDS) (10%、wt/巻) の硫酸塩しよく混ぜます。
注: この PBS バッファーは 4 ° C で最大 6 ヶ月間保存できます。既成の換散バッファーは、最大 2 ヶ月間常温保存できます。 - セル死亡を確認し、セルをカウントします。ライブは、> 85%、総細胞数は 30 × 106を確認します。
注: このプロトコルで使用される細胞は、1000 ゲノム コンソーシアム構造変化分析のため広くプエルトリコ出身のヒトリンパ芽球細胞株 HG00733 細胞株 (注文情報用材料の表を参照)、所属国際ゲノム サンプル リソース。 - RT。 破棄中で 5 分間 200 × gで遠心分離によって細胞を収集し、1 x PBS バッファーの 5 ml (30 x 106細胞) 細胞ペレットを再懸濁します。再び常温 5 分 200 x gで遠心し、上澄みを廃棄します。
注: 25-35 10 x6セルはこのアプローチのため。さらにさまざまな量の使用されているセルは、最適化に必要。細胞ペレットは、−80 ° C で 6 ヶ月保存できます。 - 1 x PBS バッファーの 200 μ l 細胞ペレットを再懸濁します。凍結細胞ペレットを使用している場合は、5 mL の 1x PBS バッファーで洗浄します。遠心分離機の RT で 5 分間 200 x gでソリューション、上澄みを廃棄し、PBS バッファー x 1 の 200 μ L で細胞を再懸濁します。
- 10 mL を 50 mL のチューブでの換散バッファーを準備します。3 s. 加温 1 h 37 ° C でソリューションの換散バッファーと最高速度で渦に 200 μ L 細胞懸濁液を追加します。
- ライセートに rnase A 2 μ L (100 mg/mL) を追加します。サンプルをミックスに 50 mL のチューブを慎重に下ろします。1 h の 37 ° C でソリューションを孵化させなさい。
- ライセートに 50 μ L プロティナーゼ K (20 mg/mL) を追加します。サンプルをミックスに 50 mL のチューブを慎重に下ろします。2 h 50 ° C でソリューションを孵化させなさい。、インキュベーション中に、30 分毎にサンプルをミックス優しく。
- 50 ° C から 50 mL のチューブを外し、5 分間室温で放置します。
- ライセートにフェノール: クロロホルム: イソアミル アルコール (25:24:1、巻/巻/巻) のフェノール層の 10 mL を追加し、回転ミキサーでチューブを回転させて (材料表参照) 常温で 10 分間 20 rpm で発煙のフード ラップ漏れを防ぐためにパラフィルムでチューブ キャップ回転中に
- 右で 2 分間 1,500 × gで遠心分離 (材料の表を参照してください)、2 つの 50 mL ゲル管を準備します。
注: ゲルは、核酸を含む水相と有機溶剤の安定したバリアを形成します。 - ステップは 1.10 からゲル チューブ、50 mL の準備の一つにサンプル/フェノール溶液を注ぐ。3,000 × g 10 分間室温で溶液を遠心分離します。
- 新しい 50 mL のチューブに上清を注ぐ。フェノール: クロロホルム: イソアミル アルコール (25:24:1、巻/巻/巻) のフェノール層の 10 mL を追加し、10 分の 20 の rpm で発煙のフード RT で回転子のミキサーのチューブを回転させます。
- 2 番目に一度準備ステップ 1.11 ゲル管を繰り返します。
- 新しい 50 mL のチューブに上清を注ぐ。冷えた 100% エタノール 25 mL を追加し、DNA を沈殿させる (図 2) まで、チューブを手で優しく回転します。
注: 降水量アプローチは、高分子 DNA を安定させるために役立ちます。 - フックに 20 μ L の先端を曲げます。慎重にフックを用いて DNA を取り出し、液体のドロップはオフしましょう。
- 70% のエタノールの 40 mL を含む 50 mL のチューブに高分子 DNA を配置します。3 回チューブを軽く反転による DNA を洗浄します。
- 1.15 一度 70% エタノール チューブから DNA を収集する手順を繰り返します。
- 70% のエタノールの 1.8 mL を含む 2 mL チューブに高分子 DNA を配置します。
- 3 10,000 x gで洗った HMW DNA を遠心分離機ピペッティングにより可能な限り残留エタノールのルート削除で s。
注: 残留エタノールをピペッティング時に、DNA の餌に不可します。 - 蓋が開いた試料を乾燥させると 10 分のための 37 ° C で 2 mL チューブを孵化させなさい。
- 2.1 機械的剪断と 1 D 結紮シーケンス キット) のステップを続行、2 mL チューブに TE (10 mM トリスと 1 ミリメートルの EDTA、pH 8.0) の 1 つの mL を追加します。
- 0.02% と 10 ミリメートル トリス (pH 8.0) の 200 μ L を追加 (トランスポザーゼ ベースの断片化、急速なシーケンス キットと)、手順 2.2 を続ける場合トリトン X-100。
注:、DNA の餌が不可します。48 h のため暗闇の中で 4 ° C に立つチューブをさせる完全に再懸濁しますサンプルに役立ちます。HMW DNA は、最大 2 週間までの 4 ° C で保存できます。ストレージ時間やその他のストレージの条件より短い断片を導入可能性があります。
2. 超長い DNA ライブラリ構築
注意: ナノポア シーケンス キットと相まって 2 つ異なるせん断方法に基づいて超長い DNA ライブラリを構築する 2 つの方法があります。機械的せん断ベースのライブラリは、ライブラリ構築の約 8 時間を取る 50-70 kb の N50 を使用してデータを生成します。トランスポザーゼ断片化ベースのライブラリは、ライブラリ構築にわずか 90 分を取って 90 100 kb のデータの N50 を生成します。機械的せん断プロトコルは、入力シーケンス アダプターとナノポア フローセルの品質の同一バージョンを使用して同じ DNA からより高い利回りを与えます。
- 機械的剪断に基づく図書館の建設
- 雪解けとミックス結紮から試薬キット (材料の表を参照してください)。雪解け FFPE DNA はバッファーと氷、渦の上終わり修理/ダ-テーリング バッファーを修復し、ミックス ダウンしてスピンします。ミックス ダウン アダプター ミックス (AMX) とアダプター ビーズ結合バッファー (ABB) 氷し、ピペットとスピンを解凍します。ダウン ミックスに燃料ミックス (RBF) と連続したバッファーとスピンし渦 RT で溶出バッファー (エルプ) を解凍します。RT とピペットの使用の前に混合するライブラリのロードのビーズ (LLB) を解凍します。
- 解凍後、氷の上のすべてのキット部品を保ちます。必要なときだけ酵素を取り出してください。Rt 用磁気ビーズをもたらします。
注: 使用する磁気ビーズに関する推奨事項は、材料の表を参照してください。
- 解凍後、氷の上のすべてのキット部品を保ちます。必要なときだけ酵素を取り出してください。Rt 用磁気ビーズをもたらします。
- 1.21.1 のステップから見られる DNA の量と質を確認してください。P200 ワイドを使用して見られる DNA チューブの 3 つの異なる場所から新しい 1.5 mL チューブに DNA の 20 μ L をピペットは、ヒントを退屈させます。蛍光光度計と読んで UV を使用して品質を使用して濃度を検出する 3 つの因数から 1 μ L を取る。複数回、結果を確認するを確認します。
注:図 3 aで期待どおりの結果が表示されます。OD260/280値は約 1.9 と OD260/230値は約 2.3。 - 転送広い P1000 を 50 mL チューブ キャップに見られる DNA の残りの 940 μ L チップを産んだ。
- 1 mL 注射器針なしにすべての DNA を吸い出しなさい。
- 27 G 針を注射器に入れ、静かにゆっくりと、キャップにすべての DNA を取り出す (~ 10 秒)。注射器から 27 G 針を外してください。
- 30 パスの合計 29 回の針を介して手順 2.1.4 と 2.1.5 を繰り返します。
注: せん断破壊 DNA は 24 h. 品質管理 (QC) までは脈打フィールドゲルの電気泳動によって強くお勧めしますが、それは高価、時間のかかる、暗闇の中で 4 ° C で保存できます。自動パルス フィールド ゲル電気泳動機械の品質管理を実行している場合は、実行 20 h 5 150 kb プロトコルを使用します。期待される結果は、図 4のとおりです。 - せん断破壊 DNA の 100 μ L の追加によって 0.2 mL チューブで DNA 修復反応を準備 (20 μ g) FFPE DNA 修復バッファーの 15 μ L、FFPE DNA 修復ミックスの 12 μ L、ヌクレアーゼ フリー水の 16 μ L。6 回軽くフリックすることで反応をミックスし、削除泡までスピンします。
- 60 分転送のための 20 の ° C で反応、p200 でも大口径ヒントを新しい 1.5 mL チューブにサンプルをインキュベートします。
- ピペッティングまたはボルテックスによって磁気ビーズを再懸濁します。6 回チューブをフリックで優しく DNA 修復反応とミックスに 143 μ L ビーズ (1 x) を追加します。30 分の 20 の rpm で RT で回転子のミキサーのチューブを回転させます。
- 2 1,000 x gでサンプル スピンダウンした場所で 10 分間磁気ラックにチューブで s 磁気ラックにチューブを維持し、上澄みを廃棄します。
- 磁気ラックにチューブを維持、ペレットを乱すことがなく作りたての 70% のエタノールの 400 μ L を追加します。30 後 70% エタノールを削除 s。
- 2.1.11 の手順をもう一度繰り返します。
- 2 1,000 x gでサンプルをスピン磁気ラック背面チューブした場所で s。任意の残留エタノールを外し、空気乾燥 30 s。以上、ペレットを乾燥しないでください。
- 磁気ラックからチューブを外し、TE (10 mM トリスと 1 ミリメートルの EDTA、pH 8.0) の 103 μ L を追加します。軽くフリック ビーズ バッファーで覆われているし、軽く 30 分常温回転ミキサーで孵化させなさいようにチューブはペレットの再懸濁のためチューブ 5 分ごとをフリックします。
- P200 でも広いと溶出液の少なくとも 10 分転送 100 μ L 穴 0.2 mL チューブ先端の磁気ラックのビーズをペレットします。
- 修理 HMW DNA の 100 μ L、14 μ L エンド修理/ダ-テーリング バッファーの終わり修理/ダ-テーリング ミックスの 7 μ L を追加して終わり修理および 0.2 mL チューブで dA テーリング反応を準備します。6 回、軽くフリックすることで反応をミックスし、削除泡までスピンします。
- 60 分の 65 ° C 20 分続いて 20 ° C で反応をインキュベートし、22 ° C で押し転送 p200 でもワイドを使用して新しい 1.5 mL チューブにサンプルを退屈させるヒントします。
- ピペッティングまたはボルテックスによって磁気ビーズを再懸濁します。ビーズの 48 μ L を追加 (0.4 倍) 終わり修理/ダ-テーリング反応と静か 6 回チューブをフリックでミックスします。30 分の 20 の rpm で RT で回転子のミキサーのチューブを回転させます。
- 2.1.10-2.1.13 の手順をもう一度繰り返します。
- 磁気ラックからチューブを外し、TE (10 mM トリスと 1 ミリメートルの EDTA、pH 8.0) の 33 μ L を追加します。軽くフリック ビーズ バッファーで覆われているし、軽く 30 分常温回転ミキサーで孵化させなさいようにチューブはペレットの再懸濁のためチューブ 5 分ごとをフリックします。
- P200 でも広いと溶出液の少なくとも 10 分転送 30 μ L を新しい 1.5 mL チューブ先端の穴の磁気ラックのビーズをペレットします。蛍光光度計を用いた濃度を検出する余分 1-2 μ L を取る。
注: この手順で 5-6 μ g の回復が期待されます。 - 最後修理 HMW DNA の追加 30 μ L で 1.5 mL のサンプル チューブの ligation の反作用、アダプター ミックス (1 D)、AMX の 20 μ L、鈍/TA 結紮マスター ミックスの 50 μ L を準備します。それぞれ順次追加と削除泡までスピン間 6 回軽くフリックすることで反応をミックスします。
- 60 分間室温で反応を孵化させなさい。
- ピペッティングまたはボルテックスによって磁気ビーズを再懸濁します。40 μ L ビーズを追加 (0.4 倍) ligation の反作用と静か 6 回チューブをフリックでミックスします。30 分の 20 の rpm で RT で回転子のミキサーのチューブを回転させます。
- 2.1.10 の手順をもう一度繰り返します。
- チューブにアダプター ビーズ結合 (ABB) バッファーの 400 μ L を追加します。軽く 6 倍のチューブをフリックすると、ビードを再停止しなさい。バッファーからビーズを分離し上澄みを廃棄磁気ラックにチューブを置きます。
- 2.1.26 の手順をもう一度繰り返します。
- 2 1,000 x gでサンプルをスピン磁気ラック背面チューブした場所で s。残留バッファーを削除し、空気乾燥 30 s。以上、ペレットを乾燥しないでください。
- 磁気ラックからチューブを外し、43 μ L の溶出バッファーでペレットを再懸濁します。軽くフリック ビーズ バッファーで覆われているし、軽く 30 分常温回転ミキサーで孵化させなさいようにチューブはペレットの再懸濁のためチューブ 5 分ごとをフリックします。
- P200 でも広いと溶出液の少なくとも 10 分転送 40 μ L を新しい 1.5 mL チューブ先端の穴の磁気ラックのビーズをペレットします。蛍光光度計を用いた濃度を検出する余分 1-2 μ L を取る。
注: この手順で 1-2 μ g の回復が期待されます。機械的せん断ベース ライブラリを読み込む準備が整います。ライブラリは、必要な場合に、シーケンスの読み込みまで 2 h までの氷の上格納できます。
- 雪解けとミックス結紮から試薬キット (材料の表を参照してください)。雪解け FFPE DNA はバッファーと氷、渦の上終わり修理/ダ-テーリング バッファーを修復し、ミックス ダウンしてスピンします。ミックス ダウン アダプター ミックス (AMX) とアダプター ビーズ結合バッファー (ABB) 氷し、ピペットとスピンを解凍します。ダウン ミックスに燃料ミックス (RBF) と連続したバッファーとスピンし渦 RT で溶出バッファー (エルプ) を解凍します。RT とピペットの使用の前に混合するライブラリのロードのビーズ (LLB) を解凍します。
- トランスポザーゼ断片化ベース ライブラリの構築
- 雪解け、トランスポザーゼから試薬キット (材料の表を参照してください)。断片化ミックス (FRA) とミックスする氷とピペットの急速なアダプター (RAP) を解凍します。ビーズ (LB) を読み込み配列バッファー (SQB) を解凍、バッファー (FLB) フラッシュし RT でテザー (FLT) をフラッシュし、ミックスするピペットします。使用の前に混合する RT とピペットで読み込みビーズ (LB) を解凍します。解凍後、氷の上のすべてのキット部品を保ちます。必要なときだけ酵素を取り出してください。
- 1.21.2 のステップから見られる DNA の量と質を確認してください。P200 ワイドを使用して見られる DNA チューブの 3 つの異なる場所から新しい 1.5 mL チューブに DNA の 20 μ L をピペットは、ヒントを退屈させます。蛍光光度計と読んで UV を使用して品質を使用して濃度を検出する 3 つの因数から 1 μ L を取る。複数回、結果を確認するを確認します。
注: 期待どおりの結果が図 3 bに表示されます。OD260/280値は約 1.9 と OD260/230値は約 2.3。 - HMW DNA 断片化の 0.02% トリトン X-100 と 1 μ L とトリス (pH 8.0) ミックス (FRA) 10 mM の 1 μ L の 22 μ L の追加によって 0.2 mL チューブで DNA tagmentation 反応を準備します。P200 でもワイドでピペッティングによるミックスを結んだ先端に徐々 に可能な限り 6 回気泡を導入することの世話
- 1 分 1 分の 80 ° C に続いて 30 ° C で反応をインキュベートし、4 ° C で押し転送ミックス広い P200 を新しい 1.5 mL チューブに穴先端とすぐに次のステップに行く。
- 1.5 mL のサンプル チューブに 1 μ L の高速アダプター (RAP) を追加します。P200 でもワイドでピペッティングによるミックスを結んだ先端に徐々 に可能な限り 6 回気泡を導入することの世話
- 60 分間室温で反応を孵化させなさい。
注: トランスポザーゼ断片化ベース ライブラリを読み込む準備が整います。ライブラリは、必要な場合に、シーケンスの読み込みまで 2 h までの氷の上格納できます。
3. ナノポア デバイスの配列
- ナノ細孔シーケンシング デバイスを確認 (材料の表を参照してください)。ソフトウェアとハードウェアが動作と十分な空き容量があることを確認します。
- フロー ・ セルを確認します。新しいフローセルを開き、ナノポア デバイスのフロー ・ セルに挿入します。(X1 X5) 挿入位置フロー ・ セルのボックスを確認します。正しいフロー セルの種類を選択します。流れ電池の確認ワークフローをクリックします。フローセル QC 分析を開始する [テストの開始] ボタンをクリックします。
注: 報告された合計アクティブな孔数が 800 未満の場合は、配列の異なる新しいフローセルを使用します。 - プライミング バッファーを準備します。機械的せん断ベースのライブラリでは、実行中の燃料ミックス (RBF) とバッファーと 624 576 μ L を追加 1.5 mL チューブにヌクレアーゼ フリー水の μ L。渦とミックス プライミング バッファーにスピン。トランスポザーゼ断片化ベースのライブラリでは、フラッシュ バッファー (FLB) のチューブにフラッシュ テザー (FLT) の 30 μ L を追加します。渦とミックス プライミング バッファーにスピン。
- フロー ・ セルのプライミング ポートを公開する時計回りにプライミング ポートカバーを移動します。
- P1000 ピペットを 100 μ L に設定し、先端をプライミング ポートに挿入します。少量のフロー ・ セルから任意の気泡を削除するバッファー (未満 30 μ L) を引きます。先端に入ると黄色い液の少量をピペッティングを停止します。
- P1000 ピペットを使用して、プライミング ポート経由のフロー ・ セルにプライミング ミックスの 800 μ L を読み込みます。気泡を導入することを回避するには、最初、プライミング ポートの上部を覆う、プライミング ポートに先端を挿入し、ゆっくりとプライミング ミックスの残りの部分を追加するためにプライミング ミックスの 30 μ L を追加します。約 50 μ L 左がある場合は、先端を取り出してください。プライミング プライミング ポートの上にミックスの残りの部分を追加します。流体は、自分自身で内部を行くでしょう。
- 5 分間インキュベートするセットアップを残します。一方で、ライブラリを含む 1.5 mL チューブにライブラリ ミックスを準備します。
注: 機械的せん断ベース ライブラリ DNA ライブラリの 40 μ L に燃料ミックス (RBF) と連続したバッファーの 35 μ L を追加します。トランスポザーゼ断片化ベース ライブラリの追加シーケンス バッファー (SQB) の 34 μ L と 16 μ L の DNA ライブラリを 25 μ l 添加するヌクレアーゼ フリー水。 - サンプル ポートを公開する優しく流れ細胞サンプル ポート カバーを開きます。P1000 ピペットを使用して、3.5 の手順で説明したようのフロー ・ セルにプライミング ポートを介してプライミング ミックスの 200 μ L を追加します。サンプル ポートを介してフロー ・ セルにプライミング ミックスが読み込まれていないことを確認します。
- P200 ピペットを 80 μ L に設定します。読み込み直前 6 回上下ピペッティングによる大口径先端穏やかライブラリを組合せ。
- フロー ・ セルに、サンプル ポートを介して滴下ライブラリ ミックスをロードします。以前のドロップがポートに完全にロードされた後にのみ、それぞれのドロップを追加します。
- サンプル ポート カバーをそっと戻さし、サンプル ポートは完全に覆われているかどうかを確認します。プライミング ポート カバーをプライミング ポートをカバーする反時計回りに移動します。デバイスを閉じた。
- 新しい実験ワークフローをクリックします。ライブラリの名前を入力、使用されるプロシージャに従って正しいキットを選択し、設定が正しい (48 h 実行、リアルタイム ベースの呼び出しに) であることを確認します。
- 実行を開始] をクリックします。10 分後、フロー セル ID 情報から実行アクティブ ナノポア数 (合計数と各 4 つのグループの番号) を記録します。
- データ分析。いつでもシーケンス実行が完了すると、ローカル コンピューターまたはクラスターにデータをコピーします。Minimap216 (https://github.com/lh3/minimap2) を使用して、参照ゲノム シーケンス データを揃えます。NanoPlot17 (https://github.com/wdecoster/NanoPlot) によって塩基配列データ線形からシーケンスのパフォーマンスをまとめたものです。
結果
超長い DNA シーケンス プロトコルは、ライブラリ構築に高分子 DNA を適用します。したがって、それはライブの比率でよく培養細胞を選択する重要です > ステップを収穫セルで 85%。DNA の抽出に使用される細胞の量は、品質と見られる DNA の量に影響します。セル換散は、あまりにも多くの細胞で始まる場合にも動作しません。あまりにもいくつかの細胞を使用しても十分な DNA ライブラリ構築のため生成しません HMW DNA の沈殿物が高速遠心分離ではなく手で緩やかな回転を使用して実行されるため。冷えた 100% エタノールを追加し、回転は図 2に白い綿のような沈殿物として表示されます後 HMW DNA の例です。
ライブラリーの構築を開始する前に入力 DNA の品質をチェックすることが重要です。劣化、不適切な数量、汚染 (例えば、蛋白質、Rna、洗剤、界面活性剤と残留フェノールまたはエタノール)、低分子量 DNA は、以後の手順と長さを読んで最終的に大きく影響を持つことができます。高分子 DNA を含むチューブの 3 つの異なる場所から DNA を使った品質管理分析を実行をお勧めします。UV HMW DNA の結果を読んでから OD260/OD280値は約 1.9 と OD260/OD230値は約 2.3 (図 3 aB)。これらの比の値は良い HMW DNA サンプルの 3 つのテスト間で一貫しています。異なるせん断方法入力 DNA の別のボリュームが必要です。高分子 DNA 濃度は、する必要があります > それはする必要があります一方、機械的剪断の 200 ng/μ L > トランスポザーゼ断片化を 1 μ g/μ L。蛍光光度計で検出された濃度は紫外線を読むより少し低いです。ただし、同じ HMW DNA サンプルの濃度の変動係数は、蛍光光度計とアッセイを読んで UV 15% 未満をする必要です。高分子 DNA を破る針通過数はせん断の DNA のサイズに影響を与えるし、最終的な長さの読み出しに針と注射器を適用する機械的剪断します。針 HMW の DNA の大部分を確保するため剪断が図 4に示すように 50 kb を超えるサイズ QC を実行することをお勧めします。機械的せん断法では、30 のパスは、距離と出力の両方を考慮し、最高の結果を配列を生成しました。
機械的せん断ベース ライブラリの N50 は 50-70 kb トランスポザーゼ断片化ベース ライブラリは 90-100 kb です。HG00733 細胞株を用いた 4 つの実行の結果は、表 1のとおりです。すべての 4 失点がある 100 kb より長い長さで 2,300 以上の読み込み。最大長は長くトランスポザーゼ断片化ベース ライブラリで (455 kb、489 kb) (348 kb、387 kb) 機械的せん断ベース ライブラリと比較して後者を生産合計読み取りより高い利回りを示します。トランスポザーゼ断片化ベース ライブラリ構築、少数のステップおよび短い準備時間少ない短い断片を紹介します。トランスポザーゼを使用して 2 つの実行がより長い平均長さを持っている (> 30 kb) と中央の長さ (> 10 kb)。さらに、データは (平均品質スコアは基本精度約 10.0、~ 90%) のすべての実行で一貫した高品質を示しています。総基地の 97% 以上がデフォルトの設定で Minimap216を使用して人間の参照ゲノム (hg19) に配置されます。Raw 読み取りの予想サイズ分布は、図 5のとおりです。すべての実行トランスポザーゼ断片化ベースのライブラリが超長い読み取り (例えば > 100 kb) の高い比率を持っている 50 kb 以上のデータの大きい割合があります。このプロトコルは、複数のひと細胞株 (補足表 1) に正常に適用されています。
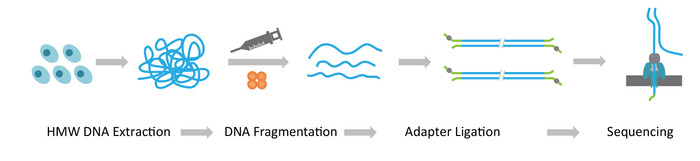
図 1: ナノポア長いリード シーケンス (seq NLR) ワークフローの図式的な概観します。オレンジ、複雑な転移。黄緑色、ナノポア アダプター。この図の拡大版を表示するのにはここをクリックしてください。

図 2: フェノール-クロロホルム抽出法から代表的な DNA の沈殿物です。白い矢印は、高分子 DNA を示します。この図の拡大版を表示するのにはここをクリックしてください。

図 3: UV 読書から見られる DNA の例 QC 結果。(A) HMW DNA ステップ 1.21.1 機械的剪断に基づく図書館の建設の準備ができてから。(B) HMW DNA 1.21.2 トランスポザーゼ断片化ベースのライブラリ構築のためのステップから。この図の拡大版を表示するのにはここをクリックしてください。

図 4: 針の品質管理結果が脈打フィールドゲルの電気泳動による高分子 DNA をせん断します。L1: クイック ロード 1 kb の DNA の梯子;L2: クイック ロード 1 kb 拡張 DNA の梯子。1-8: 針シャーリングにより異なる通過タイムが付いている DNA。1-3、せん断;4、10 回5、20 回6、30 回7、40 倍8、50 回。この QC 手順はオプションです。この図の拡大版を表示するのにはここをクリックしてください。

図 5: ナノポア超長い DNA ライブラリのサイズ分布を予想します。MS、機械的せん断ベース ライブラリ。TF、トランスポザーゼ断片化ベース ライブラリ。この図の拡大版を表示するのにはここをクリックしてください。
| 機械 shearing_rep1 | 機械 shearing_rep2 | トランスポザーゼ fragmentation_rep1 | トランスポザーゼ fragmentation_rep2 | |
| セルライン | HG00733 | HG00733 | HG00733 | HG00733 |
| 読み取りの N50 | 55,180 | 63,007 | 98,237 | 95,629 |
| 100 Kb を超える読み取りの数 | 2,500 | 3,082 | 2,386 | 2,355 |
| 合計読み取りの数 | 97,859 | 80,465 | 24,166 | 21,032 |
| 最大長さ (bp) | 348,482 | 387,113 | 454,660 | 489,426 |
| 平均長さ (bp) | 17,861 | 20,395 | 33,528 | 38,175 |
| 中央の長さ (bp) | 5,335 | 5,894 | 10,249 | 15,656 |
| 読み取りの平均品質 | 10.0 | 10.1 | 9.9 | 10.0 |
| Raw 読み取りの総基盤 | 1,747,849,822 | 1,641,058,932 | 810,229,733 | 802,886,304 |
| 一直線に並べられた読み取りの総基盤 | 1,693,300,832 | 1,607,975,925 | 791,422,077 | 778,417,627 |
| マップされた比総基盤 (hg19、Minimap2) | 96.9% | 98.0% | 97.7% | 97.0% |
| アクティブな毛穴の数 | 1225:480, 402, 254, 89 | 1058:480, 356, 176, 46 | 958:452, 328 148, 30 | 1092:487 367, 195, 43 |
表 1: パフォーマンス メトリックの概要は、せん断の異なるプロトコルで実行されます。
| ライブラリ 1 | ライブラリ 2 | |
| セルライン | K562 | GM19240 |
| 発注情報を携帯します。 | ATCC、猫。違います。CCL-243 | コーリエル医学研究所は、猫。違います。GM19240 |
| プロトコル | 機械的剪断 | 機械的剪断 |
| 読み取りの N50 | 60,063 | 55,295 |
| 合計読み取りの数 | 193,783 | 120,807 |
| 中央の長さ (bp) | 1,843 | 4,688 |
| 平均長さ (bp) | 9,825 | 17,408 |
| 最大長さ (bp) | 548,780 | 212,338 |
| Raw 読み取りの総基盤 | 1,903,989,686 | 2,103,015,331 |
| 一直線に並べられた読み取りの総基盤 | 1,837,350,047 | 1,997,419,761 |
| マップされた比総基盤 (hg19、Minimap2) | 96.6% | 95.0% |
| アクティブな毛穴の数 | 1111:482, 371, 203, 55 | 1032:447, 333, 196, 56 |
補足の表 1: 機械的せん断プロトコルと他のセルラインを使用して 2 つの NLR seq 実行の概要。
ディスカッション
原則として、ナノ細孔配列は長さ11,12,13megabase の読み取りに 100 kb を生成すること。4 つの主要な要因はシーケンス処理の実行とデータの品質のパフォーマンスに影響します: 1) アクティブな孔数と; 毛穴の活動2) モーター蛋白質; ナノポアを通過する DNA の速度を制御します。3) DNA のテンプレート (長さ、純度、品質、質量);4) シーケンス アダプター結紮効率は、入力サンプルから使用可能な DNA を決定します。最初の 2 つの要因は、フローセルと、製造元によって提供されるシーケンス キットのバージョンによって異なります。次の 2 つの要因は、このプロトコル (HMW DNA の抽出、せん断および結紮術) で重要な手順です。
このプロトコルには、忍耐と練習が必要です。高分子 DNA の品質は、超長い DNA ライブラリ6にとって重要です。プロトコルは高い生存率と細胞から始まります (> 85% 実行可能なセル優先)、死んだ細胞から劣化 DNA を制限します。避けるべきである (例えば、強い不穏な揺れ、渦、複数ピペッティング、繰り返し凍結、融解) DNA に損傷を引き起こす可能性があります任意の過酷なプロセス。プロトコルの設計、我々 は DNA の抽出のプロセス全体でピペッティングを省略します。大口径のヒントは、ピペッティングが図書館の建設中に機械的剪断とシーケンスの後必要なときに使用する必要があります。孔は商工会議所バッファー12化学物質に敏感な必要がありますいくつかの残留汚染物質 (例えば、洗剤、界面活性剤、フェノール、エタノール、蛋白質 Rna 等) 可能な DNA の。長さと収量を考慮するフェノール抽出法は、これまでテスト複数の異なる抽出法と比較して最高と最も再現性のある結果を示しています。
長い読み取りシーケンスを生成するこのプロトコルの能力、にもかかわらず、いくつかの制限は残っています。まず、このプロトコルに最適化されたパブリケーションの時に利用できるナノ細孔シーケンシング デバイスに基づいてしたがって、ナノポア ベースのシーケンスの選択的な化学に制限され、長いリード シーケンス デバイスの他の種類で実行すると最適ではない可能性があります。第二に、結果は (組織または細胞) の原料から抽出した DNA の品質に大きく依存です。開始の DNA は既に劣化または破損した場合の長さの読み取りが低下します。第三に、QC の複数のステップは DNA の品質をチェックするプロトコルに組み込まれているが最終的な収量と読み取りの長さ、フローセルを受けます、間隙ナノ細孔シーケンシング プラットフォームのこの初期段階で変数となる活動開発。
説明されたプロトコルはここで DNA の抽出のため人間サスペンションのセル行のサンプルを使用します。針シャーリング、トランスポザーゼと結紮時間記述結果を見られる DNA の比率で通過時間を最適化します。プロトコルは、4 つの方法で拡張できます。まず、ユーザーは、他の哺乳類細胞と細胞、組織、臨床サンプル、または他の生物とは異なる金額を開始できます。遠心分離や反応量溶解インキュベーション時間をさらに最適化が必要となります。第二に、超長い読み込みシーケンスのターゲット サイズを予測するは難しいです。読み取りの長さが予想よりも短く、ユーザーは機械的剪断に基づく方法で通過時刻を調整したり、トランスポザーゼ トランスポザーゼの断片化法で見られる DNA の比率を変更できます。クリーンアップ手順を実行時にバインドと溶出時間は、高分子 DNA は粘性の高いので便利です。第三に、異なるナノ細孔シーケンシング デバイスと 1 つは、量と DNA シーケンサーの基準を満たすためのボリュームを調整できます。第四に、シーケンス アダプターに結紮 DNA のみをシーケンスに配置されます。結紮術の効率のさらに 1 つはアダプターとリガーゼ濃度を滴定しなさいを試行できます。変更された結紮時間とペグ18など分子混み合いエージェントは、将来的に適用できます。CRISPR19,20と組み合わせて超長い DNA シーケンス プロトコル可能性がありますターゲット濃縮シーケンスのための効果的なツールを提供しています。
開示事項
著者は、彼らは競合する金銭的な利益があることを宣言します。
謝辞
著者は Y. 朱の原稿で彼女のコメントをありがちましょう。この出版物で報告された研究は、賞を受賞番号 P30CA034196 の下で健康の国民の協会の国立癌研究所で部分的に支持されました。内容は著者の責任と国立衛生研究所の公式見解を必ずしも表さない。
資料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Absolute ethanol | Sigma-Aldrich | E7023 | |
| Agencourt AMPure XPbeads | Beckman | A63881 | magnetic beads for cleanup |
| BD conventional needles | Becton Dickinson | 305136 | 27G, for mechanical shearing |
| BD Luer-Lok syringe | Becton Dickinson | 309628 | for mechanical shearing |
| Blunt/TA Ligase Master Mix | NEB | M0367S | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | for cell counting |
| EDTA | Invitrogen | AM9261 | pH 8.0, 0.5 M, 500 mL |
| Flow Cell | Oxford Nanopore Technologies | FLO-MIN106 | R9.4.1 |
| HG00773 cells | Coriell Institute | HG00733 | cells used in this protocol |
| Ligation Sequencing Kit 1D | Oxford Nanopore Technologies | SQK-LSK108 | nanopore ligation kit |
| MaXtract High Density tubes | Qiagen | 129073 | gel tubes |
| NEBNext FFPE DNA Repair Mix | NEB | M6630S | |
| NEBNext Ultra II End Repair/dA-Tailing Module | NEB | M7546S | |
| Nuclease-free water | Invitrogen | AM9937 | |
| Phosphate-Buffered Saline, PBS | Gibco | 70011044 | 10X, pH 7.4 |
| Phenol:chloroform:IAA | Invitrogen | AM9730 | |
| Proteinase K | Qiagen | 19131 | 20 mg/mL |
| Qubit dsDNA BR Assay Kit | Invitrogen | Q32850 | fluorometer assays for DNA quantification |
| Rapid Sequencing Kit | Oxford Nanopore Technologies | SQK-RAD004 | nanopore transposase kit |
| RNase A | Qiagen | 19101 | 100 mg/mL |
| SDS | Invitrogen | AM9822 | 10% (wt/vol) |
| Sodium chloride solution | Invitrogen | AM9759 | 5.0 M |
| TE buffer | Invitrogen | AM9849 | pH 8.0 |
| Tris | Invitrogen | AM9856 | pH 8.0, 1 M |
| Triton X-100 solution | Sigma-Aldrich | 93443 | ~10% |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Bio-Rad C1000 Thermal Cycler | Bio-Rad | 1851196EDU | |
| Centrifuge 5810R | Eppendorf | 22628180 | |
| Countess II FL Automated Cell Counter | Life Technologies | AMQAF1000 | for cell counting |
| DynaMag-2 Magnet | Life Technologies | 12321D | magnetic rack |
| Eppendorf ThermoMixer | Eppendorf | 5382000023 | for incubation |
| Freezer | LabRepCo | LHP-5-UFMB | |
| GridION | Oxford Nanopore Technologies | GridION X5 | nanopore device used in this protocol |
| HulaMixer Sample Mixer | Thermo Fisher Scientific | 15920D | rotator mixer |
| MicroCentrifuge | Benchmark Scientific | C1012 | |
| NanoDrop ND-1000 Spectrophotometer | Thermo Fisher Scientific | ND-1000 | for UV reading |
| Pippin Pulse | Sage Science | PPI0200 | pulsed-field gel electrophoresis instrument |
| Qubit 3.0 Fluorometer | Invitrogen | Q33216 | fluorometer |
| Refrigerator | LabRepCo | LABHP-5-URBSS | |
| Vortex-Genie 2 | Scientific Industries | SI-A236 | |
| Water bath | VWR | 89501-464 |
参考文献
- Mardis, E. R. Next-generation sequencing platforms. Annual Review of Analytical Chemistry. 6, 287-303 (2013).
- Goodwin, S., McPherson, J. D., McCombie, W. R. Coming of age: ten years of next-generation sequencing technologies. Nature Reviews Genetics. 17 (6), 333-351 (2016).
- Shendure, J., et al. DNA sequencing at 40: past, present and future. Nature. 550 (7676), 345-353 (2017).
- Alkan, C., Coe, B. P., Eichler, E. E. Genome structural variation discovery and genotyping. Nature Reviews Genetics. 12 (5), 363-376 (2011).
- Weischenfeldt, J., Symmons, O., Spitz, F., Korbel, J. O. Phenotypic impact of genomic structural variation: insights from and for human disease. Nature Reviews Genetics. 14 (2), 125-138 (2013).
- Pollard, M. O., Gurdasani, D., Mentzer, A. J., Porter, T., Sandhu, M. S. Long reads: their purpose and place. Human Molecular Genetics. 27 (R2), R234-R241 (2018).
- Cretu Stancu, M., et al. Mapping and phasing of structural variation in patient genomes using nanopore sequencing. Nature Communications. 8 (1), 1326 (2017).
- Gong, L., et al. Picky comprehensively detects high-resolution structural variants in nanopore long reads. Nature Methods. 15 (6), 455-460 (2018).
- Sedlazeck, F. J., et al. Accurate detection of complex structural variations using single-molecule sequencing. Nature Methods. 15 (6), 461-468 (2018).
- Jain, M., et al. Nanopore sequencing and assembly of a human genome with ultra-long reads. Nature Biotechnology. 36 (4), 338-345 (2018).
- Jain, M., et al. Improved data analysis for the MinION nanopore sequencer. Nature Methods. 12 (4), 351-356 (2015).
- Deamer, D., Akeson, M., Branton, D. Three decades of nanopore sequencing. Nature Biotechnology. 34 (5), 518-524 (2016).
- Jain, M., Olsen, H. E., Paten, B., Akeson, M. The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community. Genome Biology. 17 (1), 239 (2016).
- Editorial, The long view on sequencing. Nature Biotechnology. 36 (4), 287 (2018).
- Jain, M., et al. Linear assembly of a human centromere on the Y chromosome. Nature Biotechnology. 36 (4), 321-323 (2018).
- Li, H. Minimap2: pairwise alignment for nucleotide sequences. Bioinformatics. 34, 3094-3100 (2018).
- De Coster, W., D'Hert, S., Schultz, D. T., Cruts, M., Van Broeckhoven, C. NanoPack: visualizing and processing long-read sequencing data. Bioinformatics. 34, 2666-2669 (2018).
- Akabayov, B., Akabayov, S. R., Lee, S. J., Wagner, G., Richardson, C. C. Impact of macromolecular crowding on DNA replication. Nature Communications. 4, 1615 (2013).
- Gabrieli, T., Sharim, H., Michaeli, Y., Ebenstein, Y. Cas9-Assisted Targeting of CHromosome segments (CATCH) for targeted nanopore sequencing and optical genome mapping. bioRxiv. , (2017).
- Gabrieli, T., et al. Selective nanopore sequencing of human BRCA1 by Cas9-assisted targeting of chromosome segments (CATCH). Nucleic Acids Research. , (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved