Method Article
רצף קריאה ארוכים במיוחד עבור ניתוח הדנ א הגנומי לגמרי
In This Article
Summary
רצפי זמן-קריאה מאוד להקל על ההרכבה של הגנום מורכבים ואפיון של וריאציה מבנית. אנו מתארים שיטה כדי ליצור רצפים ארוכים במיוחד על ידי רצף nanopore מבוססי פלטפורמות. הגישה מאמצת הוצאה DNA ממוטבת ואחריו ההכנות ספריית ששונה כדי להפיק מאות קריאות kilobase עם כיסוי מתון של תאים אנושיים.
Abstract
דור שלישי יחיד מולקולה DNA רצף טכנולוגיות מציעים באופן משמעותי יותר לקרוא אורך זה יכול להקל על ההרכבה של הגנום מורכבים וניתוח של משתנים מבניים מורכבים. Nanopore לבצע רצף מולקולה בודדת על-ידי ישירות מדידת השינויים הנוכחיים מתווכת על-ידי ה-DNA מעבר דרך הנקבוביות ופלטפורמות יכול להפיק מאות קריאות kilobase (kb) במינימום עלות ההון. פלטפורמה זו אומץ על ידי חוקרים רבים למגוון רחב של יישומים. השגת עוד רצף אורכי קריאה היא הגורם הקריטי ביותר למנף את הערך של nanopore רצף פלטפורמות. כדי להפיק קריאות ארוכים במיוחד, שיקולים מיוחדים נדרשת כדי למנוע ה-DNA לנזקי השבירה ולהשיג יעילות כדי ליצור רצף פרודוקטיבי תבניות. כאן, אנו מספקים את פרוטוקול מפורט של רצפי DNA ארוכים במיוחד כולל משקל מולקולרי גבוה (HMW) ה-DNA מיצוי מתאי טרי או קפוא, ספריית בנייה על ידי הטיית מכני או פיצול transposase, וסדר במכשיר nanopore. µG 20-25 של ה-DNA HMW, השיטה ניתן להשיג N50 לקרוא באורך של 50-70 kb עם הטיית מכני, N50 של 90-100 kb לקרוא אורך עם transposase מתווכת פיצול. הפרוטוקול ניתן ליישם DNA מופק בתרבית של תאים כדי לבצע רצף הגנום כולו איתור משתנים מבניים והרכבה הגנום. שיפורים נוספים על מיצוי הדנ א ואת תגובות אנזימטיות נוספת להאריך את אורך הקריאה, להרחיב את השירות שלה.
Introduction
בעשור האחרון, בנפט מקבילי ומונהג טכנולוגיות ומדויקים רצף תפוקה גבוהה הדור השני יש פיצוץ של גילוי הביו-רפואית של חדשנות טכנולוגית1,2,3. למרות ההתקדמות הטכנית, את הנתונים קצר-קריאה שנוצר על ידי הדור השני פלטפורמות אינם יעילים בפתרון מורכב אזורים גנומית ואת מוגבלים זיהוי של משתנים מבניים גנומית (SVs), ממלאים תפקיד חשוב-אנוש האבולוציה ומחלות4,5. יתר על כן, קצר-קריאת נתונים אינן מצליחות לפתור וריאציה. אני חוזר, הזדהמו אניני טעם haplotype בהדרגה של וריאציות גנטיות6.
התקדמות אחרונים מולקולה בודדת רצף מציע באופן משמעותי יותר לקרוא את אורך, דבר היכול להקל על הזיהוי את מלוא הספקטרום של SVs7,8,9, והרכבה הצעות מלאים ומדויקים של המתחם הגנום של חיידקים, בתרבית של6,10. פלטפורמת nanopore מבצעת רצף מולקולה בודדת על-ידי ישירות מדידת השינויים הנוכחיים מתווכת על-ידי ה-DNA מעבר דרך הנקבוביות11,12,13. בניגוד ביננו כימיה רצפי DNA הקיים, nanopore הרצף ניתן להפיק קריאות רב (עשרות אלפי kilobases) בזמן אמת בלי להסתמך על פולימראז קינטיקה או מלאכותי הגברה דגימת דנ א. לכן, nanopore לונג-קריאה רצף (NLR-seq) מביאות הבטחה גדולה ליצירת באורכים ארוכים במיוחד לקרוא הרבה מעבר 100 kb, אשר במידה רבה מראש ניתוח גנומי וביו14, בפרט המורכבות נמוך או חוזר-עשיר אזורים של הגנום15.
התכונה הייחודית של רצף nanopore הוא הפוטנציאל ליצירת זמן קורא ללא מגבלה תאורטית אורך. לכן, האורך קריאה הוא תלוי אורך הפיזית של ה-DNA אשר מושפע ישירות איכות תבנית ה-DNA תקינות ורצף. יתר על כן, בהתאם להיקף של מספר הצעדים מעורב, כגון pipetting כוחות ומצבים החילוץ מניפולציה, האיכות של ה-DNA זה משתנה מאוד. לכן, זה מאתגר עבור אחד להניב קריאות ארוך על-ידי החלת רק הפרוטוקולים מיצוי DNA סטנדרטיים ושיטות הבנייה של היצרן ספריית שסופקו. לצורך כך, פיתחנו חזקים של שיטה להפקת ארוכים במיוחד לקרוא (מאות kilobases) רצף נתונים החל מתא שנקטפו כדורי. אימצנו מספר שיפורים בהליכים DNA החילוץ וספריית הכנה. אנחנו אווירודינמית פרוטוקול להוציא נהלים מיותרות הגורמות DNA השפלה ונזקים. פרוטוקול זה מורכב משקל מולקולרי גבוה (HMW) הפקת דנ א, בנייה ספריית הדי ארוכים במיוחד, רצף על פלטפורמה nanopore. עבור ביולוג מולקולרי מאומנים היטב, זה בדרך כלל לוקח 6 שעות מתא הקטיף ועד לסיום של HMW DNA החילוץ, 90 דקות או 8 שעות לבנייה ספריה בהתאם לשיטת ההטיה, ועד של 48 שעות נוספות עבור רצפי DNA. השימוש בפרוטוקול אטיל הקהילה גנומיקה לשפר את ההבנה שלנו של הגנום המורכבות ולהשיג תובנה חדשה הגנום וריאציה של מחלות אנושיות.
Protocol
הערה: פרוטוקול NLR-seq מורכב משלושה שלבים עוקבים: 1) הפקת גבוהה-מולקולרית משקל DNA גנומי (HMW); 2) ארוכים במיוחד בניה ספריית דנ א, הכוללת פרגמנטציה של ה-DNA HMW לתוך הגדלים הרצויים, מצדו של מתאמי רצף ה-DNA מסתיימת; ו 3) טעינת ה-dna ובין אם-לא מתאם אל מערכי nanopores (איור 1).
1. HMW DNA החילוץ
- ריאגנט כיוונון. להפוך 1 x פוספט תמיסת מלח במאגר (PBS) מאגר (1,000 מ ל) על-ידי הוספת 100 מ של PBS (10 x) עד 900 מ"ל של מים, לערבב היטב. לבצע פירוק מאגר (50 מ"ל) על-ידי הוספת 43.5 מ ל מים צינור 50 מ. להוסיף 500 μL של טריס (1 מ', pH 8.0), 1 מ"ל של נתרן כלורי (NaCl) (5 מ'), 2.5 מ ל חומצה ethylenediaminetetraacetic (EDTA) (0.5 M, pH 8.0), 2.5 מ של dodecyl נתרן גופרתי (מרחביות) (10%, wt/כרך) אל הצינור ומערבבים היטב.
הערה: מאגר PBS שניתן לאחסן ב 4 ° C עד 6 חודשים. ניתן לאחסן את המאגר פירוק premade ב RT עד 2 חודשים. - בדוק את התמותה תא ולספור את התאים. ודא היחס לחיות הוא > 85% מספר התאים הכולל הוא 30 x 106.
הערה: התאים המשמשים פרוטוקול זה הם משורת תא HG00733, קו תא lymphoblastoid אדם ממוצא פורטוריקני בשימוש נרחב האיחוד הגנום 1000 לניתוח וריאציה מבניים (ראה טבלה של חומרים עבור הרכישה), אשר שייך למשאב מדגם הגנום בינלאומיים. - לאסוף את התאים על ידי צריך שתוציאו ב x 200 גרם במשך 5 דקות ב RT. להשליך המדיום, resuspend בגדר תא (30 x 106 תאים) עם 5 מ ל 1 x PBS המאגר. צנטריפוגה שוב ב x 200 גרם במשך 5 דקות ב RT וזורקים את תגובת שיקוע.
הערה: 25-35 x 106 תאים מקובלים על גישה זו. עוד וריאציה של תאים בשימוש יהיה עליך עוד יותר אופטימיזציה. ניתן לאחסן בגדר תא-−80 ° C עד 6 חודשים. - Resuspend בגדר תא בμl 200 1 x PBS המאגר. אם משתמש גלולה תא קפוא, לשטוף עם 5 מ ל 1 x PBS המאגר. Centrifuge הפתרון ב x 200 גרם במשך 5 דקות ב RT, למחוק את תגובת שיקוע, resuspend תאי μL 200 1 x מאגר PBS.
- הכינו 10 מ"ל פירוק המאגר בצינור 50 מ. להוסיף התליה תא 200 μL פירוק מאגר ו מערבולת במהירות הגבוהה ביותר עבור Incubate ס' 3 הפתרון ב 37 ° C עבור 1 h.
- להוסיף 2 μL של RNase A (100 מ"ג/מ"ל) lysate. סובב בעדינות את הצינור 50 מ לערבב את הדגימה. דגירה הפתרון ב 37 ° C עבור 1 h.
- להוסיף μL 50 proteinase K (20 מ"ג/מ"ל) lysate. סובב בעדינות את הצינור 50 מ לערבב את הדגימה. דגירה הפתרון ב 50 מעלות צלזיוס במשך שעתיים. במהלך הדגירה, בעדינות לערבב המדגם בכל 30 דקות.
- הסר את הצינור 50 מ של 50 ° C ומשהים ב RT 5 דקות.
- להוסיף 10 מ של השכבה פנול של אלכוהול פנול: כלורופורם: isoamyl (25:24:1, כרך/כרך/כרך) lysate, לסובב את הצינורית על מערבל מסובב (ראה טבלה של חומרים) ב RT בשכונה fume ב rpm 20 במשך 10 דקות לעטוף את הצינורית עם מצלמות-מיקרוסקופים כדי למנוע דליפה במהלך הסיבוב.
- להכין שני צינורות 50 מ ל ג'ל (ראה טבלה של חומרים) על-ידי צריך שתוציאו ב x 1,500 g למשך 2 דקות ב- RT.
הערה: הג'ל יוצר מחסום יציב בין השלב מימית המכילה חומצות גרעין הממס האורגני. - שופכים הפתרון מדגם/פנול לתוך אחד מוכן 50 מ ל ג'ל צינורות. שלב 1.10. Centrifuge הפתרון ב x 3000 g 10 דקות ב- RT.
- שופכים את תגובת שיקוע לתוך צינור 50 מ. להוסיף 10 מ של השכבה פנול של אלכוהול פנול: כלורופורם: isoamyl (25:24:1, כרך/כרך/כרך) ולסובב את הצינורית על מערבל מסובב ב RT בשכונה fume ב 20 rpm למשך 10 דקות.
- חזור על שלב 1.11 פעם אחת עם השנייה מוכן שפופרת ג'ל.
- שופכים את תגובת שיקוע לתוך צינור 50 מ. להוסיף 25 מ של אתנול 100% קר כקרח, סובב בעדינות את הצינורית ביד עד ה-DNA ארגונייט שוקע (איור 2).
הערה: הגישה משקעים מסייע לייצב את הדנ א HMW. - כיפוף טיפ 20 μL כדי להפוך קרס. בזהירות להוציא את ה-DNA HMW עם הקרס, שחררו את המסירה נוזלי.
- המקום ה-DNA HMW לתוך צינור 50 מ ל המכיל 40 מ"ל אתנול 70%. לשטוף את ה-DNA על-ידי היפוך בעדינות את הצינור 3 פעמים.
- חזור על שלב 1.15 פעם לאסוף DNA מהצינור 70% אתנול.
- המקום ה-DNA HMW לתוך צינור 2 מ"ל המכיל מ 1.8 ל-70% אתנול.
- Centrifuge ה-DNA HMW שטף ב x 10,000 g 3 s ב RT. הסר כמות האתנול שיורית ככל האפשר על-ידי pipetting.
הערה: נא לא להפריע בגדר DNA כאשר pipetting האתנול שיורית. - דגירה הצינור 2 מ"ל ב 37 מעלות צלזיוס למשך 10 דקות עם מכסה פתוח כדי לייבש את הדגימה.
- אם ממשיכים עם 2.1 (עם הטיית מכני ו 1 ערכת עריכה ברצף של מצדו D), להוסיף 1 מ"ל של TE (10 מ מ EDTA טריס ו- 1 מ מ, pH 8.0) הצינור 2 מ"ל.
- אם ממשיכים עם 2.2 שלב (עם רסס מבוססי transposase, ערכת רצף מהיר), הוסף 200 μL 10 מ מ טריס (pH 8.0) 0.02% טריטון X-100.
הערה: נא לא להפריע בגדר ה-DNA. נותן את הצינור עמוד 4 ° C בחושך במשך 48 שעות יעזור המדגם באופן מלא resuspend. ניתן לאחסן את הדנ א HMW ב 4 ° C עד 2 שבועות. זמן אחסון רב יותר או תנאים אחסון אחרים עשויים להציג קטעים קצרים יותר.
2. ארוכים במיוחד הבנייה ספריית דנ א
הערה: יש שתי דרכים כדי לבנות את ספריות DNA ארוכים במיוחד מבוסס על שתי שיטות ההטיה שונות בשילוב עם ערכות רצף nanopore. ספריה מבוסס על חיתוך מכני מפיק נתונים עם N50 של 50-70 kb, לוקח בערך 8 שעות להקמת ספריה. ספרייה המבוססת על פיצול transposase מייצרת של N50 של 90-100 kb נתונים, לוקח רק 90 דקות להקמת ספריה. פרוטוקול חיתוך מכני נותן תשואה גבוהה יותר מ דנ א קלט זהה בגירסאות של רצף מתאם ואיכות של תאים זרימה nanopore.
- בניית ספריה מבוסס על חיתוך מכני
- הפשרה, תערובת ריאגנטים מ מצדו קיט (ראה טבלה של חומרים). הפשרה FFPE DNA תיקון מאגר, סוף מאגר תיקון/דה-עוקב על קרח, ואז המערבולת ו ספין למטה כדי לערבב. הפשרת מתאם שילוב (AMX) ומאגר מתאם חרוז מחייב (ABB) על קרח, ואז פיפטה ו ספין למטה כדי לערבב. הפשרה פועל מאגר עם תערובת דלק (RBF) ו • תנאי מאגר (ELB)-RT, ואז מערבולת ספין כדי לערבב. הפשרת ספריית טעינת חרוזים (LLB) RT, פיפטה לערבב לפני השימוש.
- ברגע הקרת, לשמור על כל הרכיבים קיט על קרח. להוציא את אנזימי רק בעת הצורך. להביא את החרוזים מגנטי RT לשימוש.
הערה: לקבלת המלצות על החרוזים מגנטיים לשימוש ראה את הטבלה של חומרים.
- ברגע הקרת, לשמור על כל הרכיבים קיט על קרח. להוציא את אנזימי רק בעת הצורך. להביא את החרוזים מגנטי RT לשימוש.
- בדוק את האיכות והכמות של ה-DNA HMW מהשלב 1.21.1. פיפטה החוצה 20 μL של ה-DNA לתוך צינורות חדשים 1.5 mL ממקומות שונים שלושה בצינור HMW DNA באמצעות P200 רחב נשא טיפים. קח μL 1 מ aliquots שלושה כדי לזהות את הריכוז באמצעות fluorometer ואת איכות באמצעות אולטרה קריאה. בדוק מספר פעמים כדי לאשר את התוצאות.
הערה: התוצאות הצפויות מוצגים באיור 3 א. הערך260/280 OD הוא כ 1.9 והוא הערך260/230 OD כ 2.3. - העברת μL 940 הנותרים של ה-DNA HMW לתוך כובע שפופרת 50 מ עם P1000 רחב נשא עצה.
- תשאף כל DNA לתוך מזרק 1 מ"ל ללא המחט.
- לשים את המחט 27 G על גבי המזרק והוצא כל DNA לתוך הכיפה ובאיטיות (~ 10 s). . תוריד את המחט 27 G מן המזרק
- חזור על הצעדים 2.1.4 ו 2.1.5 29 פעמים עבור סכום כולל של 30 עובר דרך המחט.
הערה: ה-DNA הוטו HMW ניתן לאחסן ב 4 ° C בחושך עד ה 24 בקרת איכות (QC) מומלץ בחום על ידי אלקטרופורזה בג'ל פעמו-שדה, אבל זה יקר ומבזבז זמן. אם מבצע QC במחשב הדופק אוטומטית שדה ג'ל אלקטרופורזה להשתמש בפרוטוקול 5-150 kb עבור ה 20 לרוץ. התוצאות הצפויות מוצגים באיור4. - הכנת התגובה תיקון ה-DNA בשפופרת 0.2 מ"ל על-ידי הוספת μL 100 של DNA HMW הוטו (20 μg), μL 15 FFPE DNA תיקון מאגר μL 12 של מיקס תיקון ה-DNA FFPE, 16 μL של נוקלאז ללא מים. לערבב את התגובה על ידי מצליף בעדינות 6 פעמים, ספין למטה כדי להסיר בועות.
- דגירה התגובה ב 20 מעלות צלזיוס במשך 60 דק העברה המדגם לתוך צינור 1.5 mL החדש עם טיפ P200 נשא רחב.
- Resuspend את החרוזים מגנטי pipetting או vortexing. הוסף μL 143 חרוזים (1 x) התגובה תיקון ה-DNA, לערבב בעדינות על ידי מצליף הצינור 6 פעמים. לסובב את הצינורית על מערבל מסובב ב RT-סל ד 20 למשך 30 דקות.
- בדוגמה ב x 1000 g 2 s במקום RT. הצינורית על מתלה מגנטי במשך 10 דקות לשמור את הצינורית על מתלה מגנטי ולמחוק את תגובת שיקוע.
- שמירה על הצינור על מתלה מגנטי, להוסיף μL 400 של אתנול 70% שזה עתה הוכנו מבלי להפריע בגדר. להסיר את אתנול 70% לאחר 30 s.
- חזור על שלב 2.1.11 פעם אחת.
- בדוגמה ב x 1000 g 2 s במקום RT. הצינור בחזרה על המדף מגנטי. להסיר כל אתנול שיורית, אוויר יבש ב-30 s. לא מעל יבשה בגדר.
- להסיר את הצינור מהמדף מגנטי ולהוסיף μL 103 של TE (10 מ מ EDTA טריס ו- 1 מ מ, pH 8.0). בעדינות פליק צינור כדי להבטיח כי חרוזים מכוסים המאגר, דגירה על מערבל מסובב ב RT במשך 30 דקות בעדינות קפיצי הצינור כל 5 דקות כדי לסייע resuspension של בגדר.
- גלולה החרוזים על מתלה מגנטי במשך לפחות 10 דקות להעביר 100 μL של eluate עם P200 רחב נשא עצה לתוך צינור 0.2 מ"ל.
- הכנת התגובה דה-עוקב בשפופרת 0.2 מ"ל ותיקון קצה על-ידי הוספת μL 100 של לתיקון DNA HMW, 14 μL סוף תיקון/דה-עוקב מאגר, 7 μL של סוף תיקון/דה-עוקב מיקס. לערבב את התגובה על ידי מצליף בעדינות 6 פעמים, ספין למטה כדי להסיר בועות.
- דגירה התגובה ב 20 מעלות צלזיוס למשך 60 דקות ולאחריו 65 מעלות צלזיוס למשך 20 דקות ולאחר מכן החזק-22 מעלות צלזיוס. העברת הדגימה לתוך צינור 1.5 mL החדש באמצעות P200 רחב נשא עצה.
- Resuspend את החרוזים מגנטי pipetting או vortexing. להוסיף μL 48 של חרוזים (0.4 x) סוף תיקון/דה-עוקב התגובה, לערבב בעדינות על ידי מצליף הצינור 6 פעמים. לסובב את הצינורית על מערבל מסובב ב RT-סל ד 20 למשך 30 דקות.
- חזור על צעדים 2.1.10-2.1.13 פעם.
- להסיר את הצינור מהמדף מגנטי ולהוסיף μL 33 של TE (10 מ מ EDTA טריס ו- 1 מ מ, pH 8.0). בעדינות פליק צינור כדי להבטיח כי חרוזים מכוסים המאגר, דגירה על מערבל מסובב ב RT במשך 30 דקות בעדינות קפיצי הצינור כל 5 דקות כדי לסייע resuspension של בגדר.
- גלולה החרוזים על מתלה מגנטי במשך לפחות 10 דקות להעביר 30 μL של eluate עם P200 רחב נשא עצה לתוך צינור 1.5 mL החדש. . קח את μL עוד 1-2 כדי לזהות את הריכוז באמצעות fluorometer
הערה: שחזור של 5-6 μg בשלב זה צפוי. - להכין את התגובה מצדו בצינור מדגם 1.5 mL על-ידי הוספת μL 30 של סוף לתיקון DNA HMW, μL 20 של מתאם שילוב (AMX 1 י), μL 50 של בלאנט/ת א מצדו מיקס מאסטר. מערבבים את התגובה על ידי מצליף בעדינות 6 פעמים בין כל תוספת רציפים ו ספין למטה כדי להסיר בועות.
- דגירה התגובה ב RT עבור 60 דקות.
- Resuspend את החרוזים מגנטי pipetting או vortexing. להוסיף 40 חרוזים μL (0.4 x) מצדו תגובה, לערבב בעדינות על ידי מצליף הצינור 6 פעמים. לסובב את הצינורית על מערבל מסובב ב RT-סל ד 20 למשך 30 דקות.
- חזור על שלב 2.1.10 פעם אחת.
- להוסיף 400 μL מתאם חרוז מחייב (ABB) מאגר לתוך הצינור. קפיצי הצינור בעדינות 6 פעמים כדי resuspend את החרוזים. מקם את הצינור בחזרה על המדף מגנטי כדי להפריד את החרוזים מתוך המאגר ולמחוק תגובת שיקוע.
- חזור על שלב 2.1.26 פעם.
- בדוגמה ב x 1000 g 2 s במקום RT. הצינור בחזרה על המדף מגנטי. להסיר את כל מאגר שיורית, אוויר יבש ב-30 s. לא מעל יבשה בגדר.
- הסר את הצינור מהמדף מגנטי, resuspend בגדר ב μL 43 • תנאי המאגר. בעדינות פליק צינור כדי להבטיח חרוזים מכוסים המאגר דגירה על מערבל מסובב ב RT במשך 30 דקות בעדינות קפיצי הצינור כל 5 דקות כדי לסייע resuspension של בגדר.
- גלולה החרוזים על מתלה מגנטי במשך לפחות 10 דקות להעביר 40 μL של eluate עם P200 רחב נשא עצה לתוך צינור 1.5 mL החדש. . קח את μL עוד 1-2 כדי לזהות את הריכוז באמצעות fluorometer
הערה: שחזור של 1-2 μg בשלב זה צפוי. הספרייה מבוססת על הטיית מכני מוכן להעלאה. ניתן לאחסן בספריה על קרח עבור עד 2 שעות עד טעינה עבור רצף במידת הצורך.
- הפשרה, תערובת ריאגנטים מ מצדו קיט (ראה טבלה של חומרים). הפשרה FFPE DNA תיקון מאגר, סוף מאגר תיקון/דה-עוקב על קרח, ואז המערבולת ו ספין למטה כדי לערבב. הפשרת מתאם שילוב (AMX) ומאגר מתאם חרוז מחייב (ABB) על קרח, ואז פיפטה ו ספין למטה כדי לערבב. הפשרה פועל מאגר עם תערובת דלק (RBF) ו • תנאי מאגר (ELB)-RT, ואז מערבולת ספין כדי לערבב. הפשרת ספריית טעינת חרוזים (LLB) RT, פיפטה לערבב לפני השימוש.
- בניין הספרייה מבוססת-פיצול Transposase
- הפשרה ריאגנטים מ transposase קיט (ראה טבלה של חומרים). הפשרת פיצול מיקס (FRA), מתאם מהירה (ראפ) על קרח, פיפטה לערבב. הפשרת רצף מאגר (SQB), טוען חרוזים (LB), ריקון מאגר (FLB) ריקון הקשר (טיסה)-RT, פיפטה לערבב. הפשרת הטעינה חרוזים (LB) RT, פיפטה לערבב לפני השימוש. ברגע הקרת, לשמור על כל הרכיבים קיט על קרח. להוציא את אנזימי רק בעת הצורך.
- בדוק את האיכות והכמות של ה-DNA HMW מהשלב 1.21.2. פיפטה החוצה 20 μL של ה-DNA לתוך צינורות חדשים 1.5 mL ממקומות שונים שלושה בצינור HMW DNA באמצעות P200 רחב נשא טיפים. קח μL 1 מ aliquots שלושה כדי לזהות את הריכוז באמצעות fluorometer ואת איכות באמצעות אולטרה קריאה. בדוק מספר פעמים כדי לאשר את התוצאות.
הערה: התוצאות הצפויות מוצגים באיור 3B. הערך260/280 OD הוא כ 1.9 והוא הערך260/230 OD כ 2.3. - הכינו את התגובה tagmentation DNA בשפופרת 0.2 מ"ל על-ידי הוספת μL 22 של DNA HMW, 1 μL 10 מ מ טריס (pH 8.0) עם 0.02% טריטון μL X-100 ו- 1 של פיצול לערבב (FRA). מיקס על ידי pipetting עם P200 רחב נשא עצה לאט ככל האפשר 6 פעמים, מטפל לא להכיר בועות.
- דגירה התגובה ב 30 ° C עבור 1 דקות ולאחריה 80 ° C עבור 1 דקות ולאחר מכן החזק ב 4 º C. להעביר התערובת לתוך צינור 1.5 mL החדש עם P200 רחב נשא עצה ועבור אל הצעד הבא מיד.
- להוסיף 1 μL של מתאם מהירה (ראפ) הצינור מדגם 1.5 mL. מיקס על ידי pipetting עם P200 רחב נשא עצה לאט ככל האפשר 6 פעמים, מטפל לא להכיר בועות.
- דגירה התגובה ב RT עבור 60 דקות.
הערה: הספרייה מבוססת-פיצול transposase מוכן להעלאה. ניתן לאחסן בספריה על קרח עבור עד 2 שעות עד טעינה עבור רצף במידת הצורך.
3. קביעת רצף על המכשיר nanopore
- בדוק את ההתקן רצף nanopore (ראה טבלה של חומרים). ודא כי התוכנה והחומרה פועלים וגם די שטח אחסון.
- בדוק את התא זרימה. לפתוח תא זרם חדש והכנס את התא זרימה לתוך המכשיר nanopore. בדוק שבתיבת מיקום התא זרימה הוכנס (X1-X5). בחר את סוג התא זרימה נכונה. לחץ על בדוק תאים זרימה זרימת העבודה. לחץ על הלחצן Test להתחיל להפעיל את התא זרימה QC ניתוח.
הערה: אם המספר שדווח נקבובית פעיל הכולל פחות מ 800, השתמש תא זרימה שונים חדש עבור רצף. - הכנת המאגר לקרקע. עבור ספריית מבוסס על חיתוך מכני, להוסיף μL 576 של הפעלת מאגר עם תערובת דלק (RBF) ו- 624 μL של נוקלאז ללא מים לתוך צינור 1.5 מ ל. מערבולת, ספין למטה כדי לערבב החומר הממתן לקרקע. עבור ספריית transposase המבוסס על פיצול, להוסיף 30 μL של הקשר סומק (FLT) הצינור מאגר סומק (FLB). מערבולת, ספין למטה כדי לערבב החומר הממתן לקרקע.
- על התא זרימה, להעביר את המכסה לקרקע בכיוון השעון, כדי לחשוף את היציאה לקרקע.
- מוגדר על פיפטה P1000 100 μL והכנס את הקצה ליציאת ה-לקרקע. לסגת נפח קטן של מאגר (פחות מ- 30 μL) כדי להסיר את כל הבועות מהתא זרימה. להפסיק pipetting פעם כמות קטנה של נוזל צהוב מזין את הטיפ.
- השתמש פיפטה P1000 כדי לטעון μL 800 של המיקס לקרקע לתוך התא זרימה דרך הנמל לקרקע. כדי להימנע היכרות עם בועות, להוסיף 30 μL של המיקס לקרקע כדי לכסות את החלק העליון של הנמל לקרקע קודם, ואז להוסיף את הטיפ ליציאת ה-לקרקע ולהוסיף לאט שאר המיקס לקרקע. להוציא את הטיפ כאשר יש כ-50 μL משמאל. מוסיפים את שארית תערובת לקרקע בראש הנמל לקרקע. הנוזל יעבור בתוך עצמו.
- להשאיר את ההתקנה עד תקופת דגירה של 5 דקות. בינתיים, הכנת המיקס ספריית בצינור 1.5 mL המכיל את הספרייה.
הערה: עבור ספריית מבוסס על חיתוך מכני להוסיף µL 35 של הפעלת מאגר עם תערובת דלק (RBF) כדי µL 40 של ספריית ה-DNA. עבור ספריית המבוסס על פיצול transposase להוסיף µL 34 רצף מאגר (SQB) ו-16 µL של מים נטולי נוקלאז µL 25 של ספריית ה-DNA. - פתח את המכסה יציאת זרימה מדגם תא בעדינות כדי לחשוף את היציאה מדגם. השתמש פיפטה P1000 כדי להוסיף 200 μL של המיקס לקרקע באמצעות היציאה לקרקע לתוך התא זרימה כפי שמתואר בשלב 3.5. ודא כי השילוב לקרקע אינו נטען לתוך התא זרימה דרך היציאה הדגימה.
- הגדר של פיפטה P200 80 μL. מערבבים הספרייה בעדינות עם טיפ נשא רחב על-ידי pipetting למעלה ולמטה 6 פעמים בדיוק לפני הטעינה.
- לטעון את התמהיל ספריית dropwise דרך היציאה מדגם לתוך התא זרימה. הוסף כל ירידה רק לאחר ההעברה הקודמת היא טעינתו היציאה.
- להחזיר את המכסה מדגם בעדינות ולוודא שיציאת מדגם מכוסה. הזז את המכסה לקרקע התרחקותו כדי לכסות את היציאה לקרקע. . סגור את המכסה התקן
- לחץ על זרימת העבודה התנסות חדשה . הקלד את שם הספריה, בחר ערכת הנכונה על פי הנהלים הנהוגים ולאחר לבדוק שההגדרות הן נכונות (48 h לרוץ, על בסיס שיחות בזמן אמת).
- לחץ על התחל הפעלה. לאחר 10 דקות, הקלט המזהה את התא זרימה, המספרים nanopore פעיל (סה כ ומספרים כל ארבע קבוצות) מן המידע לרוץ.
- ניתוח נתונים. העתק את נתוני מחשב מקומי או אשכול בכל עת רצף או בסיום המרוץ. להשתמש Minimap216 (https://github.com/lh3/minimap2) כדי ליישר את הנתונים רצף הגנום הפניה. מסכמים את הביצועים רצף הנתונים הגולמיים רצף, במערכים את לפי NanoPlot17 (https://github.com/wdecoster/NanoPlot).
תוצאות
פרוטוקול ארוכים במיוחד של רצפי דנ א חל HMW DNA לבנייה הספרייה. לכן, חשוב לבחור תאים בתרבית היטב עם יחס בשידור חי > 85% מהתא קציר שלב. כמות התאים המשמש להפקת DNA ישפיעו על האיכות והכמות של ה-DNA HMW. פירוק התא לא עובד טוב אם מתחיל עם תאים רבים מדי. באמצעות תאים מעט מדי אינו יוצר מספיק דנ"א לבנייה ספריית כי המשקעים HMW DNA מתבצע באמצעות סיבוב עדין בעבודת יד במקום צנטריפוגה במהירות גבוהה. דוגמה של ה-DNA HMW לאחר הוספת אתנול 100% קר כקרח וסיבוב הוא כמוצג את התמיסה כמו כותנה לבן באיור2.
חשוב לבדוק את איכות הקלט DNA לפני תחילת בניית ספריה. השפלה, כימות שגוי, זיהום (למשל, חלבונים, RNAs, חומרי ניקוי, חומרים פעילי שטח, ו שיורית פנול או אתנול) ו- DNA משקל מולקולרי נמוך יכולה להיות השפעה משמעותית על ההליכים הבאים ועל הגמר לקרוא אורך. אנו ממליצים על ביצוע הניתוח QC באמצעות ה-DNA ממקומות שונים שלושה בצינור המכילה HMW DNA. מ- UV קריאת תוצאות ה-DNA HMW, הערך280 OD /OD260הוא כ 1.9, הערך230 260OD /OD הוא כ 2.3 (איור 3 אב'). ערכים אלה יחס הם עקביים בין שלושת המבחנים דגימת HMW DNA טוב. שיטות ההטיה שונים דורש בנפחים שונים של DNA קלט. הריכוז של ה-DNA HMW צריך להיות > 200 ng/µL עבור הגז מכני בזמן זה צריך להיות > 1 µg µL transposase שאין בהם פיצול. ריכוז שזוהה על-ידי fluorometer הוא מעט נמוך יותר UV קריאה. עם זאת, המקדם של וריאציה של ריכוז HMW לדנ אותו נדרש להיות פחות מ- 15% את fluorometer וגם את UV קריאת מבחני. הטיית מכני חלה מזרק עם מחט כדי לשבור את ה-DNA HMW כך מספר עובר דרך המחט תשפיע על הגודל של ה-DNA הוטו ולקרוא הסופי אורך. מומלץ לבצע גודל QC לאחר המחט הטיה על מנת להבטיח את הרוב של ה-DNA HMW הוא גדול יותר מאשר 50 קילובייט כמופיע באיור4. שיטת חיתוך מכני, עובר 30 שנוצר התוצאות רצף בהתחשב באורך והן פלט.
N50 של ספריה מבוסס על חיתוך מכני הוא 50-70 kb אמנם ספרייה המבוססת על פיצול transposase 90-100 kb. התוצאות של ארבע ריצות באמצעות קו תא HG00733 מוצגים בטבלה 1. כל ארבע ריצות יש קריאות מעל 2,300 עם אורך יותר מ 100 kb. האורך המקסימלי הוא ארוך יותר בספריות transposase המבוסס על פיצול (455 kb, 489 kb) בהשוואה עם ספריות מבוסס על חיתוך מכני (348 kb ו- 387 kb) בזמן האחרון המיוצר קריאות סכום נוסף, המציין תשואה גבוהה יותר. בניית הספרייה מבוססת-פיצול transposase יש לו פחות שלבים, זמן הכנה קצר יותר כך זה תציג פחות קטעים קצרים. שתי ריצות באמצעות transposase יש אורך מרושע יותר (> 30 kb) ואורך החציוני (> 10 kb). בנוסף, מציג הנתונים גבוהה ועקבית של איכות כל הרצפים (ציון איכות רשע הוא כ 10.0, ~ 90% הבסיס דיוק). יותר מ- 97% הבסיסים סה כ היו המיושר הגנום האנושי הפניה (hg19) באמצעות Minimap216 עם הגדרות ברירת המחדל. ההפצות הגודל הצפוי הקריאות raw מוצגים באיור5. כל הרצפים יש אחוז גדול של נתונים מעל 50 kb transposase המבוסס על פיצול ספריות יש יחס גבוה יותר של קריאות ארוכים במיוחד (למשל > 100 kb). פרוטוקול זה יושם בהצלחה במספר שורות תאים אנושיים (משלים טבלה 1).
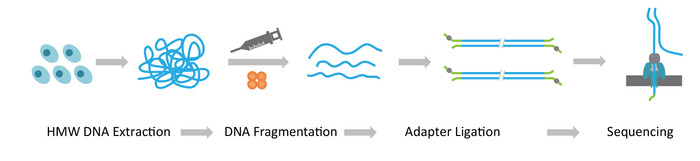
איור 1: סקירה סכמטי של זרימת העבודה רצף ארוך-קריאה (NLR-seq) nanopore. כתום, transposase מורכבים. צהוב ירוק, המתאם nanopore. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 2: נציג ה-DNA משקעים של שיטת החילוץ פנול-כלורופורם. החץ הלבן מציין את ה-DNA HMW. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 3: דוגמה QC תוצאות ה-dna HMW מקריאה UV. דנ א מ שלב 1.21.1 מוכן לבנייה ספריית מבוסס על חיתוך מכני (א) HMW. דנ א מהשלב 1.21.2 לבנייה הספרייה מבוססת-פיצול transposase (B) HMW. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 4: QC תוצאות של המחט לכסנתם HMW DNA על-ידי שדה פעמו בג'ל. L1: מטען מהיר 1 kb סולם הדנ א; L2: מטען מהיר 1 kb להרחיב סולם הדנ א. 1-8: DNA עם פטירתו שונה פעמים דרך המחט אמש. 1-3, לא הטיה; 4, 10 פעמים; 5, 20 פעמים; 6. 30 פעמים; 7, 40 פעמים; 8, 50 פעמים. שלב זה QC היא אופציונלית. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 5: צפוי הפצות גודל הספריות הדי ארוכים במיוחד של nanopore. MS, ספריות מבוסס על חיתוך מכני. TF, transposase מבוסס על פיצול ספריות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| Shearing_rep1 מכני | Shearing_rep2 מכני | Transposase fragmentation_rep1 | Transposase fragmentation_rep2 | |
| תא קו | HG00733 | HG00733 | HG00733 | HG00733 |
| N50 הקריאות | 55,180 | 63,007 | 98,237 | 95,629 |
| מספר הקריאות יותר מ-100 Kb | 2,500 | 3,082 | 2,386 | 2,355 |
| מספר הקריאות סה | 97,859 | 80,465 | 24,166 | 21,032 |
| האורך המרבי (bp) | 348,482 | 387,113 | 454,660 | 489,426 |
| כלומר אורך (bp) | 17,861 | 20,395 | 33,528 | 38,175 |
| אורך החציוני (bp) | 5,335 | 5,894 | 10,249 | 15,656 |
| זאת אומרת איכות הקריאות | 10.0 | 10.1 | 9.9 | 10.0 |
| סה כ בסיסים הקריאות raw | 1,747,849,822 | 1,641,058,932 | 810,229,733 | 802,886,304 |
| סה כ בסיסים הקריאות מיושר | 1,693,300,832 | 1,607,975,925 | 791,422,077 | 778,417,627 |
| יחס ממופה של בסיסים הכולל (hg19, Minimap2) | 96.9% | 98.0% | 97.7% | 97.0% |
| מספר הנקבוביות פעיל | 1225: 480, 402, 254, 89 | 1058: 480, 356, 176, 46 | 958: 452, 328, 148, 30 | 1092: 487, 367, 195, 43 |
טבלה 1: סיכום של מדדי ביצועים פועל עם פרוטוקולים חיתוך שונות.
| ספריית 1 | ספריית 2 | |
| תא קו | K562 | GM19240 |
| תא פרטי הזמנה | בקרת האוויר, החתול. לא. CCL-243 | מכון Coriell, חתול. לא. GM19240 |
| פרוטוקול | הטיית מכני | הטיית מכני |
| N50 הקריאות | 60,063 | 55,295 |
| מספר הקריאות סה | 193,783 | 120,807 |
| אורך החציוני (bp) | 1,843 | 4,688 |
| כלומר אורך (bp) | 9,825 | 17,408 |
| האורך המרבי (bp) | 548,780 | 212,338 |
| סה כ בסיסים הקריאות raw | 1,903,989,686 | 2,103,015,331 |
| סה כ בסיסים הקריאות מיושר | 1,837,350,047 | 1,997,419,761 |
| יחס ממופה של בסיסים הכולל (hg19, Minimap2) | 96.6% | 95.0% |
| מספר הנקבוביות פעיל | 1111: 482, 371, 203, 55 | 1032: 447, 333, 196, 56 |
משלים טבלה 1: סיכום של שתי ריצות NLR-seq באמצעות שורות תאים אחרים עם פרוטוקול חיתוך מכני.
Discussion
בעקרון, רצף nanopore הוא מסוגל לייצר 100 kb כדי קריאות megabase אורך11,12,13. ארבעה גורמים עיקריים ישפיע על הביצועים של רצף הפעלה ונתונים האיכות: 1) נקבובית פעיל מספרים וכן את פעילותם של הנקבוביות; 2) חלבון מנוע, אשר שולט על המהירות של ה-DNA עובר nanopore; 3) תבנית DNA (אורך, טוהר, איכות, המוני); 4) רצף מתאם יעילות מצדו, הקובע את ה-DNA שמיש מדגם קלט. הגורמים הראשונים תלויים בגירסה של התא זרימה, ערכת רצף המסופקים על ידי היצרן. שנית משני הגורמים הם השלבים הקריטיים פרוטוקול זה (מיצוי HMW DNA, הטיה, מצדו).
פרוטוקול זה דורש אימון וסבלנות. האיכות של ה-DNA HMW חשוב עבור ספריות DNA6ארוכים במיוחד. הפרוטוקול מתחיל עם תאים נאספו עם הכדאיות גבוהה (> תא קיימא 85% העדיפו), הגבלת את הדנ א פגום של תאים מתים. יש להימנע כל תהליך קשה אשר עלולה להציג נזק ל- DNA (למשל, חזק מפריע, רעידות, מערבולת, מרובות pipetting, חוזרות ונשנות הקפאה והפשרה של). עיצוב של הפרוטוקול, אנחנו להשמיט pipetting בתוך כל התהליך של הפקת דנ א. טיפים נשא רחב צריך לשמש כאשר pipetting יש צורך אחרי חיתוך מכני במהלך הבנייה ספריית רצף. כמו nanopores רגישים בדיקות, הביוכימיה מאגר קאמרית12, צריך להיות כמה מזהמים שיורית (למשל, חומרי ניקוי, פיתחה, פנול, אתנול, חלבונים RNAs, וכו ') ככל האפשר ב- DNA. בהתחשב האורך ואת התשואה, שיטת החילוץ פנול מראה את התוצאות הטוב ביותר ביותר לשחזור לעומת בשיטות חילוץ שונות מרובות נבדקו עד כה.
למרות היכולת של פרוטוקול זה לייצר רצף ארוך-קריאה, מספר מגבלות עדיין נשארים. ראשית, פרוטוקול זה היה מותאם המבוססת על רצף nanopore המכשיר זמין בזמן הפרסום; לפיכך, הוא מוגבל הכימיה רצף המבוסס על nanopore סלקטיבית, יכול להיות שיוצרת כאשר מבוצעת בסוגים אחרים של התקני רצף ארוך-קריאה. שנית, התוצאה תלויה מאוד האיכות של ה-DNA שחולצו מן חומר המוצא (רקמות או תאים). אורך לקריאה יסוכל אם ה-DNA מתחיל כבר מושפל או פגום. שלישית, למרות מספר השלבים QC משולבים בפרוטוקול כדי לבדוק את איכות ה-DNA, התשואה הסופית ואורך הקריאות עשויה להיות מושפעת התא זרימה ונקבוביות פעילות, אשר יכול להיות משתנה בשלב מוקדם זה של פלטפורמת רצף nanopore פיתוח.
הפרוטוקול המתואר כאן משתמש ההשעיה האדם תא קו דגימות להפקת DNA. לנו יש אופטימיזציה פעמים עובר את המחט הטיה, היחס של ה-DNA HMW transposase ואת הזמן מצדו כדי להפיק את התוצאות המתואר. ניתן להרחיב את הפרוטוקול בארבע דרכים. ראשית, משתמשים יכולים להפעיל אחרים בתרבית של תאים בתרבית, עם כמות שונה של תאים, רקמות, דוגמאות קליניות או אורגניזמים אחרים. יהיה צורך נוספים מיטוב על זמן הדגירה פירוק, התגובה נפח צנטריפוגה. שנית, קשה לחזות את גודל היעד עבור רצף קריאה ארוכים במיוחד. אם המרחק קריאה קצרה יותר מהצפוי, המשתמשים יכולים להתאים טיימס שעובר בהשיטה מבוססת על הטיית מכני או שינוי היחס של ה-DNA HMW transposase בשיטה מבוססת-פיצול transposase. זמן מחייבים, • תנאי יותר במהלך ניקוי צעדים מועילים בגלל ה-DNA HMW בעלי צמיגות גבוהה. שלישית, עם התקנים רצף nanopore שונים, אחד ניתן להתאים את כמות ונפח של ה-DNA כדי לענות על הקריטריונים של הרצפים (sequencer). רביעית, רק אלה DNA מאתרים למתאמים רצף להיות רציף. כדי לשפר את יעילות מצדו, אחד יכול לנסות titrate ריכוז המתאם, ליגאז. זמן מצדו ששונה וסוכנים המתגודדים מולקולרי כגון פג18 ניתן להחיל בעתיד. פרוטוקול רצפי DNA ארוכים במיוחד בשילוב עם CRISPR19,20 עשוי להציע כלי יעיל עבור היעד העשרה רצף.
Disclosures
המחברים מצהירים כי יש להם אינטרסים כלכליים אין מתחרים.
Acknowledgements
המחברים מודים ז'ו י' על הערות על כתב היד. מחקר דיווח בפרסום זה מומן בחלקו על ידי המכון הלאומי לסרטן של מכוני הבריאות הלאומיים תחת פרס מספר P30CA034196. התוכן הוא אך ורק באחריות המחברים, ואינם מייצגים בהכרח את הנופים הרשמי של מכוני הבריאות הלאומיים.
Materials
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Absolute ethanol | Sigma-Aldrich | E7023 | |
| Agencourt AMPure XPbeads | Beckman | A63881 | magnetic beads for cleanup |
| BD conventional needles | Becton Dickinson | 305136 | 27G, for mechanical shearing |
| BD Luer-Lok syringe | Becton Dickinson | 309628 | for mechanical shearing |
| Blunt/TA Ligase Master Mix | NEB | M0367S | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | for cell counting |
| EDTA | Invitrogen | AM9261 | pH 8.0, 0.5 M, 500 mL |
| Flow Cell | Oxford Nanopore Technologies | FLO-MIN106 | R9.4.1 |
| HG00773 cells | Coriell Institute | HG00733 | cells used in this protocol |
| Ligation Sequencing Kit 1D | Oxford Nanopore Technologies | SQK-LSK108 | nanopore ligation kit |
| MaXtract High Density tubes | Qiagen | 129073 | gel tubes |
| NEBNext FFPE DNA Repair Mix | NEB | M6630S | |
| NEBNext Ultra II End Repair/dA-Tailing Module | NEB | M7546S | |
| Nuclease-free water | Invitrogen | AM9937 | |
| Phosphate-Buffered Saline, PBS | Gibco | 70011044 | 10X, pH 7.4 |
| Phenol:chloroform:IAA | Invitrogen | AM9730 | |
| Proteinase K | Qiagen | 19131 | 20 mg/mL |
| Qubit dsDNA BR Assay Kit | Invitrogen | Q32850 | fluorometer assays for DNA quantification |
| Rapid Sequencing Kit | Oxford Nanopore Technologies | SQK-RAD004 | nanopore transposase kit |
| RNase A | Qiagen | 19101 | 100 mg/mL |
| SDS | Invitrogen | AM9822 | 10% (wt/vol) |
| Sodium chloride solution | Invitrogen | AM9759 | 5.0 M |
| TE buffer | Invitrogen | AM9849 | pH 8.0 |
| Tris | Invitrogen | AM9856 | pH 8.0, 1 M |
| Triton X-100 solution | Sigma-Aldrich | 93443 | ~10% |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Bio-Rad C1000 Thermal Cycler | Bio-Rad | 1851196EDU | |
| Centrifuge 5810R | Eppendorf | 22628180 | |
| Countess II FL Automated Cell Counter | Life Technologies | AMQAF1000 | for cell counting |
| DynaMag-2 Magnet | Life Technologies | 12321D | magnetic rack |
| Eppendorf ThermoMixer | Eppendorf | 5382000023 | for incubation |
| Freezer | LabRepCo | LHP-5-UFMB | |
| GridION | Oxford Nanopore Technologies | GridION X5 | nanopore device used in this protocol |
| HulaMixer Sample Mixer | Thermo Fisher Scientific | 15920D | rotator mixer |
| MicroCentrifuge | Benchmark Scientific | C1012 | |
| NanoDrop ND-1000 Spectrophotometer | Thermo Fisher Scientific | ND-1000 | for UV reading |
| Pippin Pulse | Sage Science | PPI0200 | pulsed-field gel electrophoresis instrument |
| Qubit 3.0 Fluorometer | Invitrogen | Q33216 | fluorometer |
| Refrigerator | LabRepCo | LABHP-5-URBSS | |
| Vortex-Genie 2 | Scientific Industries | SI-A236 | |
| Water bath | VWR | 89501-464 |
References
- Mardis, E. R. Next-generation sequencing platforms. Annual Review of Analytical Chemistry. 6, 287-303 (2013).
- Goodwin, S., McPherson, J. D., McCombie, W. R. Coming of age: ten years of next-generation sequencing technologies. Nature Reviews Genetics. 17 (6), 333-351 (2016).
- Shendure, J., et al. DNA sequencing at 40: past, present and future. Nature. 550 (7676), 345-353 (2017).
- Alkan, C., Coe, B. P., Eichler, E. E. Genome structural variation discovery and genotyping. Nature Reviews Genetics. 12 (5), 363-376 (2011).
- Weischenfeldt, J., Symmons, O., Spitz, F., Korbel, J. O. Phenotypic impact of genomic structural variation: insights from and for human disease. Nature Reviews Genetics. 14 (2), 125-138 (2013).
- Pollard, M. O., Gurdasani, D., Mentzer, A. J., Porter, T., Sandhu, M. S. Long reads: their purpose and place. Human Molecular Genetics. 27 (R2), R234-R241 (2018).
- Cretu Stancu, M., et al. Mapping and phasing of structural variation in patient genomes using nanopore sequencing. Nature Communications. 8 (1), 1326 (2017).
- Gong, L., et al. Picky comprehensively detects high-resolution structural variants in nanopore long reads. Nature Methods. 15 (6), 455-460 (2018).
- Sedlazeck, F. J., et al. Accurate detection of complex structural variations using single-molecule sequencing. Nature Methods. 15 (6), 461-468 (2018).
- Jain, M., et al. Nanopore sequencing and assembly of a human genome with ultra-long reads. Nature Biotechnology. 36 (4), 338-345 (2018).
- Jain, M., et al. Improved data analysis for the MinION nanopore sequencer. Nature Methods. 12 (4), 351-356 (2015).
- Deamer, D., Akeson, M., Branton, D. Three decades of nanopore sequencing. Nature Biotechnology. 34 (5), 518-524 (2016).
- Jain, M., Olsen, H. E., Paten, B., Akeson, M. The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community. Genome Biology. 17 (1), 239 (2016).
- Editorial, The long view on sequencing. Nature Biotechnology. 36 (4), 287 (2018).
- Jain, M., et al. Linear assembly of a human centromere on the Y chromosome. Nature Biotechnology. 36 (4), 321-323 (2018).
- Li, H. Minimap2: pairwise alignment for nucleotide sequences. Bioinformatics. 34, 3094-3100 (2018).
- De Coster, W., D'Hert, S., Schultz, D. T., Cruts, M., Van Broeckhoven, C. NanoPack: visualizing and processing long-read sequencing data. Bioinformatics. 34, 2666-2669 (2018).
- Akabayov, B., Akabayov, S. R., Lee, S. J., Wagner, G., Richardson, C. C. Impact of macromolecular crowding on DNA replication. Nature Communications. 4, 1615 (2013).
- Gabrieli, T., Sharim, H., Michaeli, Y., Ebenstein, Y. Cas9-Assisted Targeting of CHromosome segments (CATCH) for targeted nanopore sequencing and optical genome mapping. bioRxiv. , (2017).
- Gabrieli, T., et al. Selective nanopore sequencing of human BRCA1 by Cas9-assisted targeting of chromosome segments (CATCH). Nucleic Acids Research. , (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved