需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过质谱学研究蛋白质高阶结构,与二乙基碳酸酯共价标签
摘要
介绍了在质谱检测中进行基于二乙基碳酸酯的共价贴标的实验程序。二甲基碳酸酯只是与感兴趣的蛋白质或蛋白质复合物混合,导致溶剂可获取氨基酸残留物的修饰。经蛋白解消化和液相色谱/质谱分析后,可识别修改后的残留物。
摘要
描述蛋白质的更高阶结构对于理解其功能至关重要。质谱仪(MS)已成为这一目的的有力工具,尤其是对于传统方法难以研究的蛋白质系统。为了研究MS的蛋白质结构,在将蛋白质的结构信息编码到蛋白质质量的溶液中执行特定的化学反应。一个特别有效的方法是使用可共效地修改溶剂可访问氨基酸侧链的试剂。这些反应导致质量增加,当与蛋白解消化和串联质谱法相结合时,可以用残留水平分辨率进行局部化。在这里,我们描述与使用二乙基碳酸酯 (DEPC) 作为共价标记试剂以及 MS 检测相关的协议。DEPC 是一种高度电亲的分子,能够标记平均蛋白质中高达 30% 的残留物,从而提供出色的结构分辨率。DEPC 已与 MS 一起成功用于获取小单域蛋白质(如 β2 微球蛋白)到大型多域蛋白(如单克隆抗体)的结构信息。
引言
蛋白质是几乎所有生理过程中必不可少的生物分子。蛋白质所执行的各种功能是可能的,因为它们采用的结构和它们与其他生物分子的相互作用。为了更深入地了解蛋白质的功能,需要生化和生物物理工具来阐明这些重要的结构特征和相互作用。传统上,X射线晶体学、低温电子显微镜和核磁共振(NMR)光谱学为揭示蛋白质结构提供了所需的原子级细节。然而,由于结晶行为不良、蛋白质可用性有限、样品异质性过大或分子重量限制,许多蛋白质系统无法通过这些技术进行审问。因此,出现了克服这些限制的较新的分析方法。提供蛋白质结构信息的新兴技术包括质谱法。
质谱测量 (MS) 测量分子的质量对电荷 (m/z) 比率,因此必须通过将所需的结构信息编码到蛋白质的质量中来获取蛋白质高阶结构信息。已开发出几种编码这些信息的方法,包括氢-铀交换(HDX)1、2、3、4、化学交联(XL)5、6和共价标签(CL)7、8、9、10。在HDX中,骨干氢气的交换量略大,取决于溶剂的可及性和H-粘结程度。HDX的范围可以通过快速消化蛋白质到肽碎片,然后通过质谱仪分离和测量这些片段,或通过自上而下的实验分离蛋白质来本地化。确定汇率有助于进一步深入了解蛋白质动态。HDX 已被证明是一个有价值的工具,用于描述蛋白质结构,尽管与背部交换相关的挑战和需要专门设备,以最大限度地提高可重复性。在 XL-MS 中,蛋白质与双功能试剂发生反应,双功能试剂可共价地将给定蛋白质内或两种蛋白质之间的相邻残留侧链连接起来。这样,XL-MS 可以提供可用于描述蛋白质结构的距离限制。蛋白质相互连接的区域可以通过蛋白质分析消化和液相色谱(LC)-MS分析来识别。虽然 XL-MS 是一种多功能工具,用于研究各种蛋白质复合物(包括细胞内部),但 XL 产品的识别具有挑战性,需要专门的软件。
CL-MS最近已成为一种补充性的,有时是替代基于MS的工具,研究蛋白质结构和相互作用。在CL-MS中,蛋白质或蛋白质复合物与单功能试剂共效修改,可与溶剂暴露的侧链(图1)做出反应。通过比较不同条件下蛋白质或蛋白质复合物的修改范围,可以揭示出构象变化、结合位点和蛋白质-蛋白质接口。CL反应后,通常位于单一氨基酸水平的站点特定信息可以通过典型的自下而上的蛋白质组学工作流程获得,其中蛋白质被蛋白质蛋白解消化,肽片段由LC分离,并且使用串联MS(MS/MS)识别修改后的位点。丰富的生物结合化学历史为CL-MS实验提供了许多试剂。CL 试剂分为两个一般类别:(一) 特定和 (ii) 非特定。特定试剂与单个功能组(如免费胺)8、10反应,易于实现,但往往提供有限的结构信息。非特异性试剂与各种侧链发生反应,但通常需要激光或同步加速器源等专门设备才能产生这些高度反应的物种。羟基基是最常用的非特异性试剂,已应用于羟基基足迹(HRF)7、11、12、13实验,研究各种条件下的各种蛋白质和蛋白质复合物。
我们的研究小组在CL-MS实验14、15、16、17、18、19、20、21、22、23、24、25的背景下,成功地使用了另一种相对非特异性试剂,称为二乙基碳酸酯(DEPC)来研究蛋白质结构和相互作用。DEPC 提供特定标签试剂的简单性(即无需任何专门设备来执行反应),同时对普通蛋白质中高达 30% 的氨基酸做出反应。作为一种高度电效试剂,DEPC 能够与 N 端和西施泰因、西斯蒂丁、赖氨酸、酪氨酸、血清和三氨酸残留物的核亲侧链做出反应。通常,产生这些反应的单个产品,导致质量增加 72.02 Da。这种单一类型的产品与多达55种不同的产品形成对比,当蛋白质与羟基反应时,这些产品可以产生7种。这种简单的化学成分有助于识别标记的地点。
在这里,我们提供使用基于DEPC的CL-MS来研究蛋白质结构和相互作用的协议。详细介绍了与试剂制备、DEPC-蛋白质反应、蛋白质消化条件、LC-MS 和 MS/MS 参数以及数据分析相关的详细信息。我们还通过提供蛋白质金属、蛋白质-脂和蛋白质-蛋白质相互作用以及加热后发生结构变化的蛋白质提供示例结果,来证明DEPC标签的效用。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 蛋白质和试剂制剂
注:此协议包括一个用 DEPC 标记蛋白质的示例工作流。列出的某些条件和试剂浓度可能因所选蛋白质而异。
- 在 1.5 mL 微中福格管中准备所有试剂解决方案。
- 在pH 7.4的10mM 3-(N-莫索利诺)丙烷磺酸(MOPS)缓冲器中,准备一种所需的浓度蛋白质溶液,通常在数十微米范围内。或者,如果样品中含有与DEPC有反应的核嗜血缓冲液,则用10mm pH 7.4 MOPS制备缓冲交换现有蛋白质溶液。其他缓冲剂(如磷酸盐缓冲盐水)也可以使用,只要它们没有核嗜血功能组。
- 通过将库存 6.9 M DEPC 溶液中的 1.45 μL 管道输送到 ACN 的 98.55 μL 中,在干乙酰氨基三酯 (ACN) 中准备 100 mM DEPC 解决方案。
注:没有一组浓度可以与每种蛋白质配合使用,但最佳浓度可以根据他和Lys残留物23的数量来估算。例如,使用 50μM β2-微球蛋白溶液中的 50 μL,使用 100 mM DEPC 的 0.2 μL 对蛋白质做出反应,最终 DEPC 浓度为 200 μM(相当于蛋白质浓度的 4 倍),以确保使用此通用示例(表 1)所需的摩尔比。DEPC 标签是一种二 阶反应,因此改变反应混合物中蛋白质或 DEPC 的浓度将更改标签速率。 - 通过称出 10 毫克的伊米达佐尔并在 146.9 μL 的 HPLC 级水中溶解,准备 1 M imidazole 溶液。
2. 完整蛋白质的共价标签
- 将水浴温度设置为 37 °C,等待浴缸达到稳定的温度。
注:试剂浓度和体积的示例标记协议可以在 表 1中找到。 - 在一个新的微中量管中,将MOPS缓冲器和蛋白质溶液混合在 表1中列出的体积中。
- 在蛋白质和缓冲区中加入 0.2μL 的 DEPC 溶液,确保将生成的溶液正确混合,然后将含有反应混合物的管子放入 37 °C 水浴中 1 分钟。
注意:添加的 ACN 体积不应超过总反应量的 1%,以避免在标记反应期间蛋白质结构的扰动。反应时间由用户决定,尽管在示例条件下的 1 分钟反应可最大限度地减少重叠和 DEPC14的潜在水解。 - 1 分钟后,从水浴中取出含有反应混合物的管子,用 1 M imidazole 溶液中的 1μL 淬气反应,以清除剩余的未反应 DEPC。
注:反应混合物中二氧化二甲苯的最终浓度应等于反应混合物中DEPC浓度的50倍。这将确保清除剩余未反应的 DEPC。
3. 为自下而上的LC-MS准备蛋白质消化
注意:选择符合兴趣蛋白的消化条件。常见的步骤包括展开蛋白质,减少和碱化任何脱硫键。
- 通过在反应混合物中加入适当的展开试剂来展开蛋白质。
注:常见的展开剂包括ACN、尿素和盐酸瓜尼丁(GuHCl)。 - 通过称出每个 5 毫克,并在 174.4 和 270.3 μL 的 10 mM pH 7.4 MOPS 缓冲器中分别溶解新的微中微富支管,分别准备三合院(2-卡盒乙基)磷酸 (TCEP) 和碘乙酰胺 (IAM) 的解决方案,用于减少和分解步骤。
- 通过在反应混合物中加入 100 mM TCEP 中的 2μL(反应混合物中最终浓度为 2 mM)溶液,并在室温下反应 3 分钟,减少脱硫键。
注:TCEP的最终浓度应等于溶液中每份脱硫键蛋白质浓度的40倍。 - 烷基酸酯在黑暗中用100m IAM溶液中的4μL(反应混合物的最终浓度为4mM)减少半胱氨酸30分钟。IAM 对光敏感,会在直接光下分解。
注:IAM在溶液中的最终浓度应是 TCEP 浓度的两倍,或每脱硫键蛋白质浓度的 80 倍。 - 用适当的酶(如三氯辛或肌氨酸)消化蛋白质。10:1 蛋白质:在 37 °C 下消化 3 小时的酶比,以 300 冲程/分钟的摇动速度与固定酶进行 3 小时消化,通常足以获得 DEPC 标记的蛋白质。请参阅讨论。
- 消化后,以12,000 rpm的速度以离心机将固定酶与消化肽分离5分钟。
- 立即通过LC-MS/MS分析样品,或用液氮将样品闪冻,以尽量减少样品降解和标签损失。将闪冻结样品存放在<-20 °C,直到准备好进行LC-MS/MS分析。
4. LC-MS/MS 分析
注:自下而上的蛋白质组学标准LC-MS/MS参数可用于识别蛋白解肽片段上的标记位点。下面概述了一个通用示例。
- 使用倒相 C18 静止相位分离 DEPC 标记肽。使用两种溶剂的典型LC移动相:(A) 水 + 0.1% 用于微酸,(B) ACN + 0.1% 用于梯度(如 图 2),以实现肽的最佳分离。
注意:可根据样品的复杂性优化分离时间,移动相流速取决于是否使用毛细纸或纳米LC。 - 使用能够在线 LC-MS 和 MS/MS 的质谱仪来识别肽上的 DEPC 修改站点。在我们的实验中,我们成功地使用了几种类型的质谱仪。在LC-MS分析过程中,任何能够自动执行许多肽MS/MS的质谱仪都应该是合适的。相关 MS 参数包括:常规 ESI 的 ESI 源电压 = -4000 V;-2000 V表示纳米喷雾:轨道拉普分辨率 = 60,000;动态排除持续时间 = 30s;MS/MS 激活类型:CID、ETD 或两者兼有;质量扫描范围 = 200-2,000;自动增益控制 = 4.0E5(轨道圈中的 MS1)和 5.0E4(线性四足离子陷阱中的 MS2)。
- 将消化的、标记的蛋白质样本加载并注入LC系统,并开始LC-MS/MS采集。如果样品已闪冻结,在分析前解冻。将LC废水分流到废物中前5分钟,以避免过量的盐进入 ESI 源。
注意:通常使用 5 μL 注射回路,允许向 LC-MS/MS 注射约 2.5 微克蛋白质。这取决于LC的加载条件,以不堵塞样品喷油器。
5. 数据分析
- 识别DEPC标签站点,并使用用于所用质谱仪的适当软件量化肽峰值区域。
- 包括DEPC添加(72.02 Da)和卡巴米多姆甲基化(57.02 Da)作为可变修改。MS / MS 分析的其他搜索参数如下:最大未通过的 = 3 ;碎片离子类型=b和y:前体m/z公差 = 10ppm(如果使用四足离子陷阱质谱仪,此值应更高);碎片 m/z 公差 = 0.5 Da(如果用于产品离子扫描的高分辨率质谱仪,此值应较低);前体电荷=1-4。
注意:不同的数据库搜索算法有不同的评分系统,并且许多算法可能难以识别DEPC修改的肽,因为修改水平可能较低。调整分数截止点可能需要识别更多标记的肽。如果是这样,则应使用手动询问 MS/MS 数据来验证低分肽。DEPC 标签的水解产品不包括在数据搜索中,因为水解 DEPC 不再对核嗜血侧链有反应。 - 使用修改后的多肽的改性和未修改版本的色谱峰值区域确定残留级修改百分比。
注意:任何含有修改后的兴趣残留物的肽必须予以考虑,所有测量样本中必须包含所有充电状态。肽具有不同的电热效率和在不同的时间洗脱导致此值是一个相对的,而不是对特定站点的修改的绝对衡量。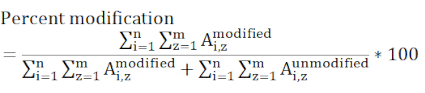
其中 Ai.z 表示任何特定肽 (i) 的峰值区域,该肽 (i) 包含利息残留物,并考虑所有检测到的电荷状态 (z)。 - 使用统计评估确定控制样本和实验样本之间的标签变化是否显著。每个样本的三个复制测量是典型的,t 测试最常用于 95 或 99% 的置信间隔。
Access restricted. Please log in or start a trial to view this content.
结果
识别 DEPC 修改站点和修改百分比
由于共价标签而增加的质量可以测量在 (a) 完整的蛋白质和 (b) 肽水平8,9。在完好无损的水平上,可以通过直接分析或贴有标签的蛋白质样本的LC-MS获得具有不同标签数的蛋白质物种的分布。要获得更高的分辨率结构信息(即特定于站点的标记数据),必须在肽级别进行测量。在标记和淬火步骤之后,标记...
Access restricted. Please log in or start a trial to view this content.
讨论
关键步骤
应考虑有关实验设计的若干点,以确保可靠的标记结果。首先,为了最大限度地提高蛋白质标签,有必要避免缓冲具有强烈核嗜血组(如Tris),因为它们可以与DEPC发生反应并降低标签的程度。也可以想象,这种缓冲器可能与标记的残留物发生反应,导致标签被移除,从而丢失结构信息。我们建议MOPS作为缓冲,但磷酸盐缓冲盐水也有效。其次,应避免使用二硫化物来减少二硫化...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
作者感谢国家卫生研究院(NIH)在格兰特R01转基因075092下的支持。用于获取此处描述的一些数据的温度轨道拉普融合质谱仪是从国家卫生研究院赠款 S10OD010645 获得的资金。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Thermo Fisher Scientific | 3448 | |
| 3-(N-morpholino)propanesulfonic acid | Millipore Sigma | M1254 | |
| 3-(N-morpholino)propanesulfonic acid sodium salt | Millipore Sigma | M9381 | |
| Acclaim PepMap RSLC C18 Column | Thermo Scientific | 164537 | 300 μm x 15 cm, C18, 2 μm, 100 A |
| Acetonitrile | Fisher Scientific | A998-1 | |
| Diethylpyrocarbonate | Millipore Sigma | D5758 | |
| HPLC-grade water | Fisher Scientific | W5-1 | |
| Imidazole | Millipore Sigma | I5513 | |
| Immobilized chymotrypsin | ProteoChem | g4105 | |
| Immobilized trypsin, TPCK Treated | Thermo Fisher Scientific | 20230 | |
| Iodoacetamide | Millipore Sigma | I1149 | |
| Tris(2-carboxyethyl)phosphine | Millipore Sigma | C4706 |
参考文献
- Katta, V., Chait, B. T., Carr, S. Conformational Changes in Proteins Probed by Hydrogen-exchange Electrospray-ionization. Rapid Communications in Mass Spectrometry. 5, 214-217 (1991).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25, 158-170 (2006).
- Pirrone, G. F., Iacob, R. E., Engen, J. R. Applications of hydrogen/deuterium exchange MS from 2012 to 2014. Analytical Chemistry. 87, 99-118 (2015).
- Oganesyan, I., Lento, C., Wilson, D. J. Contemporary hydrogen deuterium exchange mass spectrometry. Methods. 144, 27-42 (2018).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrometry Reviews. 25, 663-682 (2006).
- Holding, A. N. XL-MS: Protein cross-linking coupled with mass spectrometry. Methods. 89, 54-63 (2015).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107, 3514-3543 (2007).
- Mendoza, V. L., Vachet, R. W. Probing Protein Structure by Amino Acid-Specific Covalent Labeling and Mass Spectrometry. Mass Spectrometry Reviews. 28, 785-815 (2009).
- Limpikirati, P., Liu, T., Vachet, R. W. Covalent labeling-mass spectrometry with non-specific reagents for studying protein structure and interactions. Methods. 144, 79-93 (2018).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemistry Reviews. 120, 4335(2020).
- Maleknia, S. D., Brenowitz, M., Chance, M. R. Millisecond radiolytic modification of peptides by synchrotron X-rays identified by mass spectrometry. Analytical Chemistry. 71, 3965-3973 (1999).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77, 5814-5822 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society of Mass Spectrometry. 16, 2057-2063 (2005).
- Mendoza, V. L., Vachet, R. W. Protein surface mapping using diethylpyrocarbonate with mass spectrometric detection. Analytical Chemistry. 80, 2895-2904 (2008).
- Mendoza, V. L., Antwi, K., Barón-rodríguez, M. A., Blanco, C., Vachet, R. W. Structure of the Pre-amyloid Dimer of β-2-microglobulin from Covalent Labeling and Mass Spectrometry. Biochemistry. 49, 1522-1532 (2010).
- Mendoza, V. L., Barón-Rodríguez, M. A., Blanco, C., Vachet, R. W. Structural insights into the pre-amyloid tetramer of β-2-microglobulin from covalent labeling and mass spectrometry. Biochemistry. 50, 6711-6722 (2011).
- Zhou, Y., Vachet, R. W. Increased protein structural resolution from diethylpyrocarbonate-based covalent labeling and mass spectrometric detection. Journal of the American Society of Mass Spectrometry. 23, 708-717 (2012).
- Borotto, N. B., et al. Investigating Therapeutic Protein Structure with Diethylpyrocarbonate Labeling and Mass Spectrometry. Analytical Chemistry. 87, 10627-10634 (2015).
- Liu, T., Marcinko, T. M., Kiefer, P. A., Vachet, R. W. Using Covalent Labeling and Mass Spectrometry To Study Protein Binding Sites of Amyloid Inhibiting Molecules. Analytical Chemistry. 89, 11583-11591 (2017).
- Limpikirati, P., et al. Covalent labeling and mass spectrometry reveal subtle higher order structural changes for antibody therapeutics. MAbs. 11, 463-476 (2019).
- Limpikirati, P., Pan, X., Vachet, R. W. Covalent Labeling with Diethylpyrocarbonate: Sensitive to the Residue Microenvironment, Providing Improved Analysis of Protein Higher Order Structure by Mass Spectrometry. Analytical Chemistry. 91, 8516-8523 (2019).
- Liu, T., Limpikirati, P., Vachet, R. W. Synergistic Structural Information from Covalent Labeling and Hydrogen-Deuterium Exchange Mass Spectrometry for Protein-Ligand Interactions. Analytical Chemistry. 91, 15248-15254 (2019).
- Pan, X., Limpikirati, P., Chen, H., Liu, T., Vachet, R. W. Higher-Order Structure Influences the Kinetics of Diethylpyrocarbonate Covalent Labeling of Proteins. Journal of the American Society of Mass Spectrometry. 31, 658-665 (2020).
- Limpikirati, P. K., Zhao, B., Pan, X., Eyles, S. J., Vachet, R. W. Covalent Labeling/Mass Spectrometry of Monoclonal Antibodies with Diethylpyrocarbonate: Reaction Kinetics for Ensuring Protein Structural Integrity. Journal of the American Society of Mass Spectrometry. 31, 1223-1232 (2020).
- Liu, T., Marcinko, T. M., Vachet, R. W. Protein-Ligand Affinity Determinations Using Covalent Labeling-Mass Spectrometry. Journal of the American Society of Mass Spectrometry. 31, 1544-1553 (2020).
- Srikanth, R., Mendoza, V. L., Bridgewater, J. D., Zhang, G., Vachet, R. W. Copper Binding to β-2-Microglobulin and its Pre-Amyloid Oligomers. Biochemistry. 48, 9871-9881 (2009).
- Lim, J., Vachet, R. W. Using mass spectrometry to study copper-protein binding under native and non-native conditions: β-2-microglobulin. Analytical Chemistry. 76, 3498-3504 (2004).
- Lindsley, C. W. Predictions and Statistics for the Best-Selling Drugs Globally and in the United States in 2018 and a Look Forward to 2024 Projections. ACS Chemical Neuroscience. 10, 1115(2019).
- Floege, J., Ketteler, M. β2-Microglobulin-derived amyloidosis: An update. Kidney International. 59, 164(2001).
- Antwi, K., et al. Cu (II) organizes β-2-microglobulin oligomers but is released upon amyloid formation. Protein Science. 17, 748-759 (2008).
- Dong, J., et al. Unique Effect of Cu(II) in the Metal-Induced Amyloid Formation of β-2-Microglobulin. Biochemistry. 53, 1263-1274 (2014).
- Marcinko, T. M., Drews, T., Liu, T., Vachet, R. W. Epigallocatechin-3-gallate Inhibits Cu(II)-Induced β-2-Microglobulin Amyloid Formation by Binding to the Edge of Its β-Sheets. Biochemistry. 59, 1093-1103 (2020).
- Zhou, Y., Vachet, R. W. Diethylpyrocarbonate Labeling for the Structural Analysis of Proteins: Label Scrambling in Solution and How to Avoid it. Journal of the American Society of Mass Spectrometry. 23, 899-907 (2012).
- Borotto, N. B., Degraan-Weber, N., Zhou, Y., Vachet, R. W. Label scrambling during CID of covalently labeled peptide ions. Journal of the American Society of Mass Spectrometry. 25, 1739-1746 (2014).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical Chemistry. 90, 7721-7729 (2018).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Analytical Chemistry. 89, 1459-1468 (2017).
- Zheng, X., Wintrode, P. L., Chance, M. R. Complementary Structural Mass Spectrometry Techniques Reveal Local Dynamics in Functionally Important Regions of a Metastable Serpin. Structure. 16, 38-51 (2008).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by H/D exchange and covalent labeling: The glycerol facilitator. Journal of Molecular Biology. 416, 400-413 (2012).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical Chemistry. 89, 2250-2258 (2017).
- Borotto, N. B., Zhang, Z., Dong, J., Burant, B., Vachet, R. W. Increased β-Sheet Dynamics and D-E Loop Repositioning Are Necessary for Cu(II)-Induced Amyloid Formation by β-2-Microglobulin. Biochemistry. 56, 1095-1104 (2017).
- Shi, L., Liu, T., Gross, M. L., Huang, Y. Recognition of Human IgG1 by Fcγ Receptors: Structural Insights from Hydrogen-Deuterium Exchange and Fast Photochemical Oxidation of Proteins Coupled with Mass Spectrometry. Biochemistry. 58, 1074-1080 (2019).
- Gerega, S. K., Downard, K. M. PROXIMO - A new docking algorithm to model protein complexes using data from radical probe mass spectrometry (RP-MS). Bioinformatics. 22, 1702-1709 (2006).
- Kamal, J. K. A., Chance, M. R. Modeling of protein binary complexes using structural mass spectrometry data. Protein Science. 17, 79-94 (2007).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。