Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Kütle Spektrometresi ile Protein Yüksek Sıralı Yapısını incelemek için Diethylpyrocarbonate ile Kovalent Etiketleme
Bu Makalede
Özet
Kütle spektrometrik tespiti ile dietilptirokarbonat bazlı kovalent etiketlemenin gerçekleştirilmesi için deneysel prosedürler açıklanmıştır. Dietilpyrokarbonat, solvent erişilebilir amino asit kalıntılarının değiştirilmesine yol açan, ilgi çekici protein veya protein kompleksi ile basitçe karıştırılır. Modifiye kalıntılar proteolitik sindirim ve sıvı kromatografisi/kütle spektrometresi analizinden sonra tanımlanabilir.
Özet
Bir proteinin yüksek sıralı yapısını karakterize etmek, işlevini anlamak için gereklidir. Kütle spektrometresi (MS), özellikle geleneksel yöntemlerle incelenmesi zor protein sistemleri için bu amaç için güçlü bir araç olarak ortaya çıkmıştır. Bir proteinin yapısını MS ile incelemek için, bir proteinin yapısal bilgilerini kütlesine kodlayan çözeltide spesifik kimyasal reaksiyonlar gerçekleştirilir. Özellikle etkili bir yaklaşım, solvent erişilebilir amino asit yan zincirlerini kovalent olarak değiştiren reaktifler kullanmaktır. Bu reaksiyonlar, proteolitik sindirim ve tandem kütle spektrometresi ile birleştirildiğinde kalıntı düzeyinde çözünürlükle lokalize edilebilen kütle artışlarına yol açar. Burada, dietilpyrokarbonat (DEPC) kullanımıyla ilişkili protokolleri MS algılaması ile birlikte bir kurdent etiketleme reaktifi olarak açıklıyoruz. DEPC, ortalama proteindeki kalıntıların% 30'una kadar etiketleme yapabilen ve böylece mükemmel yapısal çözünürlük sağlayan son derece elektrofilik bir moleküldür. DEPC, MS ile birlikte β2-mikroglobulin gibi küçük tek etki alanı proteinleri için monoklonal antikorlar gibi büyük çok alanlı proteinlere yapısal bilgi elde etmek için başarıyla kullanılmıştır.
Giriş
Proteinler hemen hemen her fizyolojik süreçte temel biyomoleküllerdir. Proteinlerin gerçekleştirdiği fonksiyonların çeşitliliği, benimsedikleri yapılar ve diğer biyomoleküllerle olan etkileşimleri nedeniyle mümkündür. Protein fonksiyonunu daha derin bir düzeyde anlamak için, bu önemli yapısal özellikleri ve etkileşimleri aydınlatmak için biyokimyasal ve biyofiziksel araçlara ihtiyaç vardır. Geleneksel olarak, X-ışını kristalografisi, kriyojenik elektron mikroskopisi ve nükleer manyetik rezonans (NMR) spektroskopisi protein yapısını ortaya çıkarmak için istenen atom seviyesi detayını sağlamıştır. Bununla birlikte, zayıf kristalleşme davranışı, sınırlı protein mevcudiyeti, aşırı numune heterojenliği veya moleküler ağırlık sınırlamaları nedeniyle çok sayıda protein sistemi bu tekniklerle sorgulanamaz. Sonuç olarak, bu sınırlamaların üstesinden gelen daha yeni analiz yöntemleri ortaya çıkmıştır. Protein yapısal bilgilerini sağlayabilecek gelişen teknikler arasında kütle spektrometresi de yer alıyor.
Kütle spektrometresi (MS) bir molekülün kütle-şarj (m/z) oranını ölçtİğİnden, istenen yapısal bilgilerin proteinin kütlesine kodlanarak protein daha yüksek sıralı yapısal bilgiler elde edilmelidir. Bu bilgileri kodlamak için hidrojen-döteryum değişimi (HDX) 1,2 ,3,4,kimyasal çapraz bağlama (XL)5,6ve kurcalama etiketleme (CL) 7 , 8,9,10dahil olmak üzere çeşitli yaklaşımlar geliştirilmiştir. HDX'te omurga ortası hidrojenler, solvent erişilebilirliğine ve H-bonding kapsamına bağlı oranlarda biraz daha büyük döteryumlarla değiştirilir. HDX'in kapsamı, bu parçaları kütle spektrometresi ile ayırmadan ve ölçmeden önce proteini peptit parçalarına hızla sindirerek veya yukarıdan aşağıya bir deneyde proteini ayırarak lokalize edilebilir. Değişim hızının belirlenmesi protein dinamikleri hakkında daha fazla bilgi sağlar. HDX, geri değişimle ilgili zorluklara ve tekrarlanabilirliği en üst düzeye çıkarmak için özel ekipman ihtiyacına rağmen protein yapısını karakterize etmek için değerli bir araç olduğunu kanıtlamıştır. XL-MS'de proteinler, belirli bir protein içindeki veya iki protein arasındaki bitişik kalıntı yan zincirlerini aktif olarak bağlayan iki fonksiyonel reaktiflerle reaksiyona sokulur. Bunu yaparken XL-MS, protein yapısını karakterize etmek için kullanılabilecek mesafe kısıtlamaları sağlayabilir. Proteinin çapraz bağlı bölgeleri proteolitik sindirim ve ardından sıvı kromatografisi (LC)-MS analizi ile tanımlanabilir. XL-MS, iç hücreler de dahil olmak üzere çeşitli protein komplekslerini incelemek için kullanılan çok yönlü bir araç olsa da, XL ürünlerinin tanımlanması zordur ve özel yazılım gerektirir.
CL-MS son zamanlarda protein yapısını ve etkileşimlerini incelemek için tamamlayıcı ve bazen alternatif MS tabanlı bir araç olarak ortaya çıkmıştır. CL-MS'te, bir protein veya protein kompleksi, solvent maruz kalan yan zincirlerle reaksiyona girebilen tek fonksiyonlu bir reaktif ile birlikte değiştirilir (Şekil 1). Bir protein veya protein kompleksinin modifikasyon kapsamları farklı koşullar altında karşılaştırılarak, konformasyon değişiklikleri, bağlama bölgeleri ve protein-protein arayüzleri ortaya çıkabilir. CL reaksiyonu sonrasında, genellikle tek amino-asit düzeyinde, proteinlerin proteolitik olarak sindirildiği, peptit parçalarının LC ile ayrıldığı ve modifiye edilen bölgelerin tandem MS (MS/MS) kullanılarak tanımladığı tipik aşağıdan yukarıya proteomik iş akışları kullanılarak meydana özgü bilgiler elde edilebilir. Biyokonjuge kimyanın zengin tarihi, CL-MS deneyleri için çok sayıda reaktifi kullanıma sunmuştur. CL reaktifleri iki genel kategoriye ayrılır: (i) spesifik ve (ii) spesifik olmayan. Spesifik reaktifler tek bir fonksiyonel grupla (örneğin, serbest aminler)8,10 ile reaksiyona girilir ve uygulanması kolaydır, ancak sınırlı yapısal bilgi sağlama eğilimindedirler. Spesifik olmayan reaktifler çok çeşitli yan zincirlerle reaksiyona girer, ancak genellikle bu yüksek reaktif türleri üretmek için lazerler veya senkrotron kaynakları gibi özel ekipmanlar gerektirir. Hidroksil radikalleri, çeşitli koşullar altında çok çeşitli proteinleri ve protein komplekslerini incelemek için hidroksil radikal ayak izi (HRF)7,11,12,13 deneylerinde uygulanan, en sık kullanılan spesifik olmayan reaktiftir.
Araştırma grubumuz, CL-MS deneyleri14 , 15 , 16 ,17 , 18 , 19,20 , 21 , 22,23,24,25bağlamında protein yapısını ve etkileşimlerini incelemek için dietilpyrokarbonat (DEPC) adı verilen nispeten spesifik olmayan başka bir reaktifibaşarıylakullanmıştır. DEPC, ortalama proteindeki amino asitlerin% 30'una kadar reaksiyona girerken, belirli etiketleme reaktiflerinin basitliğini sunar (yani, reaksiyonları gerçekleştirmek için özel bir ekipman gerekmez). Son derece elektrofilik bir reaktif olarak DEPC, sistein, histidin, lizin, tirozin, serin ve threonine kalıntılarının N-terminus ve nükleofilik yan zincirleri ile reaksiyona girer. Tipik olarak, bu reaksiyonların tek bir ürünü üretilir ve 72.02 Da'lık bir kütle artışına neden edilir. Bu tek ürün türü, proteinler hidroksil radikalleri ile reaksiyona girdiğinde üretilebilen 55 farklı ürünle tezat oluşturur7. Bu tür basit kimya, etiketli sitelerin tanımlanmasını kolaylaştırır.
Burada, protein yapısını ve etkileşimlerini incelemek için DEPC tabanlı CL-MS kullanımına yönelik protokoller sunuyoruz. Reaktif hazırlama, DEPC-protein reaksiyonları, protein sindirim koşulları, LC-MS ve MS/MS parametreleri ve veri analizi ile ilgili ayrıntılar açıklanmıştır. Ayrıca protein-metal, protein-ligand ve protein-protein etkileşimlerinin yanı sıra ısıtma sırasında yapısal değişikliklere uğrayan proteinlerden örnek sonuçlar sağlayarak DEPC etiketlemesinin faydasını gösteriyoruz.
Protokol
1. Protein ve reaktif hazırlama
NOT: Bu protokol, bir proteini DEPC ile etiketlemek için örnek bir iş akışı içerir. Listelenen bazı koşullar ve reaktif konsantrasyonları tercih proteinine göre değişebilir.
- Tüm reaktif çözümlerini 1,5 mL mikrosantrifüj tüplerde hazırlayın.
- pH 7.4'te 10 mM 3-(N-morfolino)propansülonik asit (MOPS) tamponunda, genellikle onlarca μM aralığında istenen konsantrasyonda bir protein çözeltisi hazırlayın. Alternatif olarak, numune DEPC ile reaktif olacak bir nükleofilik tampon içeriyorsa, 10 mM pH 7.4 MOPS'de bir tampon değişimi mevcut protein çözeltisi hazırlayın. Diğer tamponlar (örneğin fosfat tamponlu salin), nükleofilik fonksiyonel grupları olmadığı sürece de kullanılabilir.
- 6,9 M DEPC'lik stok çözeltisinin 1,45 μL'lik kısmını ACN'nin 98,55 μL'sine pipetle ederek kuru asetonitrilde (ACN) 100 mM DEPC çözeltisi hazırlayın.
NOT: Her proteinle çalışacak bir konsantrasyon kümesi yoktur, ancak optimal konsantrasyonlar His ve Lys kalıntılarının sayısına göre tahmin edilebilir23. Örneğin, 50 μM β2-mikroglobulin çözeltisinin 50 μL'si ile, bu genel örneği kullanarak istenen moler oranını sağlamak için 200 μM 'lik (protein konsantrasyonunun 4x'ine eşit) nihai bir DEPC konsantrasyonu için proteini100mM DEPC'nin 0,2 μL'si ile reaksiyona sokun ( Tablo 1 ). DEPC etiketleme 2nd sipariş reaksiyonudur, bu nedenle reaksiyon karışımındaki protein veya DEPC konsantrasyonunun değiştirilmesi etiketleme oranını değiştirecektir. - 10 mg imidazol tartarak ve 146,9 μL HPLC sınıfı suda çözünerek 1 M imidazol çözeltisi hazırlayın.
2. Bozulmamış proteinin kovalent etiketlemesi
- Su banyosu sıcaklığını 37 °C'ye ayarlayın ve banyonun sabit bir sıcaklığa ulaşmasını bekleyin.
NOT: Örnek bir etiketleme protokolü için reaktif konsantrasyonları ve hacimleri Tablo 1'de bulunabilir. - Yeni bir mikrosantrifüj tüpünde, MOPS tamponu ve protein çözeltisi tablo 1'delistelenen hacimlerde karıştırın.
- Protein ve tampona DEPC çözeltisinin 0,2 μL'lik kısmını ekleyin, elde edilen çözeltiyi düzgün bir şekilde karıştırdığından emin olun ve ardından reaksiyon karışımını içeren tüpü 1 dakika boyunca 37 °C su banyosuna yerleştirin.
NOT: Etiketleme reaksiyonu sırasında proteinin yapısının pertürbasyonunu önlemek için eklenen ACN hacmi toplam reaksiyon hacminin% 1'ini geçmemelidir. Reaksiyon süresi kullanıcıya bağlıdır, ancak örnek koşullar altında 1 dakikalık bir reaksiyon üst üste binmeyi ve DEPC14'ünpotansiyel hidrolizini en aza indirir. - 1 dakika sonra, reaksiyon karışımını içeren tüpü su banyosundan çıkarın ve kalan yayınlanmamış DEPC'yi temizlemek için reaksiyonun 1 M imidazol çözeltisinin 1 μL'si ile söndürülür.
NOT: Reaksiyon karışımındaki son imidazol konsantrasyonu, reaksiyon karışımındaki DEPC konsantrasyonunun 50 katına eşit olmalıdır. Bu, kalan yayınlanmamış DEPC'nin atılmasını sağlar.
3. Aşağıdan yukarıya LC-MS için protein sindiriminin hazırlanması
NOT: İlgi çekici proteine uygun sindirim koşullarını seçin. Yaygın adımlar, proteinin ortaya çıkmasını ve herhangi bir disülfid bağın azaltılmasını ve alkilasyona sokulduğlarını içerir.
- Reaksiyon karışımına uygun bir açma reaktifi ekleyerek proteini açın.
NOT: Yaygın açılma ajanları ACN, üre ve guanidin hidroklorür (GuHCl) içerir. - Azaltma ve alkilasyon adımları için sırasıyla 10 mM pH 7.4 MOPS tamponunun 174.4 ve 270.3 μL'sinde her biri 5 mg tartarak ve yeni mikrosantrifüj tüplerinde çözerek Tris (2-karboksit)fosfin (TCEP) ve iodoasetamid (IAM) çözeltileri hazırlayın.
- Reaksiyon karışımına 100 mM TCEP (reaksiyon karışımında 2 mM'lik son konsantrasyon) çözeltisinin 2 μL'sini ekleyerek ve oda sıcaklığında 3 dakika reaksiyona sokarak disülfid bağları azaltın.
NOT: TCEP'in son konsantrasyonu, çözeltide bulunan disülfid bağ başına protein konsantrasyonunun 40 kat'ına eşit olmalıdır. - Alkilat, karanlıkta 30 dakika boyunca 100 mM IAM çözeltisinin 4 μL'si (reaksiyon karışımında 4 mM'lik son konsantrasyon) ile sisteinleri azalttı. IAM ışığa duyarlıdır ve doğrudan ışık altında ayrışır.
NOT: Çözeltideki IAM'nin son konsantrasyonu, TCEP için kullanılan konsantrasyonun iki katı veya disülfid bağı başına protein konsantrasyonunun 80 katı olmalıdır. - Proteini tripsin veya chymotrypsin gibi uygun bir enzimle sindirin. 37 °C'de 3 saatlik bir sindirim için 10:1 protein:enzim oranı, 300 vuruş/dakika sallama hızında hareketsiz enzim ile tipik olarak DEPC etiketli proteinler için yeterlidir. Bkz. Tartışma.
- Sindirimden sonra, hareketsiz enzimi sindirilmiş peptitlerden 5 dakika boyunca 12.000 rpm'de santrifüjleme ile ayırın.
- Numuneyi LC-MS/MS ile hemen analiz edin veya numune bozulmasını ve etiket kaybını en aza indirmek için numuneyi sıvı nitrojenle flaşla dondurun. Flaşla dondurulmuş numuneleri LC-MS/MS analizine hazır olana kadar -20 °C'< saklayın.
4. LC-MS/MS Analizi
NOT: Aşağıdan yukarıya proteomik için standart LC-MS/MS parametreleri, proteolitik peptit parçalarındaki etiketli bölgeleri tanımlamak için kullanılabilir. Genel bir örnek aşağıda özetlenmiştir.
- TERS fazlı C18 sabit faz kullanarak DEPC etiketli peptitleri ayırın. İki çözücünün tipik bir LC mobil fazını kullanın: peptitlerin en iyi ayrımını elde etmek için bir gradyan (örneğin, Şekil 2) kullanarak su + % 0.1 formik asit ve (B) ACN +% 0.1formik asit.
NOT: Ayırma süresi numune karmaşıklığına göre optimize edilebilir ve mobil faz akış hızı kılcal veya nano LC kullanılıp kullanılmadığına bağlıdır. - Peptit üzerindeki DEPC değişiklik alanlarını tanımlamak için çevrimiçi LC-MS ve MS/MS yapabilen bir kütle spektrometresi kullanın. Deneylerimizde, çeşitli kütle spektrometrelerini başarıyla kullandık. LC-MS analizi sırasında birçok peptitin MS/MS'ini otomatik olarak gerçekleştirebilen herhangi bir kütle spektrometresi uygun olmalıdır. İlgili MS parametreleri şunlardır: Normal ESI için ESI kaynak voltajı = -4000 V; Nanospray için -2000 V; Orbitrap çözünürlüğü = 60.000; Dinamik dışlama süresi = 30 s; MS/MS etkinleştirme türü: CID, ETD veya her ikisi; Toplu tarama aralığı = 200-2.000; Otomatik Kazanç Kontrolü = 4.0E5 (Orbitrap'ta MS1) ve 5.0E4 (doğrusal dörtlü iyon tuzağında MS2).
- Sindirilmiş, etiketlenmiş protein örneğini LC sistemine yükleyip enjekte edin ve LC-MS/MS alımını başlatın. Numune flaşla dondurulduysa, analizden önce çözün. Aşırı tuzların ESI kaynağına girmesini önlemek için LC atık sularını ilk 5 dakika çöpe atın.
NOT: LC-MS/MS'e yaklaşık 2,5 μg protein enjeksiyonu için izin verilen 5 μL enjeksiyon döngüsü genellikle kullanılır. Bu, örnek enjektörü tıkamamak için LC'nin yükleme koşullarına bağlıdır.
5. Veri analizi
- DEPC etiket sitelerini tanımlayın ve kullanılan kütle spektrometresi için uygun yazılımı kullanarak peptit tepe alanlarını ölçün.
- Değişken değişiklikler olarak DEPC ekleme (72.02 Da) ve karbamidometiltion (57.02 Da) ekleyin. MS/MS analizi için ek arama parametreleri aşağıdaki gibidir: En fazla kaçırılan bölünme = 3; Parça iyon türleri = b ve y; Öncül m/z toleransı = 10 ppm (dörtlü iyon bindirme kütle spektrometresi kullanılıyorsa bu değer daha yüksek olmalıdır); Parça m/z toleransı = 0,5 Da (ürün iyon taraması için yüksek çözünürlüklü kütle spektrometresi kullanılıyorsa bu değer daha düşük olmalıdır); Öncül ücret = 1-4.
NOT: Farklı veritabanı arama algoritmaları farklı puanlama sistemlerine sahiptir ve değişiklik düzeyleri düşük olabileceği için birçoğu DEPC tarafından değiştirilmiş peptitleri tanımlamakta zorlanabilir. Daha etiketli peptitleri tanımlamak için puan kesmenin ayarlanması gerekebilir. Öyleyse, düşük puanlı peptitleri doğrulamak için MS/MS verilerinin manuel olarak sorgulanması kullanılmalıdır. DEPC etiketinin hidroliz ürünü veri aramasına dahil değildir, çünkü hidrolizli DEPC artık nükleofilik yan zincirlere karşı reaktif değildir. - Peptitlerin değiştirilmiş ve değiştirilmemiş sürümlerinin kromatografik tepe alanlarını kullanarak kalıntı düzeyinde değişiklik yüzdelerini belirleyin.
NOT: Değiştirilmiş faiz kalıntılarını içeren herhangi bir peptit dikkate alınmalı ve dahil edilen tüm ücret durumları ölçülen tüm örneklerde bulunmalıdır. Farklı iyonlaşma verimliliklerine sahip ve farklı zamanlarda elde edilmesi gereken peptitler, bu değerin belirli bir sitenin değiştirilmesinin mutlak ölçüsü yerine göreceli olmasına neden olur.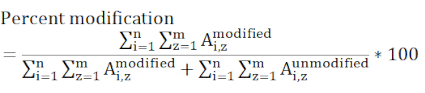
burada Ai,z, ilgi kalıntısını içeren ve algılanan tüm ücret durumlarını (z) dikkate alan herhangi bir peptitin (i) tepe alanını temsil eder. - İstatistiksel değerlendirme kullanarak denetim ve deneysel örnek arasındaki etiketleme değişikliğinin anlamlı olup olmadığını belirleyin. Her örnek için üç çoğaltma ölçümü tipiktir ve t testleri en sık %95 veya %99 güven aralıklarıyla kullanılır.
Sonuçlar
DEPC değişiklik sitelerini ve değişiklik yüzdelerini belirleme
Katvalent etiketlemeye bağlı kütle ilavesi (a) bozulmamış protein ve (b) peptit seviyeleri8,9olarak ölçülebilir. Sağlam düzeyde, etiketli protein örneklerinin doğrudan analizinden veya LC-MS'inden farklı sayıda etikete sahip protein türlerinin dağılımı elde edilebilir. Daha yüksek çözünürlüklü yapısal bilgiler (yani bölgeye özgü etiketleme verileri) elde ...
Tartışmalar
Kritik Adımlar
Güvenilir etiketleme sonuçları sağlamak için deneysel tasarımla ilgili çeşitli noktalar göz önünde bulundurulmalıdır. İlk olarak, protein etiketlemeyi en üst düzeye çıkarmak için, depc ile reaksiyona girebilecekleri ve etiketleme kapsamını düşürebilecekleri için güçlü nükleofilik gruplara (örneğin Tris) sahip tamponlardan kaçınmak gerekir. Bu tür tamponların etiketli kalıntılarla reaksiyona neden olabileceği, etiketin çıkarılmasına ve dolayısıyla yap...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Yazarlar, Grant R01 GM075092 kapsamında Ulusal Sağlık Enstitüleri'nden (NIH) destek kabul ediyorlar. Burada açıklanan verilerin bir kısmını elde etmek için kullanılan Thermo Orbitrap Fusion kütle spektrometresi, Ulusal Sağlık Enstitüleri hibesi S10OD010645'ten alınan fonlarla elde edildi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Thermo Fisher Scientific | 3448 | |
| 3-(N-morpholino)propanesulfonic acid | Millipore Sigma | M1254 | |
| 3-(N-morpholino)propanesulfonic acid sodium salt | Millipore Sigma | M9381 | |
| Acclaim PepMap RSLC C18 Column | Thermo Scientific | 164537 | 300 μm x 15 cm, C18, 2 μm, 100 A |
| Acetonitrile | Fisher Scientific | A998-1 | |
| Diethylpyrocarbonate | Millipore Sigma | D5758 | |
| HPLC-grade water | Fisher Scientific | W5-1 | |
| Imidazole | Millipore Sigma | I5513 | |
| Immobilized chymotrypsin | ProteoChem | g4105 | |
| Immobilized trypsin, TPCK Treated | Thermo Fisher Scientific | 20230 | |
| Iodoacetamide | Millipore Sigma | I1149 | |
| Tris(2-carboxyethyl)phosphine | Millipore Sigma | C4706 |
Referanslar
- Katta, V., Chait, B. T., Carr, S. Conformational Changes in Proteins Probed by Hydrogen-exchange Electrospray-ionization. Rapid Communications in Mass Spectrometry. 5, 214-217 (1991).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25, 158-170 (2006).
- Pirrone, G. F., Iacob, R. E., Engen, J. R. Applications of hydrogen/deuterium exchange MS from 2012 to 2014. Analytical Chemistry. 87, 99-118 (2015).
- Oganesyan, I., Lento, C., Wilson, D. J. Contemporary hydrogen deuterium exchange mass spectrometry. Methods. 144, 27-42 (2018).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrometry Reviews. 25, 663-682 (2006).
- Holding, A. N. XL-MS: Protein cross-linking coupled with mass spectrometry. Methods. 89, 54-63 (2015).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107, 3514-3543 (2007).
- Mendoza, V. L., Vachet, R. W. Probing Protein Structure by Amino Acid-Specific Covalent Labeling and Mass Spectrometry. Mass Spectrometry Reviews. 28, 785-815 (2009).
- Limpikirati, P., Liu, T., Vachet, R. W. Covalent labeling-mass spectrometry with non-specific reagents for studying protein structure and interactions. Methods. 144, 79-93 (2018).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemistry Reviews. 120, 4335 (2020).
- Maleknia, S. D., Brenowitz, M., Chance, M. R. Millisecond radiolytic modification of peptides by synchrotron X-rays identified by mass spectrometry. Analytical Chemistry. 71, 3965-3973 (1999).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77, 5814-5822 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society of Mass Spectrometry. 16, 2057-2063 (2005).
- Mendoza, V. L., Vachet, R. W. Protein surface mapping using diethylpyrocarbonate with mass spectrometric detection. Analytical Chemistry. 80, 2895-2904 (2008).
- Mendoza, V. L., Antwi, K., Barón-rodríguez, M. A., Blanco, C., Vachet, R. W. Structure of the Pre-amyloid Dimer of β-2-microglobulin from Covalent Labeling and Mass Spectrometry. Biochemistry. 49, 1522-1532 (2010).
- Mendoza, V. L., Barón-Rodríguez, M. A., Blanco, C., Vachet, R. W. Structural insights into the pre-amyloid tetramer of β-2-microglobulin from covalent labeling and mass spectrometry. Biochemistry. 50, 6711-6722 (2011).
- Zhou, Y., Vachet, R. W. Increased protein structural resolution from diethylpyrocarbonate-based covalent labeling and mass spectrometric detection. Journal of the American Society of Mass Spectrometry. 23, 708-717 (2012).
- Borotto, N. B., et al. Investigating Therapeutic Protein Structure with Diethylpyrocarbonate Labeling and Mass Spectrometry. Analytical Chemistry. 87, 10627-10634 (2015).
- Liu, T., Marcinko, T. M., Kiefer, P. A., Vachet, R. W. Using Covalent Labeling and Mass Spectrometry To Study Protein Binding Sites of Amyloid Inhibiting Molecules. Analytical Chemistry. 89, 11583-11591 (2017).
- Limpikirati, P., et al. Covalent labeling and mass spectrometry reveal subtle higher order structural changes for antibody therapeutics. MAbs. 11, 463-476 (2019).
- Limpikirati, P., Pan, X., Vachet, R. W. Covalent Labeling with Diethylpyrocarbonate: Sensitive to the Residue Microenvironment, Providing Improved Analysis of Protein Higher Order Structure by Mass Spectrometry. Analytical Chemistry. 91, 8516-8523 (2019).
- Liu, T., Limpikirati, P., Vachet, R. W. Synergistic Structural Information from Covalent Labeling and Hydrogen-Deuterium Exchange Mass Spectrometry for Protein-Ligand Interactions. Analytical Chemistry. 91, 15248-15254 (2019).
- Pan, X., Limpikirati, P., Chen, H., Liu, T., Vachet, R. W. Higher-Order Structure Influences the Kinetics of Diethylpyrocarbonate Covalent Labeling of Proteins. Journal of the American Society of Mass Spectrometry. 31, 658-665 (2020).
- Limpikirati, P. K., Zhao, B., Pan, X., Eyles, S. J., Vachet, R. W. Covalent Labeling/Mass Spectrometry of Monoclonal Antibodies with Diethylpyrocarbonate: Reaction Kinetics for Ensuring Protein Structural Integrity. Journal of the American Society of Mass Spectrometry. 31, 1223-1232 (2020).
- Liu, T., Marcinko, T. M., Vachet, R. W. Protein-Ligand Affinity Determinations Using Covalent Labeling-Mass Spectrometry. Journal of the American Society of Mass Spectrometry. 31, 1544-1553 (2020).
- Srikanth, R., Mendoza, V. L., Bridgewater, J. D., Zhang, G., Vachet, R. W. Copper Binding to β-2-Microglobulin and its Pre-Amyloid Oligomers. Biochemistry. 48, 9871-9881 (2009).
- Lim, J., Vachet, R. W. Using mass spectrometry to study copper-protein binding under native and non-native conditions: β-2-microglobulin. Analytical Chemistry. 76, 3498-3504 (2004).
- Lindsley, C. W. Predictions and Statistics for the Best-Selling Drugs Globally and in the United States in 2018 and a Look Forward to 2024 Projections. ACS Chemical Neuroscience. 10, 1115 (2019).
- Floege, J., Ketteler, M. β2-Microglobulin-derived amyloidosis: An update. Kidney International. 59, 164 (2001).
- Antwi, K., et al. Cu (II) organizes β-2-microglobulin oligomers but is released upon amyloid formation. Protein Science. 17, 748-759 (2008).
- Dong, J., et al. Unique Effect of Cu(II) in the Metal-Induced Amyloid Formation of β-2-Microglobulin. Biochemistry. 53, 1263-1274 (2014).
- Marcinko, T. M., Drews, T., Liu, T., Vachet, R. W. Epigallocatechin-3-gallate Inhibits Cu(II)-Induced β-2-Microglobulin Amyloid Formation by Binding to the Edge of Its β-Sheets. Biochemistry. 59, 1093-1103 (2020).
- Zhou, Y., Vachet, R. W. Diethylpyrocarbonate Labeling for the Structural Analysis of Proteins: Label Scrambling in Solution and How to Avoid it. Journal of the American Society of Mass Spectrometry. 23, 899-907 (2012).
- Borotto, N. B., Degraan-Weber, N., Zhou, Y., Vachet, R. W. Label scrambling during CID of covalently labeled peptide ions. Journal of the American Society of Mass Spectrometry. 25, 1739-1746 (2014).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical Chemistry. 90, 7721-7729 (2018).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Analytical Chemistry. 89, 1459-1468 (2017).
- Zheng, X., Wintrode, P. L., Chance, M. R. Complementary Structural Mass Spectrometry Techniques Reveal Local Dynamics in Functionally Important Regions of a Metastable Serpin. Structure. 16, 38-51 (2008).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by H/D exchange and covalent labeling: The glycerol facilitator. Journal of Molecular Biology. 416, 400-413 (2012).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical Chemistry. 89, 2250-2258 (2017).
- Borotto, N. B., Zhang, Z., Dong, J., Burant, B., Vachet, R. W. Increased β-Sheet Dynamics and D-E Loop Repositioning Are Necessary for Cu(II)-Induced Amyloid Formation by β-2-Microglobulin. Biochemistry. 56, 1095-1104 (2017).
- Shi, L., Liu, T., Gross, M. L., Huang, Y. Recognition of Human IgG1 by Fcγ Receptors: Structural Insights from Hydrogen-Deuterium Exchange and Fast Photochemical Oxidation of Proteins Coupled with Mass Spectrometry. Biochemistry. 58, 1074-1080 (2019).
- Gerega, S. K., Downard, K. M. PROXIMO - A new docking algorithm to model protein complexes using data from radical probe mass spectrometry (RP-MS). Bioinformatics. 22, 1702-1709 (2006).
- Kamal, J. K. A., Chance, M. R. Modeling of protein binary complexes using structural mass spectrometry data. Protein Science. 17, 79-94 (2007).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır