É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Rotulagem covalente com dietilpirocarbonato para estudar estrutura de alta ordem de proteína por espectrometria de massa
Neste Artigo
Resumo
Os procedimentos experimentais para a realização de rotulagem covalente à base de diethylpirocarbonato com detecção espectrométrica em massa são descritos. O dietilpirocarbonato é simplesmente misturado com o complexo de proteínas ou proteínas de interesse, levando à modificação de resíduos de aminoácidos acessíveis solventes. Os resíduos modificados podem ser identificados após a digestão proteolítica e a análise da cromatografia líquida/espectrometria de massa.
Resumo
Caracterizar a estrutura de alta ordem de uma proteína é essencial para entender sua função. A espectrometria de massa (MS) emergiu como uma ferramenta poderosa para este fim, especialmente para sistemas proteicos difíceis de estudar pelos métodos tradicionais. Para estudar a estrutura de uma proteína por MS, reações químicas específicas são realizadas em solução que codifica as informações estruturais de uma proteína em sua massa. Uma abordagem particularmente eficaz é o uso de reagentes que modificam covalentemente cadeias laterais de aminoácidos acessíveis de solvente. Essas reações levam a aumentos de massa que podem ser localizados com resolução de nível de resíduo quando combinados com digestão proteolítica e espectrometria de massa tandem. Aqui, descrevemos os protocolos associados ao uso de diethylpirocarbonato (DEPC) como um reagente de rotulagem covalente juntamente com a detecção de MS. O DEPC é uma molécula altamente eletrofílica capaz de rotular até 30% dos resíduos na proteína média, proporcionando assim excelente resolução estrutural. O DEPC tem sido usado com sucesso junto com a MS para obter informações estruturais para pequenas proteínas de domínio único, como β2-microglobulina, para grandes proteínas multi-domínio, como anticorpos monoclonais.
Introdução
Proteínas são biomoléculas essenciais em praticamente todos os processos fisiológicos. A variedade de funções que as proteínas realizam são possíveis devido às estruturas que adotam e às interações que têm com outras biomoléculas. Para entender a função proteica em um nível mais profundo, ferramentas bioquímicas e biofísicas são necessárias para elucidar essas importantes características e interações estruturais. Tradicionalmente, a cristalografia de raios-X, a microscopia eletrônica criogênica e a espectroscopia de ressonância magnética nuclear (RMN) forneceram o detalhe desejado do nível atômico para revelar a estrutura proteica. No entanto, inúmeros sistemas proteicos não podem ser interrogados por essas técnicas devido ao mau comportamento de cristalização, disponibilidade limitada de proteínas, heterogeneidade excessiva da amostra ou limitações de peso molecular. Consequentemente, surgiram novos métodos de análise que superam essas limitações. Entre as técnicas emergentes que podem fornecer informações estruturais proteicas está a espectrometria de massa.
A espectrometria de massa (MS) mede a relação massa-carga (m/z) de uma molécula, de modo que informações estruturais de alta ordem proteica devem ser obtidas codificando as informações estruturais desejadas na massa da proteína. Várias abordagens para codificar essas informações foram desenvolvidas, incluindo troca de deutério de hidrogênio (HDX)1,2,3,4, crosslinking químico (XL)5,6, e rotulagem covalente (CL)7,8,9,10. Em HDX, os hidrogênios de amido de espinha dorsal são trocados por deutérios ligeiramente mais massivos a taxas que dependem da acessibilidade solvente e da extensão de ligação H. A extensão do HDX pode ser localizada digerindo rapidamente a proteína em fragmentos de peptídeos antes de separar e medir esses fragmentos pelo espectrômetro de massa ou dissolvendo a proteína em um experimento de cima para baixo. Determinar a taxa de câmbio fornece mais informações sobre a dinâmica das proteínas. A HDX provou ser uma ferramenta valiosa para caracterizar a estrutura proteica, apesar dos desafios associados à troca traseira e da necessidade de equipamentos especializados para maximizar a reprodutibilidade. No XL-MS, as proteínas são reagidas com reagentes bifuncionais que ligam covalentemente cadeias laterais de resíduos dentro de uma determinada proteína ou entre duas proteínas. Ao fazer isso, o XL-MS pode fornecer restrições de distância que podem ser usadas para caracterizar a estrutura proteica. As regiões da proteína que estão cruzadas podem ser identificadas pela digestão proteolítica seguida de cromatografia líquida (LC)-MS. Embora o XL-MS seja uma ferramenta versátil que tem sido usada para estudar uma variedade de complexos proteicos, incluindo células internas, a identificação dos produtos XL é desafiadora e requer software especializado.
O CL-MS emergiu recentemente como uma ferramenta complementar e às vezes alternativa baseada em MS para estudar a estrutura e as interações proteicas. Em CL-MS, um complexo proteico ou proteico é covalentemente modificado com um reagente monofuncional que pode reagir com cadeias laterais expostas a solventes(Figura 1). Comparando as extensões de modificação de um complexo proteico ou proteico em diferentes condições, mudanças de conformação, locais de ligação e interfaces proteína-proteína podem ser reveladas. Após a reação cl, informações específicas do local, muitas vezes no nível único de aminoácidos, podem ser obtidas usando fluxos de trabalho típicos de proteômica de baixo para cima em que as proteínas são digeridas proteolíticos, fragmentos de peptídeos são separados por LC e locais modificados são identificados usando ms tandem (MS/MS). A rica história da química bioconjugada disponibilizou numerosos reagentes para experimentos cl-ms. Os reagentes CL se enquadram em duas categorias gerais: (i) específicas e (ii) não específicas. Reagentes específicos reagem com um único grupo funcional (por exemplo, aminas livres)8,10 e são fáceis de implementar, mas tendem a fornecer informações estruturais limitadas. Reagentes não específicos reagem com uma ampla gama de cadeias laterais, mas muitas vezes requerem equipamentos especializados, como lasers ou fontes síncrotrons para produzir essas espécies altamente reativas. Os radicais hidroxil são os reagentes não específicos mais utilizados, tendo sido aplicados em pegada radical hidroxil (HRF)7,11,12,13 experimentos para estudar uma ampla gama de proteínas e complexos proteicos sob uma variedade de condições.
Nosso grupo de pesquisa utilizou com sucesso outro reagente relativamente não específico chamado dietilpiroto (DEPC) para estudar estrutura proteica e interações no contexto dos experimentos cl-ms14,15,16,17,18,19,20,21,22,23,24,25. O DEPC oferece a simplicidade de reagentes de rotulagem específicos (ou seja, nenhum equipamento especializado é necessário para realizar as reações), ao mesmo tempo em que reage com até 30% dos aminoácidos na proteína média. Como um reagente altamente eletrofílico, o DEPC é capaz de reagir com os resíduos n-terminus e as cadeias laterais nucleofílicas de cisteína, histidina, lise, tyrosina, serina e resíduos de threonina. Normalmente, um único produto dessas reações é gerado, resultando em um aumento em massa de 72,02 Da. Este único tipo de produto contrasta com os até 55 produtos diferentes que podem ser produzidos quando as proteínas reagem com radicais hidroxil7. Tal química simples facilita a identificação de locais rotulados.
Aqui, fornecemos protocolos para o uso do CL-MS baseado em DEPC para estudar estrutura e interações proteicas. Detalhes associados à preparação de reagentes, reações de proteínas DEPC, condições de digestão proteica, parâmetros LC-MS e MS/MS e análise de dados são descritos. Também demonstramos a utilidade da rotulagem DEPC, fornecendo exemplos de interações proteína-metal, proteína-ligante e proteína-proteína, bem como proteínas submetidas a mudanças estruturais após o aquecimento.
Protocolo
1. Preparação de proteínas e reagentes
NOTA: Este protocolo inclui um fluxo de trabalho de exemplo para rotular uma proteína com DEPC. Algumas condições e concentrações de reagentes listadas podem variar de acordo com a proteína escolhida.
- Prepare todas as soluções de reagente em tubos de microcentrifus de 1,5 mL.
- Prepare uma solução proteica de concentração desejada, geralmente na faixa de dezenas de μM, em um tampão de ácido propanesulfônico de 10 mM 3-(N-morpholino)em pH 7.4. Alternativamente, prepare uma solução proteica existente de troca de buffer em 10 mM pH 7.4 MOPS se a amostra contiver um tampão nucleofílico que seria reativo com DEPC. Outros buffers (por exemplo, soro fisiológico tampão fosfato) também podem ser usados, desde que não tenham grupos funcionais nucleofílicos.
- Prepare uma solução DEPC de 100 mM em acetonitrilo seco (ACN) pipetando 1,45 μL da solução de 6,9 M DEPC em 98,55 μL da ACN.
NOTA: Não há um conjunto de concentrações que funcionem com todas as proteínas, embora as concentrações ideais possam ser estimadas com base no número de resíduos dele e lys23. Por exemplo, com 50 μL de uma solução de 50 μM β2-microglobulina, reaja a proteína com 0,2 μL do DEPC de 100 mM para uma concentração final de DEPC de 200 μM (igual a 4x a concentração de proteína) para garantir a razão molar desejada usando este exemplo geral(Tabela 1). A rotulagem DEPC é uma reaçãode 2ª ordem, portanto, mudar a concentração de proteína ou DEPC na mistura de reação mudará a taxa de rotulagem. - Prepare a solução de imidazol de 1 M, pesando 10 mg de imidazol e dissolvendo-se em 146,9 μL de água de grau HPLC.
2. Rotulagem covalente de proteína intacta
- Coloque a temperatura do banho de água para 37 °C e espere o banho atingir uma temperatura estável.
NOTA: Concentrações e volumes de reagentes para um protocolo de rotulagem de exemplo podem ser encontrados na Tabela 1. - Em um novo tubo de microcentrifuuge, misture o tampão MOPS e a solução de proteínas em volumes listados na Tabela 1.
- À proteína e ao tampão adicione 0,2 μL da solução DEPC, certificando-se de misturar adequadamente a solução resultante e, em seguida, coloque o tubo contendo a mistura de reação no banho de água de 37 °C por 1 minuto.
NOTA: O volume adicionado pela ACN não deve exceder 1% do volume total de reação para evitar perturbação da estrutura da proteína durante a reação de rotulagem. O tempo de reação cabe ao usuário, embora uma reação de 1 minuto sob as condições de exemplo minimize o sobrelabelamento e a potencial hidrólise do DEPC14. - Após 1 minuto, remova o tubo contendo a mistura de reação do banho de água e sacie a reação com 1 μL da solução de imidazol de 1 M para ressarcê-lo o DEPC não redigido restante.
NOTA: A concentração final de imidazol na mistura de reação deve ser igual a 50x da concentração de DEPC na mistura de reação. Isso garantirá que o DEPC não redigido seja redigido.
3. Preparação de digestão de proteína para LC-MS de baixo para cima
NOTA: Escolha condições de digestão que sejam favoráveis à proteína de interesse. As etapas comuns envolvem o desdobramento da proteína e a redução e aprisão de quaisquer ligações de dissulfeto.
- Desdobre a proteína adicionando um reagente desdobrado apropriado à mistura de reação.
NOTA: Os agentes comuns incluem acn, ureia e cloridrato de guanidina (GuHCl). - Prepare soluções de Tris(2-carboxyethyl)fospfina (TCEP) e iodoacetamida (IAM) pesando 5 mg de cada e dissolvendo-as em novos tubos de microcentrifuuge em 174,4 e 270,3 μL de 10 mM pH 7,4 mops tampão, respectivamente, para a redução e 270,3 μL de 10 mM pH 7,4 mops tampão, respectivamente, para a redução e a alquilação.
- Reduza as ligações de dissulfeto adicionando 2 μL da solução de 100 mM TCEP (concentração final de 2 mM na mistura de reação) à mistura de reação e reagindo por 3 minutos à temperatura ambiente.
NOTA: A concentração final de TCEP deve ser igual a 40x da concentração proteica por ligação dissulfeto presente na solução. - Os cisteínas reduzidas de alquila com 4 μL da solução IAM de 100 mM (concentração final de 4 mM na mistura de reação) por 30 minutos no escuro. O IAM é sensível à luz e se decompõe sob luz direta.
NOTA: A concentração final do IAM na solução deve ser o dobro da concentração utilizada para TCEP, ou 80x a concentração de proteína por ligação de dissulfeto. - Digerir a proteína com uma enzima apropriada, como trippsina ou chymotripsina. Uma razão de proteína de 10:1:enzima para uma digestão de 3 horas a 37 °C com enzima imobilizada a uma taxa de agitação de 300 golpes/min é tipicamente suficiente para proteínas rotuladas pelo DEPC. Veja discussão.
- Após a digestão, separe a enzima imobilizada dos peptídeos digeridos por centrifugação a 12.000 rpm por 5 minutos.
- Analise a amostra imediatamente por LC-MS/MS ou congele flash a amostra com nitrogênio líquido para minimizar a degradação da amostra e a perda do rótulo. Armazene as amostras congeladas a < -20 °C até estar pronta para análise de LC-MS/MS.
4. Análise LC-MS/MS
NOTA: Os parâmetros padrão LC-MS/MS para proteômica de baixo para cima podem ser usados para identificar locais rotulados nos fragmentos de peptídeos protelíticos. Um exemplo geral é descrito abaixo.
- Separe os peptídeos rotulados pelo DEPC usando uma fase inversa C18 estacionária. Use uma fase móvel LC típica de dois solventes: (A) água + 0,1% ácido fórmico e (B) ACN + 0,1% ácido fórmico usando um gradiente (por exemplo, Figura 2) para alcançar a melhor separação dos peptídeos.
NOTA: O tempo de separação pode ser otimizado com base na complexidade da amostra, e a taxa de fluxo de fase móvel depende se o capilar ou nano LC é usado. - Use um espectrômetro de massa capaz de fazer LC-MS e MS/MS on-line para identificar os locais de modificação do DEPC no peptídeo. Em nossos experimentos, usamos com sucesso vários tipos de espectrômetros de massa. Qualquer espectrômetro de massa capaz de realizar automaticamente MS/MS de muitos peptídeos durante uma análise LC-MS deve ser adequado. Os parâmetros de MS relevantes incluem: tensão de origem ESI = -4000 V para ESI regular; -2000 V para nanospray; Resolução Orbitrap = 60.000; Duração de exclusão dinâmica = 30 s; Tipo de ativação MS/MS: CID, ETD ou ambos; Faixa de varredura em massa = 200-2.000; Controle automático de ganho = 4.0E5 (MS1 no Orbitrap) e 5.0E4 (MS2 na armadilha de íon quadrupole linear).
- Carregue e injete a amostra de proteína digerida e rotulada no sistema LC e inicie a aquisição da LC-MS/MS. Se a amostra tiver sido congelada, descongele antes da análise. Desvie o efluente LC para o lixo durante os primeiros 5 minutos para evitar que sais excessivos entrem na fonte ESI.
NOTA: Um laço de injeção de 5 μL é geralmente utilizado, permitindo a injeção de aproximadamente 2,5 μg de proteína para o LC-MS/MS. Isso depende das condições de carregamento da LC para não entupir o injetor amostral.
5. Análise de dados
- Identifique os sites de etiquetas DEPC e quantifique as áreas de pico do peptídeo usando um software apropriado para o espectrômetro de massa que é usado.
- Inclua adição de DEPC (72.02 Da) e carbamidometilação (57,02 Da) como modificações variáveis. Os parâmetros adicionais de pesquisa para a análise de MS/MS são os seguintes: Decotes máximos perdidos = 3; Tipos de íons de fragmentação = b e y; Tolerância precursora m/z = 10 ppm (este valor deve ser maior se um espectrômetro de massa de armadilha de íon quadrupole for usado); Tolerância de fragmento m/z = 0,5 Da (este valor deve ser menor se um espectrômetro de massa de alta resolução for usado para uma varredura de íons do produto); Carga precursora = 1-4.
NOTA: Diferentes algoritmos de pesquisa de banco de dados têm sistemas de pontuação diferentes, e muitos podem ter dificuldade em identificar peptídeos modificados pelo DEPC porque os níveis de modificação podem ser baixos. O ajuste do corte de pontuação pode ser necessário para identificar peptídeos mais rotulados. Nesse caso, o interrogatório manual dos dados de MS/MS deve ser usado para verificar peptídeos de baixa pontuação. O produto da hidrólise do selo DEPC não está incluído na pesquisa de dados porque o DEPC hidrolisado não é mais reativo em relação às cadeias laterais nucleofílicas. - Determine percentuais de modificação em nível de resíduo utilizando as áreas de pico cromatográficas das versões modificadas e não modificadas dos peptídeos.
NOTA: Qualquer peptídeo contendo o resíduo modificado de interesse deve ser considerado e todos os estados de carga que estão incluídos devem estar presentes em todas as amostras medidas. Peptídeos com diferentes eficiências de ionização e eluição em diferentes momentos faz com que esse valor seja uma medida relativa e não absoluta da modificação de um local específico.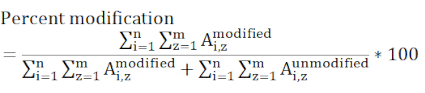
onde Ai,z representa área de pico de qualquer peptídeo (i) que contenha o resíduo de interesse e considere todos os estados de carga detectados (z). - Determinar se uma mudança de rotulagem entre uma amostra de controle e experimental é significativa por meio da avaliação estatística. Três medidas de replicação para cada amostra são típicas, e os testes t são mais comumente utilizados com intervalos de confiança de 95 ou 99%.
Resultados
Identificação de locais de modificação do DEPC e percentagens de modificação
A adição em massa devido à rotulagem covalente pode ser medida nos (a) níveis de proteína intacta e (b) peptídeo8,9. No nível intacto, uma distribuição de espécies proteicas com diferentes números de rótulos pode ser obtida a partir de análise direta ou LC-MS de amostras de proteínas rotuladas. Para obter informações estruturais de maior resolução (ou ...
Discussão
Passos críticos
Vários pontos relativos ao design experimental devem ser considerados para garantir resultados confiáveis de rotulagem. Primeiro, para maximizar a rotulagem de proteínas, é necessário evitar buffers com grupos fortemente nucleofílicos (por exemplo, Tris) porque eles podem reagir com o DEPC e diminuir a extensão da rotulagem. Também é concebível que tais tampões possam reagir com resíduos rotulados, causando a remoção do rótulo e, portanto, a perda de informações estruturais. ...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores reconhecem o apoio dos Institutos Nacionais de Saúde (NIH) sob o Grant R01 GM075092. O espectrômetro de massa Thermo Orbitrap Fusion usado para adquirir alguns dos dados aqui descritos foi adquirido com recursos do Instituto Nacional de Saúde s10OD010645.
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Thermo Fisher Scientific | 3448 | |
| 3-(N-morpholino)propanesulfonic acid | Millipore Sigma | M1254 | |
| 3-(N-morpholino)propanesulfonic acid sodium salt | Millipore Sigma | M9381 | |
| Acclaim PepMap RSLC C18 Column | Thermo Scientific | 164537 | 300 μm x 15 cm, C18, 2 μm, 100 A |
| Acetonitrile | Fisher Scientific | A998-1 | |
| Diethylpyrocarbonate | Millipore Sigma | D5758 | |
| HPLC-grade water | Fisher Scientific | W5-1 | |
| Imidazole | Millipore Sigma | I5513 | |
| Immobilized chymotrypsin | ProteoChem | g4105 | |
| Immobilized trypsin, TPCK Treated | Thermo Fisher Scientific | 20230 | |
| Iodoacetamide | Millipore Sigma | I1149 | |
| Tris(2-carboxyethyl)phosphine | Millipore Sigma | C4706 |
Referências
- Katta, V., Chait, B. T., Carr, S. Conformational Changes in Proteins Probed by Hydrogen-exchange Electrospray-ionization. Rapid Communications in Mass Spectrometry. 5, 214-217 (1991).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25, 158-170 (2006).
- Pirrone, G. F., Iacob, R. E., Engen, J. R. Applications of hydrogen/deuterium exchange MS from 2012 to 2014. Analytical Chemistry. 87, 99-118 (2015).
- Oganesyan, I., Lento, C., Wilson, D. J. Contemporary hydrogen deuterium exchange mass spectrometry. Methods. 144, 27-42 (2018).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrometry Reviews. 25, 663-682 (2006).
- Holding, A. N. XL-MS: Protein cross-linking coupled with mass spectrometry. Methods. 89, 54-63 (2015).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107, 3514-3543 (2007).
- Mendoza, V. L., Vachet, R. W. Probing Protein Structure by Amino Acid-Specific Covalent Labeling and Mass Spectrometry. Mass Spectrometry Reviews. 28, 785-815 (2009).
- Limpikirati, P., Liu, T., Vachet, R. W. Covalent labeling-mass spectrometry with non-specific reagents for studying protein structure and interactions. Methods. 144, 79-93 (2018).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemistry Reviews. 120, 4335 (2020).
- Maleknia, S. D., Brenowitz, M., Chance, M. R. Millisecond radiolytic modification of peptides by synchrotron X-rays identified by mass spectrometry. Analytical Chemistry. 71, 3965-3973 (1999).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77, 5814-5822 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society of Mass Spectrometry. 16, 2057-2063 (2005).
- Mendoza, V. L., Vachet, R. W. Protein surface mapping using diethylpyrocarbonate with mass spectrometric detection. Analytical Chemistry. 80, 2895-2904 (2008).
- Mendoza, V. L., Antwi, K., Barón-rodríguez, M. A., Blanco, C., Vachet, R. W. Structure of the Pre-amyloid Dimer of β-2-microglobulin from Covalent Labeling and Mass Spectrometry. Biochemistry. 49, 1522-1532 (2010).
- Mendoza, V. L., Barón-Rodríguez, M. A., Blanco, C., Vachet, R. W. Structural insights into the pre-amyloid tetramer of β-2-microglobulin from covalent labeling and mass spectrometry. Biochemistry. 50, 6711-6722 (2011).
- Zhou, Y., Vachet, R. W. Increased protein structural resolution from diethylpyrocarbonate-based covalent labeling and mass spectrometric detection. Journal of the American Society of Mass Spectrometry. 23, 708-717 (2012).
- Borotto, N. B., et al. Investigating Therapeutic Protein Structure with Diethylpyrocarbonate Labeling and Mass Spectrometry. Analytical Chemistry. 87, 10627-10634 (2015).
- Liu, T., Marcinko, T. M., Kiefer, P. A., Vachet, R. W. Using Covalent Labeling and Mass Spectrometry To Study Protein Binding Sites of Amyloid Inhibiting Molecules. Analytical Chemistry. 89, 11583-11591 (2017).
- Limpikirati, P., et al. Covalent labeling and mass spectrometry reveal subtle higher order structural changes for antibody therapeutics. MAbs. 11, 463-476 (2019).
- Limpikirati, P., Pan, X., Vachet, R. W. Covalent Labeling with Diethylpyrocarbonate: Sensitive to the Residue Microenvironment, Providing Improved Analysis of Protein Higher Order Structure by Mass Spectrometry. Analytical Chemistry. 91, 8516-8523 (2019).
- Liu, T., Limpikirati, P., Vachet, R. W. Synergistic Structural Information from Covalent Labeling and Hydrogen-Deuterium Exchange Mass Spectrometry for Protein-Ligand Interactions. Analytical Chemistry. 91, 15248-15254 (2019).
- Pan, X., Limpikirati, P., Chen, H., Liu, T., Vachet, R. W. Higher-Order Structure Influences the Kinetics of Diethylpyrocarbonate Covalent Labeling of Proteins. Journal of the American Society of Mass Spectrometry. 31, 658-665 (2020).
- Limpikirati, P. K., Zhao, B., Pan, X., Eyles, S. J., Vachet, R. W. Covalent Labeling/Mass Spectrometry of Monoclonal Antibodies with Diethylpyrocarbonate: Reaction Kinetics for Ensuring Protein Structural Integrity. Journal of the American Society of Mass Spectrometry. 31, 1223-1232 (2020).
- Liu, T., Marcinko, T. M., Vachet, R. W. Protein-Ligand Affinity Determinations Using Covalent Labeling-Mass Spectrometry. Journal of the American Society of Mass Spectrometry. 31, 1544-1553 (2020).
- Srikanth, R., Mendoza, V. L., Bridgewater, J. D., Zhang, G., Vachet, R. W. Copper Binding to β-2-Microglobulin and its Pre-Amyloid Oligomers. Biochemistry. 48, 9871-9881 (2009).
- Lim, J., Vachet, R. W. Using mass spectrometry to study copper-protein binding under native and non-native conditions: β-2-microglobulin. Analytical Chemistry. 76, 3498-3504 (2004).
- Lindsley, C. W. Predictions and Statistics for the Best-Selling Drugs Globally and in the United States in 2018 and a Look Forward to 2024 Projections. ACS Chemical Neuroscience. 10, 1115 (2019).
- Floege, J., Ketteler, M. β2-Microglobulin-derived amyloidosis: An update. Kidney International. 59, 164 (2001).
- Antwi, K., et al. Cu (II) organizes β-2-microglobulin oligomers but is released upon amyloid formation. Protein Science. 17, 748-759 (2008).
- Dong, J., et al. Unique Effect of Cu(II) in the Metal-Induced Amyloid Formation of β-2-Microglobulin. Biochemistry. 53, 1263-1274 (2014).
- Marcinko, T. M., Drews, T., Liu, T., Vachet, R. W. Epigallocatechin-3-gallate Inhibits Cu(II)-Induced β-2-Microglobulin Amyloid Formation by Binding to the Edge of Its β-Sheets. Biochemistry. 59, 1093-1103 (2020).
- Zhou, Y., Vachet, R. W. Diethylpyrocarbonate Labeling for the Structural Analysis of Proteins: Label Scrambling in Solution and How to Avoid it. Journal of the American Society of Mass Spectrometry. 23, 899-907 (2012).
- Borotto, N. B., Degraan-Weber, N., Zhou, Y., Vachet, R. W. Label scrambling during CID of covalently labeled peptide ions. Journal of the American Society of Mass Spectrometry. 25, 1739-1746 (2014).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical Chemistry. 90, 7721-7729 (2018).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Analytical Chemistry. 89, 1459-1468 (2017).
- Zheng, X., Wintrode, P. L., Chance, M. R. Complementary Structural Mass Spectrometry Techniques Reveal Local Dynamics in Functionally Important Regions of a Metastable Serpin. Structure. 16, 38-51 (2008).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by H/D exchange and covalent labeling: The glycerol facilitator. Journal of Molecular Biology. 416, 400-413 (2012).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical Chemistry. 89, 2250-2258 (2017).
- Borotto, N. B., Zhang, Z., Dong, J., Burant, B., Vachet, R. W. Increased β-Sheet Dynamics and D-E Loop Repositioning Are Necessary for Cu(II)-Induced Amyloid Formation by β-2-Microglobulin. Biochemistry. 56, 1095-1104 (2017).
- Shi, L., Liu, T., Gross, M. L., Huang, Y. Recognition of Human IgG1 by Fcγ Receptors: Structural Insights from Hydrogen-Deuterium Exchange and Fast Photochemical Oxidation of Proteins Coupled with Mass Spectrometry. Biochemistry. 58, 1074-1080 (2019).
- Gerega, S. K., Downard, K. M. PROXIMO - A new docking algorithm to model protein complexes using data from radical probe mass spectrometry (RP-MS). Bioinformatics. 22, 1702-1709 (2006).
- Kamal, J. K. A., Chance, M. R. Modeling of protein binary complexes using structural mass spectrometry data. Protein Science. 17, 79-94 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados