Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Marquage covalent avec diéthylpyrocarbonate pour l’étude de la structure d’ordre supérieur des protéines par spectrométrie de masse
Dans cet article
Résumé
Les procédures expérimentales pour effectuer un marquage covalent à base de diéthylpyrocarbonate avec détection spectrométrique de masse sont décrites. Le diéthylpyrocarbonate est simplement mélangé avec la protéine ou le complexe protéique d’intérêt, conduisant à la modification des résidus d’acides aminés accessibles aux solvants. Les résidus modifiés peuvent être identifiés après digestion protéolytique et analyse chromatographie liquide/spectrométrie de masse.
Résumé
La caractérisation de la structure d’ordre supérieur d’une protéine est essentielle pour comprendre sa fonction. La spectrométrie de masse (SEP) est apparue comme un outil puissant à cette fin, en particulier pour les systèmes protéiques difficiles à étudier par des méthodes traditionnelles. Pour étudier la structure d’une protéine par MS, des réactions chimiques spécifiques sont réalisées en solution qui codent l’information structurelle d’une protéine dans sa masse. Une approche particulièrement efficace consiste à utiliser des réactifs qui modifient de manière covalente les chaînes latérales d’acides aminés accessibles aux solvants. Ces réactions conduisent à des augmentations de masse qui peuvent être localisées avec une résolution au niveau des résidus lorsqu’elles sont combinées à la digestion protéolytique et à la spectrométrie de masse en tandem. Ici, nous décrivons les protocoles associés à l’utilisation du diéthylpyrocarbonate (DEPC) comme réactif de étiquetage covalent avec la détection de milliseconde. Le DEPC est une molécule hautement électrophile capable de étiqueter jusqu’à 30% des résidus dans la protéine moyenne, offrant ainsi une excellente résolution structurelle. Le DEPC a été utilisé avec succès avec ms pour obtenir des informations structurelles pour de petites protéines à domaine unique, telles que la β2-microglobuline, aux grandes protéines multi-domaines, telles que les anticorps monoclonaux.
Introduction
Les protéines sont des biomolécules essentielles dans pratiquement tous les processus physiologiques. La variété des fonctions que les protéines remplissent est possible en raison des structures qu’elles adoptent et des interactions qu’elles ont avec d’autres biomolécules. Pour comprendre la fonction des protéines à un niveau plus profond, des outils biochimiques et biophysiques sont nécessaires pour élucider ces caractéristiques et interactions structurelles importantes. Traditionnellement, la cristallographie aux rayons X, la microscopie électronique cryogénique et la spectroscopie de résonance magnétique nucléaire (RMN) ont fourni le détail de niveau atomique souhaité pour révéler la structure des protéines. Cependant, de nombreux systèmes protéiques ne peuvent pas être interrogés par ces techniques en raison d’un comportement de cristallisation médiocre, d’une disponibilité limitée des protéines, d’une hétérogénéité excessive des échantillons ou de limitations de poids moléculaire. Par conséquent, de nouvelles méthodes d’analyse ont vu le jour pour surmonter ces limites. Parmi les techniques émergentes qui peuvent fournir des informations structurelles sur les protéines, il y a la spectrométrie de masse.
La spectrométrie de masse (MS) mesure le rapport masse/charge (m/z) d’une molécule, de sorte que les informations structurelles d’ordre supérieur des protéines doivent être obtenues en codant les informations structurelles souhaitées dans la masse de la protéine. Plusieurs approches pour coder ces informations ont été développées, notamment l’échange hydrogène-deutérium (HDX)1,2,3,4,la réticulation chimique (XL)5,6,et le étiquetage covalent (CL)7,8,9,10. Dans HDX, les hydrogènes d’amide de base sont échangés par des deutériums légèrement plus massifs à des vitesses qui dépendent de l’accessibilité des solvants et de l’étendue de la liaison H. L’étendue de HDX peut être localisée en digérant rapidement la protéine en fragments peptidiques avant de séparer et de mesurer ces fragments par le spectromètre de masse ou en dissociant la protéine dans une expérience descendante. La détermination du taux de change fournit un aperçu supplémentaire de la dynamique des protéines. HDX s’est avéré être un outil précieux pour caractériser la structure des protéines malgré les défis associés à l’échange de dos et le besoin d’équipement spécialisé pour maximiser la reproductibilité. Dans XL-MS, les protéines réagissent avec des réactifs bi-fonctionnels qui lient de manière covalent les chaînes latérales de résidus adjacentes au sein d’une protéine donnée ou entre deux protéines. Ce faisant, XL-MS peut fournir des contraintes de distance qui peuvent être utilisées pour caractériser la structure des protéines. Les régions de la protéine qui sont réticulées peuvent être identifiées par digestion protéolytique suivie d’une analyse par chromatographie liquide (LC)-MS. Bien que XL-MS soit un outil polyvalent qui a été utilisé pour étudier une variété de complexes protéiques, y compris les cellules internes, l’identification des produits XL est difficile et nécessite un logiciel spécialisé.
La CL-MS est apparue récemment comme un outil complémentaire et parfois alternatif basé sur la SEP pour étudier la structure et les interactions des protéines. Dans la CL-MS, une protéine ou un complexe protéique est modifié de manière covalente avec un réactif mono-fonctionnel qui peut réagir avec des chaînes latérales exposées au solvant (Figure 1). En comparant les degrés de modification d’une protéine ou d’un complexe protéique dans différentes conditions, des changements de conformation, des sites de liaison et des interfaces protéine-protéine peuvent être révélés. Après la réaction CL, des informations spécifiques au site, souvent au niveau d’un seul acide aminé, peuvent être obtenues à l’aide de flux de travail protéomiques ascendants typiques dans lesquels les protéines sont digérées protéolytiquement, les fragments peptidiques sont séparés par LC et les sites modifiés sont identifiés à l’aide de la SP en tandem (MS / MS). La riche histoire de la chimie bioconjuguée a rendu de nombreux réactifs disponibles pour les expériences cl-ms. Les réactifs cl se répartissent en deux grandes catégories: (i) spécifique et (ii) non spécifique. Des réactifs spécifiques réagissent avec un seul groupe fonctionnel (p. ex. amines libres)8,10 et sont faciles à mettre en œuvre, mais ils ont tendance à fournir des informations structurelles limitées. Les réactifs non spécifiques réagissent avec une large gamme de chaînes latérales, mais nécessitent souvent des équipements spécialisés tels que des lasers ou des sources synchrotron pour produire ces espèces hautement réactives. Les radicaux hydroxyles sont le réactif non spécifique le plus couramment utilisé, ayant été appliqué dans les expériences d’empreintes de radicaux hydroxyles (HRF)7,11,12,13 pour étudier un large éventail de protéines et de complexes protéiques dans diverses conditions.
Notre groupe de recherche a utilisé avec succès un autre réactif relativement non spécifique appelé diéthylpyrocarbonate (DEPC) pour étudier la structure et les interactions des protéines dans le contexte des expériences CL-MS14,15,16,17,18,19,20,21,22,23,24,25. DEPC offre la simplicité de réactifs d’étiquetage spécifiques (c’est-à-dire qu’aucun équipement spécialisé n’est nécessaire pour effectuer les réactions), tout en réagissant avec jusqu’à 30% d’acides aminés dans la protéine moyenne. En tant que réactif hautement électrophile, DEPC est capable de réagir avec le N-terminus et les chaînes latérales nucléophiles de la cystéine, de l’histidine, de la lysine, de la tyrosine, de la sérine et des résidus de thréonine. Typiquement, un seul produit de ces réactions est généré, ce qui entraîne une augmentation de masse de 72,02 Da. Ce type unique de produit contraste avec les jusqu’à 55 produits différents qui peuvent être produits lorsque les protéines réagissent avec les radicaux hydroxyles7. Une telle chimie simple facilite l’identification des sites étiquetés.
Ici, nous fournissons des protocoles pour l’utilisation de CL-MS à base de DEPC pour étudier la structure et les interactions des protéines. Les détails associés à la préparation des réactifs, aux réactions des protéines DEPC, aux conditions de digestion des protéines, aux paramètres LC-MS et MS/MS, ainsi qu’à l’analyse des données sont décrits. Nous démontrons également l’utilité du étiquetage DEPC en fournissant des exemples de résultats d’interactions protéine-métal, protéine-ligand et protéine-protéine ainsi que de protéines subissant des changements structurels lors du chauffage.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparation de protéines et de réactifs
Remarque : ce protocole inclut un exemple de flux de travail pour le étiquetage d’une protéine avec DEPC. Certaines conditions et concentrations de réactifs énumérées peuvent varier en fonction de la protéine de choix.
- Préparer toutes les solutions réactives dans des tubes de microcentrifugation de 1,5 mL.
- Préparer une solution protéique de la concentration désirée, habituellement de l’ordre de dizaines de μM, dans un tampon d’acide 3-(N-morpholino)propanesulfonique (MOPS) de 10 mM à un pH de 7,4. Alternativement, préparer une solution protéique existante d’échange tampon dans un pH de 10 mM de 7,4 MOPS si l’échantillon contient un tampon nucléophile qui serait réactif avec le DEPC. D’autres tampons (p. ex. solution saline tamponnée au phosphate) peuvent également être utilisés, à condition qu’ils n’aient pas de groupes fonctionnels nucléophiles.
- Préparer une solution de DEPC de 100 mM dans de l’acétonitrile sec (ACN) en piper 1,45 μL de la solution de DEPC mère de 6,9 M dans 98,55 μL de l’ACN.
REMARQUE: Il n’y a pas un seul ensemble de concentrations qui fonctionnera avec chaque protéine, bien que les concentrations optimales puissent être estimées en fonction du nombre de résidus His et Lys23. Par exemple, avec 50 μL d’une solution de β2-microglobuline de 50 μM, réagissez la protéine avec 0,2 μL du DEPC de 100 mM pour une concentration finale de DEPC de 200 μM (égale à 4x la concentration en protéines) pour assurer le rapport molaire souhaité en utilisant cet exemple général(tableau 1). Le étiquetage DEPC est une réaction d’ordre 2nd, donc changer la concentration de protéines ou de DEPC dans le mélange réactionnel changera le taux d’étiquetage. - Préparer une solution d’imidazole 1 M en pesant 10 mg d’imidazole et en les dissolvant dans 146,9 μL d’eau de qualité CLHP.
2. Étiquetage covalent des protéines intactes
- Réglez la température du bain d’eau à 37 °C et attendez que le bain atteigne une température stable.
REMARQUE : Les concentrations et les volumes de réactifs pour un exemple de protocole d’étiquetage se trouvent dans le tableau 1. - Dans un nouveau tube à microcentrifugation, mélanger le tampon MOPS et la solution protéique dans les volumes énumérés au tableau 1.
- À la protéine et au tampon, ajoutez 0,2 μL de la solution de DEPC, en vous assurant de bien mélanger la solution résultante, puis placez le tube contenant le mélange réactionnel dans le bain-marie à 37 °C pendant 1 minute.
REMARQUE: Le volume d’ACN ajouté ne doit pas dépasser 1% du volume total de réaction pour éviter la perturbation de la structure de la protéine pendant la réaction de étiquetage. Le temps de réaction est à la charge de l’utilisateur, bien qu’une réaction de 1 minute dans les conditions d’exemple minimise le surétiquetage et l’hydrolyse potentielle de DEPC14. - Après 1 minute, retirer le tube contenant le mélange réactionnel du bain-marie et éteindre la réaction avec 1 μL de la solution d’imidazole 1 M pour récupérer le DEPC restant qui n’a pas réagi.
NOTA: La concentration finale d’imidazole dans le mélange réactionnel doit être égale à 50 fois la concentration de DEPC dans le mélange réactionnel. Cela garantira que le DEPC restant n’ayant pas réagi est récupéré.
3. Préparation du digeste protéique pour la LC-MS ascendante
REMARQUE: Choisissez des conditions de digestion qui se prêtent à la protéine d’intérêt. Les étapes courantes consistent à déplier la protéine et à réduire et à alkyler les liaisons disulfures.
- Dépliez la protéine en ajoutant un réactif de dépliage approprié au mélange réactionnel.
REMARQUE: Les agents de dépliage courants comprennent l’ACN, l’urée et le chlorhydrate de guanidine (GuHCl). - Préparer des solutions de tris(2-carboxyéthyl)phosphine (TCEP) et d’iodoacétamide (IAM) en pesant 5 mg de chacune et en les dissolvant dans de nouveaux tubes microcentrifugés dans 174,4 et 270,3 μL de tampon MOPS de 10 mM pH 7,4, respectivement, pour les étapes de réduction et d’alkylation.
- Réduire les liaisons disulfures en ajoutant 2 μL de la solution de PTCE (concentration finale de 2 mM dans le mélange réactionnel) de 100 mM au mélange réactionnel et en réagissant pendant 3 minutes à température ambiante.
NOTA : La concentration finale de PTCE doit être égale à 40 fois la concentration en protéines par liaison disulfure présente dans la solution. - L’alkylate a réduit les cystéines avec 4 μL de la solution IAM de 100 mM (concentration finale de 4 mM dans le mélange réactionnel) pendant 30 minutes dans l’obscurité. IAM est sensible à la lumière et se décompose sous la lumière directe.
NOTA : La concentration finale d’IAM en solution devrait être deux fois plus forte que celle utilisée pour le PTCE, ou 80 fois la concentration protéique par liaison disulfure. - Digérer la protéine avec une enzyme appropriée telle que la trypsine ou la chymotrypsine. Un rapport protéines/enzymes de 10:1 pour une digestion de 3 heures à 37 °C avec une enzyme immobilisée à un taux de secousse de 300 coups/min est généralement suffisant pour les protéines marqués par DEPC. Voir discussion.
- Après digestion, séparer l’enzyme immobilisée des peptides digérés par centrifugation à 12 000 rpm pendant 5 minutes.
- Analyser l’échantillon immédiatement par CL-MS/MS ou congeler l’échantillon avec de l’azote liquide pour minimiser la dégradation de l’échantillon et la perte de l’étiquette. Conserver les échantillons congelés instantanément à < -20 °C jusqu’à ce qu’ils soient prêts pour l’analyse LC-MS/MS.
4. Analyse LC-MS/MS
REMARQUE : Les paramètres LC-MS/MS standard pour la protéomique ascendante peuvent être utilisés pour identifier les sites marqués sur les fragments peptidiques protéolytiques. Un exemple général est présenté ci-dessous.
- Séparez les peptides marqués par le DEPC à l’aide d’une phase stationnaire C18 en phase inversée. Utiliser une phase mobile LC typique de deux solvants : (A) eau + 0,1 % d’acide formique et (B) ACN + 0,1 % d’acide formique à l’aide d’un gradient (p. ex. figure 2)pour obtenir la meilleure séparation des peptides.
REMARQUE: Le temps de séparation peut être optimisé en fonction de la complexité de l’échantillon, et le débit de phase mobile dépend de l’utilisation d’un capillar ou d’un nano LC. - Utilisez un spectromètre de masse capable de faire de la LC-MS et de la MS/MS en ligne pour identifier les sites de modification de LAPC sur le peptide. Dans nos expériences, nous avons utilisé avec succès plusieurs types de spectromètres de masse. Tout spectromètre de masse capable d’effectuer automatiquement des MS/MS de nombreux peptides au cours d’une analyse LC-MS devrait convenir. Les paramètres MS pertinents incluent: tension source ESI = -4000 V pour ESI régulier; -2000 V pour nanospray; Résolution Orbitrap = 60 000; Durée d’exclusion dynamique = 30 s; Type d’activation MS/MS : CID, ETD ou les deux ; Plage de balayage de masse = 200-2 000; Contrôle automatique du gain = 4.0E5 (MS1 dans Orbitrap) et 5.0E4 (MS2 dans le piège à ions quadripôle linéaire).
- Chargez et injectez l’échantillon de protéines digérées et étiquetées dans le système LC et commencez l’acquisition de LC-MS/MS. Si l’échantillon a été congelé à l’éclair, décongeler avant l’analyse. Détourner l’effluent de LC en déchets pendant les 5 premières minutes pour éviter que des sels excessifs ne pénètrent dans la source ESI.
NOTA : Une boucle d’injection de 5 μL est généralement utilisée, ce qui permet l’injection d’environ 2,5 μg de protéines dans la LC-MS/MS. Cela dépend des conditions de chargement du LC pour ne pas obstruer l’injecteur d’échantillon.
5. Analyse des données
- Identifier les sites d’étiquette DEPC et quantifier les zones de pic peptidique à l’aide du logiciel approprié pour le spectromètre de masse utilisé.
- Inclure l’ajout de DEPC (72,02 Da) et la carbamidométhylation (57,02 Da) comme modifications variables. Voici d’autres paramètres de recherche pour l’analyse ms/ms : clivages manqués maximum = 3; Types d’ions fragments = b et y; Tolérance m/z du précurseur = 10 ppm (cette valeur devrait être plus élevée si un spectromètre de masse à piège ionique quadripolaire est utilisé); Tolérance au fragment m/z = 0,5 Da (cette valeur doit être inférieure si un spectromètre de masse à haute résolution est utilisé pour un balayage ionique de produit); Charge de précurseur = 1-4.
REMARQUE: Différents algorithmes de recherche de base de données ont différents systèmes de notation, et beaucoup peuvent avoir des difficultés à identifier les peptides modifiés par DEPC car les niveaux de modification peuvent être faibles. L’ajustement de la limite du score peut être nécessaire pour identifier plus de peptides marqués. Si c’est le cas, l’interrogation manuelle des données MS/MS devrait être utilisée pour vérifier les peptides à faible score. Le produit de l’hydrolyse de l’étiquette DEPC n’est pas inclus dans la recherche de données parce que le DEPC hydrolysé n’est plus réactif envers les chaînes latérales nucléophiles. - Déterminer les pourcentages de modification au niveau des résidus à l’aide des zones de pic chromatographique des versions modifiées et non modifiées des peptides.
NOTA : Tout peptide contenant le résidu modifié d’intérêt doit être pris en compte et tous les états d’accusation qui sont inclus doivent être présents dans tous les échantillons mesurés. Les peptides ayant des efficacités d’ionisation différentes et éluant à différents moments fait que cette valeur est une mesure relative plutôt qu’absolue de la modification d’un site spécifique.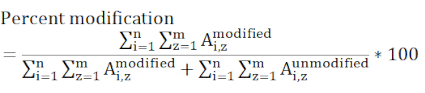
où Ai,z représente la surface de crête d’un peptide donné (i) qui contient le résidu d’intérêt et prend en compte tous les états de charge détectés (z). - Déterminer si un changement d’étiquetage entre un échantillon témoin et un échantillon expérimental est significatif à l’aide d’une évaluation statistique. Trois mesures répétées pour chaque échantillon sont typiques, et les tests t sont le plus souvent utilisés avec des intervalles de confiance de 95 ou 99 %.
Access restricted. Please log in or start a trial to view this content.
Résultats
Identification des sites de modification de DEPC et des pourcentages de modification
L’addition de masse due au marquage covalent peut être mesurée aux niveaux (a) de protéines intactes et (b) depeptides 8,9. Au niveau intact, une distribution d’espèces protéiques avec différents nombres d’étiquettes peut être obtenue à partir d’une analyse directe ou de LC-MS d’échantillons de protéines marqués. Pour obtenir des renseignements s...
Access restricted. Please log in or start a trial to view this content.
Discussion
Étapes critiques
Plusieurs points concernant la conception expérimentale devraient être pris en compte pour assurer des résultats d’étiquetage fiables. Tout d’abord, pour maximiser l’étiquetage des protéines, il est nécessaire d’éviter les tampons avec des groupes fortement nucléophiles (par exemple, Tris) car ils peuvent réagir avec le DEPC et réduire l’étendue de l’étiquetage. Il est également concevable que de tels tampons puissent réagir avec des résidus étiquetés, entraîna...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs reconnaissent le soutien des National Institutes of Health (NIH) dans le cadre de la subvention R01 GM075092. Le spectromètre de masse Thermo Orbitrap Fusion utilisé pour acquérir certaines des données décrites ici a été acquis avec des fonds de la subvention S10OD010645 des National Institutes of Health.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Thermo Fisher Scientific | 3448 | |
| 3-(N-morpholino)propanesulfonic acid | Millipore Sigma | M1254 | |
| 3-(N-morpholino)propanesulfonic acid sodium salt | Millipore Sigma | M9381 | |
| Acclaim PepMap RSLC C18 Column | Thermo Scientific | 164537 | 300 μm x 15 cm, C18, 2 μm, 100 A |
| Acetonitrile | Fisher Scientific | A998-1 | |
| Diethylpyrocarbonate | Millipore Sigma | D5758 | |
| HPLC-grade water | Fisher Scientific | W5-1 | |
| Imidazole | Millipore Sigma | I5513 | |
| Immobilized chymotrypsin | ProteoChem | g4105 | |
| Immobilized trypsin, TPCK Treated | Thermo Fisher Scientific | 20230 | |
| Iodoacetamide | Millipore Sigma | I1149 | |
| Tris(2-carboxyethyl)phosphine | Millipore Sigma | C4706 |
Références
- Katta, V., Chait, B. T., Carr, S. Conformational Changes in Proteins Probed by Hydrogen-exchange Electrospray-ionization. Rapid Communications in Mass Spectrometry. 5, 214-217 (1991).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25, 158-170 (2006).
- Pirrone, G. F., Iacob, R. E., Engen, J. R. Applications of hydrogen/deuterium exchange MS from 2012 to 2014. Analytical Chemistry. 87, 99-118 (2015).
- Oganesyan, I., Lento, C., Wilson, D. J. Contemporary hydrogen deuterium exchange mass spectrometry. Methods. 144, 27-42 (2018).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrometry Reviews. 25, 663-682 (2006).
- Holding, A. N. XL-MS: Protein cross-linking coupled with mass spectrometry. Methods. 89, 54-63 (2015).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107, 3514-3543 (2007).
- Mendoza, V. L., Vachet, R. W. Probing Protein Structure by Amino Acid-Specific Covalent Labeling and Mass Spectrometry. Mass Spectrometry Reviews. 28, 785-815 (2009).
- Limpikirati, P., Liu, T., Vachet, R. W. Covalent labeling-mass spectrometry with non-specific reagents for studying protein structure and interactions. Methods. 144, 79-93 (2018).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemistry Reviews. 120, 4335(2020).
- Maleknia, S. D., Brenowitz, M., Chance, M. R. Millisecond radiolytic modification of peptides by synchrotron X-rays identified by mass spectrometry. Analytical Chemistry. 71, 3965-3973 (1999).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77, 5814-5822 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society of Mass Spectrometry. 16, 2057-2063 (2005).
- Mendoza, V. L., Vachet, R. W. Protein surface mapping using diethylpyrocarbonate with mass spectrometric detection. Analytical Chemistry. 80, 2895-2904 (2008).
- Mendoza, V. L., Antwi, K., Barón-rodríguez, M. A., Blanco, C., Vachet, R. W. Structure of the Pre-amyloid Dimer of β-2-microglobulin from Covalent Labeling and Mass Spectrometry. Biochemistry. 49, 1522-1532 (2010).
- Mendoza, V. L., Barón-Rodríguez, M. A., Blanco, C., Vachet, R. W. Structural insights into the pre-amyloid tetramer of β-2-microglobulin from covalent labeling and mass spectrometry. Biochemistry. 50, 6711-6722 (2011).
- Zhou, Y., Vachet, R. W. Increased protein structural resolution from diethylpyrocarbonate-based covalent labeling and mass spectrometric detection. Journal of the American Society of Mass Spectrometry. 23, 708-717 (2012).
- Borotto, N. B., et al. Investigating Therapeutic Protein Structure with Diethylpyrocarbonate Labeling and Mass Spectrometry. Analytical Chemistry. 87, 10627-10634 (2015).
- Liu, T., Marcinko, T. M., Kiefer, P. A., Vachet, R. W. Using Covalent Labeling and Mass Spectrometry To Study Protein Binding Sites of Amyloid Inhibiting Molecules. Analytical Chemistry. 89, 11583-11591 (2017).
- Limpikirati, P., et al. Covalent labeling and mass spectrometry reveal subtle higher order structural changes for antibody therapeutics. MAbs. 11, 463-476 (2019).
- Limpikirati, P., Pan, X., Vachet, R. W. Covalent Labeling with Diethylpyrocarbonate: Sensitive to the Residue Microenvironment, Providing Improved Analysis of Protein Higher Order Structure by Mass Spectrometry. Analytical Chemistry. 91, 8516-8523 (2019).
- Liu, T., Limpikirati, P., Vachet, R. W. Synergistic Structural Information from Covalent Labeling and Hydrogen-Deuterium Exchange Mass Spectrometry for Protein-Ligand Interactions. Analytical Chemistry. 91, 15248-15254 (2019).
- Pan, X., Limpikirati, P., Chen, H., Liu, T., Vachet, R. W. Higher-Order Structure Influences the Kinetics of Diethylpyrocarbonate Covalent Labeling of Proteins. Journal of the American Society of Mass Spectrometry. 31, 658-665 (2020).
- Limpikirati, P. K., Zhao, B., Pan, X., Eyles, S. J., Vachet, R. W. Covalent Labeling/Mass Spectrometry of Monoclonal Antibodies with Diethylpyrocarbonate: Reaction Kinetics for Ensuring Protein Structural Integrity. Journal of the American Society of Mass Spectrometry. 31, 1223-1232 (2020).
- Liu, T., Marcinko, T. M., Vachet, R. W. Protein-Ligand Affinity Determinations Using Covalent Labeling-Mass Spectrometry. Journal of the American Society of Mass Spectrometry. 31, 1544-1553 (2020).
- Srikanth, R., Mendoza, V. L., Bridgewater, J. D., Zhang, G., Vachet, R. W. Copper Binding to β-2-Microglobulin and its Pre-Amyloid Oligomers. Biochemistry. 48, 9871-9881 (2009).
- Lim, J., Vachet, R. W. Using mass spectrometry to study copper-protein binding under native and non-native conditions: β-2-microglobulin. Analytical Chemistry. 76, 3498-3504 (2004).
- Lindsley, C. W. Predictions and Statistics for the Best-Selling Drugs Globally and in the United States in 2018 and a Look Forward to 2024 Projections. ACS Chemical Neuroscience. 10, 1115(2019).
- Floege, J., Ketteler, M. β2-Microglobulin-derived amyloidosis: An update. Kidney International. 59, 164(2001).
- Antwi, K., et al. Cu (II) organizes β-2-microglobulin oligomers but is released upon amyloid formation. Protein Science. 17, 748-759 (2008).
- Dong, J., et al. Unique Effect of Cu(II) in the Metal-Induced Amyloid Formation of β-2-Microglobulin. Biochemistry. 53, 1263-1274 (2014).
- Marcinko, T. M., Drews, T., Liu, T., Vachet, R. W. Epigallocatechin-3-gallate Inhibits Cu(II)-Induced β-2-Microglobulin Amyloid Formation by Binding to the Edge of Its β-Sheets. Biochemistry. 59, 1093-1103 (2020).
- Zhou, Y., Vachet, R. W. Diethylpyrocarbonate Labeling for the Structural Analysis of Proteins: Label Scrambling in Solution and How to Avoid it. Journal of the American Society of Mass Spectrometry. 23, 899-907 (2012).
- Borotto, N. B., Degraan-Weber, N., Zhou, Y., Vachet, R. W. Label scrambling during CID of covalently labeled peptide ions. Journal of the American Society of Mass Spectrometry. 25, 1739-1746 (2014).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical Chemistry. 90, 7721-7729 (2018).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Analytical Chemistry. 89, 1459-1468 (2017).
- Zheng, X., Wintrode, P. L., Chance, M. R. Complementary Structural Mass Spectrometry Techniques Reveal Local Dynamics in Functionally Important Regions of a Metastable Serpin. Structure. 16, 38-51 (2008).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by H/D exchange and covalent labeling: The glycerol facilitator. Journal of Molecular Biology. 416, 400-413 (2012).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical Chemistry. 89, 2250-2258 (2017).
- Borotto, N. B., Zhang, Z., Dong, J., Burant, B., Vachet, R. W. Increased β-Sheet Dynamics and D-E Loop Repositioning Are Necessary for Cu(II)-Induced Amyloid Formation by β-2-Microglobulin. Biochemistry. 56, 1095-1104 (2017).
- Shi, L., Liu, T., Gross, M. L., Huang, Y. Recognition of Human IgG1 by Fcγ Receptors: Structural Insights from Hydrogen-Deuterium Exchange and Fast Photochemical Oxidation of Proteins Coupled with Mass Spectrometry. Biochemistry. 58, 1074-1080 (2019).
- Gerega, S. K., Downard, K. M. PROXIMO - A new docking algorithm to model protein complexes using data from radical probe mass spectrometry (RP-MS). Bioinformatics. 22, 1702-1709 (2006).
- Kamal, J. K. A., Chance, M. R. Modeling of protein binary complexes using structural mass spectrometry data. Protein Science. 17, 79-94 (2007).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon