このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
質量分析によるタンパク質高次構造の研究のためのジエチルピロカーボネートとの共有結合標識
要約
質量分析検出を用いてジエチルピロカーボネートベースの共有標識を行うための実験的手順について説明する。ジエチルピロカーボネートは、単に目的のタンパク質またはタンパク質複合体と混合され、溶媒にアクセス可能なアミノ酸残基の修飾につながる。修飾残基は、タンパク質分解と液体クロマトグラフィー/質量分析分析の後に同定することができる。
要約
タンパク質の高次構造を特徴付けて、その機能を理解するためには不可欠です。質量分析(MS)は、特に従来の方法では研究が困難なタンパク質系に対して、この目的のための強力なツールとして登場しました。MSによるタンパク質の構造を研究するために、タンパク質の構造情報をその質量にコードする溶液中で特定の化学反応が行われます。特に効果的なアプローチの1つは、溶媒のアクセス可能なアミノ酸側鎖を共有的に修飾する試薬を使用することです。これらの反応は、タンパク質分解性消化およびタンデム質量分析と組み合わせると残留レベルの分解能に局在化できる質量増加を招く。ここでは、MS検出と共に共有標識試薬としてのジエチルピロカーボネート(DEPC)の使用に関連するプロトコルについて説明する。DEPCは、平均タンパク質中の残基の30%まで標識することができる高い電気電子分子であり、それにより優れた構造分解能を提供する。DEPCはMSと共に、β2-ミクログロブリンなどの小さな単一ドメインタンパク質の構造情報をモノクローナル抗体などの大きな多ドメインタンパク質に対して得るのに成功しています。
概要
タンパク質は、事実上すべての生理学的プロセスにおいて不可欠な生体分子です。タンパク質が採用する構造と他の生体分子との相互作用のために、タンパク質が果たす様々な機能が可能です。タンパク質の機能を深く理解するためには、これらの重要な構造特徴と相互作用を解明するために生化学的および生物物理学的ツールが必要です。従来、X線結晶学、極低温電子顕微鏡、核磁気共鳴(NMR)分光法は、タンパク質構造を明らかにするために所望の原子レベルの詳細を提供してきました。しかし、結晶化の挙動、タンパク質の入手可能性の制限、過剰なサンプルの不均一性、分子量の制限など、これらの手法では、多数のタンパク質システムを問い合せることはできません。その結果、これらの制限を克服する新しい分析方法が登場しました。タンパク質の構造情報を提供できる新たな技術の中には、質量分析があります。
質量分析(MS)は分子の質量電荷(m/z)比を測定するため、所望の構造情報をタンパク質の質量にコードすることにより、タンパク質の高次構造情報を取得する必要があります。この情報をコード化するためのいくつかのアプローチは、水素重水素交換(HDX)1、2、3、4、化学架橋(XL)5、6、および共有結合標識(CL)7、8、9、10を含む開発された。HDXでは、溶媒の入手可能性とH結合の範囲に依存する速度でわずかに大きい重水素によって、骨格アミド水素が交換されます。HDXの範囲は、タンパク質をペプチド断片に素早く消化してから、質量分析計でこれらの断片を分離して測定するか、またはトップダウン実験でタンパク質を解離することによって局在化することができる。交換速度を決定することで、タンパク質のダイナミクスに関するさらなる洞察が得られます。HDXは、バック交換に伴う課題や、再現性を最大限に高める特殊な機器の必要性にも関わらず、タンパク質構造を特徴付けるための貴重なツールであることが証明されています。XL-MSでは、タンパク質は、特定のタンパク質内または2つのタンパク質間で隣接する残基側鎖を共有結合する二機能性試薬と反応する。その際、XL-MSは、タンパク質構造を特徴付けるために使用できる距離制約を提供することができます。架橋されたタンパク質の領域は、タンパク質分解消化と続く液体クロマトグラフィー(LC)-MS分析によって同定することができる。XL-MSは、細胞内を含む様々なタンパク質複合体を研究するために使用されてきた汎用性の高いツールですが、XL製品の同定は困難であり、特殊なソフトウェアが必要です。
CL-MSは、タンパク質の構造と相互作用を研究するための補完的で、時には代替MSベースのツールとして最近登場しました。CL-MSにおいて、タンパク質またはタンパク質複合体は、溶媒暴露側鎖と反応し得る単一機能試薬で共有修飾される(図1)。異なる条件下でタンパク質またはタンパク質複合体の修飾範囲を比較することにより、立体構造変化、結合部位、およびタンパク質-タンパク質のインターフェースが明らかになる。CL反応後、部位特異的情報は、多くの場合、単一アミノ酸レベルで、タンパク質がタンパク質分解され、ペプチド断片がLCによって分離され、改変部位がタンデムMS(MS/MS)を使用して同定される典型的なボトムアッププロテオミクスワークフローを使用して得ることができる。バイオコンジュゲート化学の豊富な歴史により、CL-MS実験に数多くの試薬が利用可能になりました。CL試薬は、(i)特異的および(ii)非特異的の2つの一般的なカテゴリーに分類される。特定の試薬は、単一の官能基(例えば、遊離アミン)8、10と反応し、実装が容易であるが、それらは限られた構造情報を提供する傾向がある。非特異的試薬は、広範囲の側鎖と反応しますが、多くの場合、これらの反応性の高い種を生成するためにレーザーやシンクロトロン源などの特殊な機器が必要です。ヒドロキシルラジカルは、最も一般的に使用される非特異的試薬であり、ヒドロキシルラジカルフットプリント(HRF)7、11、12、13実験を行い、様々な条件下で幅広いタンパク質およびタンパク質複合体を研究した。
我々の研究グループは、CL-MS実験14、15、16、17、18、19、20、21、22、23、24、25の文脈におけるタンパク質構造および相互作用を研究するために、ジエチルピロカーボネート(DEPC)と呼ばれる別の比較的非特異的な試薬を使用することに成功した。DEPCは、特定の標識試薬のシンプルさを提供し(すなわち、反応を行うために特別な装置は必要ありません)、一方で、平均タンパク質中のアミノ酸の最大30%と反応します。高い電気反応試薬として、DEPCはシステイン、ヒスチジン、リジン、チロシン、セリン、スレオニン残基のN末端および求核側鎖と反応することができる。典型的には、これらの反応の単一の産物が生成され、結果として72.02Daの質量増加が生じる。この単一タイプの製品は、タンパク質がヒドロキシルラジカルと反応するときに生成することができる最大55種類の製品と対比する7。このような単純な化学は、標識された部位の同定を容易にする。
ここでは、DEPCベースのCL-MSを用いてタンパク質の構造と相互作用を研究するためのプロトコルを提供します。試薬調製、DEPC-タンパク質反応、タンパク質消化条件、LC-MSおよびMS/MSパラメータ、およびデータ分析に関連する詳細が記載されています。また、タンパク質-金属、タンパクリガンド、タンパク質-タンパク質相互作用、加熱時に構造変化を起こしているタンパク質の結果の例を提供することで、DEPC標識の有用性を実証します。
プロトコル
1. タンパク質および試薬の調製
注: このプロトコルには、DEPC でタンパク質を標識するワークフローの例が含まれています。一部の条件および試薬濃度は、選択したタンパク質に基づいて異なる場合があります。
- 1.5 mLマイクロ遠心チューブですべての試薬溶液を調製します。
- pH 7.4で所望の濃度のタンパク質溶液を、通常は数十μMの範囲で10 mM 3-(N-モルフォリノ)プロパンスルホン酸(MOPS)緩衝液で調製します。あるいは、サンプルにDEPCで反応性となる求核バッファーが含まれている場合は、10 mM pH 7.4 MOPS でバッファー交換既存のタンパク質溶液を調製します。他の緩衝剤(例えば、リン酸緩衝食塩基)も、求核性官能基を有しないものであれば使用することができる。
- 1.45 μLのストック6.9 M DEPC溶液をACNの98.55 μLにピペット処理して、乾燥アセトニトリル(ACN)に100 mM DEPC溶液を調製します。
注:すべてのタンパク質で動作する濃度のセットはありませんが、最適な濃度は、HisとLys残基の数に基づいて推定することができます23.例えば、50μM β2マイクログロブリン溶液の50μLで、100mM DEPCの0.2 μLでタンパク質を反応させて、最終的なDEPC濃度200μM(4倍のタンパク質濃度に等しい)を用いて、この一般的な例を用いて所望のモル比を確保する(表1)。DEPC標識は2次 反応であるため、反応混合物中のタンパク質またはDEPCの濃度を変化させると、標識速度が変化する。 - 10 mgのイミダゾールを計量し、146.9 μLのHPLC級水に溶解して、1 Mイミダゾール溶液を調製します。
2. インタクトタンパク質のコバレント標識
- 水浴温度を37°Cに設定し、お風呂が安定した温度に達するのを待ちます。
注: 例示のラベル付けプロトコルの試薬濃度と容積は 、表 1に記載されています。 - 新しいマイクロ遠心管で、 表1に記載されているボリュームにMOPSバッファーとタンパク質溶液を混ぜ合わせる。
- タンパク質とバッファーに、DEPC溶液の0.2 μLを加え、得られた溶液を適切に混合し、その後、反応混合物を含むチューブを37°Cの水浴に1分間入れます。
注:ACNの添加量は、標識反応中にタンパク質の構造が摂動するのを避けるために、全反応量の1%を超えてはならない。反応時間はユーザまでであり、例条件下での1分間の反応は、DEPC14の過剰標識および電位加水分解を最小限に抑える。 - 1分後、水浴から反応混合物を含むチューブを取り出し、1Mイミダゾール溶液の1μLで反応を焼き付け、残りの未反応DEPCを清掃する。
注:反応混合物中のイミダゾールの最終濃度は、反応混合物中のDEPCの濃度の50倍に等しくする必要があります。これにより、残りの未反応 DEPC が清掃されます。
3. ボトムアップLC-MS用タンパク質消化の調製
注: 目的のタンパク質に適した消化条件を選択してください。一般的なステップは、タンパク質を展開し、任意の二硫化物結合を減少させ、アルキル化することを含む。
- 反応混合物に適当な展開試薬を加えてタンパク質を展開する。
注:一般的な展開剤には、ACN、尿素、グアニジン塩酸塩(GuHCl)が含まれます。 - トリス(2-カルボクセチル)ホスフィン(TCEP)とヨウオアセトアミド(IAM)の溶液をそれぞれ5mgの計量し、10mM pH 7.4 MOPSバッファーの174.4および270.3 μLの新しいマイクロ遠心チューブに溶解して、還元およびアルキリエーションステップの手順を調製します。
- 反応混合物に2μLの100 mM TCEP(反応混合物中の2mMの最終濃度)溶液を加え、室温で3分間反応させることにより、二硫化結合を低減します。
注: TCEP の最終的な濃度は、溶液中に存在する二硫化物結合あたりのタンパク質濃度の 40 倍に等しいはずです。 - アルキル酸は、100 mMのIAM溶液(反応混合物中の4 mMの最終濃度)を暗い中で30分間4μLで減少させたシステインを減少させた。IAMは光に敏感で、直接光の下で分解します。
注: 溶液中の IAM の最終的な濃度は、TCEP に使用される濃度の 2 倍、つまり、200% の結合あたりのタンパク質濃度の 80 倍である必要があります。 - トリプシンやキモトリプシンなどの適切な酵素でタンパク質を消化します。300ストローク/分の揺れ速度で固定化酵素を用いた37°Cでの3時間消化に対する10:1タンパク質:酵素比は、通常、DEPC標識タンパク質に十分である。「ディスカッション」を参照してください。
- 消化後、消化したペプチドから固定化した酵素を12,000rpmで5分間遠心分離して分離する。
- サンプルをLC-MS/MSで直ちに分析するか、液体窒素でサンプルをフラッシュフリーズして、サンプルの劣化とラベルの損失を最小限に抑えます。フラッシュ冷凍サンプルは、LC-MS/MS分析の準備ができるまで、< -20 °Cで保管してください。
4. LC-MS/MS分析
注: ボトムアッププロテオミクスの標準 LC-MS/MS パラメータを使用して、タンパク質分解ペプチドフラグメント上の標識部位を同定することができます。一般的な例を以下に示します。
- 逆相C18定常相を用いてDEPC標識ペプチドを分離する。典型的なLC移動相の2つの溶媒を使用する:(A)水+0.1%のギ酸および(B)ACN+0.1%のギ酸を使用して、勾配(例えば、 図2)を使用してペプチドの最良の分離を達成する。
注:分離時間はサンプルの複雑さに基づいて最適化することができ、移動相流量は毛細管またはナノLCが使用されているかどうかによって異なります。 - オンラインLC-MSおよびMS/MSを行うことができる質量分析計を使用して、ペプチド上のDEPC修飾部位を同定する。我々の実験では、質量分析計のいくつかのタイプを使用することに成功しました。LC-MS分析の過程で多くのペプチドのMS/MSを自動的に実行できる質量分析計は、適切であるべきです。関連するMSパラメータには、ESIソース電圧= -4000 V(通常のESI用)が含まれます。ナノスプレー用-2000 V;軌道上解像度 = 60,000;動的除外期間 = 30 s;MS/MS アクティベーション タイプ: CID、ETD、またはその両方。質量スキャン範囲 = 200-2,000;自動ゲイン制御 = 4.0E5 (オービトラップの MS1)および 5.0E4 (線形四重極イオントラップの MS2)
- 消化された標識タンパク質サンプルをLCシステムにロードして注入し、LC-MS/MSの取得を開始します。サンプルがフラッシュ凍結されている場合は、分析前に解凍します。過度の塩がESI源に入るのを避けるために、LCの排水を最初の5分間無駄にします。
注意:5 μLの注入ループは一般に利用され、およそ2.5 μgのタンパク質をLC-MS/MSに注入することを可能にします。これは、サンプルインジェクタを詰まらせないように、LCの負荷条件に依存する。
5. データ分析
- 使用する質量分析計に適したソフトウェアを使用して、DEPCラベル部位を特定し、ペプチドピーク領域を定量化します。
- DEPC添加(72.02 Da)およびカルバミドメチル化(57.02 Da)を可変的な変更として含める。MS/MS 分析の追加の検索パラメーターは次のとおりです: 最大切断を逃した = 3;フラグメントイオンタイプ = bおよびy;前駆体 m/z 許容値 = 10 ppm (四重極イオントラップ質量分析計を使用する場合、この値は高くなる必要があります)。フラグメント m/z 許容値 = 0.5 Da (製品イオンスキャンに高解像度の質量分析計を使用する場合、この値は低くなる必要があります)。前駆体電荷 = 1-4.
注: 異なるデータベース検索アルゴリズムは、異なるスコアリングシステムを持っており、多くは、変更レベルが低い可能性があるため、DEPC修飾ペプチドを識別することが困難な場合があります。スコアカットオフを調整することは、より多くの標識ペプチドを同定するために必要である場合がある。その場合は、MS/MSデータの手動による問い合わせを使用して、低スコアペプチドを検証する必要があります。加水分解されたDEPCが求核側鎖に対して反応性を持たなくなったため、DEPCラベルの加水分解の産物はデータ検索に含まれない。 - ペプチドの修飾および未改変バージョンのクロマトグラフィーピーク領域を使用して、残基レベルの改変率を決定します。
注: 対象の修飾残基を含むペプチドは考慮する必要があり、含まれるすべての電荷状態は、すべての測定サンプルに存在する必要があります。異なるイオン化効率を有し、異なる時間に溶出するペプチドは、この値が特定の部位の修飾の絶対的尺度ではなく相対的なものである。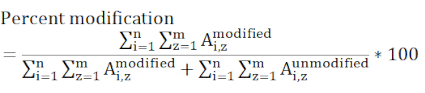
ここで、A,zは 、対象の残基を含む任意のペプチド(i)のピーク面積を表し、すべての検出された電荷状態(z)を考慮します。 - 統計的評価を使用して、コントロールと実験サンプルの間のラベリングの変化が有意かどうかを判断します。各サンプルの3つの反復測定が典型的であり、t検定は95または99%の信頼区間で最も一般的に利用される。
結果
DEPC 変更サイトと変更率の特定
共有結合標識による質量付加は、(a)インタクトタンパク質及び(b)ペプチドレベル8,9で測定することができる。無傷レベルでは、標識数の異なるタンパク質種の分布を、直接分析またはLC-MSの標識タンパク質サンプルから得ることができます。より高い分解能構造情報(すなわち、部位特異的標識データ)を得る?...
ディスカッション
重要なステップ
実験計画に関するいくつかの点を考慮して、信頼性の高いラベル作成結果を確保する必要があります。まず、タンパク質標識を最大化するためには、DEPCと反応して標識の程度を低下させることができるため、強い求核基(例えば、Tris)を有する緩衝液を避ける必要がある。また、このようなバッファーが標識残基と反応し、ラベルの除去を引き起こし、したがって構?...
開示事項
著者らは開示するものは何もない。
謝辞
著者らは、グラントR01 GM075092の下で国立衛生研究所(NIH)からの支援を認めている。ここで説明したデータの一部を取得するために使用されるサーモオービトラップ核質量分析計は、国立衛生研究所の助成金S10OD010645からの資金で取得されました。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Thermo Fisher Scientific | 3448 | |
| 3-(N-morpholino)propanesulfonic acid | Millipore Sigma | M1254 | |
| 3-(N-morpholino)propanesulfonic acid sodium salt | Millipore Sigma | M9381 | |
| Acclaim PepMap RSLC C18 Column | Thermo Scientific | 164537 | 300 μm x 15 cm, C18, 2 μm, 100 A |
| Acetonitrile | Fisher Scientific | A998-1 | |
| Diethylpyrocarbonate | Millipore Sigma | D5758 | |
| HPLC-grade water | Fisher Scientific | W5-1 | |
| Imidazole | Millipore Sigma | I5513 | |
| Immobilized chymotrypsin | ProteoChem | g4105 | |
| Immobilized trypsin, TPCK Treated | Thermo Fisher Scientific | 20230 | |
| Iodoacetamide | Millipore Sigma | I1149 | |
| Tris(2-carboxyethyl)phosphine | Millipore Sigma | C4706 |
参考文献
- Katta, V., Chait, B. T., Carr, S. Conformational Changes in Proteins Probed by Hydrogen-exchange Electrospray-ionization. Rapid Communications in Mass Spectrometry. 5, 214-217 (1991).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25, 158-170 (2006).
- Pirrone, G. F., Iacob, R. E., Engen, J. R. Applications of hydrogen/deuterium exchange MS from 2012 to 2014. Analytical Chemistry. 87, 99-118 (2015).
- Oganesyan, I., Lento, C., Wilson, D. J. Contemporary hydrogen deuterium exchange mass spectrometry. Methods. 144, 27-42 (2018).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrometry Reviews. 25, 663-682 (2006).
- Holding, A. N. XL-MS: Protein cross-linking coupled with mass spectrometry. Methods. 89, 54-63 (2015).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107, 3514-3543 (2007).
- Mendoza, V. L., Vachet, R. W. Probing Protein Structure by Amino Acid-Specific Covalent Labeling and Mass Spectrometry. Mass Spectrometry Reviews. 28, 785-815 (2009).
- Limpikirati, P., Liu, T., Vachet, R. W. Covalent labeling-mass spectrometry with non-specific reagents for studying protein structure and interactions. Methods. 144, 79-93 (2018).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemistry Reviews. 120, 4335 (2020).
- Maleknia, S. D., Brenowitz, M., Chance, M. R. Millisecond radiolytic modification of peptides by synchrotron X-rays identified by mass spectrometry. Analytical Chemistry. 71, 3965-3973 (1999).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77, 5814-5822 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society of Mass Spectrometry. 16, 2057-2063 (2005).
- Mendoza, V. L., Vachet, R. W. Protein surface mapping using diethylpyrocarbonate with mass spectrometric detection. Analytical Chemistry. 80, 2895-2904 (2008).
- Mendoza, V. L., Antwi, K., Barón-rodríguez, M. A., Blanco, C., Vachet, R. W. Structure of the Pre-amyloid Dimer of β-2-microglobulin from Covalent Labeling and Mass Spectrometry. Biochemistry. 49, 1522-1532 (2010).
- Mendoza, V. L., Barón-Rodríguez, M. A., Blanco, C., Vachet, R. W. Structural insights into the pre-amyloid tetramer of β-2-microglobulin from covalent labeling and mass spectrometry. Biochemistry. 50, 6711-6722 (2011).
- Zhou, Y., Vachet, R. W. Increased protein structural resolution from diethylpyrocarbonate-based covalent labeling and mass spectrometric detection. Journal of the American Society of Mass Spectrometry. 23, 708-717 (2012).
- Borotto, N. B., et al. Investigating Therapeutic Protein Structure with Diethylpyrocarbonate Labeling and Mass Spectrometry. Analytical Chemistry. 87, 10627-10634 (2015).
- Liu, T., Marcinko, T. M., Kiefer, P. A., Vachet, R. W. Using Covalent Labeling and Mass Spectrometry To Study Protein Binding Sites of Amyloid Inhibiting Molecules. Analytical Chemistry. 89, 11583-11591 (2017).
- Limpikirati, P., et al. Covalent labeling and mass spectrometry reveal subtle higher order structural changes for antibody therapeutics. MAbs. 11, 463-476 (2019).
- Limpikirati, P., Pan, X., Vachet, R. W. Covalent Labeling with Diethylpyrocarbonate: Sensitive to the Residue Microenvironment, Providing Improved Analysis of Protein Higher Order Structure by Mass Spectrometry. Analytical Chemistry. 91, 8516-8523 (2019).
- Liu, T., Limpikirati, P., Vachet, R. W. Synergistic Structural Information from Covalent Labeling and Hydrogen-Deuterium Exchange Mass Spectrometry for Protein-Ligand Interactions. Analytical Chemistry. 91, 15248-15254 (2019).
- Pan, X., Limpikirati, P., Chen, H., Liu, T., Vachet, R. W. Higher-Order Structure Influences the Kinetics of Diethylpyrocarbonate Covalent Labeling of Proteins. Journal of the American Society of Mass Spectrometry. 31, 658-665 (2020).
- Limpikirati, P. K., Zhao, B., Pan, X., Eyles, S. J., Vachet, R. W. Covalent Labeling/Mass Spectrometry of Monoclonal Antibodies with Diethylpyrocarbonate: Reaction Kinetics for Ensuring Protein Structural Integrity. Journal of the American Society of Mass Spectrometry. 31, 1223-1232 (2020).
- Liu, T., Marcinko, T. M., Vachet, R. W. Protein-Ligand Affinity Determinations Using Covalent Labeling-Mass Spectrometry. Journal of the American Society of Mass Spectrometry. 31, 1544-1553 (2020).
- Srikanth, R., Mendoza, V. L., Bridgewater, J. D., Zhang, G., Vachet, R. W. Copper Binding to β-2-Microglobulin and its Pre-Amyloid Oligomers. Biochemistry. 48, 9871-9881 (2009).
- Lim, J., Vachet, R. W. Using mass spectrometry to study copper-protein binding under native and non-native conditions: β-2-microglobulin. Analytical Chemistry. 76, 3498-3504 (2004).
- Lindsley, C. W. Predictions and Statistics for the Best-Selling Drugs Globally and in the United States in 2018 and a Look Forward to 2024 Projections. ACS Chemical Neuroscience. 10, 1115 (2019).
- Floege, J., Ketteler, M. β2-Microglobulin-derived amyloidosis: An update. Kidney International. 59, 164 (2001).
- Antwi, K., et al. Cu (II) organizes β-2-microglobulin oligomers but is released upon amyloid formation. Protein Science. 17, 748-759 (2008).
- Dong, J., et al. Unique Effect of Cu(II) in the Metal-Induced Amyloid Formation of β-2-Microglobulin. Biochemistry. 53, 1263-1274 (2014).
- Marcinko, T. M., Drews, T., Liu, T., Vachet, R. W. Epigallocatechin-3-gallate Inhibits Cu(II)-Induced β-2-Microglobulin Amyloid Formation by Binding to the Edge of Its β-Sheets. Biochemistry. 59, 1093-1103 (2020).
- Zhou, Y., Vachet, R. W. Diethylpyrocarbonate Labeling for the Structural Analysis of Proteins: Label Scrambling in Solution and How to Avoid it. Journal of the American Society of Mass Spectrometry. 23, 899-907 (2012).
- Borotto, N. B., Degraan-Weber, N., Zhou, Y., Vachet, R. W. Label scrambling during CID of covalently labeled peptide ions. Journal of the American Society of Mass Spectrometry. 25, 1739-1746 (2014).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical Chemistry. 90, 7721-7729 (2018).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Analytical Chemistry. 89, 1459-1468 (2017).
- Zheng, X., Wintrode, P. L., Chance, M. R. Complementary Structural Mass Spectrometry Techniques Reveal Local Dynamics in Functionally Important Regions of a Metastable Serpin. Structure. 16, 38-51 (2008).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by H/D exchange and covalent labeling: The glycerol facilitator. Journal of Molecular Biology. 416, 400-413 (2012).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical Chemistry. 89, 2250-2258 (2017).
- Borotto, N. B., Zhang, Z., Dong, J., Burant, B., Vachet, R. W. Increased β-Sheet Dynamics and D-E Loop Repositioning Are Necessary for Cu(II)-Induced Amyloid Formation by β-2-Microglobulin. Biochemistry. 56, 1095-1104 (2017).
- Shi, L., Liu, T., Gross, M. L., Huang, Y. Recognition of Human IgG1 by Fcγ Receptors: Structural Insights from Hydrogen-Deuterium Exchange and Fast Photochemical Oxidation of Proteins Coupled with Mass Spectrometry. Biochemistry. 58, 1074-1080 (2019).
- Gerega, S. K., Downard, K. M. PROXIMO - A new docking algorithm to model protein complexes using data from radical probe mass spectrometry (RP-MS). Bioinformatics. 22, 1702-1709 (2006).
- Kamal, J. K. A., Chance, M. R. Modeling of protein binary complexes using structural mass spectrometry data. Protein Science. 17, 79-94 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved