È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Etichettatura covalente con dietilpirocarbonato per lo studio della struttura proteica di ordine superiore mediante spettrometria di massa
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Vengono descritte le procedure sperimentali per l'esecuzione di etichette covalenti a base di dietilpirrocarbonato con rilevamento spettrometrico di massa. Il dietilpirrocarbonato viene semplicemente miscelato con il complesso proteico o proteico di interesse, portando alla modifica di residui di amminoacidi accessibili al solvente. I residui modificati possono essere identificati dopo la digestione proteolitica e l'analisi della cromatografia liquida/spettrometria di massa.
Abstract
Caratterizzare la struttura di ordine superiore di una proteina è essenziale per comprenderne la funzione. La spettrometria di massa (SM) è emersa come un potente strumento a questo scopo, specialmente per i sistemi proteici che sono difficili da studiare con i metodi tradizionali. Per studiare la struttura di una proteina da parte della SM, vengono eseguite reazioni chimiche specifiche in soluzione che codificano le informazioni strutturali di una proteina nella sua massa. Un approccio particolarmente efficace è quello di utilizzare reagenti che modifichino covalentemente catene laterali di amminoacidi accessibili ai solventi. Queste reazioni portano ad aumenti di massa che possono essere localizzati con risoluzione a livello di residuo se combinati con digestione proteolitica e spettrometria di massa tandem. Qui descriviamo i protocolli associati all'uso del dietilpirocarbonato (DEPC) come reagente di etichettatura covalente insieme al rilevamento MS. Il DEPC è una molecola altamente elettrofila in grado di etichettare fino al 30% dei residui nella proteina media, fornendo così un'eccellente risoluzione strutturale. Il DEPC è stato utilizzato con successo insieme alla SM per ottenere informazioni strutturali per piccole proteine monodominio, come la β2-microglobulina, a grandi proteine multidominio, come gli anticorpi monoclonali.
Introduzione
Le proteine sono biomolecole essenziali praticamente in ogni processo fisiologico. La varietà di funzioni che le proteine svolgono sono possibili a causa delle strutture che adottano e delle interazioni che hanno con altre biomolecole. Per comprendere la funzione proteica a un livello più profondo, sono necessari strumenti biochimici e biofisici per chiarire queste importanti caratteristiche strutturali e interazioni. Tradizionalmente, la cristallografia a raggi X, la microscopia elettronica criogenica e la spettroscopia a risonanza magnetica nucleare (NMR) hanno fornito i dettagli desiderati a livello atomico per rivelare la struttura proteica. Tuttavia, numerosi sistemi proteici non possono essere interrogati da queste tecniche a causa dello scarso comportamento di cristallizzazione, della limitata disponibilità di proteine, dell'eccessiva eterogeneità del campione o delle limitazioni di peso molecolare. Di conseguenza, sono emersi metodi di analisi più nuovi che superano questi limiti. Tra le tecniche emergenti che possono fornire informazioni strutturali sulle proteine c'è la spettrometria di massa.
La spettrometria di massa (SM) misura il rapporto massa-carica (m/z) di una molecola, quindi le informazioni strutturali di ordine superiore delle proteine devono essere ottenute codificando le informazioni strutturali desiderate nella massa della proteina. Sono stati sviluppati diversi approcci per codificare queste informazioni, tra cui lo scambio idrogeno-deuterio (HDX)1,2,3,4, il crosslinking chimico (XL)5,6e l'etichettatura covalente (CL)7,8,9,10. In HDX, gli idrogenati di ammide dorsale vengono scambiati da deuterio leggermente più massicci a tassi che dipendono dall'accessibilità dei solventi e dall'estensione del legame H. L'estensione dell'HDX può essere localizzata digerendo rapidamente la proteina in frammenti peptidici prima di separare e misurare questi frammenti dallo spettrometro di massa o dissociando la proteina in un esperimento dall'alto verso il basso. Determinare il tasso di cambio fornisce ulteriori informazioni sulla dinamica proteica. HDX ha dimostrato di essere uno strumento prezioso per caratterizzare la struttura proteica nonostante le sfide associate allo scambio posteriore e la necessità di apparecchiature specializzate per massimizzare la riproducibilità. In XL-MS, le proteine reagisce con reagenti bifunzionali che collegano covalentemente catene laterali di residui adiacenti all'interno di una data proteina o tra due proteine. In questo modo, XL-MS può fornire vincoli di distanza che possono essere utilizzati per caratterizzare la struttura proteica. Le regioni della proteina che sono reticate possono essere identificate dalla digestione proteolitica seguita da analisi della cromatografia liquida (LC)-MS. Mentre XL-MS è uno strumento versatile che è stato utilizzato per studiare una varietà di complessi proteici, tra cui celle interne, l'identificazione dei prodotti XL è impegnativa e richiede software specializzato.
Cl-MS è emerso di recente come uno strumento complementare e talvolta alternativo basato sulla SM per studiare la struttura e le interazioni proteiche. In CL-MS, un complesso proteico o proteico viene modificato covalentemente con un reagente monofunzionale in grado di reagire con catene laterali esposte al solvente (Figura 1). Confrontando le estensioni di modifica di un complesso proteico o proteico in diverse condizioni, è possibile rivelare cambiamenti di conformazione, siti di legame e interfacce proteina-proteina. Dopo la reazione CL, le informazioni specifiche del sito, spesso a livello di singolo amminoacido, possono essere ottenute utilizzando i tipici flussi di lavoro di proteomica bottom-up in cui le proteine vengono digerite proteoliticamente, i frammenti peptidici sono separati da LC e i siti modificati vengono identificati utilizzando la SM tandem (MS/MS). La ricca storia della chimica bioconiutogata ha reso disponibili numerosi reagenti per esperimenti CL-MS. I reagenti CL si suddividono in due categorie generali: i) specifiche e (ii) non specifiche. Reagenti specifici reagiscono con un singolo gruppo funzionale (ad esempio, ammine libere)8,10 e sono facili da implementare, ma tendono a fornire informazioni strutturali limitate. Reagenti non specifici reagiscono con una vasta gamma di catene laterali, ma spesso richiedono attrezzature specializzate come laser o sorgenti di sincrotrone per produrre queste specie altamente reattive. I radicali idrossilici sono il reagente non specifico più comunemente usato, essendo stati applicati in impronte di radicali idrossilici (HRF)7,11,12,13 esperimenti per studiare una vasta gamma di proteine e complessi proteici in una varietà di condizioni.
Il nostro gruppo di ricerca ha utilizzato con successo un altro reagente relativamente non specifico chiamato dietilpirocarbonato (DEPC) per studiare la struttura proteica e le interazioni nel contesto degli esperimenti CL-MS14,15,16,17,18,19,20,21,22,23,24,25. DEPC offre la semplicità di reagenti specifici per l'etichettatura (cioè, non è necessaria alcuna apparecchiatura specializzata per eseguire le reazioni), reagendo con fino al 30% di amminoacidi nella proteina media. Come reagente altamente elettrofilo, il DEPC è in grado di reagire con il N-terminale e le catene laterali nucleofile di cisteina, istidina, lisina, tirosina, serina e residui di treonina. Tipicamente, viene generato un singolo prodotto di queste reazioni, con un aumento di massa di 72,02 Da. Questo singolo tipo di prodotto contrasta con i fino a 55 diversi prodotti che possono essere prodotti quando le proteine reagiscono con radicali idrossilici7. Tale chimica semplice facilita l'identificazione dei siti etichettati.
Qui, forniamo protocolli per l'utilizzo di CL-MS basato su DEPC per studiare la struttura e le interazioni proteiche. Vengono descritti i dettagli associati alla preparazione dei reagenti, alle reazioni depc-proteina, alle condizioni di digestione proteica, ai parametri LC-MS e MS/MS e all'analisi dei dati. Dimostriamo anche l'utilità dell'etichettatura DEPC fornendo risultati di esempio dalle interazioni proteina-metallo, proteina-ligando e proteina-proteina, nonché proteine che subiscono cambiamenti strutturali al riscaldamento.
Protocollo
1. Preparazione di proteine e reagenti
NOTA: questo protocollo include un flusso di lavoro di esempio per l'etichettatura di una proteina con DEPC. Alcune condizioni e concentrazioni di reagenti elencate possono variare in base alla proteina scelta.
- Preparare tutte le soluzioni di reagente in tubi microcentrifugo da 1,5 ml.
- Preparare una soluzione proteica della concentrazione desiderata, di solito nell'intervallo di decine di μM, in un tampone di acido propanosolfonico (MOPS) 10 mM 3-(N-morfolino) a pH 7,4. In alternativa, preparare una soluzione proteica esistente a scambio tampone in MOPS 7.4 pH 10 mM se il campione contiene un tampone nucleofilo che sarebbe reattivo con DEPC. Possono essere utilizzati anche altri tamponi (ad esempio, salina tamponata con fosfato), purché non abbiano gruppi funzionali nucleofili.
- Preparare una soluzione DEPC da 100 mM in acetonitrile secco (ACN) pipettando 1,45 μL della soluzione DEPC stock 6.9 M in 98,55 μL dell'ACN.
NOTA: Non esiste un insieme di concentrazioni che funzioni con ogni proteina, anche se le concentrazioni ottimali possono essere stimate in base al numero di residui di His e Lys23. Ad esempio, con 50 μL di una soluzione di β2-microglobulina da 50 μM, reagire alla proteina con 0,2 μL del DEPC da 100 mM per una concentrazione finale di DEPC di 200 μM (pari a 4 volte la concentrazione proteica) per garantire il rapporto molare desiderato utilizzando questo esempio generale(tabella 1). L'etichettatura DEPC è una reazionedi 2 ° ordine, quindi cambiare la concentrazione di proteine o DEPC nella miscela di reazione cambierà il tasso di etichettatura. - Preparare 1 M di soluzione di imidazolo pesando 10 mg di imidazolo e sciogliendo in 146,9 μL di acqua di grado HPLC.
2. Etichettatura covalente di proteine intatte
- Impostare la temperatura del bagno d'acqua a 37 °C e attendere che il bagno raggiunga una temperatura stabile.
NOTA: Le concentrazioni e i volumi dei reagenti per un protocollo di etichettatura di esempio sono disponibili nella tabella 1. - In un nuovo tubo di microcentrifugo, mescolare tampone MOPS e soluzione proteica in volumi elencati nella tabella 1.
- Alla proteina e al tampone aggiungere 0,2 μL della soluzione DEPC, assicurandosi di mescolare correttamente la soluzione risultante, quindi posizionare il tubo contenente la miscela di reazione nel bagno d'acqua di 37 °C per 1 minuto.
NOTA: Il volume di ACN aggiunto non deve superare l'1% del volume totale di reazione per evitare perturbazioni della struttura della proteina durante la reazione di etichettatura. Il tempo di reazione è all'utente, anche se una reazione di 1 minuto nelle condizioni di esempio riduce al minimo l'etichettatura eccessiva e la potenziale idrolisi di DEPC14. - Dopo 1 minuto, rimuovere il tubo contenente la miscela di reazione dal bagno d'acqua e spegnere la reazione con 1 μL della soluzione di imidazolo da 1 M per scavengere il DEPC non reatto rimanente.
NOTA: La concentrazione finale di imidazolo nella miscela di reazione deve essere pari a 50 volte la concentrazione di DEPC nella miscela di reazione. Ciò garantirà che il DEPC non transazionato rimanga scavenged.
3. Preparazione del digestione proteica per l'LC-MS dal basso verso l'alto
NOTA: Scegliere condizioni di digestione che siano suscettibili alla proteina di interesse. I passi comuni prevedono lo spiegamento della proteina e la riduzione e l'alchilazione di eventuali legami disolfuro.
- Aprire la proteina aggiungendo un reagente dispiegabile appropriato alla miscela di reazione.
NOTA: Gli agenti di spiegamento comuni includono ACN, urea e cloridrato di guanidina (GuHCl). - Preparare soluzioni di Tris(2-carbossietile)fosfina (TCEP) e iodoacetamide (IAM) pesando 5 mg ciascuno e sciogliendoli in nuovi tubi a microcentrifugo rispettivamente in 174,4 e 270,3 μL di tampone MOPS da 10 mM pH 7,4 per le fasi di riduzione e alchilazione.
- Ridurre i legami disolfuro aggiungendo 2 μL della soluzione TCEP da 100 mM (concentrazione finale di 2 mM in miscela di reazione) alla miscela di reazione e reagendo per 3 minuti a temperatura ambiente.
NOTA: La concentrazione finale di TCEP deve essere pari a 40 volte la concentrazione proteica per legame disolfuro presente nella soluzione. - Alchilato ridotto cisteine con 4 μL della soluzione IAM da 100 mM (concentrazione finale di 4 mM in miscela di reazione) per 30 minuti al buio. L'IAM è sensibile alla luce e si decompone sotto la luce diretta.
NOTA: La concentrazione finale di IAM in soluzione deve essere il doppio della concentrazione utilizzata per il TCEP, o 80 volte la concentrazione proteica per legame disolfuro. - Digerire la proteina con un enzima appropriato come la trippatina o la chimotripsina. Un rapporto proteina-enzima di 10:1 per una digestione di 3 ore a 37 °C con enzima immobilizzato ad una velocità di scuotimento di 300 colpi/min è tipicamente sufficiente per le proteine etichettate DEPC. Vedi Discussione.
- Dopo la digestione, separare l'enzima immobilizzato dai peptidi digeriti per centrifugazione a 12.000 giri/min per 5 minuti.
- Analizzare immediatamente il campione tramite LC-MS/MS o congelare il campione con azoto liquido per ridurre al minimo la degradazione del campione e la perdita di etichette. Conservare i campioni congelati a < -20 °C fino a quando non sono pronti per l'analisi LC-MS/MS.
4. Analisi LC-MS/MS
NOTA: I parametri standard LC-MS/MS per la proteomica dal basso verso l'alto possono essere utilizzati per identificare siti etichettati sui frammenti peptidici proteolitici. Di seguito viene illustrato un esempio generale.
- Separare i peptidi etichettati DEPC utilizzando una fase stazionaria C18 in fase invertita. Utilizzare una tipica fase mobile LC di due solventi: (A) acqua + 0,1% acido formico e (B) ACN + 0,1% acido formico utilizzando un gradiente (ad esempio, figura 2)per ottenere la migliore separazione dei peptidi.
NOTA: Il tempo di separazione può essere ottimizzato in base alla complessità del campione e la portata di fase mobile dipende dall'utilizzo di capillare o nano LC. - Utilizzare uno spettrometro di massa in grado di eseguire LC-MS on-line e MS/MS per identificare i siti di modifica DEPC sul peptide. Nei nostri esperimenti, abbiamo utilizzato con successo diversi tipi di spettrometri di massa. Qualsiasi spettrometro di massa in grado di eseguire automaticamente MS/MS di molti peptidi nel corso di un'analisi LC-MS dovrebbe essere adatto. I parametri MS rilevanti includono: tensione di sorgente ESI = -4000 V per l'ESI regolare; -2000 V per nanospray; Risoluzione orbitrap = 60.000; Durata di esclusione dinamica = 30 s; Tipo di attivazione MS/MS: CID, ETD o entrambi; Intervallo di scansione di massa = 200-2.000; Controllo automatico del guadagno = 4,0E5 (MS1 in Orbitrap) e 5,0E4 (MS2 nella trappola ionica quadrupolo lineare).
- Caricare e iniettare il campione proteico digerito ed etichettato nel sistema LC e avviare l'acquisizione LC-MS/MS. Se il campione è stato congelato in flash, scongelare prima dell'analisi. Deviare l'effluente LC in rifiuti per i primi 5 minuti per evitare che sali eccessivi possano entrare nella fonte ESI.
NOTA: Un circuito di iniezione di 5 μL è generalmente utilizzato, consentendo l'iniezione di circa 2,5 μg di proteine alla LC-MS/MS. Ciò dipende dalle condizioni di carico dell'LC per non ostruire l'iniettore del campione.
5. Analisi dei dati
- Identificare i siti di etichette DEPC e quantificare le aree di picco peptidice utilizzando un software appropriato per lo spettrometro di massa utilizzato.
- Includere l'addizione DEPC (72,02 Da) e la carbamidometilazione (57,02 Da) come modifiche variabili. Ulteriori parametri di ricerca per l'analisi MS/MS sono i seguenti: Scollatura massima persa = 3; Tipi di ioni frammentazione = b e y; Tolleranza precursore m/z = 10 ppm (questo valore dovrebbe essere più alto se viene utilizzato uno spettrometro di massa a trappola ionica quadrupolo); Tolleranza m/z del frammento = 0,5 Da (questo valore deve essere inferiore se per una scansione ionica del prodotto viene utilizzato uno spettrometro di massa ad alta risoluzione); Carica precursore = 1-4.
NOTA: diversi algoritmi di ricerca di database hanno sistemi di punteggio diversi e molti possono avere difficoltà a identificare peptidi modificati da DEPC perché i livelli di modifica possono essere bassi. La regolazione del taglio del punteggio può essere necessaria per identificare peptidi più etichettati. In tal caso, l'interrogazione manuale dei dati MS/MS deve essere utilizzata per verificare peptidi a basso punteggio. Il prodotto dell'idrolisi dell'etichetta DEPC non è incluso nella ricerca dei dati perché il DEPC idrolizzato non è più reattivo verso le catene laterali nucleofile. - Determinare le percentuali di modifica del livello di residui utilizzando le aree di picco cromatografiche delle versioni modificate e non modificate dei peptidi.
NOTA: Qualsiasi peptide contenente il residuo di interesse modificato deve essere preso in considerazione e tutti gli stati di carica inclusi devono essere presenti in tutti i campioni misurati. I peptidi con diverse efficienze di ionizzazione ed eluizione in momenti diversi fa sì che questo valore sia una misura relativa piuttosto che assoluta della modifica di un sito specifico.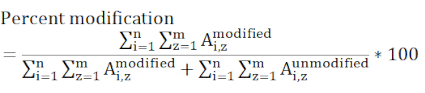
dove Ai,z rappresenta l'area di picco di un dato peptide (i) che contiene il residuo di interesse e considera tutti gli stati di carica rilevati (z). - Determinare se una modifica dell'etichettatura tra un controllo e un campione sperimentale è significativa utilizzando la valutazione statistica. Tre misurazioni di replica per ogni campione sono tipiche e i test t sono più comunemente utilizzati con intervalli di confidenza del 95 o 99%.
Risultati
Identificazione dei siti di modifica DEPC e delle percentuali di modifica
L'aggiunta di massa dovuta all'etichettatura covalente può essere misurata ai livelli di (a) proteina intatta e (b) peptidici8,9. A livello intatto, una distribuzione di specie proteiche con un numero diverso di etichette può essere ottenuta dall'analisi diretta o LC-MS di campioni proteici etichettati. Per ottenere informazioni strutturali a risoluzione più elevata (ad esemp...
Discussione
Passaggi critici
Per quanto riguarda la progettazione sperimentale, si dovrebbero prendere in considerazione diversi punti per garantire risultati di etichettatura affidabili. In primo luogo, per massimizzare l'etichettatura delle proteine, è necessario evitare tamponi con gruppi fortemente nucleofili (ad esempio Tris) perché possono reagire con DEPC e abbassare l'estensione dell'etichettatura. È anche concepibile che tali tamponi possano reagire con residui etichettati, causando la rimozione dell'etichett...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori riconoscono il sostegno dei National Institutes of Health (NIH) nell'ambito di Grant R01 GM075092. Lo spettrometro di massa Thermo Orbitrap Fusion utilizzato per acquisire alcuni dei dati qui descritti è stato acquisito con fondi della sovvenzione dei National Institutes of Health S10OD010645.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Thermo Fisher Scientific | 3448 | |
| 3-(N-morpholino)propanesulfonic acid | Millipore Sigma | M1254 | |
| 3-(N-morpholino)propanesulfonic acid sodium salt | Millipore Sigma | M9381 | |
| Acclaim PepMap RSLC C18 Column | Thermo Scientific | 164537 | 300 μm x 15 cm, C18, 2 μm, 100 A |
| Acetonitrile | Fisher Scientific | A998-1 | |
| Diethylpyrocarbonate | Millipore Sigma | D5758 | |
| HPLC-grade water | Fisher Scientific | W5-1 | |
| Imidazole | Millipore Sigma | I5513 | |
| Immobilized chymotrypsin | ProteoChem | g4105 | |
| Immobilized trypsin, TPCK Treated | Thermo Fisher Scientific | 20230 | |
| Iodoacetamide | Millipore Sigma | I1149 | |
| Tris(2-carboxyethyl)phosphine | Millipore Sigma | C4706 |
Riferimenti
- Katta, V., Chait, B. T., Carr, S. Conformational Changes in Proteins Probed by Hydrogen-exchange Electrospray-ionization. Rapid Communications in Mass Spectrometry. 5, 214-217 (1991).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25, 158-170 (2006).
- Pirrone, G. F., Iacob, R. E., Engen, J. R. Applications of hydrogen/deuterium exchange MS from 2012 to 2014. Analytical Chemistry. 87, 99-118 (2015).
- Oganesyan, I., Lento, C., Wilson, D. J. Contemporary hydrogen deuterium exchange mass spectrometry. Methods. 144, 27-42 (2018).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrometry Reviews. 25, 663-682 (2006).
- Holding, A. N. XL-MS: Protein cross-linking coupled with mass spectrometry. Methods. 89, 54-63 (2015).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107, 3514-3543 (2007).
- Mendoza, V. L., Vachet, R. W. Probing Protein Structure by Amino Acid-Specific Covalent Labeling and Mass Spectrometry. Mass Spectrometry Reviews. 28, 785-815 (2009).
- Limpikirati, P., Liu, T., Vachet, R. W. Covalent labeling-mass spectrometry with non-specific reagents for studying protein structure and interactions. Methods. 144, 79-93 (2018).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemistry Reviews. 120, 4335 (2020).
- Maleknia, S. D., Brenowitz, M., Chance, M. R. Millisecond radiolytic modification of peptides by synchrotron X-rays identified by mass spectrometry. Analytical Chemistry. 71, 3965-3973 (1999).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77, 5814-5822 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society of Mass Spectrometry. 16, 2057-2063 (2005).
- Mendoza, V. L., Vachet, R. W. Protein surface mapping using diethylpyrocarbonate with mass spectrometric detection. Analytical Chemistry. 80, 2895-2904 (2008).
- Mendoza, V. L., Antwi, K., Barón-rodríguez, M. A., Blanco, C., Vachet, R. W. Structure of the Pre-amyloid Dimer of β-2-microglobulin from Covalent Labeling and Mass Spectrometry. Biochemistry. 49, 1522-1532 (2010).
- Mendoza, V. L., Barón-Rodríguez, M. A., Blanco, C., Vachet, R. W. Structural insights into the pre-amyloid tetramer of β-2-microglobulin from covalent labeling and mass spectrometry. Biochemistry. 50, 6711-6722 (2011).
- Zhou, Y., Vachet, R. W. Increased protein structural resolution from diethylpyrocarbonate-based covalent labeling and mass spectrometric detection. Journal of the American Society of Mass Spectrometry. 23, 708-717 (2012).
- Borotto, N. B., et al. Investigating Therapeutic Protein Structure with Diethylpyrocarbonate Labeling and Mass Spectrometry. Analytical Chemistry. 87, 10627-10634 (2015).
- Liu, T., Marcinko, T. M., Kiefer, P. A., Vachet, R. W. Using Covalent Labeling and Mass Spectrometry To Study Protein Binding Sites of Amyloid Inhibiting Molecules. Analytical Chemistry. 89, 11583-11591 (2017).
- Limpikirati, P., et al. Covalent labeling and mass spectrometry reveal subtle higher order structural changes for antibody therapeutics. MAbs. 11, 463-476 (2019).
- Limpikirati, P., Pan, X., Vachet, R. W. Covalent Labeling with Diethylpyrocarbonate: Sensitive to the Residue Microenvironment, Providing Improved Analysis of Protein Higher Order Structure by Mass Spectrometry. Analytical Chemistry. 91, 8516-8523 (2019).
- Liu, T., Limpikirati, P., Vachet, R. W. Synergistic Structural Information from Covalent Labeling and Hydrogen-Deuterium Exchange Mass Spectrometry for Protein-Ligand Interactions. Analytical Chemistry. 91, 15248-15254 (2019).
- Pan, X., Limpikirati, P., Chen, H., Liu, T., Vachet, R. W. Higher-Order Structure Influences the Kinetics of Diethylpyrocarbonate Covalent Labeling of Proteins. Journal of the American Society of Mass Spectrometry. 31, 658-665 (2020).
- Limpikirati, P. K., Zhao, B., Pan, X., Eyles, S. J., Vachet, R. W. Covalent Labeling/Mass Spectrometry of Monoclonal Antibodies with Diethylpyrocarbonate: Reaction Kinetics for Ensuring Protein Structural Integrity. Journal of the American Society of Mass Spectrometry. 31, 1223-1232 (2020).
- Liu, T., Marcinko, T. M., Vachet, R. W. Protein-Ligand Affinity Determinations Using Covalent Labeling-Mass Spectrometry. Journal of the American Society of Mass Spectrometry. 31, 1544-1553 (2020).
- Srikanth, R., Mendoza, V. L., Bridgewater, J. D., Zhang, G., Vachet, R. W. Copper Binding to β-2-Microglobulin and its Pre-Amyloid Oligomers. Biochemistry. 48, 9871-9881 (2009).
- Lim, J., Vachet, R. W. Using mass spectrometry to study copper-protein binding under native and non-native conditions: β-2-microglobulin. Analytical Chemistry. 76, 3498-3504 (2004).
- Lindsley, C. W. Predictions and Statistics for the Best-Selling Drugs Globally and in the United States in 2018 and a Look Forward to 2024 Projections. ACS Chemical Neuroscience. 10, 1115 (2019).
- Floege, J., Ketteler, M. β2-Microglobulin-derived amyloidosis: An update. Kidney International. 59, 164 (2001).
- Antwi, K., et al. Cu (II) organizes β-2-microglobulin oligomers but is released upon amyloid formation. Protein Science. 17, 748-759 (2008).
- Dong, J., et al. Unique Effect of Cu(II) in the Metal-Induced Amyloid Formation of β-2-Microglobulin. Biochemistry. 53, 1263-1274 (2014).
- Marcinko, T. M., Drews, T., Liu, T., Vachet, R. W. Epigallocatechin-3-gallate Inhibits Cu(II)-Induced β-2-Microglobulin Amyloid Formation by Binding to the Edge of Its β-Sheets. Biochemistry. 59, 1093-1103 (2020).
- Zhou, Y., Vachet, R. W. Diethylpyrocarbonate Labeling for the Structural Analysis of Proteins: Label Scrambling in Solution and How to Avoid it. Journal of the American Society of Mass Spectrometry. 23, 899-907 (2012).
- Borotto, N. B., Degraan-Weber, N., Zhou, Y., Vachet, R. W. Label scrambling during CID of covalently labeled peptide ions. Journal of the American Society of Mass Spectrometry. 25, 1739-1746 (2014).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical Chemistry. 90, 7721-7729 (2018).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Analytical Chemistry. 89, 1459-1468 (2017).
- Zheng, X., Wintrode, P. L., Chance, M. R. Complementary Structural Mass Spectrometry Techniques Reveal Local Dynamics in Functionally Important Regions of a Metastable Serpin. Structure. 16, 38-51 (2008).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by H/D exchange and covalent labeling: The glycerol facilitator. Journal of Molecular Biology. 416, 400-413 (2012).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical Chemistry. 89, 2250-2258 (2017).
- Borotto, N. B., Zhang, Z., Dong, J., Burant, B., Vachet, R. W. Increased β-Sheet Dynamics and D-E Loop Repositioning Are Necessary for Cu(II)-Induced Amyloid Formation by β-2-Microglobulin. Biochemistry. 56, 1095-1104 (2017).
- Shi, L., Liu, T., Gross, M. L., Huang, Y. Recognition of Human IgG1 by Fcγ Receptors: Structural Insights from Hydrogen-Deuterium Exchange and Fast Photochemical Oxidation of Proteins Coupled with Mass Spectrometry. Biochemistry. 58, 1074-1080 (2019).
- Gerega, S. K., Downard, K. M. PROXIMO - A new docking algorithm to model protein complexes using data from radical probe mass spectrometry (RP-MS). Bioinformatics. 22, 1702-1709 (2006).
- Kamal, J. K. A., Chance, M. R. Modeling of protein binary complexes using structural mass spectrometry data. Protein Science. 17, 79-94 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon